原核生物蛋白质N-糖基化修饰的研究进展
谭亚红, 高利娟, 宋文霞, 卢雪梅
(山东大学微生物技术国家重点实验室, 青岛266237)
蛋白质糖基化是碳水化合物与氨基酸残基共价结合的过程,是自然界中最丰富的翻译后修饰[1]。蛋白质糖基化主要包括聚糖共价连接到Asn残基(有的物种连接到Arg残基[2])的N-糖基化[3],或者连接到Ser或Thr残基的O-糖基化[4]。蛋白质糖基化过程一般有两种途径:一种是依赖OST(oligosaccharyltransferase, OST)将组装在脂质载体上的寡糖(lipid-linked oligosaccharide, LLO)整体转移到受体蛋白上;另一种是不依赖OST而是由N-或O-糖基转移酶将单糖依次添加到受体蛋白上。
N-糖基化是一种常见的糖基化类型。在真核生物中,N-糖基化影响蛋白质的折叠、稳定性、分选和运输等[5],对蛋白质发挥功能十分重要。在细菌中,N-糖基化主要与细菌的致病性、黏附性和环境适应性等有关[6]。在古菌中,N-糖基化在菌毛[7]和S-层蛋白的组装以及适应环境变化方面起着重要作用[8]。随着对N-糖基化的重要作用的认识,相关领域逐渐成为研究者们关注的热点。虽然蛋白质N-糖基化在真核生物中已广泛研究,但原核生物比真核生物具有更多样的糖基化途径以及更复杂的聚糖结构,对原核生物蛋白质N-糖基化的了解仍然有限。现就原核生物蛋白质N-糖基化的研究进展进行综述。
1 细菌的N-糖基化
1.1 聚糖连接到Asn残基的N-糖基化
1.1.1 依赖OST的N-糖基化
迄今为止,细菌中研究最多的是Campylobacterjejuni的N-糖基化系统。C.jejuni是革兰氏阴性菌,属于ε-变形菌纲,是人类肠胃炎的病原体。C.jejuni是第一个被发现存在N-糖基化系统的细菌,有80多种蛋白质被N-糖基化[9]。C.jejuni的蛋白N-糖基化过程:细胞质中的UDP-GlcNAc依次经过脱水酶PglF、氨基转移酶PglE、乙酰基转移酶PglD催化生成UDP-2,4-diacetamido-Bacillosamine(也称为UDP-diNAcBac),再由糖基转移酶PglC转移到脂质载体十一烯基焦磷酸(undecaprenyl pyrophosphate, Und-PP),随后糖基转移酶PglA、PglJ、PglH以及PglI向其依次添加不同的单糖形成LLO[Und-PP-GlcGalNAc5Bac],LLO通过翻转酶PglK转移到周质空间后,由寡糖基转移酶PglB将寡糖释放到周质中[10]或者转移到受体蛋白序列Asp/Glu-X1-Asn-X2-Ser/Thr(X1和X2是除Pro以外的任意氨基酸残基)的Asn残基上[11][图1(a)]。这些参与N-糖基化过程的酶由pgl基因簇编码。在C.jejuni中,N-糖基化途径的破坏会影响其致病性、硝酸还原酶活性、蛋白质稳定性、营养转运、应激反应和抗菌素耐药性[12-13]。

(a)Campylobacter jejuni的N-糖基化途径;(b)Haemophilus influenzae的N-糖基化途径;(c)NleB/SseK介导的精氨酸N-GlcNAc糖基化途径;(d)Shewanellaoneidensis的N-糖基化途径。图1 细菌的蛋白质N-糖基化途径Figure 1 Overview of the pathway of bacterial protein N-glycosylation
在其他的ε-变形菌中也发现了与C.jejuni同源的pgl基因簇,包括一些Campylobacter和3个Helicobacter物种(Helicobacterpullorum、Helicobactercanadensis、Helicobacterwinghamensis)[14],与Campylobacter明显不同的是Helicobacter的pgl基因分散在5个基因簇中,并且有2个pglB基因,研究还发现Helicobacter可能存在2种N-糖基化途径:一种是与C.jejuni类似的依赖PglB1的N-糖基化途径;另一种是依赖PglB2的糖基化方式[15]。这暗示了Helicobacter的N-糖基化系统可能比C.jejuni更为复杂。此外,在Desulfovibriodesulfuricans[16]、Nitratiruptortergarcus和Sulfurovumlithotrophicum[17]中也发现了PglB的同源蛋白,但这些PglBs识别的受体蛋白序列和聚糖结构有所不同。尚未在革兰氏阳性菌中发现类似的N-糖基化系统。
1.1.2 不依赖OST的N-糖基化
与ε-变形菌纲中发现的N-糖基化系统不同,β-和γ-变形菌纲中存在另一种N-糖基化系统。在病原菌Haemophilusinfluenzae的细胞质中,糖基转移酶HMW1C将UDP-Glc和UDP-Gal转移至黏附素HMW1的Asn-X-Ser/Thr(X≠Pro)序列上使HMW1发生N-糖基化修饰[18][图1(b)]。病原菌Actinobacilluspleuropneumoniae的N-糖基化途径与H.influenzae相似,区别仅在于H.influenzae的HMW1C既可催化UDP-Glc或UDP-Gal与 HMW1的连接,又能在HMW1连接的Glc上连接另一个己糖,而A.pleuropneumoniae存在共转录的N-糖基转移酶(N-linking glycosyltransferase,NGT)和辅助糖基转移酶(accessory glycosyltransferase,AGT),其中NGT 利用Glc对黏附蛋白进行糖基化,AGT则负责将己糖添加到糖蛋白连接的Glc上[19]。其他病原菌如Kingellakingae和Aggregatibacteraphrophilus也具有HMW1C同源蛋白,能将己糖转移到黏附蛋白上[20]。这些病原菌中修饰蛋白的碳水化合物是己糖而不是N-乙酰化糖,并且糖基化的缺失会影响病原菌对宿主细胞的黏附。
1.2 N-GlcNAc或Rha连接到Arg残基的N-糖基化
1.2.1 N-GlcNAc糖基化
许多革兰氏阴性病原菌通过Ⅲ型分泌系统将具有糖基转移酶活性的效应子注入宿主细胞的细胞质中,包括存在Citrobacterrodentium和肠致病性Escherichiacoli中的NleB以及存在Salmonellaentericaserovar Typhimurium的SseK[2]。NleB/SseK在宿主细胞中将UDP-GlcNAc共价连接到宿主含有死亡结构域衔接蛋白的保守Arg残基上[图1(c)],蛋白被糖基化后不能形成死亡诱导信号复合物,使宿主细胞凋亡或坏死的信号传导受阻[21]。此外,NleB/SseK催化的Arg糖基化还能促进病原菌在肠道上皮附着[22]。
1.2.2 Rha糖基化
在核糖体将mRNA序列翻译成多肽链的过程中,多聚Pro序列会导致翻译停滞,细菌中存在翻译延伸因子P(elongation factor P, EF-P),EF-P可以与核糖体结合来刺激肽键的形成从而使翻译继续进行[23]。Shewanellaoneidensis存在一种独特修饰策略激活EF-P:糖基转移酶EarP将dTDP-β-L-Rha转移到EF-P的Arg32以激活EF-P的功能[图1(d)][24]。在Pseudomonasaeruginosa[25]和Neisseriameningitidis[26]中也存在这种修饰。研究表明EF-P和EarP的缺失会影响病原菌毒力因子的翻译从而导致病原菌毒性降低。此外,研究还发现EF-P的三维结构主要由凸面组成,而EarP具有底物结合口袋,更易与抗菌药物结合[27]。因此,将EarP作为治疗靶点进行研究不失为一个对抗病原菌感染的新思路。
1.3 Cytophaga hutchinsonii中的N-糖基化
此前,细菌N-糖基化系统仅在变形菌门中发现,最近在研究中发现拟杆菌门中的C.hutchinsonii也具有N-糖基化系统[28]。研究发现参与C.hutchinsonii蛋白质N-糖基化的糖基转移酶chu_3842是C.jejuni的pglA的同源基因,并且C.hutchinsonii的N-糖基化也发生在周质空间。C.hutchinsonii的N-糖蛋白对能切割N-糖链的N-糖苷酶敏感,而C.jejuni的N-糖蛋白连接的寡糖链含有独特的diNAcBac故不能被N-糖苷酶切割。研究表明C.hutchinsonii的N-糖基化受体序列是Asn-X-Ser/Thr(X≠Pro),这也与C.jejuni不同。此外,还发现C.hutchinsonii的N-糖基化对蛋白的运输和定位有重要影响,这与真核生物类似,但之前的研究显示真核生物N-糖基化的这些功能并不适用于细菌,因此,这项研究丰富了对细菌中N-糖基化的生物学作用的了解。
2 古菌的N-糖基化
古菌最为人所知的是其在极端环境中的生存能力。在极端环境中,古菌不仅能保持细胞表面完整,还能执行各种正常的生理功能。古菌的细胞表面有高度多样化的蛋白质结构,然而,对古菌中细胞表面结构的认识仅限于S-层和菌毛:S-层糖蛋白可形成一种有孔的晶格状单分子层包被在细胞外,以维持细胞形态;菌毛可以介导古菌的表面附着和运动能力,并在生物膜形成、DNA交换和细胞间通信中发挥作用[29]。古菌的细胞表面蛋白主要被N-聚糖修饰,而在细菌中O-糖基化是细胞表面蛋白糖基化的主要形式。
迄今为止,对古菌N-糖基化途径的了解仍然有限,仅对少数物种进行了描述。类似于真核生物和细菌,古菌的N-聚糖由糖基转移酶在多萜醇磷酸载体上装配,再翻转至细胞膜外侧由寡糖基转移酶AglB转至蛋白的Asn-X-Ser/Thr(X≠Pro)序列的Asn残基上[30]。然而,古菌中N-聚糖的结构和组成是高度多样化的,表明古菌N-糖基化过程的多样性[31],这是真核生物或细菌所不具有的。Haloferaxvolcanii[32]有两种不同的N-糖基化途径,并且可以用不同的聚糖同时修饰相同的糖蛋白,同一蛋白也可结合一种聚糖类型的不完全聚糖前体,这些蛋白广泛多样的糖基化修饰可能与古菌在极端的pH、温度或盐度以及其他恶劣的环境中生长有关。然而,关于这些交替的N-糖基化途径的调控尚待确定。
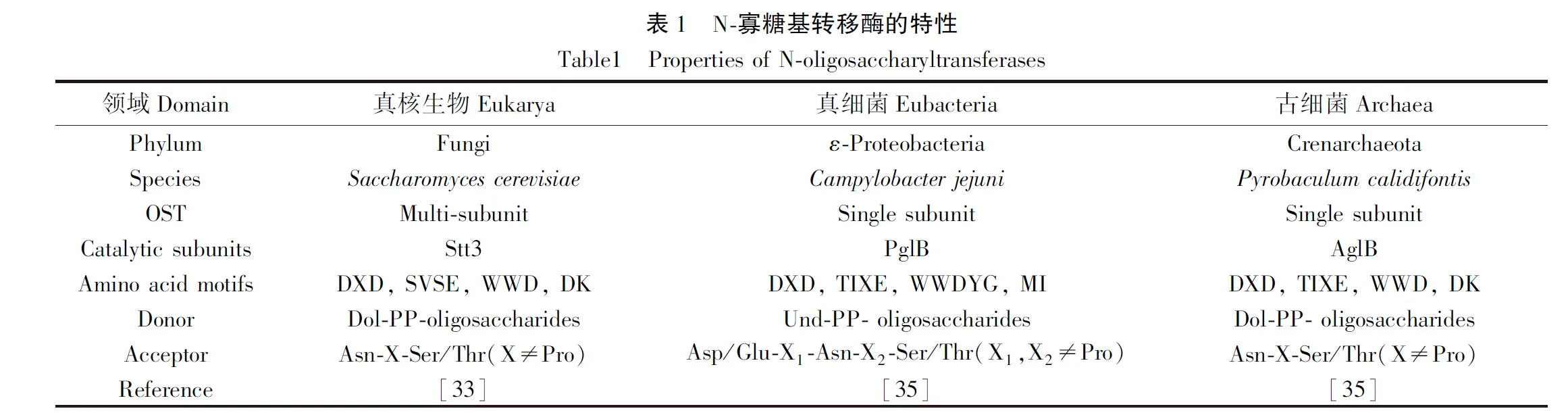
3 OST的结构
N-糖基化途径中最保守的成分是OST。真核生物的OST复合物含有多个亚基,其催化亚基为Stt3[33],与细菌和古菌中的PglB和AglB同源。尽管所有OSTs的序列保守性非常低,但在结构和寡糖转移机制上很相似。Stt3/AglB/PglB蛋白的晶体结构揭示了它们的主要序列包括N端的13个跨膜(transmembrane,TM)域和C端的可溶性球状结构域,还有一些保守的短氨基酸基序,包括N端TM域的EL1(external loop, EL)和EL2上的两个DXD基序以及EL5上的SVSE/TIXE 基序,C端球状结构域的WWD和DK/MI 基序[34](表1)。研究表明OSTs的寡糖转移机制是OSTs的WWD基序中Asp残基的羧基特异性结合受体序列Ser/Thr残基的羟基,TM域中的DXD基序与二价金属离子形成催化结构并激活受体序列Asn残基的侧链氮原子,活化的酰胺氮攻击LLO糖上的异头碳从而使聚糖连接到蛋白上[35]。PglB[36]和AglB[37]中无序的EL5结合聚糖底物后会变得有序,研究者提出PglB中无序的EL5可促进聚糖底物转移至催化位点,而酵母的聚糖底物比细菌的要大得多,不能通过无序的EL5进入催化位点[33]。
研究表明,C.jejuni的PglB也是一种糖蛋白,它能通过WWDYG基序催化自身DYNQS序列的糖基化[38],但PglB的活性不受其糖基化位点的影响,未糖基化的PglB仍然能够转移聚糖。Campylobacter属的PglB都具有WWDYG和糖基化基序,而大多数真核生物的STT3和古菌中的AglB仅含有WWD基序,其他细菌的PglB同源物也基本不存在WWD和糖基化基序。
4 N-聚糖的受体序列
细菌中Campylobacter属的N-糖基化受体序列一般为Asp/Glu-X1-Asn-X2-Ser/Thr(X1,X2≠Pro),而其他细菌和古菌的N-糖基化受体序列都是Asn-X-Ser/Thr(X≠Pro)[39]。研究表明Pro能破坏蛋白的二级结构并导致多肽链扭结,故受体序列的X位不存在Pro残基[40]。由此可以看出,Asn-X-Ser / Thr序列(X≠Pro)是蛋白质N-糖基化通用的受体序列。
5 OST的聚糖底物特异性
在大多数真核生物中,聚糖结构为Glc3Man9GlcNAc2[41]。细菌和古菌利用的聚糖类型因物种而异。细菌的PglB可以转移多种聚糖底物,但是C.jejuni的糖底物需满足以下条件:还原末端第一个糖的C2位有乙酰化修饰,并且还原末端前两个糖基的连接方式不能是β1-4连接[42]。古菌中的聚糖种类非常复杂。Archaeoglobusfulgidus的N-聚糖仅由己糖残基组成[43],此外,属于古菌泉古菌门的Pyrobaculumcalidifontis的N-聚糖是高甘露糖型寡糖[44],这与真核生物的N-聚糖结构相似。至于聚糖载体,真核生物使用十二烷醇焦磷酸(dolichol pyrophosphate,Dol-PP),细菌使用Und-PP,而古菌的广古菌门使用十二烷醇磷酸(dolichol phosphate,Dol-P),泉古菌门使用Dol-PP。泉古菌门与真核生物在聚糖结构和聚糖载体的相似性表明它们的N-糖基化系统之间可能存在密切的进化关系,这与最近的系统发育分析一致[45]。
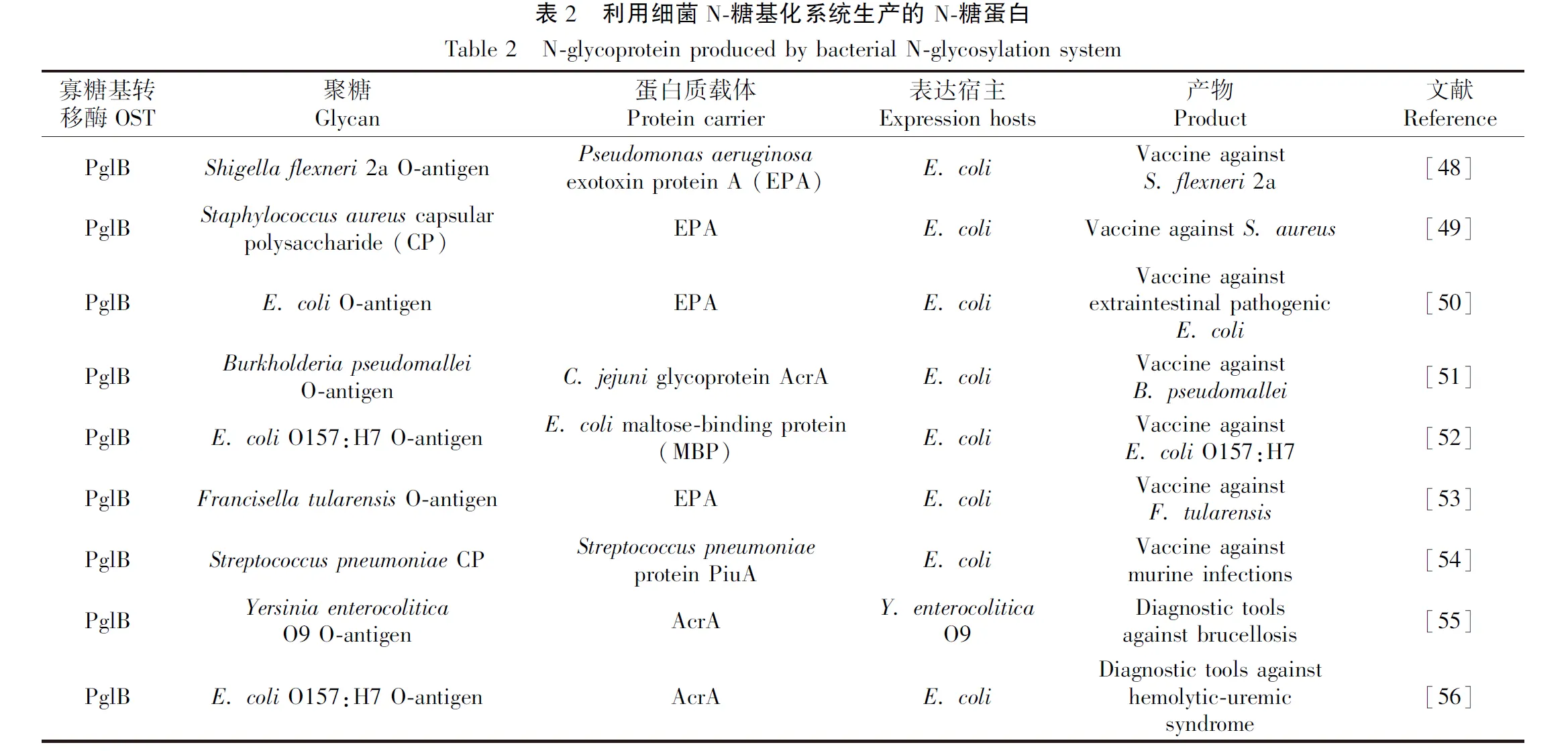
6 原核生物N-糖基化系统在糖基化工程的应用
C.jejuni的N-糖基化途径成功转移到E.coli为开发糖基化工程系统奠定了基础[46]。该系统的关键特征:C.jejuni的寡糖基转移酶PglB能够将不同来源的聚糖转移至含有特定N-糖基化位点的蛋白上,同时E.coli没有糖基化途径,故可作为合成糖蛋白的良好平台。在这个突破性的发现之后,C.jejuni的N-糖基化途径在糖基化工程中得到广泛应用[47]。目前已利用PglB合成了针对Shigellaspecies[48]、Staphylococcusaureus[49]和肠致病性E.coli[50]的糖蛋白疫苗,同时也探索了针对其他细菌的糖蛋白疫苗,例如Burkholderiapseudomallei[51]、E.coliO157[52],Francisellatularensis[53]和Streptococcuspneumoniae[54](表2)。此外,通过糖基化工程合成的糖蛋白也成功应用于布鲁氏菌病[55]和溶血性尿毒症综合征的诊断[56]。有两个条件限制C.jejuniPglB将聚糖转移到受体蛋白的能力:与其他菌中糖基化序列Asn-X-Ser/Thr(X≠Pro)相比,PglB需要一个延长的序列Asp/Glu-X1-Asn-X2-Ser/Thr(X1, X2≠Pro);只有还原端第一个糖的C2位有乙酰化修饰的聚糖才能被PglB识别[57]。为了克服这些局限性,研究者们除了改造聚糖和受体蛋白结构外,还对其他糖基转移酶进行了研究,并发现A.pleuropneumoniae的NGT也能转移到大肠杆菌中对异源蛋白进行糖基化[58]。此外,研究表明,利用NGT合成的长链聚唾液酸对治疗性蛋白质DARPin (designed ankyrin repeat protein, DARPin)进行修饰可以改善药代动力学并降低免疫原性[59],并且NGT能将人类免疫球蛋白的Fc域糖基化[60],进一步证明其在糖基化工程中的潜在用途。
与C.jejuni的PglB相比,古菌的AglB对聚糖特异性的要求更低,AglB能将多种聚糖结合到受体蛋白的Asn-X-Ser/Thr(X≠Pro)序列。Haloferaxvolcanii具有比较清楚的N-糖基化途径,能利用各种糖基转移酶使N-聚糖的组分发生广泛的变化[61],但能否在细菌或真核生物中构建古菌的N-糖基化途径来生产糖蛋白还有待进一步研究。
与传统的化学合成的糖蛋白疫苗相比,细菌糖基化工程开发的糖蛋白疫苗具有成本低、疗效突出和安全性好的优势。利用细菌糖基化工程生产糖蛋白疫苗具有广阔的发展前景。近年来,研究者们为提高糖蛋白疫苗的生产效率做了大量的研究:通过过表达E.coli的糖生物合成基因[62],将其他糖基转移酶转入E.coliK-12[47]来提高糖基化效率;通过PglB插入染色体中表达[63],将常规IPTG诱导方法改为优化的自动诱导方法[64],控制糖蛋白的易位和折叠[65],调节中心碳代谢途径增加细胞内聚糖前体库[66]、对PglB[67]和NGT[68]结构进行工程改造以及改变工程菌株的生长速率[69]来提高糖蛋白的产量;通过RecET直接克隆寡糖合成基因簇并结合PGCT(protein glycan coupling technology, PGCT)技术来加快糖蛋白的生产速率[70];通过开发无细胞糖蛋白合成平台,在体外进行N-糖基化反应[71]来克服体内生产糖蛋白的困难。这些技术手段的运用为大规模生产N-糖蛋白及糖蛋白疫苗提供了保障。
7 结论与展望
迄今为止,已经发现了原核生物的多种N-糖基化途径,并揭示了N-糖基化在原核生物的生命活动中发挥着重要作用。多种病原菌的蛋白N-糖基化修饰在病原菌的黏附及侵袭、蛋白组装和保护性免疫等过程中发挥重要作用。因此,对病原菌N-糖基化修饰的作用机制进行深入研究,既有助于更好地阐明病原菌与宿主的相互作用,也有利于探索新的方式来预防及治疗由相关病原菌引发的疾病,比如当前发展迅速的糖蛋白疫苗就是一个很好的例子。对古菌N-糖基化的研究,一方面可以揭示N-糖基化在微生物响应环境变化中的作用;另一方面古菌复杂的N-糖基化系统可以为糖基化工程系统提供多种糖合成酶类,从而有利于研发出更多在医药方面有重要意义的重组糖蛋白。可以预见,原核生物N-糖基化系统在糖基化工程中的应用将在疫苗生产、疾病诊断和治疗等方面发挥重要作用。此外,之前的研究只在细菌的变形菌门中发现有N-糖基化的存在,最近拟杆菌门Cytophagahutchinsonii中N-糖基化的发现扩大了细菌N-糖基化的研究范围,并且C.hutchinsonii的N-糖基化对蛋白定位和分泌的作用也丰富了对细菌N-糖基化功能的认识,这也说明对原核生物N-糖基化的了解还只是冰山一角,相信随着生物技术的进步,对原核生物N-糖基化系统的认识也会更全面。
随着原核生物基因组学、糖组学和蛋白组学研究的发展,对原核生物蛋白N-糖基化的研究不断增多,但这其中还存在许多未解之谜等待着去解析。因此,未来的研究应集中在以下方面:原核生物N-糖基化蛋白的结构与功能,病原菌N-糖基化与其致病机制的关系,重要的糖基转移酶的特性,构建高效简便的糖蛋白表达平台以生产糖蛋白药物,重组糖蛋白在临床方面的应用,糖蛋白药物的商业化规模生产等,这些都是研究者需要去解决的问题。

