基因加倍对棉花线粒体atpA基因RNA编辑率的影响
张晓,李才华,王婧,张兰兰,牟晓雨,王昕玥,甘刘美,周鹏展,张锐
1.长春理工大学生命科学技术学院,长春130022;
2.中国农业科学院生物技术研究所,北京100081
RNA编辑现象普遍存在于植物线粒体基因组的蛋白编码基因中,并且根据选择压强度的不同而有完全编辑和部分编辑之分。植物线粒体基因组中有的基因是多拷贝基因(加倍基因),那么同一个基因不同拷贝的RNA编辑情况如何,目前尚无相关报道。本团队前期研究发现,棉花细胞质雄性不育(cytoplasmic male sterile,CMS)系和保持系线粒体基因组中atpA基因各有两个拷贝,但每个拷贝的RNA编辑率差异尚不清楚[1]。本研究将分析两系atpA基因每个拷贝的RNA编辑率差异,这对探究植物线粒体基因表达调控(核质互作)、揭示棉花细胞质雄性不育机理具有重要意义。
植物线粒体基因组中的RNA编辑现象特指C-U RNA编辑。它是植物线粒体基因表达的必需步骤之一,属转录后加工范畴,主要发生在编码蛋白的基因中,通常位于密码子的第一、二位,导致编码的氨基酸发生改变[2]。RNA编辑后,表达的蛋白质与其他物种同源性更高[3]。目前已在甜菜[4]、油菜[5]、葡萄[6]、水稻[7]、拟南芥[8]、海枣树[9]、棉花[10]、苏铁[11]等植物线粒体基因组中分别发现了370、427、445、446、456、592、692、1 084个RNA编辑位点。拟南芥456个位点中位于蛋白编码区、非编码区(内含子、5'非翻译区和3'非翻译区)的分别是441、15个[8]。葡萄445个编辑位点中位于蛋白编码区、非编码区的分别是401、44个,位于蛋白编码区的401个位点中76%是部分编辑[6]。
PPR蛋白在RNA编辑中发挥重要作用,它们是植物中比较保守的由串联排列的35个氨基酸构成的一类蛋白[12],是目前公认的反式作用编辑因子[13],参与了细胞器RNA的成熟过程,并组成一个大的家族来负责植物细胞器中所有的RNA编辑事件。近年来,通过对突变体的研究,在拟南芥中已经发现了25种PPR蛋白参与RNA编辑[14-33]。目前,水稻、高粱和玉米等作物中也鉴定出大量的PPR蛋白,它们大多是不同物种间的同源 基 因[34]。PPR蛋 白 以 自 身 做 为RNA编 辑酶[13,35]、或充当RNA编辑酶募集者[36]的方式参与线粒体RNA编辑。
基因加倍是指在同一个基因组内存在2个以上拷贝的同源基因序列,是一种普遍的生物现象。在已完成全测序的生物基因组中,存在大量的加倍基因[37-38],它们在基因组分化和生物进化中发挥重要作用[39]。植物线粒体基因组结构复杂,存在基因重排、重组现象,导致产生较多重复序列,位于长重复序列上的基因便成为加倍基因。不同植物线粒体基因组中的加倍基因数目从一个到十几个不等[40-45]。
本团队研究发现,棉花CMS系和保持系线粒体atpA基因各有两个拷贝:一个是完整的,一个是3'截短的,属于基因加倍现象。但该基因加倍对RNA编辑的影响尚不清楚。本研究拟对两系atpA双拷贝基因进行DNA序列解析和RNA编辑分析,研究基因加倍对RNA编辑率的影响,以期为植物线粒体基因表达调控提供实验数据,并探讨RNA编辑率与棉花细胞质雄性不育之间的关系。
1 材料与方法
1.1 植物材料
陆地棉CMS系P30A,保持系P30B。P30B是陆地棉品种;P30A是P30B与104-7A多代回交得到的高代不育系;P30B与P30A的核背景一致。所有材料种植于大田或人工气候培养箱。
1.2 方法
1.2.1 总DNA提取5 g新鲜叶片用液氮速冻后,快速充分研磨成粉末,转入50 mL离心管中,加入20 mL提取液[100 mmol·L-1Tris-HCl(pH8.0),1.5 mol·L-1NaCl,20 mmol·L-1EDTA(pH8.0),2%CTAB,2%PVP40,2%β-巯基乙醇],65℃水浴40 min,期间温和颠倒混匀数次,然后加入20 mL氯仿∶异戊醇(24∶1)振荡混匀,12 000 r·min-1离心10 min,后续步骤参照张晓等[46]的方法。
1.2.2 Southern blot取40 μg总DNA,分别以300 UEcoRⅠ和HindⅢ(美国Promega公司)充分酶切,0.8%琼脂糖凝胶电泳,经脱嘌呤、变性步骤后,以10×SSC为转膜缓冲液用真空转印仪(Vacuum Blotter 785,美国Bio-Rad公司)将DNA转印到Hybond N+尼龙膜(美国Amersham Pharmacia Biotech公司)上,用紫外交联仪(CL-1000 UV Crosslinker,美国UVP公司)交联1 min(剂量约为0.1 J·cm-2)。探针标记、杂交、检测步骤参照张晓等[47]的方法。
取1 μgatpA基因DNA片段(所用引物见表1中的atpAF和atpAR)为模板进行标记。同时将Gene-ruler DNA ladder Mix SM0331(美国Thermo公司)进行标记做为分子量参照。尼龙膜在杂交炉(美国Hybaid公司)中42℃杂交至少20 h,探针浓度为25 ng·mL-1。X光胶片曝光30 min后冲洗胶片。
1.2.3 反向PCR法结合Tail-PCR法扩增atpA5'和3'侧翼序列 具体方法参照张晓等[1]的方法。
1.2.4 总RNA提取与Northern blot分析 花蕾总RNA的提取采用RNAout Plant试剂盒(Tianze,China)。进行Northern blot实验时,RNA样品在1.2%甲醛变性胶中60 V电泳2 h,后续步骤同1.2.2。
1.2.5 RT-PCR与cRT-PCR RT-PCR实 验 参 照张晓等[46]的方法。RT-PCR所用引物序列见表1。cRT-PCR参照Kuhn等[48]和张晓等[46]的方法,所用特异反向引物为表1中的atpAIF和atpAIR。

表1 研究使用的引物Table 1 Primers used in this study
2 结果与分析
2.1 棉花CMS系和保持系atpA基因结构
Southern blot实验在棉花保持系和CMS系中各获得两个atpA基因杂交条带(图1)。保持系、CMS系的EcoRⅠ限制片段长度分别是2.2和5.1 kb、2.2和3.3 kb;HindⅢ限制片段长度分别是8.5和11.7 kb、9.1和11.7 kb。通过反向PCR和Tail-PCR技术克隆到这些片段的具体序列,结果发现,两系各含有2个atpA基因拷贝:一个是完整的,一个是3'截短型的(图2)。保持系中的完整拷贝和截短型拷贝分别命名为(N)atpA-1(11 689 bp)和(N)atpA-2(8 501 bp);CMS系中相应的拷贝分别命名为(S)atpA-1(11 658 bp)和(S)atpA-2(9 139 bp)。上述4段序列具有相同的5'侧翼区(图2),并且一致性序列至少延伸到atpA起始密码子上游-4 351 bp(HindⅢ位点)处。
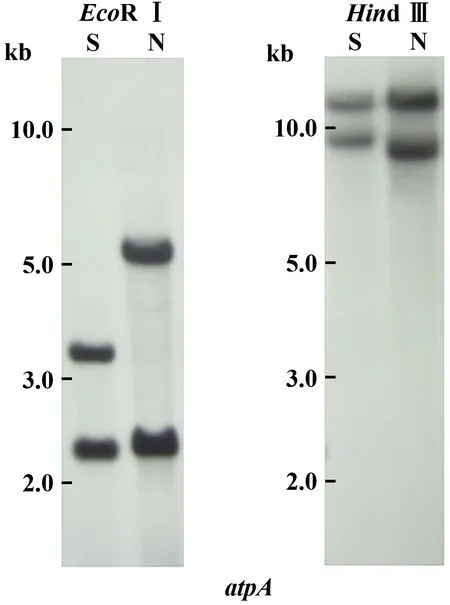
图1 陆地棉细胞质雄性不育系、保持系总DNA的Southern blot分析Fig.1 Southern blot analysis of total DNAs of CMS,maintainer line of Gossypium hirsutum L.
(N)atpA-1和(S)atpA-1含有完整的atpA基因编码序列(1 524 bp),并具有相似的3'延伸区,但是在atpA终止密码子下游161~212 bp区域,存在一个SSR位点。该位点在保持系中是(TAA)7(TA)6,在CMS中是(TAA)3(TA)2。
(N)atpA-2和(S)atpA-2含有3'截短型的atpA基因,但是断点不同:(N)atpA-2是在atpA编码序列的第1 352 bp处截断;(S)atpA-2是在第1 336 bp处截断(图2)。二者序列在atpA编码区一致,但是从断点处往下开始出现长度分别为515和555 bp的特异嵌合序列。N515和S555嵌合序列后面,又分别存在一段2 010 bp(或2 016 bp)的一致序列。在该一致序列之后,又开始出现差异,这段差异序列一直延伸到各自HindⅢ位点处,并继续往下延伸,总长度未知。经ORFfinder预测,(N)atpA-2和(S)atpA-2中atpAORF分别是1 947和1 821 bp,即终止密码子分别位于各自断点下游595、485 bp处。
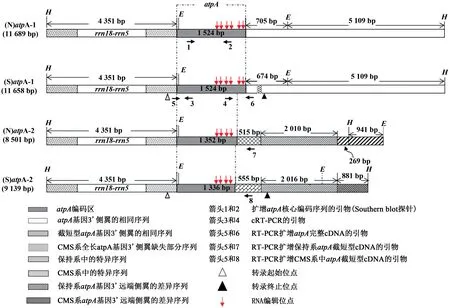
图2 棉花CMS和保持系中atpA基因HindⅢ限制性片段Fig.2 HindⅢrestriction fragments of atpA gene in CMS and maintainer line of cotton.
2.2 atpA基因的转录分析
为研究棉花两系中atpA基因的两个拷贝是否都转录,我们合成了针对每个拷贝的特异引物,并进行RT-PCR分析。结果显示,4个拷贝的cDNA都检测到,这说明尽管3'末端大约1/8的编码区被截短,但(N)atpA-2和(S)atpA-2都能够转录(图3)。两系的atpA全长拷贝都是1 524 bp;截短型拷贝在保持系中预测开放阅读框(open reading frame,ORF)是1 947 bp,在CMS系中预测开放阅读框是1 821 bp。
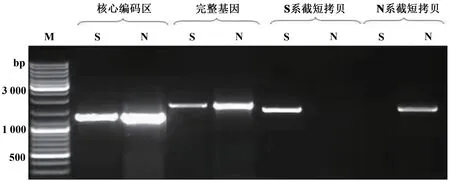
图3 棉花CMS和保持系中atpA基因的RT-PCR分析Fig.3 RT-PCR analysis of atpA gene in CMS and maintainer line of cotton
利用cRT-PCR法克隆到CMS系atpA基因两个拷贝的转录本全长。完整atpA基因的cDNA全长1 807 bp,包含1 524 bp的完整atpA基因。主要的转录起始位点位于-81 bp处(表2),主要的转录终止位点位于+200 bp处(表2)。
CMS系截短型atpA基因的cDNA全长2 150 bp,其中包含一个1 821 bp的ORF,该ORF由1 336 bp的atpA编码序列和其下游485 bp序列组成。主要的转录起始位点位于-81 bp处(表2)。主要的转录终止位点位于终止密码子(TAG)下游+249 bp处(表2)。
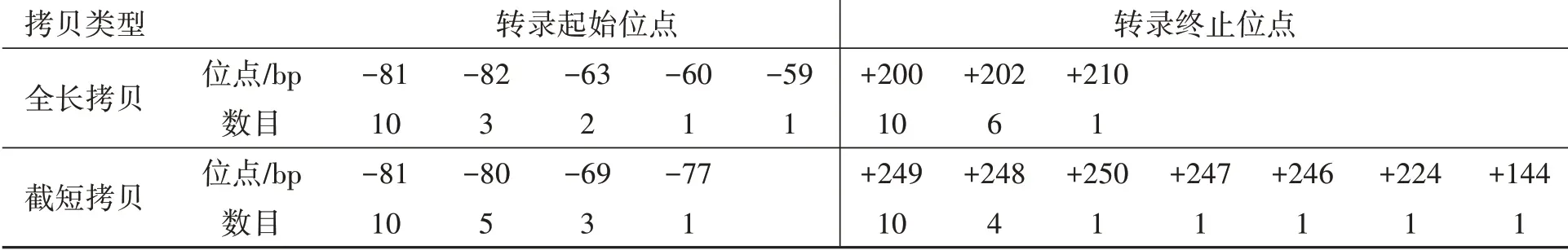
表2 棉花CMS系全长和截短型atpA基因转录起始位点和终止位点情况Table 2 Start and termination sites of transcripts of intact and truncated atpA gene in CMS line of cotton
由此可见,CMS系atpA基因两个拷贝转录起始位点均位于-81 bp附近,表明二者启动子基本相同。
2.3 atpA基因的Northern blot分析
为研究atpA基因的转录本情况,以atpA核心序列为探针进行了Northern blot分析,发现CMS系和保持系均有4个转录本:最长的1个转录本丰度最高、较短的3个转录本丰度均较低,且两系之间带型无明显差异(图4)。这说明虽然不育系P30A、保持系P30B都有特异截短型的atpA转录本,但可能由于电泳时间不够长、转录本的大小差异不明显或者表达量很低,因此用Northern blot方法检测不到差异。

图4 棉花CMS和保持系atpA基因Northern blot分析Fig.4 Northern blot analysis of atpA gene in CMS and maintainer line of cotton
2.4 棉花atpA cDNA中的RNA编辑位点
利用特异引物对P30B和P30AatpA全长和截短型拷贝进行了RT-PCR分析。将cDNA序列和基因组序列进行比对发现,atpA全长拷贝中存在6处C-U RNA编辑位点,它们的核苷酸位置(对应密码子、对应氨基酸位置、对应氨基酸变化)分别是:1 039(CCC-UCC、347、Pro-Ser)、1 064(UCGUUG、355、Ser-Leu)、1 216(CUU-UUU、406、Leu-Phe)、1 292(CCG-CUG、431、Pro-Leu)、1 415(CCACUA、472、Pro-Leu)、1 484(CCA-CUA、495、Pro-Leu)bp处(图2)。这6处位点4个发生在密码子的第二位,2个发生在密码子的第1位。这些位点对应在ATPA蛋白的C末端区域,导致6个氨基酸发生改变,其中3个脯氨酸和1个丝氨酸都变成亮氨酸,使得多肽中更容易形成α-螺旋,同时提高了棉花ATPA蛋白与其他物种ATPA蛋白的相似性。
在提莫菲维小麦、普通小麦[49]、黑小麦[50]、月见草[51]和甜菜[52]线粒体atpA转录本中也分别发现了6、6、6、4、3个RNA编辑位点。不同植物atpA转录本的RNA编辑通常发生在同样的位点,有的没有发生编辑是因为它们在基因组水平上已经完成编辑。例如,第472位氨基酸对应的序列在提莫菲维小麦、普通小麦、黑小麦基因组上已经是棉花编辑后的序列;同样在棉花基因组上第10、324、393位氨基酸对应的序列已经是提莫菲维小麦、普通小麦、黑小麦RNA编辑后的序列(表3)。
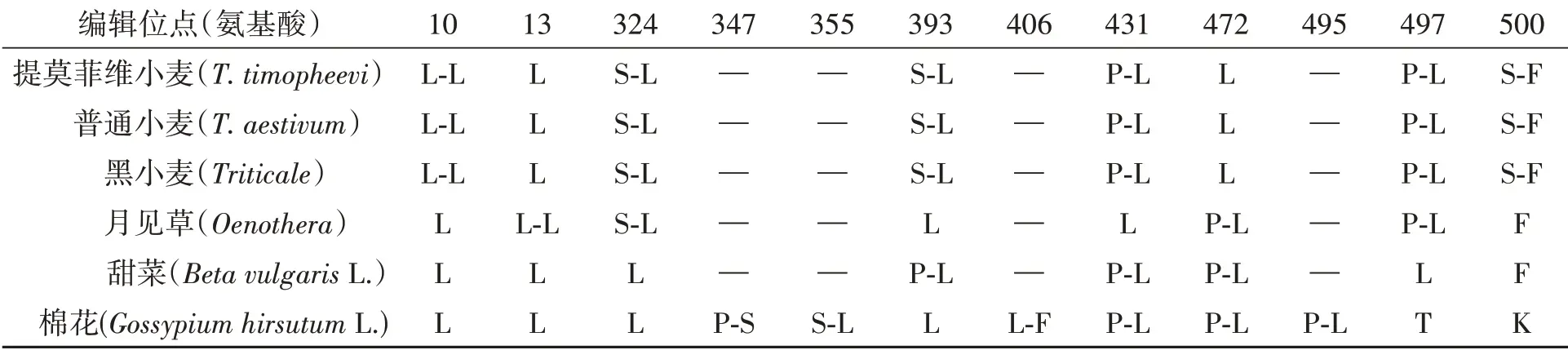
表3 棉花atpA编辑位点与其他植物的比较Table 3 Comparison of cotton atpA editing sites with those of other plants
在两系截短型拷贝中,分别存在4个RNA编辑位点:第1 039、1 064、1 216、1 292 bp。这4个位点与全长拷贝中的完全一样。
同时,在P30A全长atpA拷贝终止密码子下游+78 bp处(3'-UTR区),存在一个编辑位点,该位点的编辑率是41%(7/17),属于部分编辑。
2.5 保持系和CMS系atpA基因RNA编辑率的差异
对cDNA克隆子进行测序发现,棉花atpA基因的RNA编辑率在不同系和不同拷贝间存在明显差异(表4)。
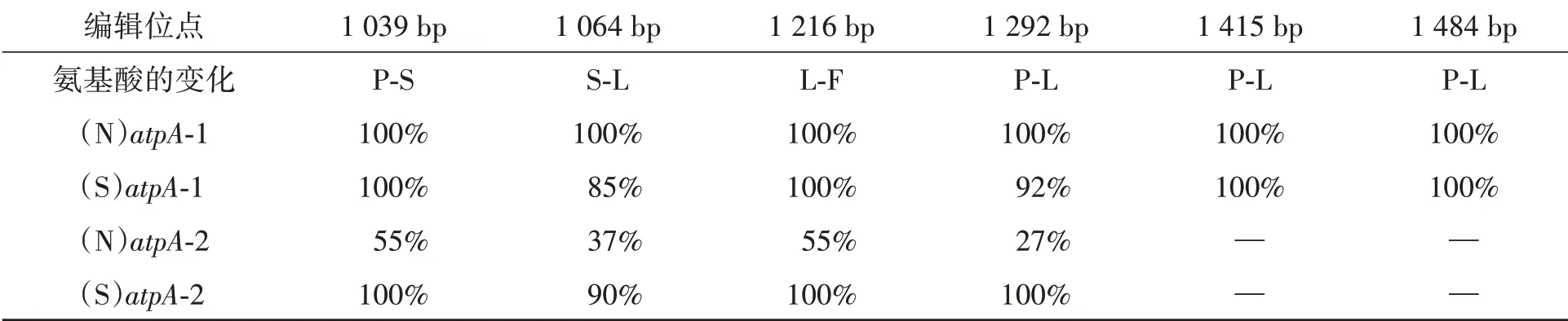
表4 棉花CMS系(S)和保持系(N)atpA基因完整拷贝和截短型拷贝的RNA编辑率比较Table 4 Comparison of RNA editing rates of intact and truncated copies of atpA gene in CMS line(S)and maintainer line(N)of cotton
(N)atpA-1中6个位点的编辑率都是100%,说明保持系中完整atpA基因是完全编辑。(S)atpA-1中6个位点的编辑率分别是100%、85%、100%、92%、100%、100%,即第2(S-L)、4(P-L)位点的编辑率不足100%。这两处编辑位点的作用都是改变氨基酸序列,特别是第4位点,能够将脯氨酸修正为亮氨酸,并且从表3可看出,每一种植物在此处都应是亮氨酸,此处编辑不足,可能导致大约8%的转录本是“不合格”的。这种“不合格率”是否会影响CMS系中线粒体ATP合酶的正常功能尚未知。
(N)atpA-2和(S)atpA-2的RNA编辑率存在明显差异。(N)atpA-2中4个位点的编辑率分别是55%、37%、55%、27%,说明保持系截短型atpA基因是部分编辑,且编辑率特别低。但(S)atpA-2中4个位点的编辑率分别是100%、90%、100%、100%,接近完全编辑,整体编辑率甚至超过(S)atpA-1。特别是第4个位点,完整拷贝中编辑率是92%,截短型拷贝中是100%。
综上,保持系中atpA基因完整拷贝编辑率较高(100%),截短型拷贝编辑率较低(60%以下)。而CMS系中完整拷贝和截短型拷贝编辑率均较高(接近100%)。据此推测,atpA基因RNA编辑率可能与棉花CMS之间有相关性。
3 讨论
高等植物线粒体基因组中的多拷贝基因通常都是位于长重复序列上[53]。本文中的棉花双拷贝atpA基因也是如此。在每个棉花品种中,全长拷贝和截短型拷贝之间的重复序列都在5 800 bp以上。在Genbank中进一步分析发现,该重复序列可能位于与亚洲棉线粒体基因组中的反向重复序列R1(63.789 kb)[54]、海岛棉线粒体基因组中的正向重复序列R1(63.904 kb)[55]、三裂棉线粒体基因组中的正向重复序列R1(12.921 kb)[54]相似的长重复序列上。atpA基因位于长重复序列的3'端,此处是重组或重排位点,CMS系和保持系在此处存在明显序列差异。棉花线粒体atpA基因是研究基因加倍的一个很好的模型,因为两个拷贝的3'末端有特异序列,所以可以设计特异引物对每一个拷贝进行扩增分析。
植物线粒体基因组结构复杂多样,有环形(主环、亚环)、线形等多种形式[56]。Iwahashi等[57]提出水稻线粒体基因组由5个亚环构成,每个亚环都与其他1、2个环共享一段同源重复序列,某些重复序列会引起分子内或分子间重组事件,使DNA主环变为2个亚环分子,或使2个亚环形成一个主环,使线粒体基因组结构呈现异质性[58-60]。目前已公布的棉属植物线粒体基因组都是一个主环结构,推测atpA基因的两个拷贝是位于同一个主环内。棉花线粒体基因组是否存在亚环,以及atpA基因是否有可能位于不同的环状基因组分子中,目前尚无相关报道。
高等植物线粒体基因组中几乎每一个编码蛋白的基因中都存在RNA编辑。RNA编辑位点绝大多数出现在基因编码区,少部分在基因间隔区或非偏码区。编码区的RNA编辑位点基本都是完全编辑,而非编码区的编辑位点通常是部分编辑[8]。这说明,编辑位点的选择遵循“节约”原则。需要编辑的地方因为具有功能所以受到的选择压较大而编辑,不需要编辑的地方因为失去功能所以受到的选择压较小而不编辑,这样的节约原则也影响了基因的表达。
CMS系和保持系中atpA基因各有两个拷贝,通过对P30A中atpA转录本5'末端进行分析表明,完整拷贝和截短型拷贝转录本的5'末端是基本相同的,说明二者启动子基本相同,即启动子对这两个拷贝的RNA编辑率的影响可能不大。
完整atpA基因的RNA编辑位点在保持系和CMS系中相同,且6个位点的编辑率都接近100%。但截短型拷贝的RNA编辑率在保持系和CMS系间差距较大:保持系的编辑率很低,在27%~55%之间;而CMS系的却很高,接近100%。加倍基因的命运通常有新功能化、亚功能化和去功能化3种[61]。保持系atpA截短型拷贝的RNA编辑率特别低,可能说明该基因(加倍基因)的功能可有可无,所受选择压很低,逐步退出调控网络,为亚功能化或去功能化;相反,CMS系中该拷贝的编辑率很高(接近100%),可能意味着该基因仍有明确的功能,所受选择压较大,为新功能化。CMS系中atpA截短型拷贝的RNA编辑率非常充分的原因,即为何CMS系体内要动用这个拷贝、对之施加这么高的选择压让其充分编辑,可能是因为CMS系中完整拷贝的6个RNA编辑位点中有2个位点的编辑率不足100%,导致不能产生足够多的正确的ATPA蛋白,需要再动员截短型拷贝来继续生产ATPA相似蛋白来补充,但具体结论需要我们进一步深入研究。
RNA编辑既受所在基因组序列的影响,也受细胞核的调控影响[62]。本研究内容属于前者,因为P30A和P30B是同核异质系,细胞核是相同的,atpA基因RNA编辑的差异主要受其所在线粒体基因组序列的影响。后续研究可以将恢复系和F1代也引入分析。比如不育系P30A和恢复系的细胞质(包括线粒体基因组)是相同的,但细胞核不同,因此未来可以将两者进行比较,来探究细胞核对RNA编辑的影响。

