基于线粒体Cytb基因和D-loop区的野生与养殖小黄鱼群体遗传多样性
郭丹丹,刘 峰,牛宝龙,楼 宝
(浙江省农业科学院 水生生物研究所,浙江 杭州 310021)
小黄鱼()属鲈形目(Perciformes)石首鱼科(Sciaenidae)黄鱼属(),在我国主要分布于渤海、东海与黄海,其作为我国“四大海产”(大黄鱼、小黄鱼、带鱼、乌贼)之一,是我国近海重要的经济鱼类。由于持续的高捕捞强度,自20世纪80年代以来小黄鱼种群趋于小型化、早熟和低龄化,种质资源面临衰退。在2016年,小黄鱼被列入《世界自然保护联盟濒危物种红色名录》。当前,开展人工繁殖和优良品种选育是减少小黄鱼野生资源被过度开发、破坏,并实现小黄鱼资源可持续利用的有效途径。2016年,楼宝等成功突破了小黄鱼全人工繁育关键技术,目前已实现了百万级小黄鱼苗种规模化繁育。
线粒体DNA(mitochondrial DNA,mtDNA)具有母系遗传、几乎不发生重组、进化速度快与种内多态性高等特点,是研究种属间群体遗传结构和亲缘关系的理想对象。小黄鱼mtDNA包括13个编码蛋白基因(7个NADH脱氢酶亚基:1、2、3、4、4、5、6;3个细胞色素氧化酶亚基:1、2、3;2个ATP酶亚基:6、8;1个细胞色素b基因:)和1个D-loop控制区。不同区域的进化速度不同,基因进化速度适中,适合用于种间和种内群体的遗传结构分析;D-loop控制区处于非编码区,进化速度最快,适用于亲缘关系较近群体间(如种内)的分析。程磊等利用线粒体D-loop区和基因对广西全州、融水、环江三地禾花鲤的遗传结构进行了分析;郑文娟等利用线粒体D-loop区分析了舟山海域带鱼种群的遗传结构;何震晗等基于线粒体D-loop区序列对8个地理群体黄鳍棘鲷的遗传多样性和遗传结构进行了分析;陈浩等基于线粒体基因和D-loop区序列分析了3条水系董氏须鳅的遗传多样性与种群结构。不同学者分别利用线粒体基因或D-loop区序列对我国不同海域野生小黄鱼的遗传结构进行了分析,但目前关于舟山嵊泗海域和象山三门口海域小黄鱼野生群体多样性,以及小黄鱼野生和养殖群体遗传多样性比较的研究尚未报道。因此,本研究基于线粒体基因和D-loop区序列,对舟山嵊泗海域(SS)、象山三门口海域(SMK)2个野生群体和1个养殖群体(YZ)进行了遗传分化与多样性分析,一方面可以丰富我国小黄鱼群体遗传多样性基础资料,另一方面为小黄鱼种质资源保护、利用与遗传育种提供科学依据。
1 材料与方法
1.1 样本采集与DNA提取
象山三门口海域小黄鱼野生群体(SMK)样本采集于浙江省宁波市象山一桥水产养殖专业合作社,舟山嵊泗海域野生群体(SS)和养殖群体(YZ)样本采集于浙江省宁波市象山港湾水产苗种有限公司,每个群体各取30尾。剪取新鲜样本的鳍条组织,并以无水乙醇固定、保存。采用酚-氯仿抽提法提取基因组DNA,经RNase处理后,分别用超微量紫外-可见光分光光度计(Therom Fisher Scientific,NanoDropOne)和琼脂糖凝胶电泳检测基因组DNA的浓度与质量,于-20 ℃冰箱保存备用。
1.2 PCR扩增与测序
根据小黄鱼线粒体基因组信息(GenBank序列号NC_013754.1)获取基因和D-loop区序列,利用Primer premier 5软件分别设计特异性引物进行PCR扩增。基因扩增引物为:Cytb-F,5′-GATGAAACTTTGGCTCCCT-3′;Cytb-R,5′-GATGAAAGGGTGTTCTACTGG-3′。D-loop区扩增引物为:D-loop-F,5′-CCATATCAATGATATCTAAGTACATAGG-3′;D-loop-R,5′-TTCCTGATCAGAAACTTAGGTGAC-3′。引物由生工生物工程(上海)股份有限公司合成。PCR扩增体系均为20 μL,其中包括10 μL 2×PCR mix(HieffDNA Polymerase、dNTP混合物、MgCl等)、8 μL DEPC HO、1 μL DNA模板、0.5 μL上游引物和0.5 μL下游引物。反应程序为:94 ℃预变性5 min;94 ℃变性30 s,56.5 ℃()或55.5 ℃(D-loop)退火30 s,72 ℃延伸30 s,35个循环;72 ℃终延伸10 min,12 ℃保存。扩增产物经1.2%的琼脂糖凝胶电泳检测后,利用ABI 3730测序仪进行双向测序(北京擎科生物科技有限公司)。
1.3 序列处理与数据分析
利用BioEdit 7.0软件对序列进行校对,确保序列的准确性。用Clustal X 1.83软件进行多序列比对后,截取同源序列。用MEGA 5.05软件进行核苷酸组成分析、遗传距离的计算,并用邻接法(Neighbor-Joining,NJ)构建单倍型间的系统发育树。用DnaSP 5.0 软件进行单倍型和遗传多样性分析,统计多态位点数()、单倍型数()、单倍型多样性()、核苷酸多样性()、平均核苷酸差异数()、Tajima’s和Fu and Li's等参数。利用Arlequin 3.5软件进行分子方差分析(AMOVA)、核苷酸错配分析与群体间遗传分化指数(-statistics,Fst)计算。
2 结果与分析
2.1 碱基组成
经引物扩增、测序与序列比对,分别获得了3个小黄鱼群体共90个样本的基因和D-loop区部分同源序列。结果显示:基因同源序列长度为841 bp,其中T、C、A、G 4种碱基所占平均比值分别为26.9%、34.8%、23.3%和15%,A+T含量(50.2%)与C+G含量(49.8%)相似;D-loop区序列经比对后,获得的同源序列长度为629~635 bp,分析发现D-loop区序列比基因更具碱基偏倚性,序列中T、C、A、G 4种碱基所占平均比值分别为28.7%、24.9%、30.2%和16.2%,A+T含量(58.9%)远高于C+G含量(41.1%),这与其他鱼类D-loop区序列碱基组成相似。
2.2 遗传多样性
基于基因,3个小黄鱼群体共确认74个多态位点(),62种单倍型(),整体单倍型多样性()、核苷酸多样性()和平均核苷酸差异数()分别为0.980、0.005 07和4.258。养殖和野生群体相比,YZ群体多态位点数(=22)、单倍型数(=12)、单倍型多样性(=0.857)、核苷酸多样性(=0.004 29)和平均核苷酸差异数(=3.600)略低于SS群体(=40,=26,=0.984,=0.004 85,=4.078)和SMK群体(=42,=27,=0.993,=0.005 53,=4.646)。2个野生群体相比,SMK群体的多态性位点、单倍型数、单倍型多样性、核苷酸多样性和平均核苷酸差异数高于SS群体(表1、表2)。
3个小黄鱼群体D-loop区序列具有66个多态位点(),65种单倍型(),整体单倍型多样性()、核苷酸多样性()和平均核苷酸差异数()分别为0.978、0.011 52和7.073。YZ群体多态位点数(=27)、单倍型数(=10)、单倍型多样性(=0.805)、核苷酸多样性(=0.009 64)和平均核苷酸差异数(=5.984)同样略低于SS群体(=44,=27,=0.993,=0.011 74,=7.301)和SMK群体(=50,=30,=1.000,=0.012 67,=7.832)。2个野生群体相比,SMK群体的多态性位点、单倍型数、单倍型多样性、核苷酸多样性与平均核苷酸差异数高于SS群体(表1、表2)。分析结果表明,3个群体均属于高单倍型多样性(>0.5);此外,YZ群体遗传多样性略低于野生群体(SS和SMK),SMK群体遗传多样性最高,SS群体次之。
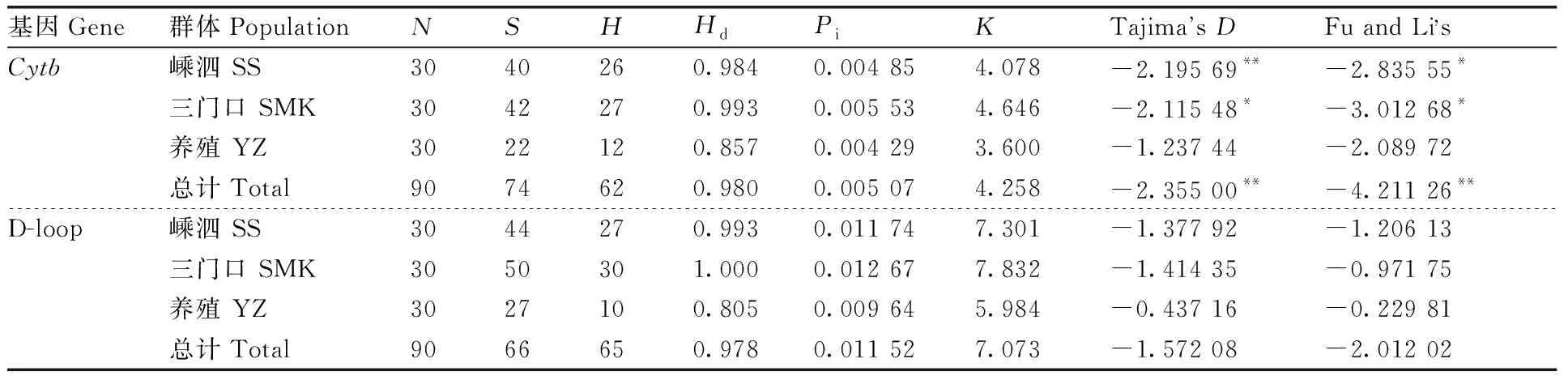
表1 三个小黄鱼群体mtDNA Cytb基因和D-loop区序列遗传多样性参数
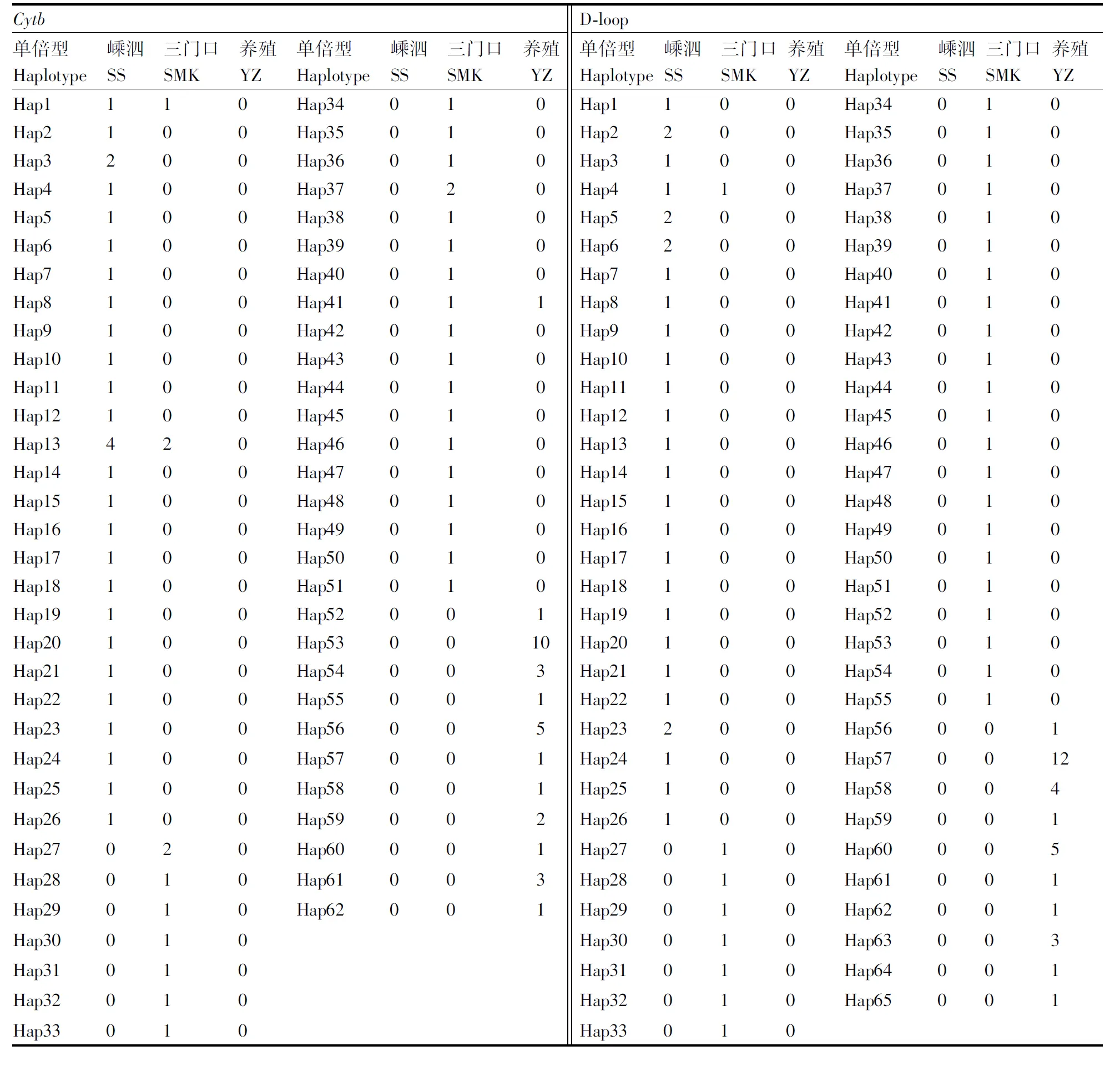
表2 Cytb基因和D-loop区单倍型的群体分布
2.3 中性检验
中性检验结果表明,基于基因和D-loop区序列,SS、SMK和YZ群体的Tajima’s值和Fu and Li's值均为负数,其中,SS和SMK群体基因中性检验的Tajima’s值和Fu and Li's值显著(<0.05)偏离中性(表1);此外,在核苷酸错配分析中,基于序列SS群体呈单峰类型,SMK和YZ群体呈现多峰,而基于D-loop区序列3个群体都呈现多峰(图1)。这说明SS和SMK群体有可能经历过群体扩张。
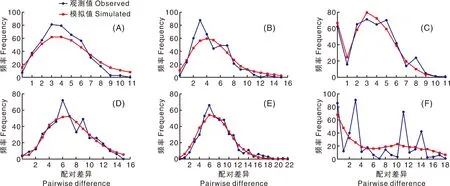
A,SS群体Cytb序列;B,SMK群体Cytb序列;C,YZ群体Cytb序列;D,SS群体D-loop区序列;E,SMK群体D-loop区序列;F,YZ群体D-loop区序列。A, Cytb sequence of SS; B, Cytb sequence of SMK; C, Cytb sequence of YZ; D, D-loop sequence of SS; E, D-loop sequence of SMK; F, D-loop sequence of YZ.图1 Cytb基因和D-loop区序列核苷酸错配分析Fig.1 Nucleotide mismatch analysis of Cytb and D-loop

表3 遗传分化与遗传距离
2.4 遗传分化
基于基因序列,SS、SMK和YZ群体内遗传距离差异较小,分别为0.005、0.006和0.004;群体间遗传距离(SS和SMK、SS和YZ、SMK和YZ)均为0.005。SS和SMK、SS和YZ、SMK和YZ群体间的遗传分化指数分别为0.012 11、0.063 39、0.078 78(表3)。AMOVA分析结果显示,群体间变异占整个变异的5.13%,群体内变异占94.87%(表4)。基于D-loop区序列,SS群体内遗传距离为0.011,SMK群体内的遗传距离为0.013,YZ群体内遗传距离为0.010。群体间遗传距离(SS和SMK、SS和YZ、SMK和YZ)均为0.012,比基因分析获得的遗传距离要远。SS和SMK、SS和YZ、SMK和YZ群体间的遗传分化指数分别为-0.002 30、0.081 04、0.050 51(表3)。群体间变异占整个变异的4.26%,群体内变异占整个变异的95.74%(表4)。由此可见,3个小黄鱼群体的遗传变异几乎全部来源于群体内。
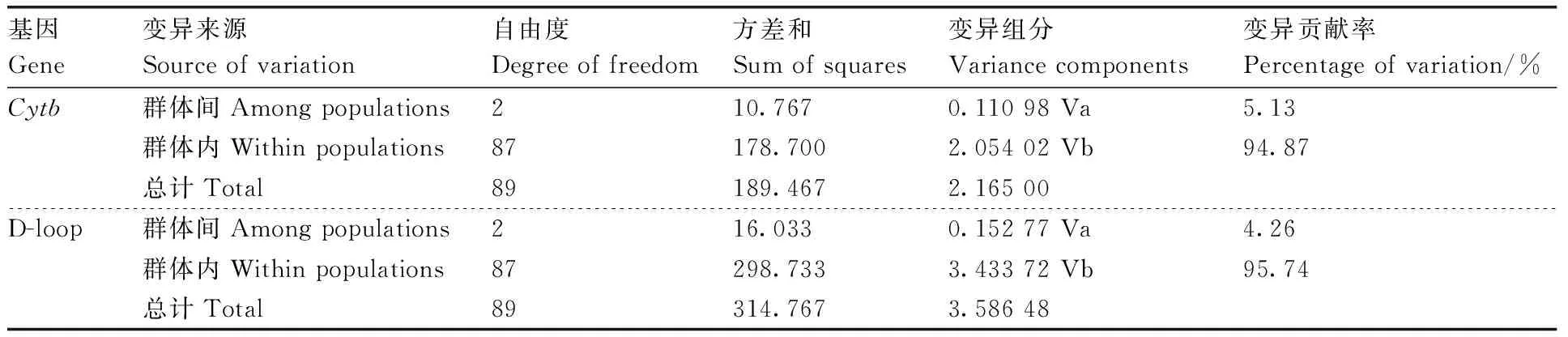
表4 分子方差分析(AMOVA)
2.5 单倍型系统发育树
对角线以下数值为群体间遗传距离,对角线上数值为群体内遗传距离,对角线以上数值为遗传分化指数。
利用邻接法(Neighbor-Joining,NJ),分别基于基因和D-loop区单倍型序列构建系统发育树(图2、图3)。结果显示,SS、SMK和YZ群体均未表现出明显的地理聚集,3个群体间互有交叉。在基因的62个单倍型中,SS和SMK群体共享2个单倍型(Hap1和Hap13),SMK和YZ群体共享1个单倍型Hap41;其中,Hap13、Hap53和Hap56为优势单倍型,24个为SS群体独有单倍型(Hap2-Hap12,Hap14-Hap26),24个为SMK群体独有单倍型(Hap27-Hap40,Hap42-Hap51),11个为YZ群体独有单倍型(Hap52-Hap62)。在D-loop的55个单倍型中,SS和SMK群体共享1个单倍型(Hap4),Hap57和Hap60为优势单倍型,25个为SS群体独有单倍型(Hap1-Hap3,Hap5-Hap26),29个为SMK群体独有单倍型(Hap27-Hap55),10个为YZ群体独有单倍型(Hap56-Hap65)。

图2 基于Cytb基因单倍型序列的系统发育树Fig.2 Phylogenetic tree based on haplotype sequences of Cytb gene
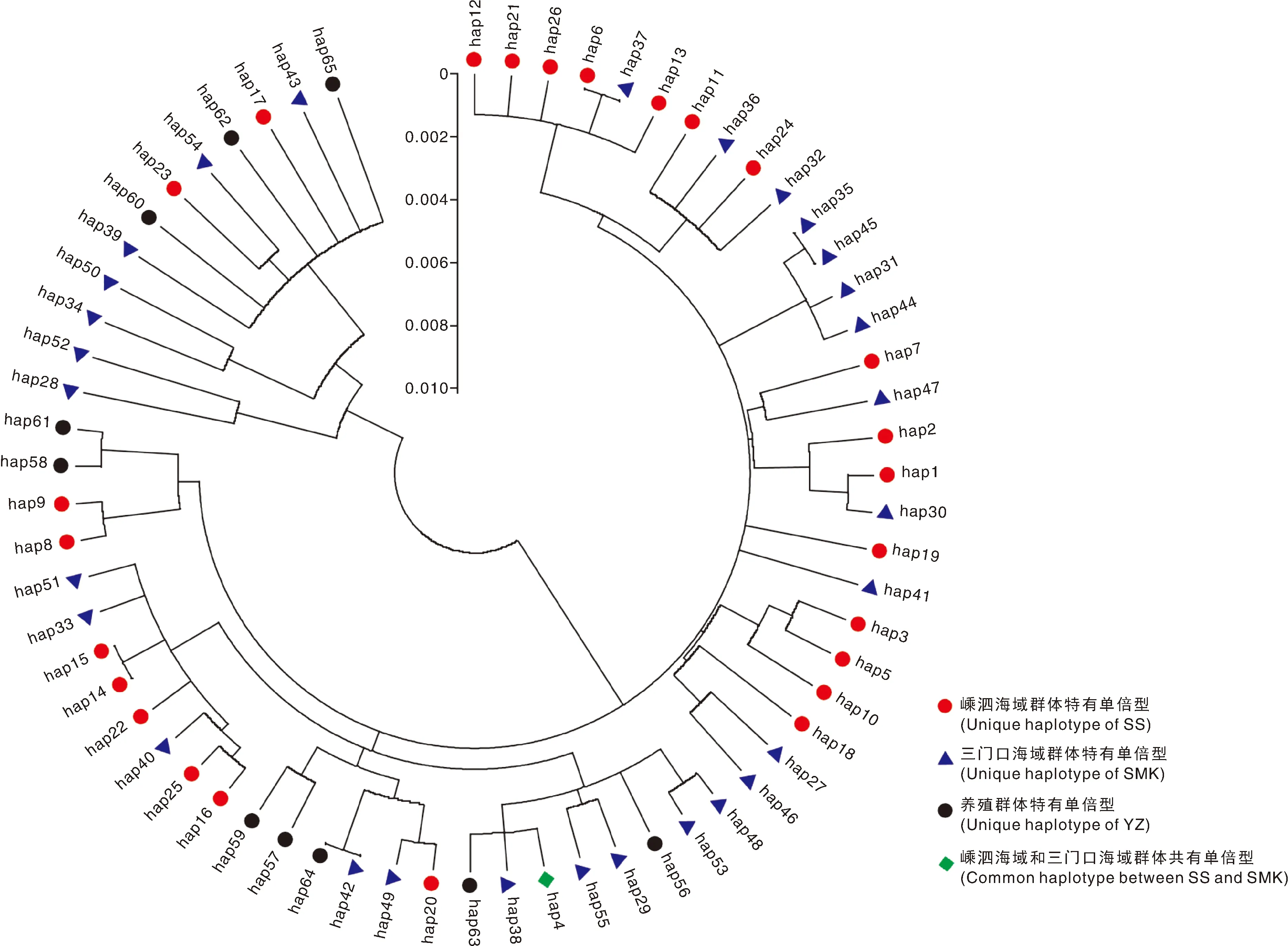
图3 基于D-loop区单倍型序列的系统发育树Fig.3 Phylogenetic tree based on haplotype sequences of D-loop region
3 讨论
本研究通过PCR扩增分别获得了3个小黄鱼群体的基因和D-loop区部分同源序列。基因同源序列中T、C、A、G所占平均比值分别为26.9%、34.8%、23.3%和15%,A+T含量(50.2%)与C+G含量(49.8%)相似,这与吴仁协等的研究结果一致。在郑文娟等对舟山附近海域小黄鱼群体遗传多样性的结果分析中,存在AT碱基远高于CG碱基的现象。同样地,本研究中D-loop区序列中T、C、A、G所占平均比值分别为28.7%、24.9%、30.2%和16.2%,其A+T含量(58.9%)远高于C+G含量(41.1%),表现出明显的AT碱基偏倚性,这符合动物线粒体基因组的共性。
遗传结构的差异存在于物种间与物种内个体间,反映了物种的遗传多样性,是评判物种对环境适应能力与能否长期存在的依据。单倍型多样性和核苷酸多样性是衡量群体遗传多样性的重要指标,单倍型多样性以0.5为临界值,核苷酸多样性以0.005为临界值,数值越大代表群体的遗传多样性越高。在本研究中,基因序列的单倍型多样性和核苷酸多样性分别为0.980(>0.5)和0.005 07(>0.005),D-loop区序列的单倍型多样性和核苷酸多样性分别为0.978(>0.5)和0.011 52(>0.005),表明3个小黄鱼群体总的遗传多样性较高,种质资源较丰富。本文基于D-loop区分析得到3个小黄鱼群体均具有高单倍型多样性和高核苷酸多样性,基于基因分析得到SMK群体属于高单倍型多样性和高核苷酸多样性,但SS和YZ群体属于高单倍型多样性和低核苷酸多样性,这一结论与黄昊的分析结果不符,这可能是由于实验中所用小黄鱼分别采集于不同的地理区域导致的。另外,D-loop区序列与基因的单倍型多样性相似,但D-loop区序列的核苷酸多样性远高于基因序列,此现象可能与D-loop控制区处于非编码区,受到的选择压力较小,变异速率较快有关,这说明mtDNA D-loop区作为反映小黄鱼群体间遗传多样性的敏感度高于基因。基于基因和D-loop区序列分析结果显示,YZ群体的单倍型多样性和核苷酸多样性略低于SS群体和SMK群体;2个野生群体相比,SMK群体单倍型多样性和核苷酸多样性高于SS群体。这表明野生群体小黄鱼多样性略高于养殖群体,SMK群体遗传多样性最高,SS群体次之。
遗传距离和遗传分化指数用来衡量群体间的分化程度。本研究中,根据基因序列分析获得的群体间遗传距离(SS和SMK、SS和YZ、SMK和YZ)均为0.005,根据D-loop区序列获得的群体间遗传距离(SS和SMK、SS和YZ、SMK和YZ)均为0.012。在群体内遗传距离方面,基于D-loop分析的SS、SMK和YZ群体内遗传距离(0.011、0.013和0.010)比基因分析的遗传距离(0.005、0.006和0.004)要远,可能是由于进化速度不同从而导致二者变异的不均一性。从遗传分化指数来看,遗传分化指数为0~0.05表示群体间存在极小分化,遗传分化指数为0.05~0.15表示群体间存在中度分化,遗传分化指数为0.15~0.25表示群体间存在较高的遗传分化。基于基因和D-loop区序列,SS和SMK群体间的遗传分化指数小于0.05,表明2个野生群体间的分化程度极小,这与此前其他不同地理区域小黄鱼野生群体的研究结果一致;而SS和YZ、SMK和YZ群体间的遗传分化指数介于0.05~0.15,表明养殖群体与野生群体间存在中度分化。AMOVA分析结果表明,群体内个体的变异是遗传变异的主要来源。由基因和D-loop区单倍型构建的系统发育树显示,3个群体间互有交叉,并未表现出明显的地理聚集,说明3个群体间的分化尚不明显。从种群历史动态来看,SS和SMK群体基因中性检验的Tajima’s值和Fu and Li's值均为负值且显著偏离了中性选择,此外,SS群体核苷酸错配分析呈单峰趋势,表明SS和SMK群体有可能经历过群体扩张。
综上所述,本研究中野生群体间的遗传分化程度较低,经过长期的驯化,养殖群体与野生群体间出现了中度分化。3个小黄鱼群体均表现出高单倍型多样性,但与野生群体相比,养殖群体的遗传多样性略低。单倍型多样性和核苷酸多样性较高的种群被认为可能是由一个大而稳定的种群经过长时间演化而来或由两个不同系群的种群二次接触所形成,而单倍型多样性和核苷酸多样性较低的种群可能发生了瓶颈效应,我们推测养殖群体遗传多样性较低可能是由于近期发生了瓶颈效应。因此,在育种过程中应适时补充不同群体作为后备亲鱼,以防止近亲繁殖与瓶颈效应,保持繁育群体的遗传多样性。本研究结果不仅丰富了我国小黄鱼野生群体遗传多样性基础资料,而且对小黄鱼野生资源的保护与合理利用,尤其对小黄鱼的遗传育种提供了参考依据。

