樟子松优树群体遗传多样性评价及指纹图谱构建
王辉丽,于树学,郭立,梁海龙,李伟
(1.北京林业大学生物科学与技术学院,林木育种国家工程实验室,林木花卉遗传育种教育部重点实验室,树木花卉育种生物工程国家林业和草原局重点实验室,北京 100083;2.河北省丰宁千松坝林场,河北 承德 068350)
樟子松(Pinussylvestrisvar.)为松科(Pinaceae)松属(Pinus)植物,是我国珍贵的沙地树种[1-2],具有抗逆性强、生长快、成材早、耐寒性和深根性等特性[3]。樟子松是我国北方地区重要的水土保持、防风固沙和水源涵养树种[4],也是东北地区主要速生用材和庭园观赏树种[5],被广泛的应用于三北防护林建设中[6],具有极高的生态效益、经济效益和社会效益[7]。从20世纪90年代起育种工作者开展樟子松遗传多样性研究,初期主要以表型测定和地理变异研究为主,秦泅华等[8]研究了樟子松生长性状的地理变异规律,表明樟子松纬向呈现负向渐变趋势,经向呈现明显的随机变异规律;肖杰等[9]通过培育不同地区樟子松幼苗并统计表型变化,认为樟子松种源间种源内都存在丰富的变异。近年来,随着分子标记技术的发展,樟子松遗传多样性研究进入了分子标记时代,周志强等[10]利用ISSR技术分析了天然樟子松林在不同海拔和群落下的遗传多样性,发现海拔和纬度差异一定程度上影响了樟子松遗传分化,而不同的群落类型对樟子松群体的遗传多样性影响较低;苗禹博等[11]利用SSR标记技术分析不同地区樟子松育种资源的遗传多样性参数以及遗传差异,发现不同樟子松群体之间的空间分布与遗传距离呈现明显的正相关关系。目前,樟子松遗传改良正处于升级换代的关键时期,在高世代种子园营建过程中,樟子松引种信息不清楚,以半同胞子代为基础的高世代育种材料亲缘关系难鉴别,高世代育种材料偏少等问题不可避免产生,这导致出现樟子松高世代种子园近交水平显著上升,近交衰退的风险增加,樟子松高世代种子园种子的遗传多样性降低等。因此,开展樟子松遗传多样性评价,建立一套快速、准确的鉴定方法,能够为樟子松引种、营建种子园以及遗传改良提供理论依据和奠定基础。
简单重复序列(simple sequence repeats,SSR)分子标记具有灵敏度高、多态性好、稳定可靠等优点[12-13],是遗传多样性分析及群体亲缘关系区分的标记方法之一[14],被广泛应用于品种鉴定、多样性分析、指纹图谱的构建等研究中[15]。指纹图谱能够利用植物某个基因型特有标记对其进行准确的鉴定,是亲本鉴定的依据,也是遗传多样性的基础[16],近些年在林木的应用也越来越多。刘超凡等[17]以141份杨树无性系为材料利用筛选出的14对引物进行遗传多样性分析,构建其DNA指纹图谱;毛秀红等[18]以49份刺槐种质为材料利用8对核心引物进行了遗传多样性分析,并完成了DNA指纹图谱的构建;雷阿娜等[19]利用22对多态性高的引物构建黄帝手植柏DNA指纹图谱;赵阿风等[20]利用8对多态性较高的引物分析了120份马尾松无性系遗传多样性,成功构建其DNA指纹图谱。而目前针对樟子松研究主要集中在遗传多样性、遗传关系的分析[21],而利用SSR分子标记对樟子松育种资源进行指纹图谱构建鲜见报道。
本研究以红花尔基天然林和丰宁、围场、章古台人工引种林,4个地区的樟子松群体作为研究对象,利用本课题组开发的12对樟子松特异SSR分子标记[22],计算优树群体的遗传多样性参数,构建不同地区樟子松优树SSR指纹图谱,分析不同地区优良单株的遗传多样性差异,为樟子松的种子园营建、遗传改良等方面提供理论依据,促进SSR分子标记在樟子松育种工作中的应用。
1 材料与方法
1.1 试验材料
材料为红花尔基(HHEJ)、章古台(ZGT)、围场(WC)、丰宁(FN)共4个地区的樟子松优良单株,其中章古台、围场、丰宁的种源来自红花尔基地区天然林的混杂种子,具有相同的遗传背景,人工林建成后,没有进行人为选择干预,所有引种樟子松均在自然选择的压力下生长、繁殖、筛选、淘汰。基本信息情况见表1。2019年10~11月,在材料采集地选取樟子松当年生松针8~10针,装入离心管中迅速置于液氮,做好标签,放入-80℃超低温冰箱中保存备用。

表1 樟子松试验材料采集地基本信息Table 1 Basic information of collection regions of Pinussylvestris var.mongolica
1.2 樟子松DNA提取与PCR扩增
所有实验材料的针叶DNA均采用艾科瑞生化科技有限公司植物基因组DNA提取试剂盒(货号AG21011)按照说明书进行提取,-20℃保存备用。PCR反应体系总体积21μL,包含1μL基因组DNA,0.3μL上游引物,1μL下游引物,0.7μL荧光引物,10μL Mix,8μL ddH2O。PCR扩增反映程序:95℃预变性5 min;95℃30 s,最适退火温度(55℃)30 s,72℃延伸30 s,10个循环;95℃30 s,最适退火温度降低2℃(53℃)30 s,72℃延伸30 s,25个循环;72℃延伸10 min;4℃保存。引物具体信息见表2。

表2 樟子松SSR多态性引物Table 2 Polymorphic primers of SSR in Pinussylvestris var.mongolica
1.3 数据分析
PCR扩增产物由北京睿博兴科生物技术有限公司用毛细管电泳法检测,利用Gene marker 2.2.0软件进行峰值读取,Excel记录整理数据;用Ge⁃nALEx 6.5软件计算平均等位基因数Na、有效等位基因数Ne和Shannon信息指数I;用power⁃makerV3.25软件计算多态性信息量PIC;利用Pow⁃erMarkerV3.25软件计算基于等位基因频率的Nei1983距离,并对Nei1983距离进行UPGMA聚类,用MEGA7.0.14查看聚类结果,根据聚类结果分析亲缘关系情况。根据每对SSR引物的带型结果,组合形成每个无性系的SSR指纹图谱,根据指纹图谱的差异,再区分樟子松优良单株。
2 结果与分析
2.1 12对引物遗传多样性分析
利用12条引物扩增的SSR位点进行计算,99个样本中共扩增出35个等位基因,其中每个位点平均扩增出5.25个等位基因,lw_isotig27940引物为最多,有15个等位基因,最少的是引物lw_isotig05123,只有2个等位基因;有效等位基因数(Ne)为1.313~10.574,平均值为3.112;Shannon’s信息指数在0.339~2.461范围内波动,平均值为1.124;不同位点的PIC值在0.202~0.932范围之间变化波动很大,平均PIC值为0.558(表3)。根据Botstein(1980)的理论[23],有1个标记为低度多态位点(PIC<0.25),4个标记为中度多态位点(0.25

表3 12个引物的SSR位点多态性分析Table 3 Polymorphic analysis of SSR locus of 12 primers
2.2 樟子松优树群体间的遗传关系分析
聚类分析是将育种资源根据它们综合性状之间的相近程度进行分类,以4个樟子松育种群体的遗传相似系数为依据,采用UPGMA法进行聚类分析,分别得到供试樟子松群体和个体的遗传关系聚类图。聚类结果显示4个群体被分为3大类群(图1)。其中,围场、丰宁2个群体遗传距离最近,两者聚为一类;其余2个种群各为一类,而红花尔基群体遗传距离相对较远,距离最远的群体是章古台群体,说明章谷台群体差异较大。从个体聚类图中可以发现(图2),各优株间都存在着不同程度的亲缘关系,各地区樟子松材料并没有明显被区分开,分散在每个类群中,来源地相同的个体并没有根据亲缘关系相似而聚为一类,表明聚类结果与樟子松种质的地域来源并没有显著的相关性。
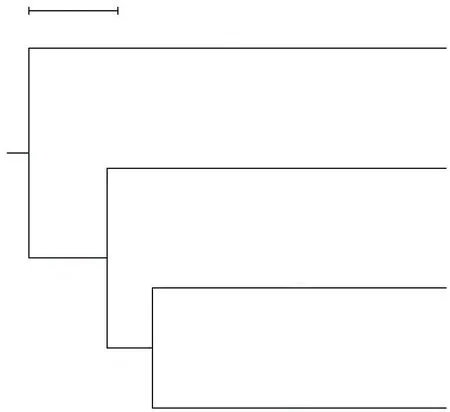
图1 4个地区的樟子松遗传相似系数聚类图Figure 1 Cluster diagram of genetic similarity coefficient of 4 regions in Pinussylvestris var.mongolica

图2 樟子松优良单株遗传相似系数聚类图Figure 2 Cluster diagram of genetic similarity coefficient of excellent individuals in Pinussylvestris var.mongolica
2.3 樟子松优树指纹图谱构建
构建种质DNA指纹数据库,最有效便捷是分子标记引物组合法[24]。所有樟子松个体并未有明显特征谱带,无法通过1对引物将其全部区分开,因此需要利用引物组合进行区分(图3~6)。结果发现,发现引物lw_isotig27940和lw_isotig21953组合的分辨率最高,能够鉴别40份樟子松优良单株材料,分辨率为40.4%;使用引物lw_isotig17679、lw_isotig27940和lw_isotig21953组合,能够区分58份樟子松优良单株,分辨率为58.5%,使用引物lw_isotig17679、lw_isotig27940、lw_isotig21953和lw_isotig00080组合,能够区分72份樟子松优株,分辨率为72.7%。最终选取7对多态性信息含量高的引物lw_is⁃otig17679、lw_isotig21953、lw_isotig27940、lw_is⁃otig04306、lw_isotig00080、lw_isotig20215、lw_is⁃otig11166进行组合用来构建樟子松SSR指纹图谱,分辨率为100%。7对引物构建樟子松优株的唯一的指纹图谱。

图3 41份围场樟子松优良单株的指纹图谱Figure 3 Fingerprint of 41 excellent individuals of Pinussylvestris var.mongolica in WC

图4 21份丰宁樟子松优良单株的指纹图谱Figure 4 Fingerprint of 21 excellent individuals of Pinussylvestris var.mongolica in FN

图5 21份红花尔基樟子松优良单株的指纹图谱Figure 5 Fingerprint of 21 excellent individuals of Pinussylvestris var.mongolica in HHEJ
3 讨论
种群的遗传多样性决定了一个种群是否能够适应复杂的环境,遗传多样性越高种群对不同环境的适应性越强,就越能抵抗环境变化所产生的冲击[25]。而亲本的遗传多样性会直接影响无性系造林、母树林、采穗圃以及种子园营建等方面。本研究利用12对SSR引物对4个群体共99份樟子松优良单株材料进行遗传多样性分析,共检测到35个等位基因,平均每个位点扩增出5.25个等位基因和3.112个有效等位基因,与王思琪[26]在红花尔基地区挑选的3个群体(平均等位基因数分别为1.154、1.202和1.196)相比,各群体的遗传组成更复杂,遗传多样性更高,也高于苗禹博[11]利用SSR标记得到的4个樟子松群体结果(平均等位基因数分别为1.852 4、1.849 8、1.999 8和2.110 5)。99份材料不同位点的Shannon’s信息指数为1.124,PIC值变化差异较大,在0.202~0.932之间,平均PIC值为0.558,也证明不同单株材料间具有较高的遗传多样性,具备一定的遗传改良育种潜力,可为营建高世代种子园提供优良丰富的原材料。同一地区的种群内变异大,说明种群内杂交程度较高,河北丰宁、河北围场以及辽宁章古台地区的人工林中林分仍有进一步选择疏伐的空间,人工引种林应采取多无性系间隔搭配以及适宜的栽培管理措施,与内蒙古红花尔基天然林内的自然杂交程度相接近。同时本研究所使用的引物从分子水平上为樟子松群体遗传多样性分析提供了参考价值,也可以为其他针叶树多样性研究提供可靠的引物资源和理论依据。

图6 16份章古台樟子松优良单株的指纹图谱Figure 6 Fingerprint of 16 excellent individuals of Pinussylvestris var.mongolica in ZGT
UPGMA聚类分析结果显示,4个群体被分为3大类群。其中,河北围场和丰宁2个群体聚成一类,遗传距离相对较近;内蒙古红花尔基群体和辽宁章古台群体各自为一类,与河北围场和丰宁遗传距离相对较远。分析结果符合地区相近遗传关系较近的规律,由于地理距离差异会存在基因交流的限制,不同种群在不同生境的长期适应最终导致各地区种群间差异大、种群间遗传分化强烈。个体聚类结果则显示各地区99份樟子松材料并没有明显被区分开,来源地相同的个体并没有根据亲缘关系相似而聚为一类。这可能由于个体间发生的遗传变异在4个地区群体中均存在或存在相似遗传变异,总体遗传变异的比例、模式相近,所以在聚类过程中不同群体内的个体并不会因为地区差异而聚成一类。
4 结论
SSR分子标记特异性强、谱带清晰、数据准确,适合大量资源的指纹图谱构建工作[27]。本研究利用12对SSR引物对4个地区种群的99份樟子松优良单株分别构建指纹图谱,由于樟子松优良单株的多态性位点丰富,1条引物无法对不同个体进行鉴别。利用2对引物以上组合可以将4个种群的个体鉴定出来,不同引物组合在鉴定不同种群时有不同的分辨 率,利 用lw_isotig17679、lw_isotig21953、lw_is⁃otig27940、lw_isotig02842、lw_isotig00080、lw_is⁃otig05123、lw_isotig02138进行组合可以将99份材料鉴定出来,分辨率为100%。樟子松指纹图谱能够使每份优良单株具有唯一的一套DNA指纹,这可为樟子松种质资源鉴定、管理与保护奠定基础和提供技术支撑,为樟子松的引种、种子园营建和遗传改良提供科学的理论依据。

