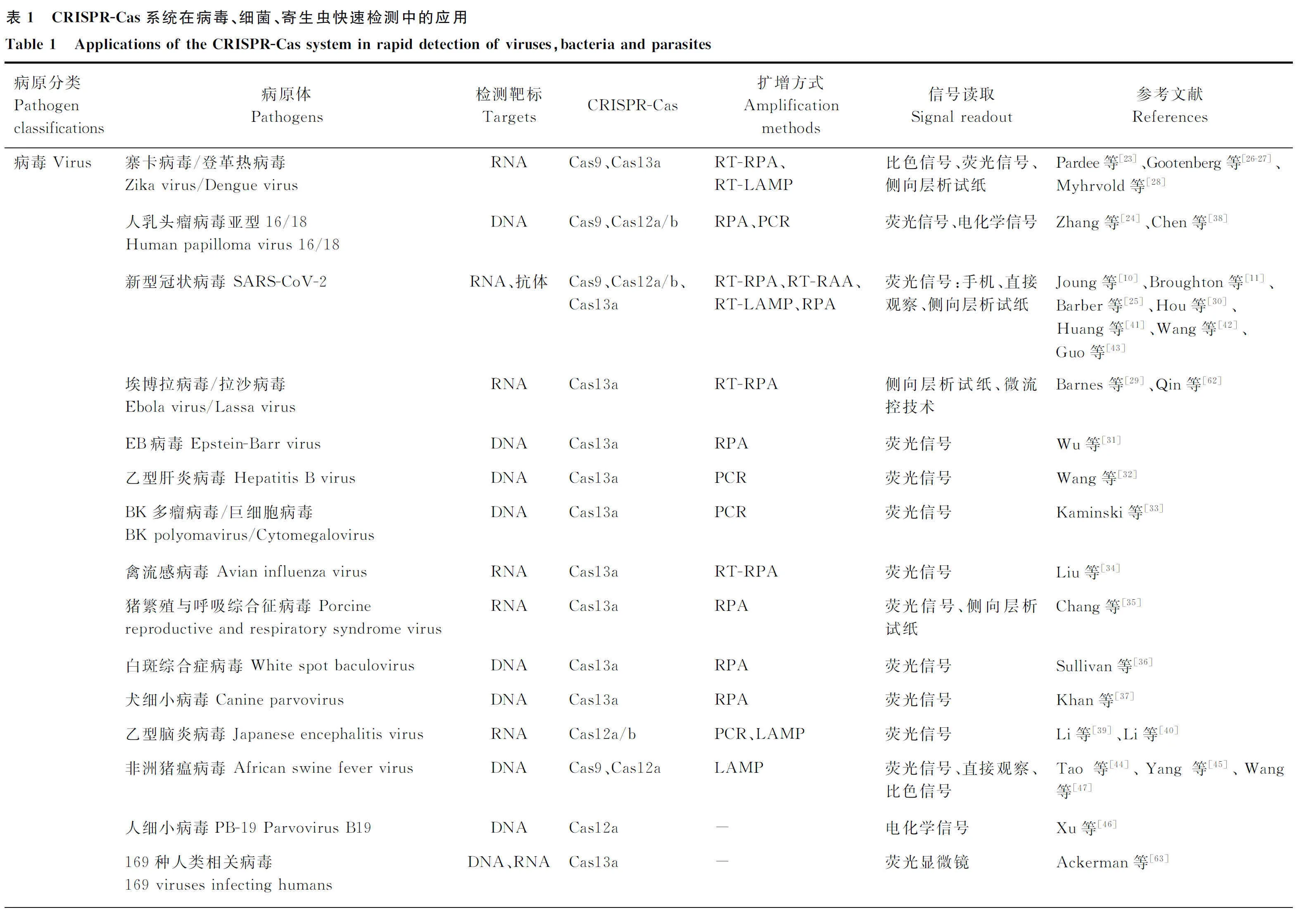
基于CRISPR-Cas系统的病原体检测研究进展
张庆勋,钟震宇,郭青云,何宏轩,白加德
(1.北京麋鹿生态实验中心,北京 100076;2.北京生物多样性保护研究中心,北京 100076;3.中国科学院动物研究所,北京 100101)
近年来,新发突发人兽共患病、动物疫病如新型冠状病毒(SARS-CoV-2)、寨卡病毒、埃博拉病毒、甲型流感病毒、非洲猪瘟病毒等,严重威胁公共卫生及兽医卫生安全[1-2]。快速、灵敏、特异的检测方法对于传染病的防控至关重要[3-4]。目前动物病原体常见的检测方法包括基于抗原抗体的免疫学方法、聚合酶链式反应(PCR)和实时荧光定量PCR方法、基因组测序方法等[5-8],但是这些方法存在一定的局限性,如非特异性的干扰、耗时、价格高、对设备的依赖性强等。病原体检测的最为理想的方法应当是快速、灵敏、特异、经济实惠、设备自由且直观的方法。
CRISPR-Cas系统源自细菌的适应性免疫系统,因其识别和剪切特定DNA或RNA序列的能力而在基因编辑领域得到广泛研究。除基因编辑功能外,Cas核酸酶(如Cas12和Cas13)在特异识别和结合靶标核酸后,表现出强烈的附属切割活性,即非特异地切割周围的RNA或DNA[9]。目前,基于CRISPR-Cas13a的特异高灵敏度酶报告系统(SHERLOCK)[10]以及基于CRISPR-Cas12a的SARS-CoV-2快速检测方法(DETECTR)[11]已被开发出来,并且相关的SARS-CoV-2检测试剂盒已获得美国食品药品监督管理局(FDA)的紧急使用授权。基于CRISPR-Cas系统的诊断方法作为新一代的诊断技术,表现出灵敏度高、特异性好等诸多优点,并且可以实现快速即时检测,在病原体检测方面具有巨大应用前景。作者将对CRISPR-Cas系统类型及其在病原体快速检测中的最新进展展开论述。
1 CRISPR-Cas系统类型及应用
CRISPR-Cas系统是细菌的一种RNA引导的适应性免疫系统,通过RNA引导Cas核酸酶与特定的核酸序列结合并切断该序列,用以抵抗外源DNA或病毒的入侵[12]。CRISPR-Cas系统可分为两大类、6种型、33种亚型。类别1包括Ⅰ、Ⅲ、Ⅳ型及16个亚型,主要由多个Cas核酸酶组成的复合物发挥作用。与类别1不同,类别2包括Ⅱ、Ⅴ、Ⅵ型及17个亚型,仅需要1个大的多功能结合域的Cas核酸酶。其中Cas9存在于所有Ⅱ型系统,Ⅴ型和Ⅵ型CRISPR-Cas系统均包含多种变体,Ⅴ型主要由DNA酶Cas12a、Cas12b和Cas14组成,Ⅵ型包括Cas13a和Cas13b,其只能切割靶标RNA序列[13-15]。
在CRISPR-Cas9系统中,CRISPR RNA(crRNA)会与反式激活crRNA(tracrRNA)结合形成指导RNA(gRNA),目前的研究已将二者整合为单一向导RNA(sgRNA)。Cas9蛋白在sgRNA的协助下精准定位到原间隔序列临近基序(PAM)靶位点,最终完成对双链DNA的精准切割(图1)。目前,CRISPR-Cas9系统已成为强有力的基因编辑工具,广泛应用于各个领域[16]。在Ⅴ型CRISPR-Cas系统中,tracrRNA能够干扰Cas12b和Cas12c,但不影响Cas12a。Cas12a在crRNA介导下靶向切割富含T碱基的PAM区的双链DNA(dsDNA),同时激活Cas12a的单链DNase活性,进而以非特异性的方式切割非靶标单链DNA(ssDNA)[17]。基于Cas12a的这种特性,CRISPR-Cas12系统被广泛应用于DNA的检测。与Cas12不同的是,Cas13和Cas14核酸酶crRNA介导下分别靶向切割目标单链RNA(ssRNA)和ssDNA,核酸酶被激活后通常表现出附属切割活性,切割非特异性ssRNA和ssDNA(图1)[18-19]。此外,Cas13和crRNA复合体切割ssRNA时,不需要tracrRNA和PAM区,但是其与ssRNA的结合需要原型间隔序列(即PFS区)[15]。
随着新的CRISPR-Cas12/Cas13系统的发现及其作用机理的逐步揭示,这些系统实现了更加精准高效的基因编辑。鉴于CRISPR-Cas12/Cas13在DNA及RNA检测中的优势,这两种系统被广泛应用于病原体快速诊断技术的研究。其中最为经典的是基于CRISPR-Cas13a的SHERLOCK以及基于CRISPR-Cas12a的DETECTR,这两种检测方法都是利用荧光信号强度来表征检测结果[10-11]。简单来说,首先构建FAM及BHQ荧光基团标记的非靶标ssRNA或dsDNA报告基因;在系统识别了目标基因以后,Cas13和Cas12的附属切割活性被激活,进而切割非靶标ssRNA或dsDNA报告基因,最终通过多功能酶标仪读取样本荧光强度。相较于传统的检测方法,CRISPR-Cas系统表现出快速、灵敏度高、特异性好、适用范围广等诸多优点,在肿瘤、单核苷酸多态性(SNP)及遗传病、病原体(病毒、细菌、寄生虫)、耐药基因、有机小分子、金属离子、水质、公共卫生等检测方面得到了广泛的应用(图1)[20-22]。
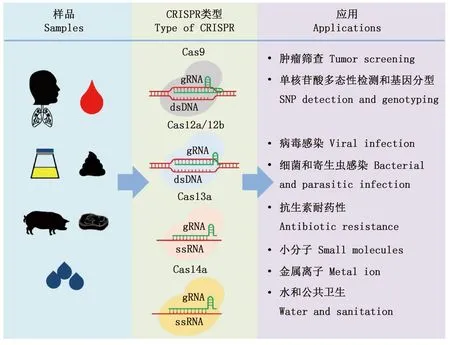
图1 CRISPR-Cas系统在快速检测中的应用Fig.1 Applications of CRISPR-Cas system in rapid detection
2 CRISPR-Cas病原体快速检测技术
2.1 CRISPR-Cas系统在病毒检测中的研究
CRISPR-Cas系统在新发突发传染病、人兽共患病、动物疫病检测中得到广泛的应用。Pardee等[23]开发了一种基于RNA等温扩增及传感器相结合的CRISPR裂解方法,并且成功从血液样本中检测具有单核苷酸多态性差异的美国和非洲寨卡病毒(ZIKA)毒株。Zhang等[24]构建了基于CRISPR-Cas9的HPV16/18的检测及分型方法,该方法首先通过PCR方法进行扩增,而后基于CRISPR-Cas9切割的荧光信号鉴定PCR产物。此外,科研人员通过改造CRISPR-Cas9系统开发了一种新型蛋白质研究技术(PICASSO),该方法将经修饰、无催化活性的Cas9蛋白(Cas9识别DNA序列但不进行切割)与定制的肽复合物相连,利用sgRNA引导该复合物与DNA自组装,用此方法构建的DNA-蛋白质微阵列能够快速识别样本中的上千种抗体。基于这种特性,PICASSO成功检测了新型冠状病毒肺炎康复者的血液中是否存在与病原体蛋白质结合的抗体[25]。
Cas13a是目前发现的唯一一个靶向ssRNA的核酸酶,并且Cas13a附属切割活性也仅针对ssRNA。Gootenberg等[26]开发出基于CRISPR-Cas13a的SHERLOCK系统后,首先将其运用于ZIKA和登革热病毒(DENV)的检测中,该方法的检测灵敏度达到阿摩尔(aM)级别。为了进一步扩展该方法的应用,研究人员对该方法进行了优化,建立了SHERLOCKv2系统。此方法利用CRISPR Ⅲ类效应器核酸酶Csm6来提高不同细菌种类Cas13酶的附带切割活性,增加其检测灵敏性;同时依托FAM-生物素系统建立了侧向层析试纸条,适用于现场的核酸可视化快速检测方法[27]。为了使检测过程更加便捷,研究者将SHERLOCK与一种对未提取的诊断样品进行加热来消除核酸酶的方法(HUDSON)联用,免去了核酸提取的步骤,实现了对DENV、埃博拉病毒、拉沙病毒的快速检测[28-29]。之后,研究人员在原有的技术基础上建立了STOPCovid.V2法,通过磁珠优化RNA的提取,不仅缩短RNA提取时间,同时提高了方法的检测灵敏度,在临床样本的检测中,该方法能够达到93.1%的灵敏度和98.5%的特异性,并且阳性样本只需15~45 min即可获得检测结果[10],目前该方法已经通过FDA批准授权使用。此外,多项研究将CRISPR-Cas13a用于SARS-CoV-2、Epstein-Barr病毒(EBV)、乙肝病毒(HBV)、BK多瘤病毒/巨细胞病毒(BKV/HCMV)、禽流感病毒(AIV)、猪繁殖与呼吸综合征病毒(PRRSV)、白斑综合症病毒(WSSV)、犬细小病毒(CPV)、非洲猪瘟病毒(ASFV)等病毒的快速检测技术研发[30-37](表1)。
CRISPR-Cas12是一种强大的基因编辑系统,研究发现毛螺旋菌科细菌(Lb)的Cas12a能够在crRNA的介导下靶向切割dsDNA或ssDNA,并且触发附属非特异性切割ssDNA荧光报告基因的能力。Chen等[38]利用LbCas12a与RPA联用建立了适用于DNA检测的DETECTR系统,该系统被首先运用于人乳头瘤病毒(HPV)的检测,能够有效检测出临床的高危亚型HPV16/18。在基于Cas12a建立JEV检测方法HOLMERS的基础上[39],Li等[40]利用Cas12b与LAMP反应联用构建了HOLMERSv2方法,该方法利用依赖RNA的DNA聚合酶,去除了逆转录的过程,使JEV的检测更加便利。进一步研究发现,Cas12a/b能够同时检测DNA和RNA靶标,基于Cas12a/b系统与多种核酸扩增方法(如RT-RPA/RT-LAMP等)联用开发出了SARS-CoV-2、ASFV、PB-19/CPV等病毒的检测方法[41-46]。
2.2 CRISPR-Cas系统在细菌检测中的研究
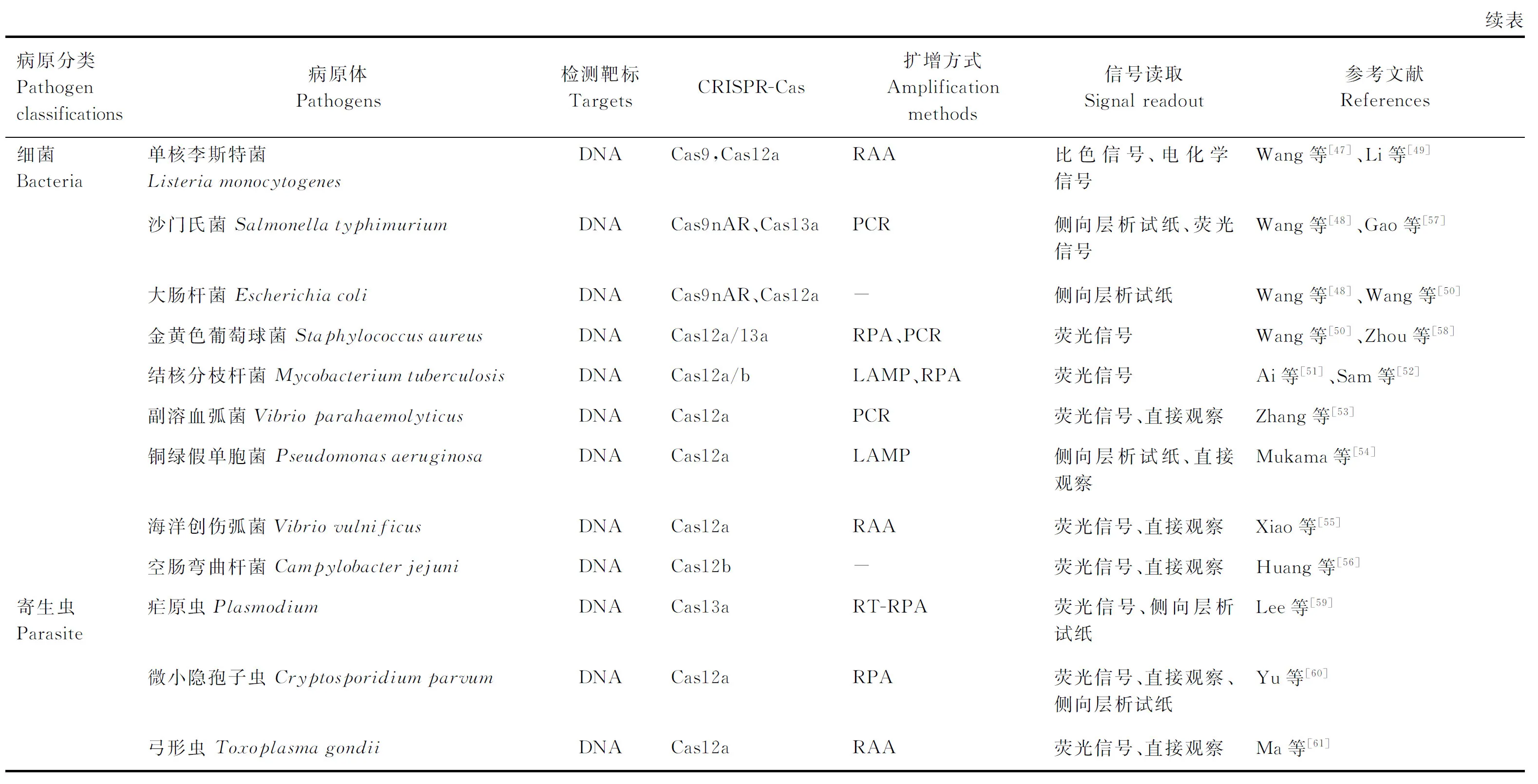
目前,在细菌快速检测方法研究中最常用的是CRISPR-Cas9/12/13系统。Wang等[47]将CRISPR-Cas9与侧向层析法相结合,构建了称为CASLFA的检测方法,该方法能够在非实验室条件下以100%的准确性检测单核李斯特菌、转基因生物、ASFV等。此外,研究人员开发了一种基于Cas9切口酶扩增的方法(Cas9nAR),与侧向层析试纸条相结合,可以作为鼠伤寒沙门菌和大肠杆菌的双重检测系统[48]。Li等[49]利用CRISPR-Cas12a与电化学信号相结合,开展了食品中单核李斯特菌的快速及现场检测。基于CRISPR-Cas12a、RPA、荧光信号检测等方法,Wang等[50]建立了金黄色葡萄球菌和大肠杆菌O157∶H7的快速检测方法-OCTOPUS,该方法能够在50 min内实现阿摩尔(aM)级的核酸检测。除了CRISPR-Cas12a-RPA以外,CRISPR-Cas12b-LAMP也适用于结核分支杆菌检测方法的开发[51-52]。越来越多的研究证明,CRISPR-Cas12/13系统能够被广泛应用于细菌快速、灵敏的现场检测,如副溶血弧菌、铜绿假单胞菌、海洋创伤弧菌、空肠弯曲杆菌、金黄色葡萄球菌等(表1)[53-58]。
2.3 CRISPR-Cas系统在寄生虫检测中的研究
随着CRISPR-Cas系统在病毒、细菌等病原体检测中的广泛应用,研究人员开始探究此方法用于寄生虫病的检测。Lee等[59]首先通过SHERLOCK检测平台建立了恶性疟原虫(即镰刀疟原虫)的诊断方法,同时该方法能够鉴别恶性疟原虫、间日疟原虫、卵形疟原虫、三日疟原虫。同时这种方法优化了核酸提取过程,能够在60 min内检测出每毫升血液中的2个疟原虫,符合了WHO建议的检测灵敏度。基于CRISPR-Cas12系统,Yu等[60]建立了另外一种原虫(微小隐孢子虫Ⅱd亚型)的快速检测方法,该方法与其他亚型的微小隐孢子虫无交叉反应,表明其特异性较好。此外,CRISPR-Cas12系统也被用于弓形虫的检测方法开发,该方法的灵敏性能够达到飞摩尔(fM)级别(表1)[61]。
2.4 基于CRISPR-Cas系统的病原体高通量及多重检测
CRISPR-Cas系统与微流控技术的结合,可以使多个样本或多个病原体的同时检测成为可能。Qin等[62]建立了一种基于CRISPR-Cas13a与自动化微流控POC技术联用的病原体核酸检测方法,该方法可以在无需核酸扩增的条件下,实现数十个样本中埃博拉病毒的高通量检测。此外,Ackerman等[63]开发了一种基于核酸多重评估的组合排列反应(CARMEN),将CARMEN与Cas13a、微孔阵列结合形成一种多通道检测方法。CARMEN-Cas13方法能够对169种人类病毒同时运行4 500次的测试,同时该平台还能够区分多种亚型的流感病毒和完成数十种耐药HIV病毒毒株的鉴定。
3 CRISPR-Cas病原体检测技术研究中的关键问题
3.1 靶标基因的序列保守性
对于Cas12,在crRNA设计时,需要考虑的问题主要是PAM区以及与crRNA互补的靶标基因的保守性,这是开发CRISPR-Cas12检测方法成功与否的关键[55]。研究显示,crRNA靶向区的碱基错配显著影响Cas核酸酶的切割效率[64]。这种特性也表明了Cas12方法的高特异性,但是同时也存在一定的问题,如较短的靶标序列或者存在碱基错配时,高度保守序列的筛选难度增加,可能影响其检测效率。在空肠弯曲杆菌CRISPR-Cas12b快速检测方法开发时,研究人员分析了1 037条空肠弯曲杆菌的全基因组序列,鉴定出了1条20 bp长度的序列在1 024条空肠弯曲杆菌的全基因组中高度保守,并且是空肠弯曲杆菌所特有的[56]。同时,为了摆脱该方法对于PAM区的依赖,研究人员设计了2种方案:一是通过PCR外源引入PAM区改进HOLMES法[39];二是设计2条不具有PAM区特性的crRNA,增加反应的检测灵敏性[65]。对于Cas13a,其crRNA设计需要涉及PFS区,然而Cas13a的这种局限很容易消除,比如选择PFS区非依赖性的LwaCas13a[26]。然而,对于易突变的RNA病毒,一旦产生突变将会影响CRISPR-Cas的检测效率。Liu等[66]利用LbuCas13a、化学修饰的RNA激活剂与TtCsm6结合,开发了一种免扩增快速实现核酸检测的方法(FIND-IT),其灵敏度和准确性和实时荧光定量PCR方法一致或更优。该方法将TtCsm6与包含8种不同crRNA的LbuCas13a效应子相结合,不仅可以最大程度地覆盖多种病毒的突变体,同时也避免了核酸扩增步骤。
3.2 靶标基因的富集与扩增
为了优化CRISPR-Cas系统的检出下限,经常与核酸扩增方法联用,比如重组酶聚合酶扩增技术(RPA/RT-RPA)、重组酶介导的扩增技术(RAA/RT-RAA)、环介导等温扩增技术(LAMP/RT-LAMP)、PCR或RT-PCR等(表1)。如在海洋创伤弧菌的检测中,RAA与CRISPR-Cas12a联用方法的检测灵敏性显著高于单独使用CRISPR-Cas12a的检测方法[55]。在上述的核酸扩增方法中,LAMP属于60~65 ℃等温扩增,但易受气溶胶污染而出现假阳性;RAA和RPA需要25~42 ℃反应,研发价格高于LAMP;而PCR需要依赖设备且扩增时间长于其他方法[1]。因此,在病原体检测技术开发中,目前最为常用的核酸扩增方法主要是RPA/RAA以及LAMP。
为了进一步缩短反应时间,研究人员开发了一系列不需要核酸扩增的方法。Fozouni等[67]通过设计多个靶向SARS-CoV-2的crRNA建立了不需要核酸扩增的CRISPR-Cas13a检测方法,该方法可直接用于临床样本的检测,其检测灵敏性可达100拷贝/μL。除了利用多个crRNA,Nguyen等[68]通过改造修饰单个crRNA如在3′-端和5′-端添加不同长度的ssDNA、ssRNA及硫代磷酸化的ssDNA,可能增加CRISPR-Cas12a的检测灵敏性。
3.3 终信号的读取方式
目前,在基于CRISPR-Cas系统的病原体检测技术研究中最为常用的终信号读取方式包括:荧光信号、比色信号、电化学信号和侧向层析分析(表1)。最具代表性的SHERLOCK和DETECTR方法采用的是基于荧光信号的读取方式[10-11],在这种信号读取的基础上,更多的研究开发出了利用LED蓝光灯、微流控技术、手机、侧向层析试纸条等读取终信号[54,62,67,69]。比色信号是一种基于金纳米颗粒的可视化分析方法,其能够通过颜色变化判断检测结果。Hu等[70]基于CRISPR-Cas12a构建了一种磁珠下拉辅助的比色分析法,称之为M-CDC。这种方法能够实现对ASFV等病原的特异性检测,同时这种比色法可广泛应用于荧光检测或侧向层析分析。电化学信号用于核酸检测的优势主要是简单、价格低廉、特异性高等。目前,基于电化学信号的CRISPR-Cas检测方法研究主要集中在HPV、人细小病毒(PB-19)、单核李斯特菌等[46,49]。
4 展 望
CRISPR-Cas系统作为新一代的可扩展性诊断技术,开创了分子诊断的新时代。该系统表现出灵敏度高、特异性好、价格低廉、操作简单,并且可以实现快速即时检测,在病原体检测方面展示出巨大的应用潜力。基于SHERLOCK、DETECTR方法的SARS-CoV-2检测试剂盒在美国相继获批,同年中国研发的基于CRISPR-Cas系统的SARS-CoV-2检测试剂盒也获批上市。迄今为止,在超过数十种人畜共患病和动物病原体中已开展了SHERLOCK、DETECTR及其相应的优化方法的研究。尽管CRISPR-Cas系统具有多种优势,其未来的发展仍面临多种挑战。如开展临床样本的大规模验证,利用测序或多种成熟的方法来确证检测结果的准确性;在不牺牲检测灵敏性的条件下,开发临床样本的预处理方法、设计多条crRNA等,优化或免除核酸的提取及扩增;CRISPR-Cas系统在更多病原体检测方面的临床应用;建立基于CRISPR-Cas系统的定制型高通量检测或多病原检测方法;更多新的Cas同源基因或CRISPR新方法的研发;CRISPR-Cas系统与自动化、人工智能等多学科的交叉。随着研究的进一步深入,未来CRISPR-Cas系统将会在病原体检测等领域得到广泛的应用,并成为强有力的新一代检测工具。