四倍体小麦株高和穗长性状的QTL定位及其遗传效应分析
陈黄鑫,李 聪,吴坤燕,王 岳,牟 杨,唐华苹,唐力为,兰秀锦,马 建
(1.四川农业大学小麦研究所,四川成都 611130; 2.四川农业大学农学院,四川成都 611130;3.攀枝花市农林科学研究院,四川攀枝花 617061)
小麦作为全球重要的粮食作物之一,其产量的提高对于解决人口剧增导致的粮食短缺问题非常关键。而株高和穗长对小麦产量的提升具有积极的促进作用。
迄今为止,在小麦染色体上已经鉴定到许多控制株高和穗长的基因/QTL。20世纪中期,()和()矮秆基因的发掘,提高了作物的抗倒伏能力,进一步提高了作物产量,掀起了矮秆育种的绿色革命浪潮;之后,Peng等在圆锥小麦Aiganfanmai的7A染色体短臂上定位到控制株高的基因;Mo等利用UC1110和PI610750构建的包含186个株系的RIL群体为材料,在小麦6A染色体短臂上定位到控制株高的基因,这些株高基因与赤霉素途径密切相关。此外,付美玉等利用矮秆突变体,使用外显子捕获测序和集群分离分析法,在2D染色体上检测到1个控制株高的QTL;Xiong等基于55K SNP高密度遗传图谱,利用eh1和轮选987构建的包含207个株系的RIL群体为材料,在2A、4A、4B和6B染色体上共鉴定到7个控制株高的QTL,可解释4.40%~ 34.40%的表型变异,在3A、4A、5B、6A、6B和7D染色体上检测到9个控制穗长的QTL,可解释 3.00%~22.00%的表型变异;Anuarbek等利用全基因组关联分析对184份原始和驯化小麦进行了SNP位点挖掘,共检测到15个控制株高的QTL和13个控制穗长的QTL;姚俭昕等利用小偃81和西农1376构建的包含120个株系的F代RIL群体为材料,共检测到3个控制穗长的QTL,分布在2B、2D和5D染色体上;Deng等利用花培3和豫麦57构建的包含168个双单倍体小麦群体为材料,在4个不同处理下共检测到11个控制穗长的QTL;严 俊等以硬粒小麦Langdon和野生二粒小麦G18-16构建的包含152个株系的F代RIL群体为材料,在5A和7A染色体上共检测到3个控制穗长的QTL。
关于小麦株高和穗长基因/QTL的研究已有较多报道,但由于六倍体普通小麦基因组庞大且复杂,同时受到现阶段分子技术限制,研究较为困难。在小麦的进化或驯化过程中,四倍体硬粒小麦(L.,2=4=28,AABB)和野生二粒小麦(ssp.,2=4=28,AABB)分别是普通小麦的重要近缘物种和祖先种,同时也是六倍体小麦重要的二级资源库。相比于六倍体小麦,四倍体小麦的基因组相对较小,染色体上也含有丰富的基因资源,研究起来相对简单,从四倍体小麦的近缘物种中挖掘鉴定可用的基因资源并运用到六倍体小麦,成为现阶段的主要育种手段之一,同时,四倍体硬粒小麦和野生二粒小麦具有一定的亲缘关系,便于深入研究株高和穗长的遗传基础,对普通小麦高产育种有着重要意义。因此,本研究以硬粒小麦矮兰麦和野生二粒小麦LM001构建的包含121个株系的F代RIL群体为材料,基于55K SNP芯片构建了遗传连锁图谱,并结合5年8个生态环境的株高和穗长表型数据,挖掘与株高和穗长相关且能够稳定表达的主效QTL,并分析其遗传效应,为其精细定位和聚合育种提供理论基础。
1 材料与方法
1.1 试验材料
试验材料为矮兰麦和LM001及其通过单籽粒传法构建的F代RIL群体。该群体包含121个株系,其亲本为四川地方硬粒小麦品种矮兰麦(母本)和野生二粒小麦LM001(父本),两者均为四倍体小麦,由四川农业大学小麦研究所收集并保存。其中,母本矮兰麦穗长较短且株高较矮。
1.2 试验方法和表型鉴定
对119份RIL群体及其亲本分别于2017-2021年种植在四川省崇州市试验基地(分别用2017CZ、2018CZ、2019CZ、2020CZ和2021CZ表示),并于2020年和2021年也种植在四川省成都市温江区试验基地(分别用2020WJ和2021WJ表示),进行株高和穗长的表型鉴定;于2020年也种植在四川省雅安市试验基地(用2020YA表示),仅进行穗长的表型鉴定。于小麦成熟期,每个株系选取生长良好且长势一致的三个单株(排除边际效应),使用米尺和直尺测量株高和穗长(不包括芒)。播种方法为单粒播种,每个株系播种一行,行长1.5 m,行距0.3 m,株距0.1 m,按当地常规小麦生产进行田间管理。
1.3 遗传图谱的构建和QTL定位
利用本课题组前期基于小麦55K SNP芯片构建的遗传连锁图谱,结合表型数据,用IciMapping 4.1 (https://www.isbreeding.net/)软件中的完备复合区间作图法(ICIM-ADD)对株高和穗长QTL进行检测,参数设置为:Step=1 cM,PIN值=0.001,LOD阈值=2.5,并计算出每个QTL的表型变异和加性效应,同时进行QTL的多环境分析,参数设置为:Step=1 cM,PIN值=0.001,LOD阈值=8。QTL命名按照国际遗传命名规则(https://wheat.pw.usda.gov/ggpages/wgc/98/Intro.htm)进行。在Graingenes 2.0(https://wheat.pw.usda.gov/gg2/index.shtml)和NCBI(https://www.ncbi.nlm.nih.gov/)网站检索前人已经报道的小麦株高和穗长QTL及其侧翼标记和基因信息,并与中国春的最新参考基因组v2.1、硬粒小麦参考基因组和野生二粒小麦参考基因组2.0进行比对,获得标记的物理位置。
1.4 统计分析
用Microsoft Excel 2016对株高和穗长的表型数据进行平均值的计算;用SAS 9.1.3对株高和穗长进行BLUP值和广义遗传力()的计算;用Origin 2021绘制株高和穗长的频率分布图和箱线图;用IBM SPSS Statistics 27进行独立样本T检验,分析显著性水平(异常值不做数据分析);根据55K SNP芯片数据查找本研究中株高和穗长QTL侧翼标记的物理位置。
2 结果与分析
2.1 亲本及其RIL群体株高和穗长的表型分布
从表1可知,亲本LM001的株高在除2018CZ之外的6个环境中均极显著高于矮兰麦;而LM001的穗长仅在2021CZ环境下显著高于矮兰麦。RIL群体株高和穗长的遗传力分别为0.79和0.70,表明它们受遗传因素影响较大,受环境影响较小。RIL群体在不同环境下的株高和穗长都存在超亲分离现象,且两者的频率分布图在每个环境下均呈近似正态分布(图1)。因此,株高和穗长性状具有典型的数量遗传学特点,可用作QTL检测。
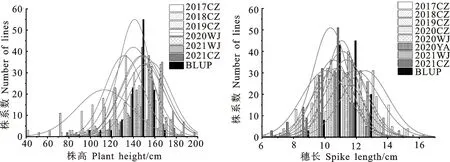
图1 不同环境下RIL群体株高和穗长的频率分布

表1 不同环境下亲本及RIL群体株高和穗长的表型分布
2.2 株高和穗长QTL的检测结果
结合本课题组前期构建的遗传连锁图谱,并结合5年8个生态环境的表型数据,共鉴定到7个株高QTL和17个穗长QTL,分布在9条染色体上(表2)。
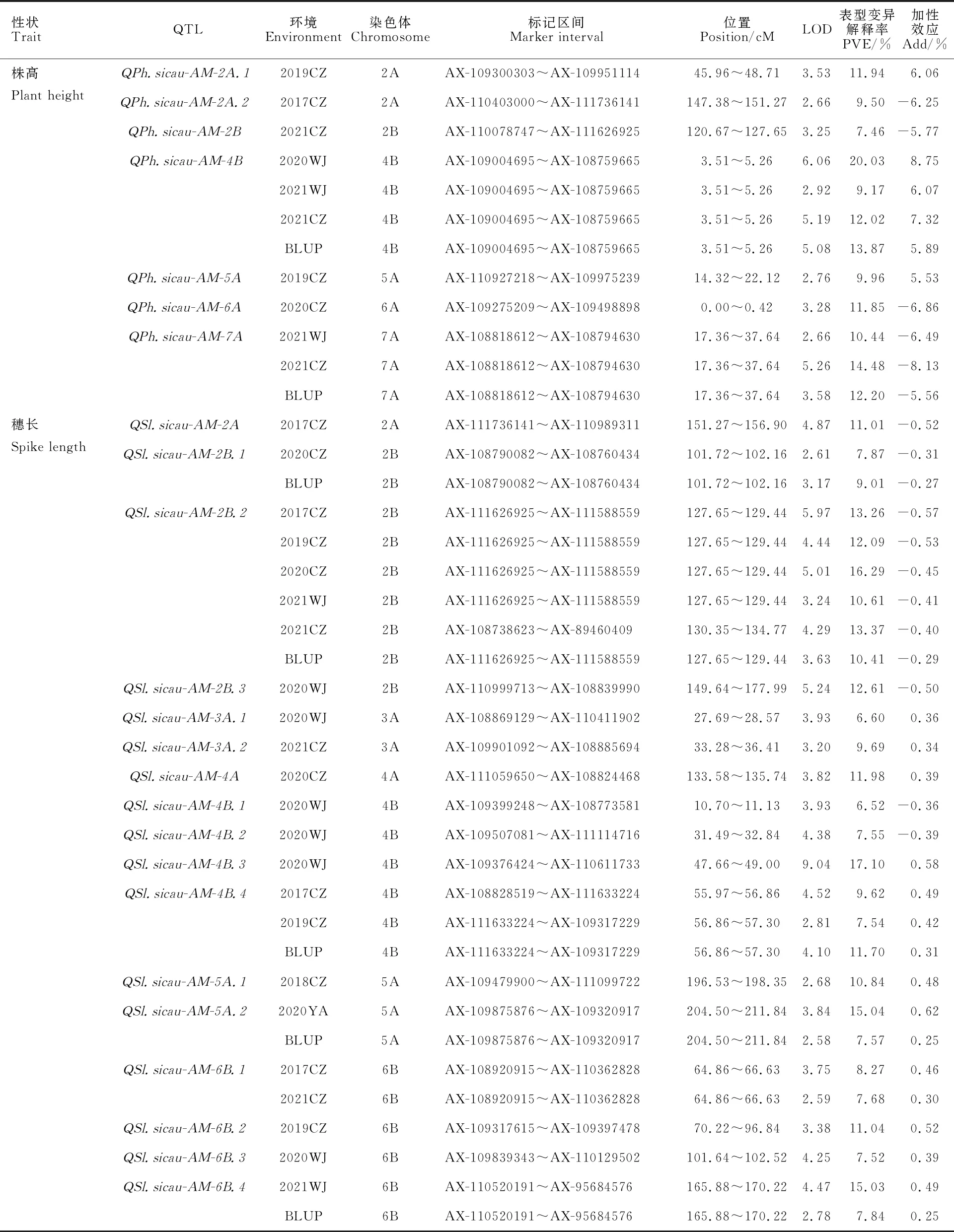
表2 基于RIL群体检测到的株高和穗长QTL
7个株高QTL分别位于2A、2B、4B、5A、6A和7A染色体上,其中,4B染色体上的在3个环境中被检测到,可解释 9.17%~20.03%的表型变异,为稳定表达的主效位点,其加性效应来源于母本矮兰麦;7A染色体上的在2个环境中被检测到,可解释10.44%~14.48%的表型变异,也为稳定表达的主效位点,其加性效应来源于父本LM001;其他5个QTL仅在单个环境中检测到,可解释7.46%~11.94%的表型变异。
17个穗长QTL分别位于2A、2B、3A、4A、4B、5A和6B染色体上,其中,2B染色体上的在5个环境中被检测到,可解释10.41%~16.29%的表型变异,为稳定表达的主效位点,其加性效应来源于父本LM001;4B染色体上的和6B染色体上的均在2个环境中被检测到,分别可解释7.54%~11.70%和7.68%~ 8.27%的表型变异,其加性效应均来源于母本矮兰麦,其中,也为稳定表达的主效位点;其他15个QTL仅在1个环境中检测到,可解释6.52%~17.10%的表型变异。
多环境分析结果(表3)表明,当LOD阈值为8时,共检测到13个QTL,其中,在单环境中稳定表达的主效QTL(、、和)在多环境中也能被检测到,进一步表明这4个QTL为稳定的QTL。

表3 多环境分析株高和穗长QTL
2.3 与株高和穗长相关且稳定表达的主效QTL的遗传效应
根据同一性状主效QTL侧翼标记的基因型和效应位点来源,将RIL群体的121个株系分 成两类,一类是仅携带相应QTL增效位点 的株系,一类是不携带相应QTL增效位点的 株系。
进一步对这两类株系进行检测,结果(表4)表明,单个生态环境条件下,对于株高,携带增效位点株系的株高在各个环境下均显著或极显著高于不携带该位点的株系,增幅为 9.60%~ 21.35%;携带增效位点株系的株高在除2020WJ和2021WJ之外的5个环境下均极显著高于不携带该位点的株系,增幅为 5.79%~18.20%。对于穗长,携带增效位点株系的穗长在除2018CZ和2020YA之外的6个环境下均极显著高于不携带该位点的株系,增幅为 0.80%~ 16.98%;携带增效位点株系的穗长在2017CZ、2020CZ和2021CZ三个环境下均显著或极显著高于不携带该位点的株系,增幅为 2.11%~11.93%。

表4 稳定表达的穗长和株高主效QTL的遗传效应
2.4 与株高和穗长相关且稳定表达的主效QTL的聚合效应
分别用株高和穗长的BLUP值,结合侧翼标记的基因型,对主效QTL进行聚合效应分析。对于株高,仅携带或增效位点株系的株高极显著高于不携带任何位点的株系,增幅分别为6.70%和 5.42%;同时携带和增效位点株系的株高也显著或极显著高于仅携带或增效位点的株系以及不携带任何位点的株系,增幅分别为3.91%、5.17%和10.87%(图2a)。对于穗长,仅携带或增效位点株系的穗长极显著高于不携带任何位点的株系,增幅分别为7.36%和 6.39%;同时携带和增效位点株系的穗长也极显著高于仅携带或的株系以及不携带任何位点的株系,增幅分别为6.19%、7.16%和14.01%(图2b)。

图中百分数表示两个株系之间株高或穗长的变幅。*和**分别表示株系之间在0.05和0.01水平上差异显著。
3 讨 论
本研究定位到的位于4B染色体短臂上,与Liu等定位到的距离较近,推测与可能是等位位点;位于7A染色体短臂上,与前人在7A染色体定位到的、和物理位置均无重叠区段,说明可能是新的控制株高的QTL;位于2B染色体长臂上,与前人定位到的QTL物理位置均无重叠,可能是新的控制穗长的QTL;位于4B染色体长臂上,与李乐晨定位到的穗长QTL位点距离较近。此外,在2B染色体上已经报道的、4B染色体上已经报道的以及7A染色体上已经报道的与本研究定位到的QTL位点区间无重叠,但是由于QTL定位精确度存在局限性和遗传背景的干扰,很难排除、和基因对、、和的影响,因此需要进一步用分子标记或精细定位来确定。
本研究在RIL群体的后代中发现了超亲分离性状,这种现象不仅有基因分离产生的作用,还存在着基因自由组合对性状的影响。和(控制株高)、和(控制穗长)效应位点分别来自两个不同的亲本,株高和穗长的聚合效应显著,同时株高和穗长的表型增加也并非简单的累加,说明多个主效QTL位点的存在可能是产生超亲性状的主要原因。聚合多个QTL可用于育种工作,能够避免单个QTL的效应不足,同时增加所聚合QTL的效应,提高分子辅助选择育种的效率。聚合多个抗病基因能够提升作物广谱抗性和持久性。例如,刘 凯等将、和基因聚合,选育出抗稻瘟病的水稻新品系盐稻 1626。本研究结果表明,聚合育种同样可以运用于株型和穗型的遗传改良,对于培育理想株型的小麦有着积极意义。但不同基因/QTL之间还可能存在位点冗余、位点掩盖、上位效应等复杂的互作机制,因此,在进行聚合分析时应进行多方面的研究。

