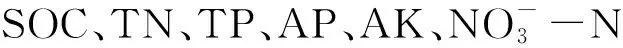秸秆还田对玉米根际氨氧化微生物群落及红壤硝化潜势的影响
郑 洁,程梦华,栾 璐,孔培君,孙 波,蒋瑀霁,*
1 土壤与农业可持续发展国家重点实验室(中国科学院南京土壤研究所), 南京 210008 2 中国科学院大学, 北京 100049 3 南京师范大学生命科学学院, 南京 210023
根际微生物作为土壤生物群落中最活跃且具有决定性影响的组分之一,在土壤-植物生态系统中参与了土壤有机质等养分循环和能量流动。揭示植物—根际微生物根际对话过程及其调控机制,对促进植物生长发育和生产力的提高至关重要[1—2]。氮是陆地生态系统中控制植物生长的关键限制营养元素,根际土壤微生物驱动着土壤—植物系统中氮素循环过程,将氮素转化为可被植物吸收的形态,满足植物的氮素需求。硝化作用是土壤氮素形态转化的关键步骤之一,我国农田生态系统硝化活性较高,以自养硝化作用为主[3—4]。作为自养硝化过程的氨氧化过程是第一和限速步骤,主要由含氨单加氧酶基因(amoA)的氨氧化古菌(ammonia-oxidizing archaea, AOA)和氨氧化细菌(ammonia-oxidizing bacteria, AOB)共同驱动,在有氧环境下将氨氧化为亚硝酸盐[5]。近年来,全程氨氧化微生物(Comammox)的发现使人们对氮循环有了全新的认识,但是深入研究发现Comammox大都来自铵贫瘠的生境,而在高底物浓度的农田土壤硝化过程中典型的氨氧化微生物(AOA和AOB)扮演的角色并不能被Comammox所取代[6—7]。农田生态系统中,环境因子(pH、NH4浓度等)和农田管理措施(种植方式、施肥管理等)决定着土壤中氨氧化微生物的生态位分异,影响群落的丰度和硝化潜势[8]。研究表明低pH和低NH4浓度条件下,AOA在氨氧化过程中发挥着主导作用,而AOB则倾向于主导中性和碱性土壤,以及高NH4浓度环境的氨氧化过程[9—10,11]。Wu等[12]和Su等[13]研究发现,不同的施肥管理方式会显著影响AOA和AOB的群落结构。可见,研究AOA和AOB群落对农田管理措施和环境因子的响应机制一直是农田生态系统研究的热点问题,对深入认识氮素转化机制具有重要意义。
红壤是中国南方地区的主要土壤类型,其分布地区气候条件优越。由于红壤酸性强,土质粘重,脱硅富铁铝化现象严重,导致土壤氮素供应失衡[11]。与此同时,农田过度施用氮肥,加剧土壤酸化,导致氮素转化功能微生物丰度和代谢活性下降,降低了作物生产力和氮素养分利用率,同时还会对环境造成负担[14]。研究表明,秸秆还田是培肥地力,改良土壤性状的有效途径之一。作为土壤养分和有机物质的重要来源,秸秆分解刺激了土壤微生物的活性,通过提高土壤氮素有效性加速氮素循环,促进作物生长发育[15]。但是秸秆还田存在腐解慢、与作物争氮和土壤疏松等不利因素,导致作物出苗困难、诱发病虫害等问题[16]。因此,揭示秸秆还田的高效激发效应,对建立贫瘠红壤快速培肥的调控技术具有重要的实践价值。
Tosti等发现秸秆猪粪配施通过调节秸秆中的碳氮比可以加快微生物的矿化作用,显著提高有机氮的矿化与矿质氮的微生物固持,进而增加作物产量[17]。Yu等发现秸秆生物炭可以增加微孔生境的可用性,提高微生物生物量和多样性[18]。不同秸秆还田方式对培育土壤肥力、增强功能微生物活性具有重要影响,但目前关于秸秆还田处理对根际氨氧化微生物群落和功能的影响机制尚不明确。为此,本研究以南方典型旱地红壤为研究对象,依托秸秆还田长期定位试验(9年),通过Illumina高通量测序技术,研究不同秸秆还田处理对玉米根际氨氧化微生物多样性和群落结构、以及硝化潜势的影响,旨在揭示不同秸秆还田处理对根际氮转化微生物调控机制,以期为建立科学高效的秸秆还田措施、提高氮肥利用率提供科学依据。
1 材料与方法
1.1 研究区概况
田间试验于2010年设置在江西省鹰潭市中国科学院红壤生态实验站(116°55E,28°13N),属中亚热带季风气候,年平均气温17.8℃,年降水量约为1795 mm,其土壤为第四纪红黏土发育的红壤(黏化湿润富铁铝土)。田间试验小区面积为100 m2(5 m×20 m)。土壤酸性较强、有机质低。试验地开始前表层土壤(0—20 cm)的基本理化性质为:pH 4.73,有机碳2.52 g/kg,全氮0.40 g/kg,全磷0.23 g/kg,碱解氮38.30 mg/kg,全钾11.95 g/kg,速效磷0.76 mg/kg,速效钾47.58 mg/kg,阳离子交换量12.12 cmol/kg。
1.2 试验材料与设计
试验共设置5个处理,分别为:不施肥(CK);氮磷钾肥(NPK);氮磷钾肥+秸秆(NPKS);氮磷钾化肥+秸秆猪粪配施(秸秆猪粪按9∶1的碳配比施用,NPKSM);氮磷钾肥+秸秆生物炭(NPKB)。试验供试作物为玉米(ZeamaysL.,苏玉24),每年4月份种植,7月份收获。每个处理设置3个重复,随机区组排列。秸秆生物炭是利用风干玉米秸秆在生物炭炉中450 ℃下通过厌氧闷烧制成。不同秸秆还田处理(NPKS、NPKSM、NPKB)每年的碳输入量为1000 kg/hm2,各处理的化肥的施入量为:尿素(N, 150 kg/hm2),钙镁磷肥(P2O5, 75 kg/hm2),无机钾肥(K2O, 60 kg/hm2),所有氮磷钾肥、秸秆、猪粪和生物炭在玉米种植前一次性施入土壤并翻耕混匀。
1.3 样品采集与土壤性质测定
1.4 高通量测序与荧光定量PCR分析
采用MoBio PowerSoil DNA(MIBIO laboratories Inc.,Carsbad,USA)试剂盒提取土壤样品DNA。通过NanoDrop®ND- 2000检测DNA浓度质量,然后进行1.2%(w/v)的琼脂糖凝胶电泳,检测DNA片段大小。AOA的扩增引物为Arch-amoA26F(5′-GACTACATATTCTACACWGACTGGGC- 3′)和Arch-amoA417R(5′-GGTGTCATATATGGAGGCAACGTTGG- 3′);AOB的扩增引物为amoA- 1F(5′-GGGGTTTCTACTGGTGGT- 3′)和amoA- 2R(5′-CCCCTCKGSAAAGCCTTCTTC- 3′)。PCR反应体系为20 μL:2×SYBR Premix 10 μL,0.8 μL上下引物,1 μL DNA模板,加ddH2O补足。PCR扩增条件为:94℃预变性2 min,94℃变性30 s,55 ℃退火30 s,72℃延伸30 s,72℃终末延伸10 min,循环30次。PCR产物纯化回收后制备测序文库,通过Illumina Miseq平台进行高通量测序。生物信息学分析采用Mothur软件包对原始序列进行质量筛选,对保留的序列进行修剪。选择与barcode完全匹配的序列,将其分配到古菌和细菌的amoA基因的单独文件中。使用FrameBot工具进一步进行筛选后,采用RDP中FunGene数据库对AOA和AOB的amoA进行相似度检索。最后,对每个样本的序列读数进行聚类,使用CD-HIT-EST得出基于97%相似度的OTU[20]。
AOA和AOB的amoA基因拷贝数用荧光定量法测定,反应在ABI StepOne TM实时荧光定量PCR仪上进行。所用引物与上述相同,反应体系为:2×SYBR®Premix Ex Taq 10 μL,上下引物各0.5 μL,DNA模板1 μL,最后用ddH2O补至20 μL。反应程序为:95℃预变性1 min,94℃变性45 s,57℃退火45 s,72℃延伸45 s,72℃终末延伸10 min,重复循环40次。每个样品3个重复,并设3个用无菌水作为空白对照。
1.5 氨氧化微生物功能的测定
1.6 数据处理
使用Chao 1指数表征丰富度,以Shannon指数表征多样性。采用主坐标分析(principal co-ordinates analysis,PCoA)计算两两样本间Bray-Curtis距离,研究样本群落组成的差异性。相关性分析和方差分析等在SPSS 24.0中完成,不同处理之间利用Tukey法进行单因素方差分析,不同字母表示处理间差异显著(P<0.05)。多元分析统计在Rstudio软件中完成,其中微生物多样性、主坐标分析(PCoA)和相似性分析(ANOSIM)通过vegan包完成。随机森林分析通过randomForest程序包计算,并通过rfUtilities和rfPermute程序包分别检验模型和每个变量的P值。结构方程模型(SEM)在AMOS 23.0软件中分析。
2 结果与分析
2.1 不同秸秆还田处理的土壤理化性质和硝化潜势
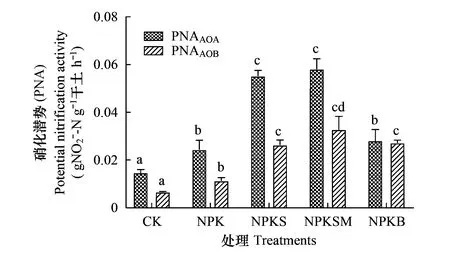
图1 不同秸秆还田处理的土壤硝化潜势 Fig.1 Potential nitrification activity under treatments with straw returning误差线表示标准误;不同小写字母表示PNAAOA和PNAAOB分别在各个处理间的差异显著性(P<0.05);CK:不施肥;NPK:氮磷钾肥;NPKS:氮磷钾肥+秸秆;NPKSM:氮磷钾肥+秸秆猪粪配施;NPKB:氮磷钾肥+秸秆生物炭AOA:氨氮氧化古菌; AOB:氨氧化细胞
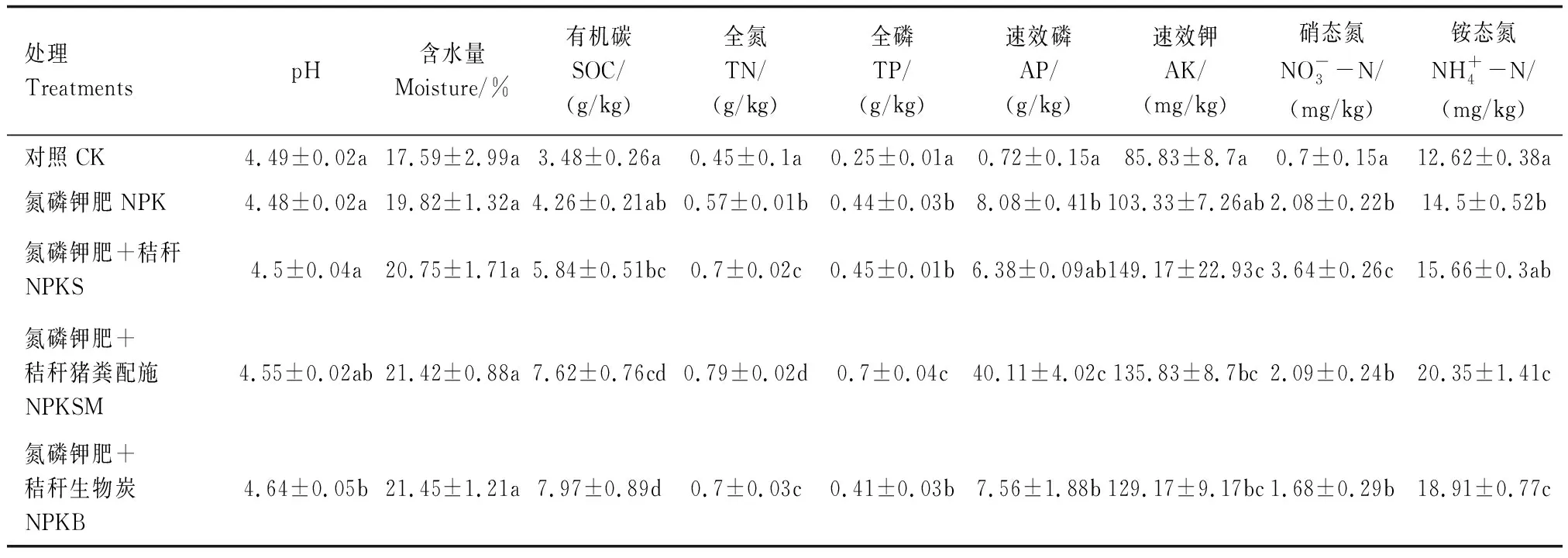
表1 不同秸秆还田处理的土壤理化性质(平均值±标准误)Table 1 Soil physic-chemical properties under different straw returning treatments(Mean±SE)
2.2 不同秸秆还田处理的氨氧化微生物amoA基因丰度
通过荧光定量PCR对不同秸秆还田处理下土壤AOA和AOB的amoA基因拷贝数进行分析,结果表明,所有处理中AOA的amoA基因的平均拷贝数(2.6×106拷贝/g)显著高于AOB(2.73×104拷贝/g)(P<0.05,图2)。AOA的amoA基因拷贝数介于0.31×105—7.16×106拷贝/g,与NPK处理比较发现,NPKS、NPKSM和NPKB处理下AOA的amoA基因拷贝数分别增加了4.88、9.64和1.93倍。AOB的amoA基因拷贝数介于0.26×104—4.93×104拷贝/g之间,NPKS、NPKSM和NPKB处理下AOB的amoA基因拷贝数分别比NPK处理增加了4.36、3.93和5.42倍。NPKSM处理下AOA的amoA基因拷贝数最高,而NPKB处理下AOB的amoA基因拷贝数最高。
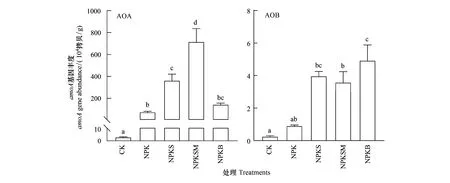
图2 不同秸秆还田处理的AOA和AOB的amoA基因丰度Fig.2 The amoA gene abundance of AOA and AOB under treatments with straw returning不同小写字母表示各处理间差异显著(P<0.0)
2.3 不同秸秆还田处理的氨氧化微生物群落多样性和结构特征
不同秸秆还田处理的AOA和AOB群落多样性的Shannon和Chao1指数在各处理间均显著改变(P<0.05,图3)。与CK处理相比,NPK处理提高了AOA的Shannon和Chao1指数,但降低了AOB的Shannon和Chao1指数。与CK和NPK处理相比,NPKS、NPKSM和NPKB处理的AOA和AOB的群落多样性指数均显著增加(P<0.05),在AOA群落中,NPKB处理的Shannon指数最高,NPKS处理的Chao1指数最高;在AOB群落中NPKS处理的Shannon指数最高,NPKSM的Chao1指数最高。
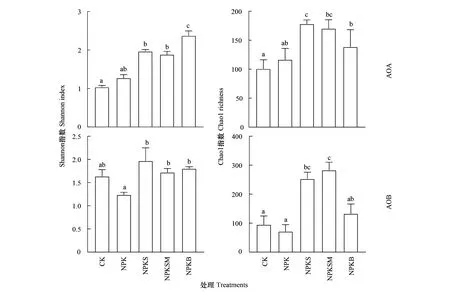
图3 不同秸秆还田处理的AOA和AOB群落的Shannon指数与Chao 1指数Fig.3 The Shannon indices and Chao 1 richness of AOA and AOB under treatments with straw returning
不同秸秆还田处理的AOA群落主要由Nitrosotalea(70.8%)、Nitrososphaera(25.1%)和Nitrosopumilus(0.24%)组成(图4),系统发育分析结果表明NitrosotaleaCluster 1.1的相对丰度占70.8%,NitrososphaeraCluster 9的相对丰度占12.3%,其余主要由NitrososphaeraCluster 1.1(4.5%)、NitrososphaeraCluster 2(3.9%)和NitrososphaeraCluster 7.2(3.1%)组成,其中NitrosotaleaCluster 1.1的相对丰度在CK处理中最高(90.1%),NitrososphaeraCluster 2在NPKSM处理中相对丰度最高(8.5%),NitrososphaeraCluster 9在NPKB处理中的相对丰度最高(37.1%)。AOB群落主要由Nitrosospira组成,其中NitrosospiraCluster 9的丰度占61.9%,NitrosospiraCluster 3a丰度占18.3%,其余主要由NitrosospiraCluster 10(6.8%)、NitrosospiraCluster 3b(3.86%)、NitrosospiraCluster 9(1.9%)等组成,NitrosospiraCluster 9在CK处理中丰度最高(7.9%),NitrosospiraCluster 12在NPK处理中丰度最高(85.8%),NitrososphaeraCluster 3a在NPKSM处理中丰度最高(36.1%),NitrosospiraCluster 10在NPKB处理中丰度最高(21.3%)。主坐标分析发现(PCoA),PCoA1轴和PCoA2轴分别解释了AOA群落结构45.27%和22.63%的差异,解释了AOB群落结构44.49%和27.82%的差异(图4)。不同秸秆还田处理下AOA和AOB群落结构在PCoA1轴和PCoA2轴上明显分异,ANOSIM分析表明不同秸秆还田处理下AOA(r=0.752,P<0.01)和AOB(r=0.834,P<0.01)群落结构的组间差异显著大于组内差异。
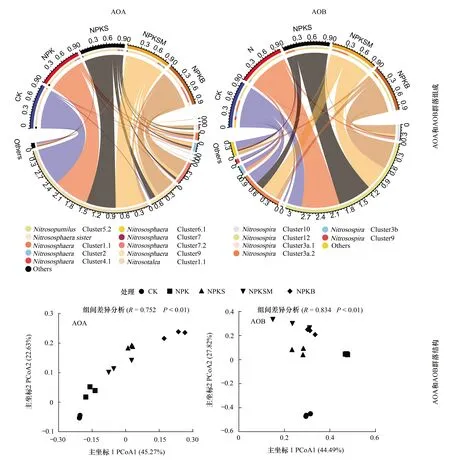
图4 不同秸秆还田处理的AOA和AOB群落组成和结构Fig.4 The composition and structure of AOA and AOB under different straw returning treatments
2.4 土壤氨氧化微生物群落与理化性质、硝化潜势的关系
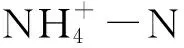
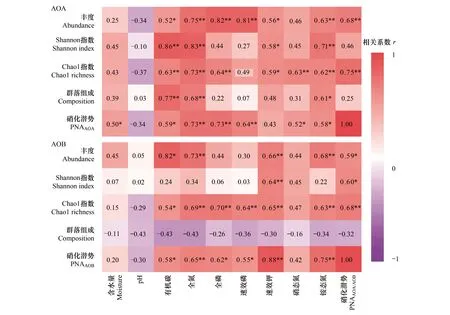
图5 土壤AOA和AOB群落与理化性质、硝化潜势的Pearson相关系数Fig.5 Pearson correlation coefficients between soil properties, PNA and AOA and AOB communityamoA基因的拷贝数代表群落丰度(Abundance);第一主坐标值(PCoA1)代表群落组成(Composition);PNAAOA代表AOA群落的硝化潜势;PNAAOB代表AOB群落的硝化潜势,PNAAOA/AOB表示在AOA热图中是PNAAOA,而在AOB热图中是PNAAOB;*:P<0.05,**:P<0.01
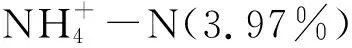
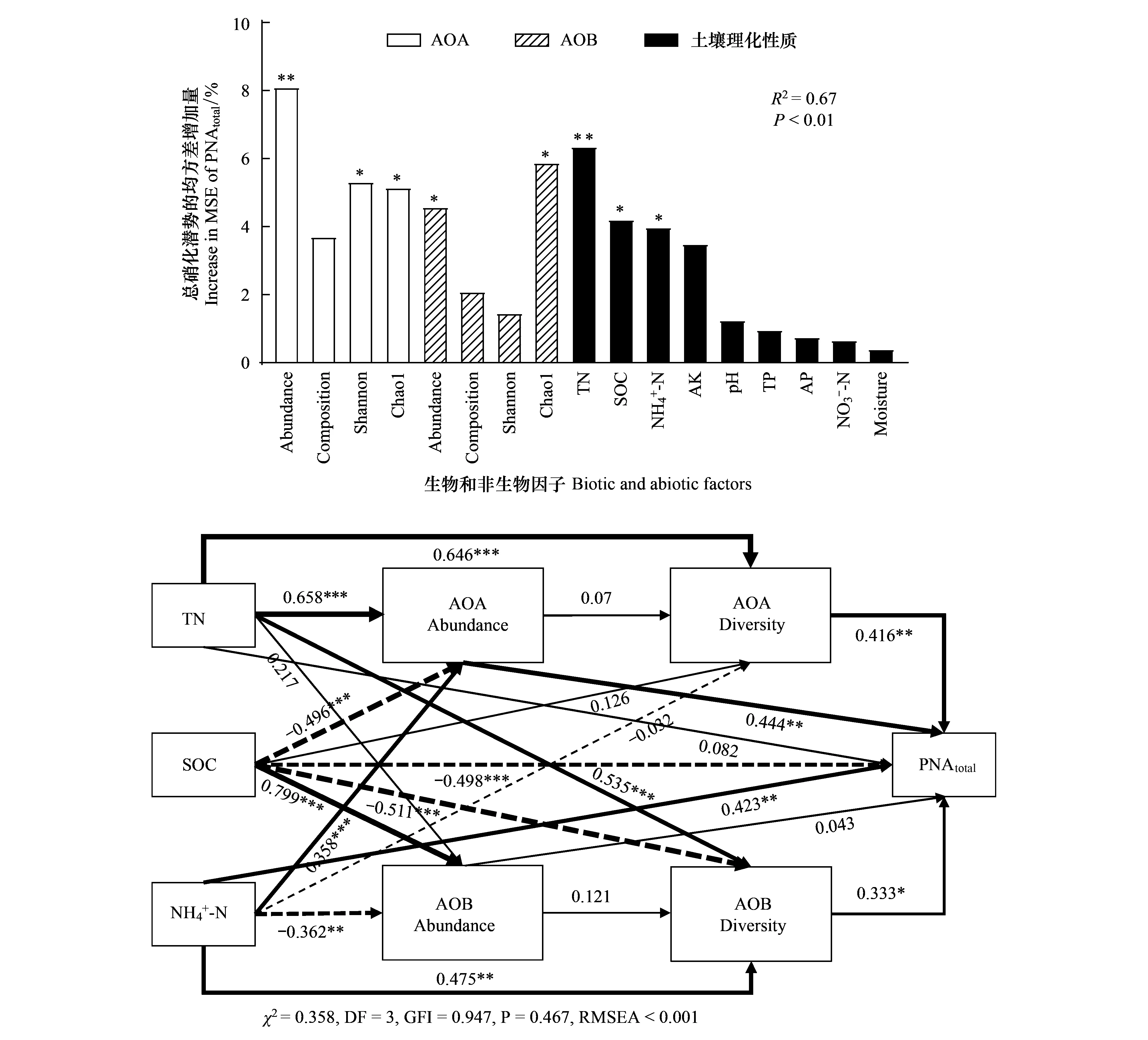
图6 随机森林分析生物因子和非生物因子对土壤硝化潜势(PNAtotal)的平均预测重要性;土壤硝化潜势(PNAtotal)影响因子的结构方程模型Fig.6 Mean predictor importance (% of increased mean square error, MSE) of abiotic and biotic factors on PNAtotal based on random forest analysis; Structural equation modelling (SEM) to predict the influence factors of PNAtotal *: P<0.05,**: P < 0.01,***: P < 0.001;单项箭头表示数据之间的因果关系,箭头上的数字表示标准化的路径系数,连线的粗细和路径系数呈比例缩放。实线表示正相关,虚线表示负相关;Abundance: amoA基因的拷贝数,Diversity:群落的Chao 1指数, PNAtotal:AOA与AOB的总硝化潜势
3 讨论
3.1 不同秸秆还田处理对土壤理化性质和硝化潜势的影响
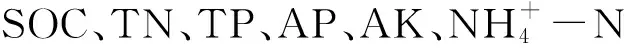
3.2 不同秸秆还田处理对根际氨氧化微生物群落结构和多样性的影响
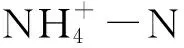
3.3 不同秸秆还田处理对根际氨氧化微生物群落功能的影响
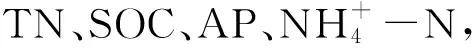
4 结论