基于线粒体Cytb基因的贵州野猪(Sus scrofa)群体遗传多样性分析
彭彩淳,李斌强,王野影,粟海军
(1.贵州大学林学院,贵阳 550025;2.贵州大学,生物多样性与自然保护研究中心,贵阳 550025;3.贵州师范大学生命科学学院,贵阳 550025)
野猪(Susscrofa)隶属于偶蹄目(Artiodactyla)猪科(Suidae)猪属(Sus),全球有25个亚种,广泛分布于亚、欧、北非大部分地区[1]。中国目前有7个亚种,分布于除干旱荒漠和高原区外的其他地区,贵州地区为华南亚种(S.s.chirodonticus)[2-3]。野猪及各地特有猪种是世界范围内一种重要遗传资源,对其展开保护与研究对于人类发展具有重要意义[4]。
遗传多样性能够反映遗传变异的水平,是衡量种群生存、进化及对环境条件适应的关键指标[5]。目前,多重微卫星标记[6]、基因扩增片段长度多态性(AFLP)分析[7]等技术早已用于野猪遗传多样性研究。线粒体DNA是动物体内长期稳定存在的核外遗传物质,线粒体Cytb基因有结构保守、扩增难度小和遗传信息丰富等特点[8],是物种系统发育、母系遗传标记以种群分类等基因组水平上遗传多样性研究中最常引用且可靠的分子标记之一[9],目前被广泛用于系统发育研究[10]。通过微卫星标记技术分析表明,中国地区野猪比国外野猪种群遗传多样性高[5,11],并使用线粒体分子标记推断出亚洲野猪种群起源于东南亚地区,且在中国内陆的野猪种群有较高的遗传多样性[12-13]。中国野猪最早可能出现在川黔湘、云贵高原等地[14-15],但相关研究还处在探索阶段。
贵州地处中国西南地区,主要以山地、丘陵为主,是世界喀斯特地貌发育最典型的地区之一[16],该地区气候温和湿润、地形地貌复杂、植被覆盖率高,以致近年多地发现野猪活动频繁[17-18]。野猪的遗传多样性对于衡量种群生存、环境适应及种群动态有着极其重要的作用。为了评估贵州野猪种群的遗传多样性,本试验对贵州野猪的11条线粒体Cytb基因进行PCR扩增和测序,以与野猪亲缘关系较近的爪哇野猪(Susverrucosus)作为外群,与GenBank中国内其他9个地区野猪种群的线粒体Cytb基因进行序列比对、系统进化树构建及历史动态分析,探究贵州野猪种群和其他地区野猪种群的遗传差异,以期为贵州野猪种群的遗传多样性保护、种质管理与利用、应对种群动态变化等提供重要依据,为遗传分化研究奠定基础。
1 材料与方法
1.1 材料
野猪组织样本均来自于2019年贵州省森林地区采样的11头野猪(猎捕及采样均获得贵州省林业局行政批准和贵州大学实验动物伦理分委员会审批),样品编号、采集地点及数量见表1。对处死后的野猪进行腿部肌肉组织采样,用无菌密封袋装集后置于冰盒中冷藏,48 h内送回实验室,-20 ℃保存备用。从GenBank数据库中下载中国部分地区野猪的55条线粒体Cytb基因序列,并下载爪哇野猪Cytb基因序列作为外群,序列信息见表2。
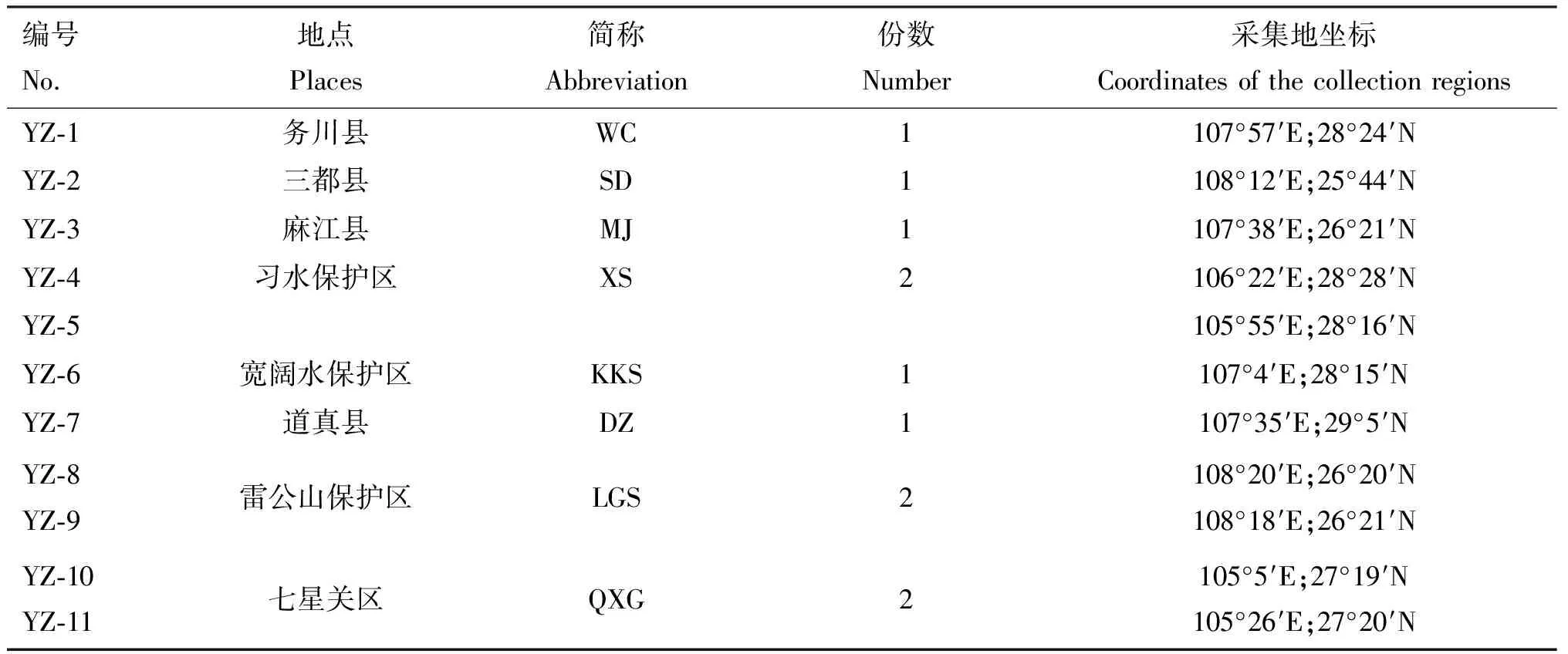
表1 贵州野猪样品采集信息
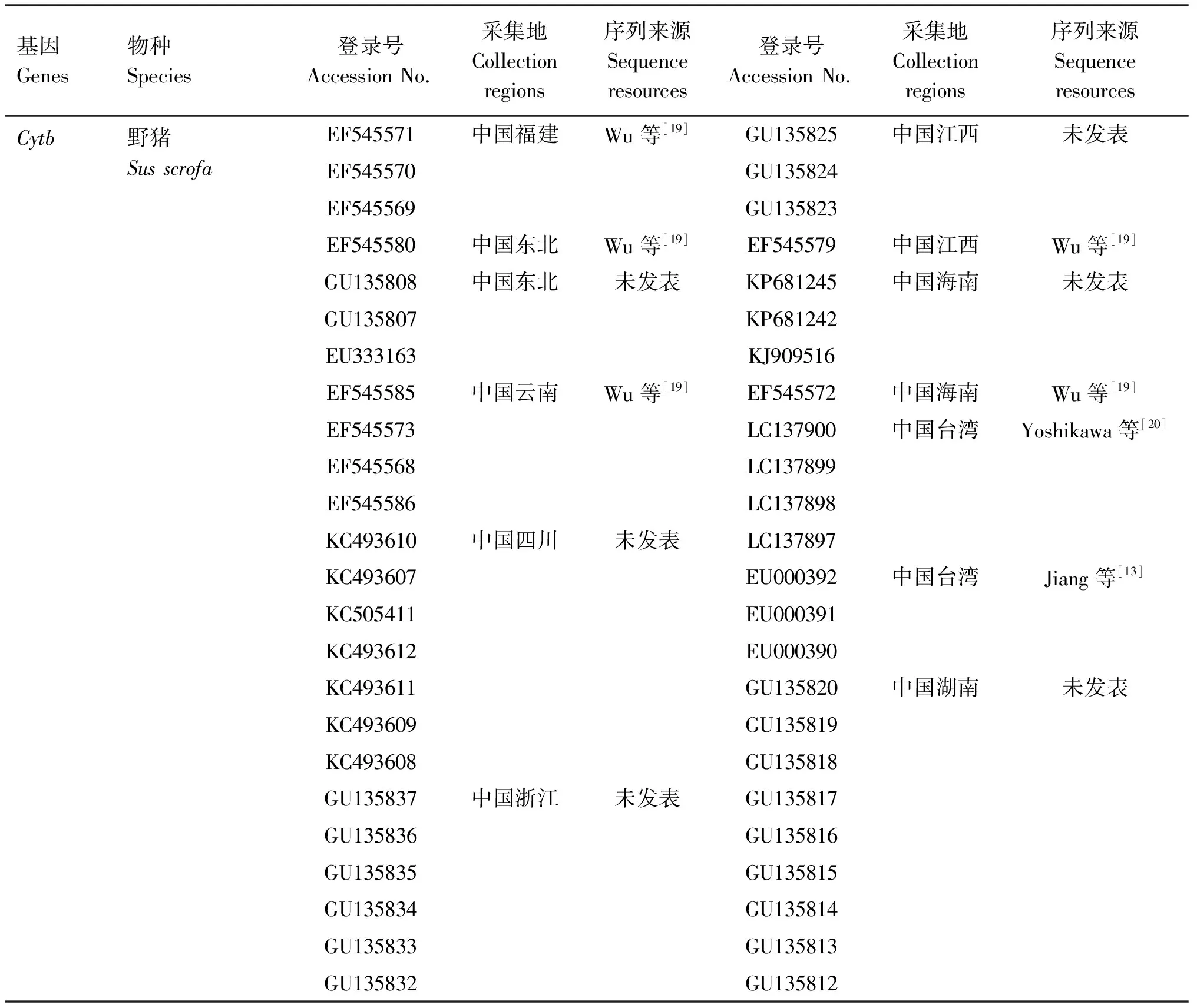
表2 中国部分地区野猪和爪哇野猪Cytb基因序列
1.2 方法
1.2.1 DNA提取、PCR扩增及测序 称取0.5 g野猪肌肉组织,参考马婷婷[22]方法提取DNA。DNA提取后加入50~100 μL去离子水溶解,使用核酸分析仪检测浓度,1.2%琼脂糖凝胶电泳中检测DNA完整性,4 ℃保存备用。
引物序列为:F:5′-CACGACCAATGACATG-AAAAATC-3′;R:5′-TGGCCCTCCTTTTCTGG-TTTAC-3′。引物由生工生物工程(上海)股份有限公司合成。
PCR反应体系25 μL:模板DNA(100 ng/μL)1 μL,上、下游引物(10 μmol/L)各1 μL,TaqPCR MasterMix(2×,Blue Dye)12.5 μL,ddH2O 9.5 μL。PCR扩增程序参考Damdin等[23]方法。取2 μL PCR扩增产物进行琼脂糖凝胶电泳,对鉴定正确的PCR产物送生工生物工程(上海)股份有限公司进行双向测序。
1.2.2 序列分析 用BioEdit v 7.0.5软件打开测序峰图,将得到的单峰序列在NCBI数据库中经BLAST比对确认是否为野猪相应序列后,将正反向引物双向测序得到的两条单峰序列使用DNAStar 7.1软件中的Seqman程序进行双向拼接,将拼接后的Cytb基因序列合并整理成Fast文件。
1.2.3 遗传多样性及系统发育分析 使用Mega 7.0[24]软件评估各地区野猪序列的保守位点(conserved sites,C)、简约信息位点(parsimony informative sites,NPi)、变异位点(variation site,V)、碱基组成、种间和种内遗传距离;用DnaSP 6.0软件[25]评估单倍型数目(number of haplotypes,H)、单倍型多样性(haplotype diversity,Hd)、核苷酸多样性(nucleotide diversity,Pi)、平均核苷酸差异(mean number of pairwise differences,K)。利用Kimura双参数距离模型以邻接法(Neighbor-Joining,NJ)构建单倍型间系统发育树,所有的计算均通过1 000次重复,贝叶斯系统发育分析采用MrBayes 3.2.1软件[26]MCMC方法,选择GTR模型,直至分析的相似度<0.01结束运算,使用FigTree v 1.4.3软件进行系统进化树查看和编辑。采用Arlequin软件进行Tajima’sD和Fu’sFs检验,并计算不同地理种群间的遗传分化系数(Fst)[27];使用Network软件中Median-Joining模型构建种群单倍型网络图,网络构建时均选择软件默认参数。
2 结 果
2.1 不同地理种群线粒体Cytb基因序列碱基组成
对测序获得的贵州野猪11条Cytb基因序列在NCBI数据库中进行BLAST比对,结果与GenBank中国内其他9个地区野猪种群线粒体Cytb基因序列的一致性>98%,确认扩增序列为野猪Cytb基因序列。使用BioEdit软件将Cytb基因序列手动补齐,补齐后序列长度为1 140 bp。贵州、台湾野猪种群该片段GC含量均为42.10%,各地区野猪Cytb基因序列都明显存在AT碱基偏好性,符合脊椎动物线粒体Cytb基因碱基组成特点,其中贵州野猪种群Cytb基因序列保守位点为1 134个,简约信息位点为1个,单一多态位点为5个(表3)。

表3 中国部分地区野猪种群的线粒体Cytb基因序列碱基组成分析
2.2 不同地理种群线粒体Cytb基因序列遗传多样性
对本研究中11条贵州野猪种群Cytb基因序列和GenBank中下载的其他地区55条Cytb基因序列进行分析发现,66条序列对应24种单倍型,高频单倍型为Hap_1和Hap_10,其中Hap_1单倍型对应7个地区的20条Cytb基因序列;Hap_10单倍型对应4个地区的16条Cytb基因序列;基于贵州野猪种群11条Cytb基因序列分析发现,贵州野猪种群变异位点为6个;单倍型数量为4个;单倍型多样性值为0.600;核苷酸多样性值为0.00118;平均核苷酸差异值为1.34545(表4)。
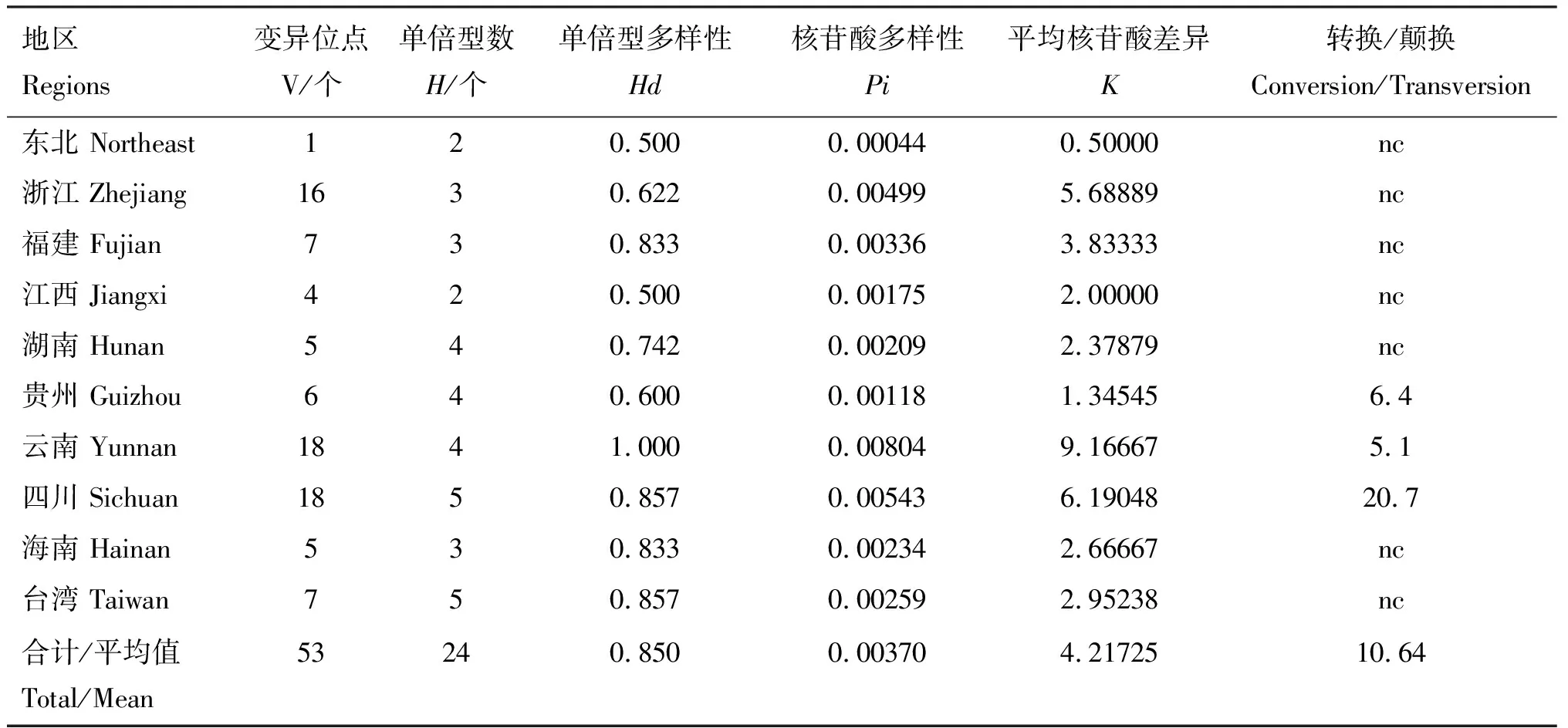
表4 中国部分地区野猪种群线粒体Cytb基因遗传多样性分析
2.3 不同地理种群野猪线粒体Cytb基因种内及种间遗传距离、遗传分化系数分析
按序列来源将野猪Cytb基因序列按照地区进行分组,分析得知各地区野猪种群种内遗传距离在0~0.008之间,贵州野猪种群种内遗传距离为0.001,东北地区野猪种群种内遗传距离为0,种内遗传距离最大为云南野猪种群(0.008);贵州野猪种群与其他地区野猪种群种间遗传距离较小,最大与云南野猪种群之间为0.006(表5)。两两种群间遗传分化系数在-0.111(湖南-台湾省)~0.424(湖南-东北地区)之间;贵州野猪种群同其他9个种群间与福建野猪种群的遗传分化系数最大(0.324),与台湾野猪种群的遗传分化系数最小(0.018);东北地区野猪种群与其他各地区种群的遗传分化系数普遍相对较大,在0.135(东北地区-贵州)~0.424(东北地区-湖南)之间,约半数的野猪种群间存在较高或高水平的遗传分化(表5)。
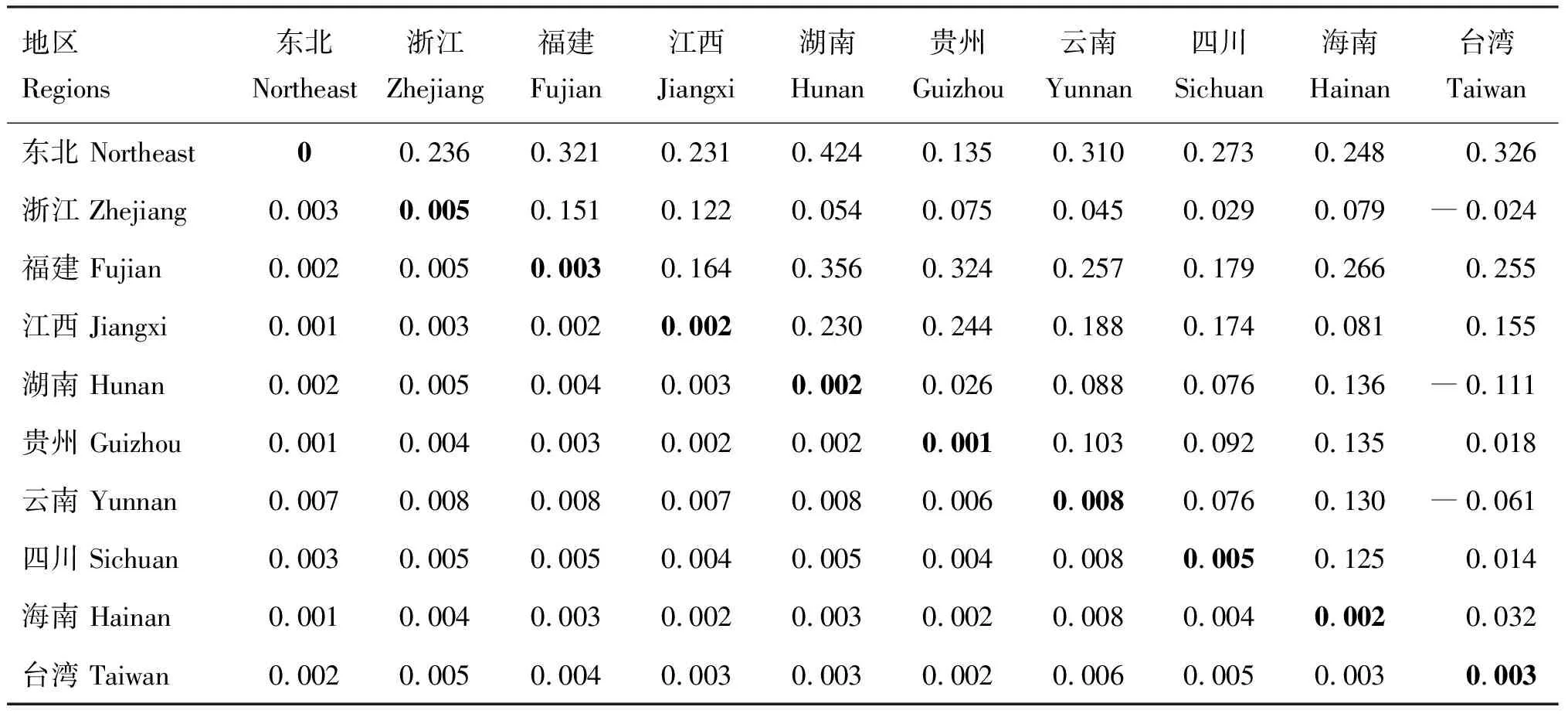
表5 中国部分地区野猪种群Cytb基因种内、种间遗传距离及遗传分化系数
2.4 种群历史动态分析
基于线粒体Cytb基因序列,利用DnaSP 6.0软件对中国不同地区野猪种群进行中性检验分析,并对贵州野猪种群进行碱基错配差异分析,结果显示,贵州野猪11条Cytb基因序列对应4种单倍型,中性检验Tajima’sD值为―1.359(P>0.10),Fu’sFs值为―0.110(P>0.10),两个值均为负值且差异不显著(表6),表明贵州野猪种群在经历种群扩张。对贵州野猪种群Cytb基因序列进行碱基差异错配分析发现,贵州野猪种群线粒体Cytb基因序列碱基差异分布呈单峰分布(图1),也表明贵州野猪种群该阶段在经历种群扩张。
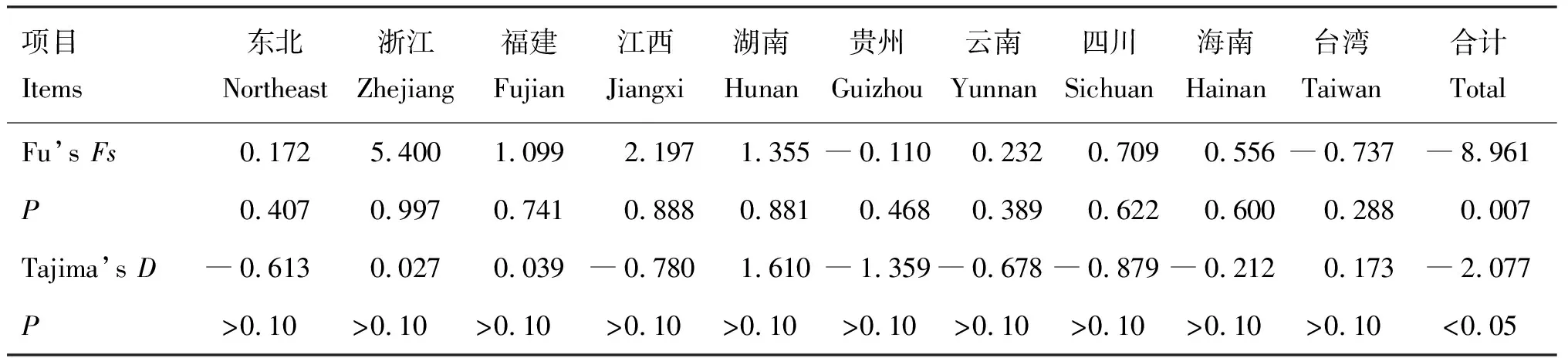
表6 中国部分地区野猪种群Cytb基因片段中性检验分析
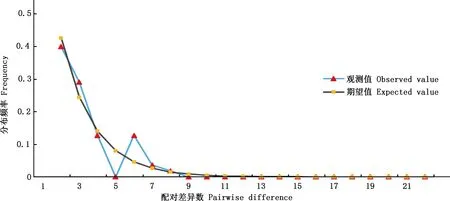
图1 贵州野猪种群Cytb基因单倍型序列错配分布
2.5 种群Cytb基因单倍型系统发育与单倍型网络分析
基于10个地区野猪Cytb基因单倍型序列,以爪哇野猪Cytb基因序列作为外群,采用邻接法(NJ)和贝叶斯方法构建系统发育树,得到了相同的结果(图2)。外群爪哇野猪与中国野猪种群之间直观地形成2个分支,国内各地区野猪种群单倍型分支混合。贵州野猪11条序列对应4种单倍型:Hap_1、Hap_10、Hap_21和Hap_22(表7),4种单倍型之间并未聚集,种群在不同时间段发生分化;四川野猪单倍型(Hap_2)、云南野猪单倍型(Hap_18、Hap_19、Hap_20)、贵州野猪单倍型(Hap_21)相对其他地区分化时间较早。通过Network软件构建各地区野猪Cytb序列单倍型网络图,其中贵州野猪单倍型(Hap_1、Hap_10)位于10个地区野猪单倍网络关系的分化中心,这2种单倍型是大部分野猪群体的优势单倍型,并与其他单倍型短支相连,在24种单倍型中相对原始,可以看作是大部分野猪种群的祖先单倍型,周围的单倍型由这2个单倍型分化衍生得到,各地区单倍型分化与地理分布之间的相关性并不明显(图3)。
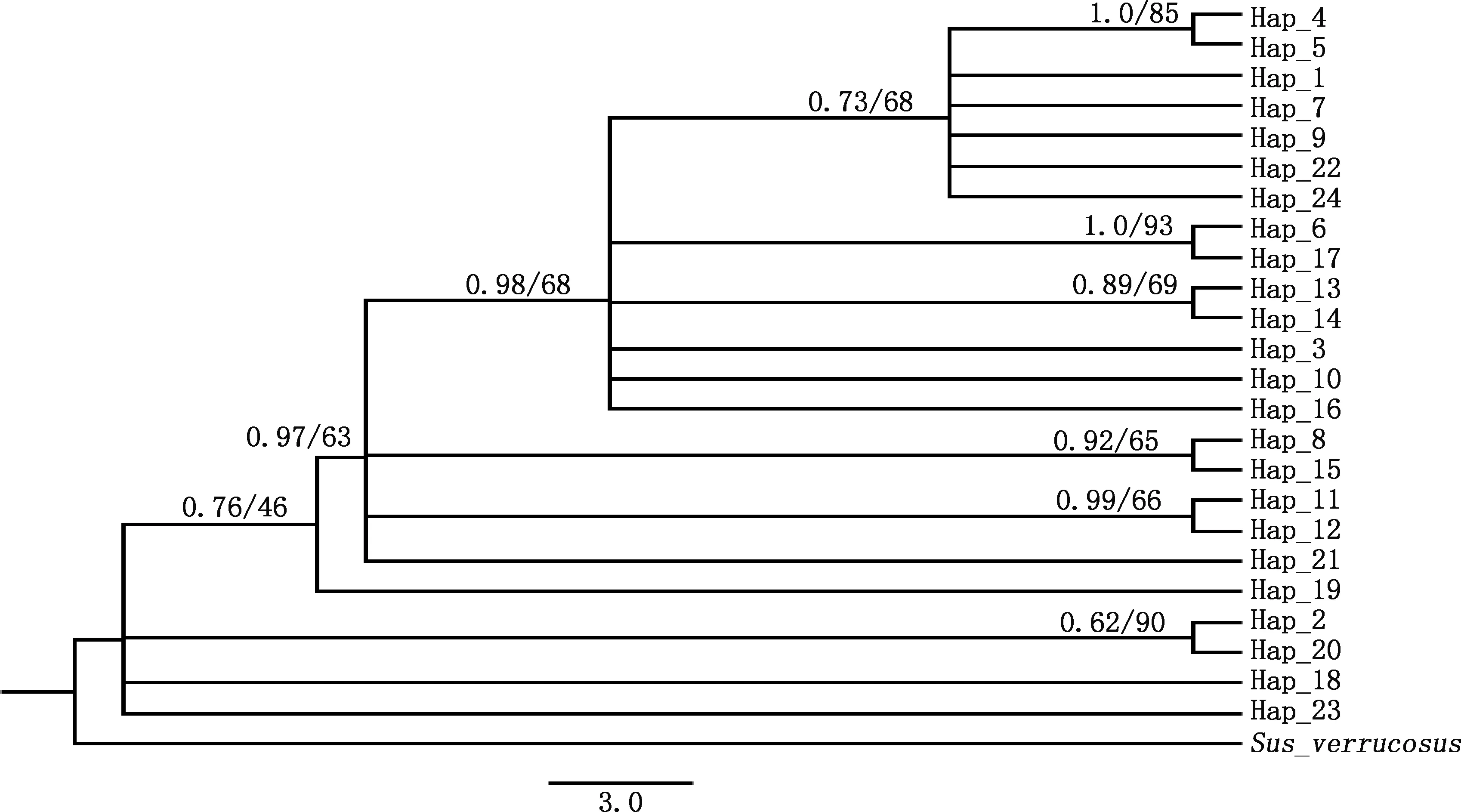
分支数字代表分支后验概率BI/NJ

表7 中国部分地区野猪种群Cytb基因对应的单倍型

图3 中国部分地区野猪群体Cytb基因的单倍型网络分析图
3 讨 论
3.1 贵州野猪种群的遗传多样性分析
遗传多样性是物种进化的重要依据保障,通常认为,物种的遗传多样性越高,其后代的适应能力越强,越容易发生扩张,反之种群的适应能力则会降低[28]。物种的单倍型多样性及核苷酸多样性是衡量物种遗传多样性的2个重要指标[29]。碱基多样性是遗传多样性的基础,研究显示,贵州野猪种群线粒体Cytb基因序列AT含量高于GC含量,与其他地区野猪种群碱基偏好性相似,符合哺乳动物线粒体DNA的进化特点,这种偏好性反映了物种受到外界自然突变与碱基选择压力的影响较高,因为线粒体基因片段中A、T 2种碱基之间键能较低,容易引起碱基发突变,导致群体性状发生变异[30]。本研究结果显示,10个地区野猪Cytb基因序列单倍型多样性在0.500~1.000之间,核苷酸多样性在0.00044~0.00804之间,贵州野猪种群Cytb基因序列片段的单倍型多样性为0.600,核苷酸多样性为0.00118,符合Grant等[31]依据物种的演化模式以线粒体单倍型多样性和核苷酸多样性推测形成种群演化经历的4种模式的种群扩张阶段,即当种群的单倍型多样性较高(>0.5)但核苷酸多样性较低(<0.5)时,种群可能正在经历由瓶颈效应后引发的快速扩张;贵州野猪种群的遗传多样性表现出高单倍型多样性、低核苷酸多样性的趋势,说明贵州地区野猪种群该阶段正经历种群扩张。
3.2 贵州野猪种群的遗传分化分析
物种种内、种间的遗传距离差异程度反映了各地理种群间亲缘关系远近或种群间遗传相似性[32]。本研究结果显示,各野猪群体间平均遗传距离范围在0.001~0.008,云南和四川野猪种群与其他地区种群间的遗传距离较大,与白立景[30]研究结果类似。贵州种群种间的遗传距离与其他地区种群相比中等,种内遗传距离(0.001)较其他地区小,说明贵州野猪种群之间亲缘关系较近,种群内基因存在相互交流;优越的栖息环境及逐渐减少的人为干扰为贵州野猪种群内的基因交流提供了良好的条件,促进了野猪种群基因交流,与胡超超[33]和白立景[30]研究结果一致。
遗传分化系数(Fst)是反映2个种群间遗传分化程度的一个指标[34]。Rousset研究认为,Fst值范围为0~1:Fst<0.05时种群间遗传分化不显著,分化程度低;0.05
3.3 贵州野猪种群的历史动态分析
种群历史演化通常采用2种方法进行检测:一种方法是通过Tajima’sD和Fu’sFs中性检验;另一种方法是通过碱基差异性分析。中性检验时,Fu’sFs检验对种群扩张的反应更加敏感[27]。本研究中Tajima’sD与Fu’sFs检验均证实各地区野猪群体遵行中性进化,通过Cytb基因序列计算贵州地区野猪Tajima’sD值和Fu’sFs值是负值且无显著差异,碱基错配分布呈单峰,说明贵州野猪种群该阶段正在经历种群扩张,与张怡[36]使用线粒体控制区得到的结果相同,遗传多样性结果也相同。基于10个地区野猪线粒体Cytb基因单倍型序列构建贝叶斯系统发育树和单倍型NJ系统发育树发现,各种单倍型之间分布混杂,其中四川、湖南、台湾部分野猪种群聚集形成分支,其余地区无明显以地理聚群形成单独的分支,与白立景[30]研究表明国内种群无明显地理划分一致。
贵州野猪种群包含的4种单倍型并未聚集在一起,而是与其他单倍型散布混杂在一起。出现以上情形可能是以下几个方面原因造成的:①野猪自身的日常生活习性不同;②不同野猪种群生活环境不同;③由于各个地理省份的分析序列局限性,无法完全覆盖地理区域内的样本。本研究中,四川野猪部分单倍型(Hap_2、Hap_4和Hap_5)与其他单倍型关系较远,与四川野猪种群作为国内野猪的分化起源中心相关,同样符合白立景[30]研究结果;贵州野猪序列中Hap_1和Hap_10属于国内野猪种群的共享单倍型,其余2种单倍型(Hap_21、Hap_22)属于贵州种群特有单倍型,说明贵州野猪种群根据自身环境发生了物种分化;单倍型网络图显示,10个地区的共享单倍型为Hap_1和Hap_10,各单倍型间亲缘关系较近,没有形成明显的系统地理结构,结果与系统发育树相似,2种共享单倍型是国内部分野猪种群较原始的祖先单倍型,与李崇奇[12]、张怡[36]研究认为川黔和云贵高原等地是亚洲野猪分化起源中心相符,且贵州野猪种群单倍型与其他单倍型之间存在相互交流的情况,导致种群遗传多样性受到影响,可能对贵州种群种质资源造成影响,并对亚洲野猪种群分化中心造成威胁。本研究中贵州野猪样本的抽样范围较为广泛,但其他9个地区的野猪样本具体来源尚不明确,因此进行比较分析可能有不足之处,但能较为客观地反映各地区野猪种群间的遗传多样性及其系统发育情况。
4 结 论
贵州野猪种群在历史上较为稳定,现处于历经瓶颈后的种群扩张阶段;种群内遗传距离较小、亲缘关系近,遗传多样性低于全国多个地区,说明贵州野猪种群发展潜力较小。因此,应采取有效的措施增加和保护其遗传多样性,进一步加强野猪种群管理和种质利用,促进野猪保护遗传学及其对环境适应机制的研究。

