利用CRISPR/Cas9技术建立OXTR基因敲除猪胎儿成纤维细胞系
刘静静 刘晓蕊 李琳 王盈 杨海元 戴一凡
(南京医科大学江苏省异种移植重点实验室,南京 211166)
自闭症是一种严重的神经发育疾病,临床表现为社会交往障碍、语言交流障碍和刻板重复的行为方式[1]。自闭症致残率较高,且患病率有逐年增长的趋势,但尚未开发出特异性的治疗药物,给患者和社会带来沉重的负担。自闭症病因和发病机制十分复杂,目前受到关注最多的是催产素系统假说。催产素是一种具有高度保守结构的神经肽,大量的动物实验表明它与情感反应和社会认知等社会行为有关。催产素的生理作用是通过其唯一的受体OXTR来介导的。人类催产素受体(OXTR)属于I类G蛋白偶联受体,位于3p25-3p26.2,由3个内含子和4个外显子组成[2]。催产素受体分布在杏仁核、纹状体、海马等调节社会认知的重要区域[3-4]。OXTR基因变异可引起大脑结构改变并与社会行为相关联,如下丘脑和杏仁核的病变以及社交缺陷等[5-8]。研究表明OXTR基因变异与自闭症存在相关性,但催产素受体影响自闭症的分子机制还不明确。
OXTR基因敲除的动物模型是揭示OXTR生理功能的重要工具。目前,已有OXTR基因敲除的小鼠、斑马鱼被用于自闭症相关行为缺陷的研究[9-10],但它们存在社交信号(发声、面部表情)和大脑活动模式有限等缺陷[11]。选择更理想的自闭症动物模型显得尤为迫切。猪和人生理特征和解剖结构更为相近,基因组大小相近、基因数量和结构类似[12-13]。猪大脑回旋类似于人的新皮质且它们灰质和白质的分布相似[14-15]。此外,猪的快速繁殖期(性成熟5-6个月)和大窝产仔数(平均7-8头仔猪),在构建人类疾病模型方面比非人类灵长类动物具有明显优势。然而,目前还未见OXTR基因敲除猪报道,限制了在猪上开展关于OXTR对于社会认知影响的实验研究。
近年来,CRISPR/Cas9基因编辑技术的不断发展极大推动了创建基因编辑猪疾病模型的研究[16-17]。本研究利用CRISPR/Cas9技术构建OXTR基因敲除的巴马公猪PFFs细胞系,为构建OXTR基因编辑猪模型进而深入研究遗传性自闭症的发病机制和防治策略奠定基础。
1 材料与方法
1.1 材料
原代巴马猪胎儿成纤维细胞由本组实验室提取培养。DH5α感受态细胞和质粒抽提试剂盒购自北京天根生化科技有限公司,pX330质粒(Addgene 42230)、Bbs I限制性内切酶、T4 DNA连接酶均购自美国New England Biolabs 公司,细胞电转仪购自德国Lonza公司,DMEM培养液、胎牛血清、G418药物均购自美国Gibco 公司,引物序列由生工生物工程(上海)股份有限公司合成。
1.2 方法
1.2.1 人/猪OXTR的同源性分析 通过NCBI数据库查找并下载人和猪等多个物种的OXTR的氨基酸序列,利用MEGA-X软件中邻接法(neighbor-joining method)构建系统进化树,自展分析1 000次用于检测系统进化树的稳定性。在线工具 iTOL(https://itol.embl.de/)进一步绘制系统进化树。
1.2.2 人/猪OXTR的结构分析 采用DNAMAN软件、DNASTAR软件对人/猪OXTR的一级、二级结构进行分析,使用BLAST中的Global Align程序计算一致性和相似性,Chou-Fasman算法预测人/猪OXTR的α螺旋、β折叠、β转角的比例。接着利用Swiss Model在 线 工 具(https://www.swissmodel.expasy.org/)对人/猪OXTR三维结构进行建模,采用PyMOL软件比较两者蛋白结构的相似度。
1.2.3 猪OXTR关键残基和结构域的鉴别 利用NCBI当中的CD-search工具搜索鉴定猪OXTR关键残基和结构域。输入猪OXTR氨基酸序列,同时在右方OPTIONS中选择数据库选择具有保守结构域数据库(conserved domain database,CCD库),提交完成后搜索匹配结果。
1.2.4 sgRNA靶点设计 根据GenBank中公布的猪的OXTR基因序列(NC_010455.5),设计引物(表1),并提取巴马猪PFFs基因组DNA进行PCR扩增,测序验证是否与公布的基因序列相符。利用在线工具CRISPOR(http://crispor.tefor.net/)针对第2外显子区筛选关键靶向位点,设计Oligo合成。
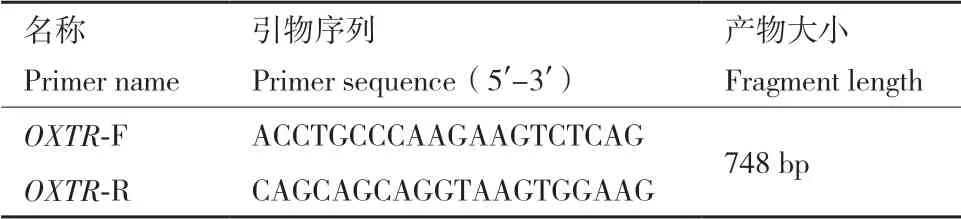
表1 OXTR基因exon 2序列扩增引物Table 1 Amplification primers for exon 2 sequene of OXTR gene
1.2.5 CRISPR/Cas9打靶载体构建 用T4 DNA连接酶将Oligo退火形成的双链二聚体与Bbs I酶切的pX330骨架质粒连接,转化入DH5α感受态细胞,均匀涂布于Ampicillin抗性的LB琼脂培养板上。挑取单菌落进行测序鉴定,扩增鉴定连接成功的菌液,并用质粒抽提试剂盒提取。
1.2.6 细胞转染 复苏冻存的野生型巴马猪原代PFFs,用细胞培养液培养至细胞长满皿底90%左右转染。利用核转染仪将打靶载体1 μg及Neomycin抗性质粒(由本实验室构建)2 μg共转染至野生型PFFs,核转染程序设定U-023。
1.2.7 单细胞克隆的筛选与鉴定 转染24 h后,用含有G418药物的完全培养液培养10 d左右,可见G418抗性单细胞克隆形成。用克隆环挑选状态佳的单克隆,转移至24孔板。第2天用含G418的培养液培养,直至细胞长满皿底,胰酶消化并传代至12孔板,待12孔板中的细胞长满后,即可冻存备用;遗留在24孔板中的细胞长满后用NP40裂解,裂解程序60℃,60 min;95℃,10 min,以裂解产物为模板PCR,并将产物送生工生物工程(上海)股份有限公司测序鉴定。
2 结果
2.1 人/猪OXTR生物信息学分析
系统进化树分析显示,猪的OXTR进化距离与人接近(图1)。经DNAMAN比对分析,人/猪OXTR氨基酸序列高度一致(一致性/Identity=91%;相似性/similarity=93%)(图2-A)。利用DNASTAR软件Protein模块中对人和猪OXTR的二级结构进行分析,Chou-Fasman算法预测人OXTR的α螺旋占30.3%,β折叠占40.6%,β转角占21.6%,猪OXTR的α螺旋占42.2%,β折叠占37.7%,β转角占19.3%(图2-B)。人和猪OXTR具有相似排布和占比的α螺旋、β折叠、β转角。而人和猪OXTR三维建模显示也具有极高相似度的三维空间排列,RMSD值为0.009(图2-C)。以上生物信息学分析表明,人/猪OXTR序列和结构具有高度的同源一致性,推测人/猪OXTR具有相似的生物学功能。
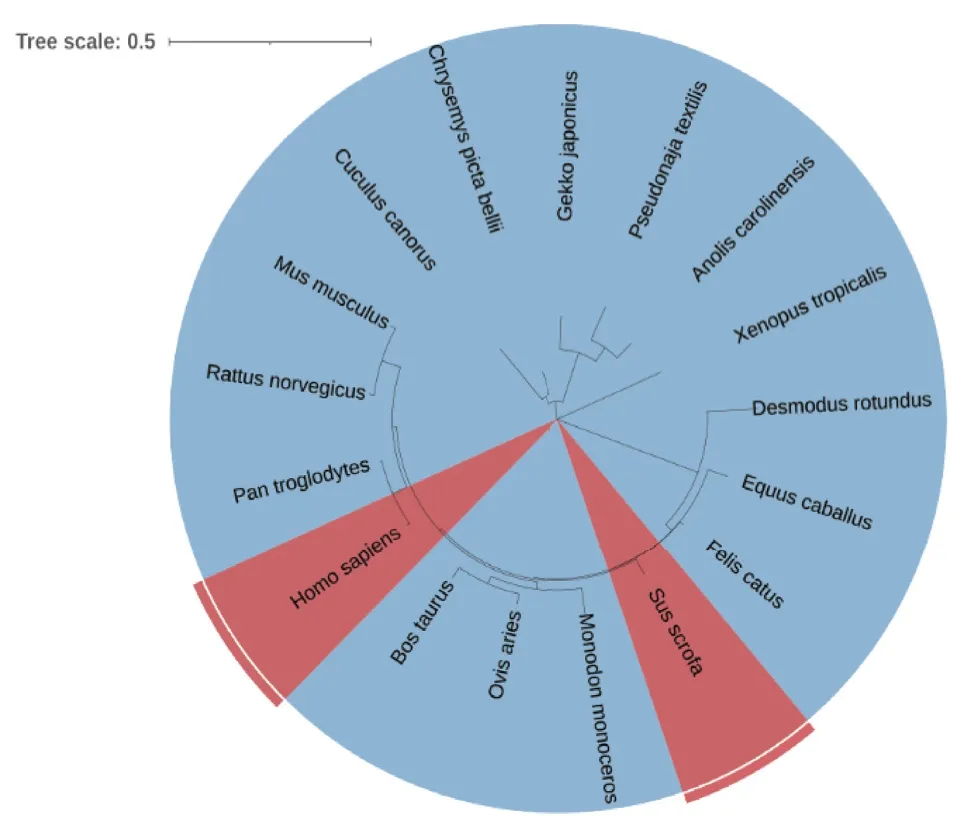
图1 不同物种OXTR序列构建的系统进化树Fig.1 Phylogenetic tree of OXTR sequences of different species
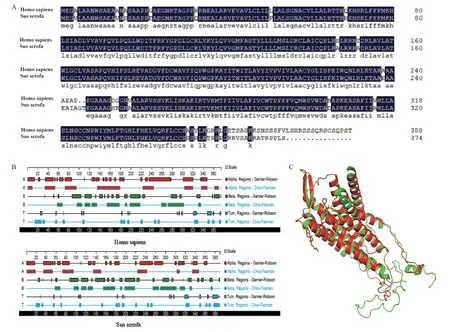
图2 人/猪OXTR的一级、二级、三级结构分析Fig.2 Analysis of primary,secondary,and tertiary structures of OXTR between humans and pigs
2.2 猪OXTR关键结构域的鉴别和靶点区域的选择
在CD-search的CDD分析中,鉴别出猪OXTR的7个α螺旋结构域,分别是41-67,74-101,112-142,152-173,197-226,270-300,310-335间的氨基酸残基(图3)。研究发现,人类OXTR的7个α螺旋结构域中,第2-5个跨膜结构域与底物的相互作用有关,其中第2个跨膜结构域还与受体的激活相关[18]。猪OXTR的第2-5个跨膜结构域由猪OXTR的第2外显子编码。因此,本研究选择猪OXTR的第2外显子作为敲除的靶点位置。

图3 猪OXTR关键残基和结构域的鉴别Fig. 3 Identification of domains and key residues of porcine OXTR
2.3 CRISPR/Cas9打靶载体的构建
针对巴马猪OXTR基因第2外显子序列,设计了2对sgRNA oligos(表2、图4-A)。测序结果显示,Oligo退火形成的双链sgRNA-1、sgRNA-2成功插入线性化的pX330骨架质粒中(图4-B-D),并用试剂盒成功提取打靶载体质粒。
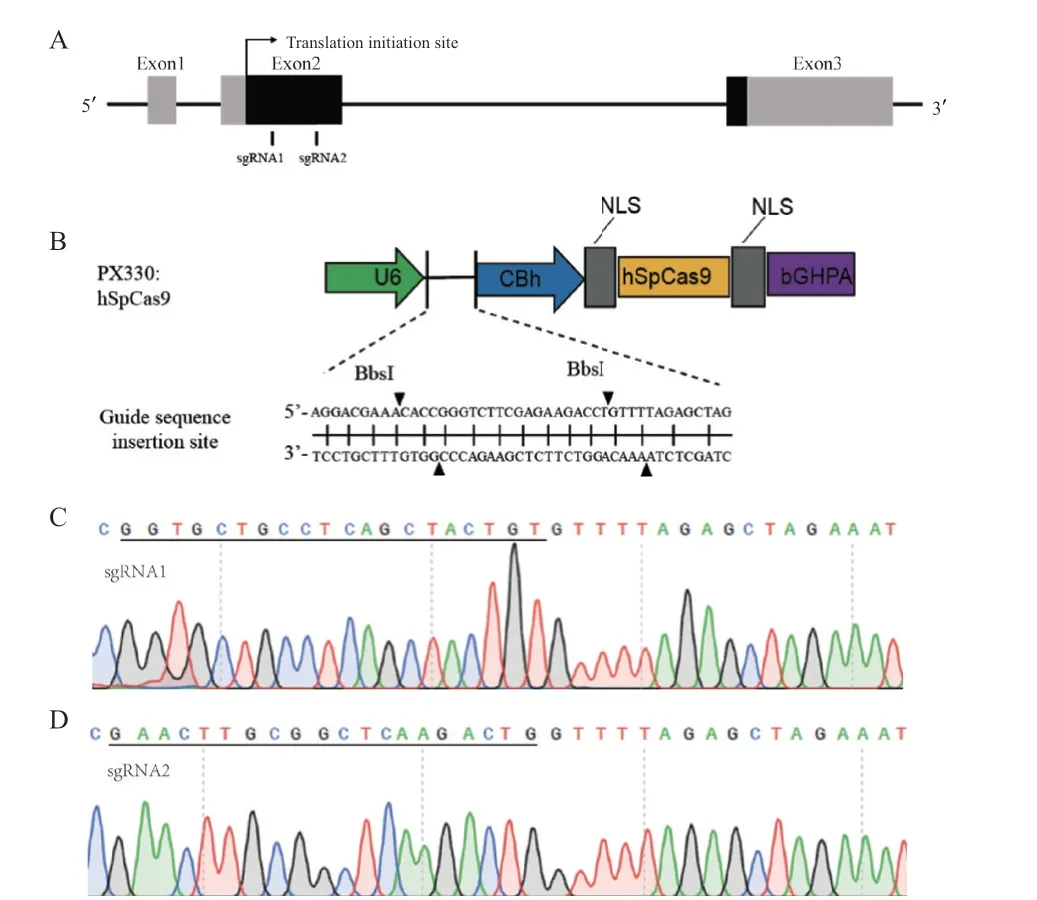
图4 OXTR基因靶点和重组载体测序Fig.4 Target of OXTR gene and sequencing of recombinant vectors
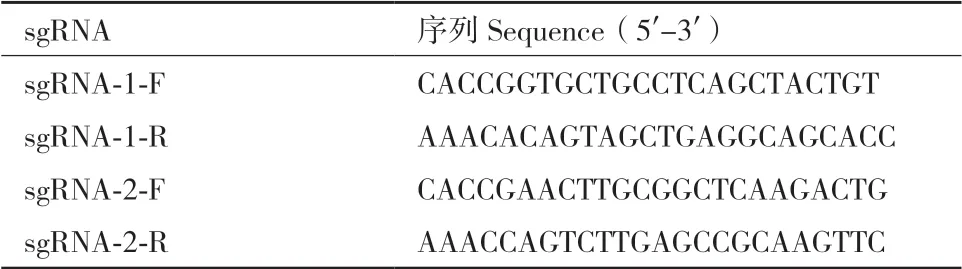
表2 OXTR基因靶向位点的sgRNA寡核苷酸序列Table 2 Oligonucleotide sequences of sgRNAs at OXTR targeting sites
2.4 OXTR基因敲除细胞系的鉴定
将靶向猪OXTR的CRISPR/Cas9质粒和Neomycin抗性表达载体共转染猪胎儿成纤维细胞,通过G418(1 mg/mL)筛选,获得到了抗性的单克隆细胞系。提取抗性的单克隆细胞的基因组DNA,PCR扩增OXTR靶点区域片段后进行测序,并与野生型猪OXTR基因序列进行比对。结果显示共获得了5个纯合的OXTR双等位基因敲除的细胞克隆,其基因型可分为3种类型,在OXTR的靶点区域产生了411 bp的缺失(mutant type 1),412 bp的缺失(mutant type 2),289 bp的缺失和289 bp的插入(mutant type 3)(图5,表3)。

表3 OXTR 敲除PFFs的基因型Table 3 Genotypes of OXTR-knockout PFFs
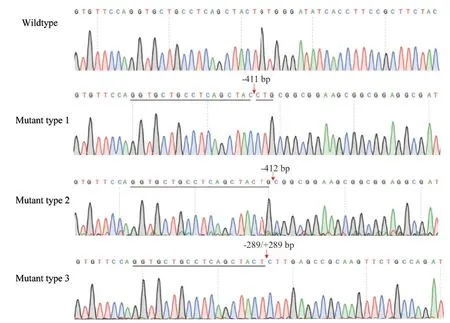
图5 OXTR敲除PFFs的测序结果Fig.5 Sequencing results of OXTR-knockout PFFs
3 讨论
自闭症是一种极其复杂的疾病,与遗传、环境等多种因素密切相关。双生子研究证实遗传因素是影响自闭症的重要因素[19]。全基因组测序确定OXTR基因是自闭症的候选基因[20]。催产素受体作为神经通路的重要环节,其异常可能造成细胞内钙离子浓度变化,导致神经元功能受损,进而影响社会认知功能[21]。因此,OXTR基因与自闭症存在高度相关性。由于自闭症涉及多种复杂的高级神经活动,要为自闭症研究和治疗提供更有利支持,更高级的OXTR基因敲除动物模型非常必要。猪被认为是理想的人类疾病动物模型,因此,本研究选择用猪胎儿成纤维细胞的OXTR基因进行编辑。
猪OXTR基因位于第13号常染色体上,生物信息学分析提示,猪OXTR基因突变后可能产生和人类似的遗传效应和病理表型。sgRNA原则上可以设计在任何外显子区域,对目标序列进行切割,通过DNA自身修复机制引入突变导致基因的敲除。由于在OXTR受体基因的5′上游区域存在许多转录因子结合位点,这些位点在调节受体转录发挥主要作用[22],结合对猪OXTR关键结构域的分析,本研究在第2外显子选择靶基因的识别序列,设计并合成sgRNA。CRISPR/Cas9优势在于操作简单、实验周期短、成本低。本课题组已利用此技术成功构建色氨酸羟化酶2(Tph2)基因敲除猪、OSBPL2敲除猪等[23-24],验证了CRISPR/Cas9技术在猪基因组修饰上的有效性。我们前期研究中选择单个sgRNA进行基因打靶,获得的细胞克隆多数为靶点区域产生小片段插入缺失,且敲除的效率相对较低[25]。本研究设计2个靶向OXTR的sgRNA进行打靶。结果显示,5个OXTR双等位基因敲除的单细胞克隆均在预期的靶点位置发生了大片段的碱基缺失或插入,确保OXTR蛋白被完全破坏,表明利用2个sgRNA同时进行基因打靶能够显著提高基因敲除的效率。
据世界卫生组织估计,全球自闭症病患病率为6.25%,男性患病率约为女性的4倍。我们推测OXTR突变的雄性巴马猪更有可能出现自闭症的病理表型,因此本研究中我们仅对雄性的巴马猪胎儿成纤维细胞进行了OXTR基因的打靶。本研究中获得的OXTR基因敲除的巴马猪胎儿成纤维细胞后续将用作SCNT(somatic cell nuclear transfer)的核供体细胞,通过胚胎移植构建OXTR敲除巴马猪疾病动物模型。
4 结论
本研究基于CRISPR/Cas9基因编辑工具在巴马公猪成纤维细胞系上高效实现了OXTR基因的打靶,成功构建了OXTR基因敲除的成纤维细胞系,为建立OXTR敲除的猪模型提供了实验材料。

