高山松地上生物量遥感估算的不确定性分析
黄屹杰,张加龙,胡耀鹏,程 滔
(1. 西南林业大学 林学院,云南 昆明 650233;2. 国家基础地理信息中心 调查监测部,北京 100830)
森林生物量是森林生态系统的最基本数量特征,是研究林业和生态问题的基础[1]。地上生物量(aboveground biomass, AGB)是森林生态系统生产力的重要指标和质量的综合体现,对研究全球气候变化具有重要意义。目前,森林地上生物量的调查方法有地面调查和遥感监测2种。传统的地面调查法耗时长、成本高,而遥感监测方法快速、无损,具有在更大尺度上应用的优势,已成为地上生物量估测的主流[2-3]。在森林调查中,常使用统计模型来预测样地内个体树木的生物量,然后对同一树种的单株地上生物量进行汇总,来作为遥感估算生物量的训练数据和精度评估数据[4]。用于地上生物量估测的遥感数据源包括光学遥感、机载雷达、激光测量数据等[5]。与成本较高的机载雷达数据相比,光学影像穿透森林冠层获取树干信息的能力较差,但随着新算法的不断改进,使用光学遥感影像进行地上生物量估算的精度也在不断提高[6]。
目前,遥感估算地上生物量在因子选取、建模方法、数据饱和等方面存在较多不确定性问题[7-8]。区域尺度森林地上生物量遥感估算包含测量和抽样、单株生物量模型、遥感因子和地形因子等不确定性来源[9-10]。其中,与测量和抽样误差相比,模型的不确定性对地上生物量估算的影响较大,如SHETTLES等[11]利用激光雷达和地面数据估测地上生物量发现:模型不确定性占总不确定性的55%。同时,用于建模的生物量数据是使用该区域树种的异速生长方程计算得出的。因此,单株生物量模型的误差会传播到样地级的生物量估测,从而影响遥感估算生物量的不确定性[12-13]。现阶段不确定性的量化方法主要分为3种[14]:过程模型分析法、随机误差传递法和Monte Carlo模拟法。
本研究基于云南省香格里拉市Landsat 8影像和外业调查数据,建立了生物量遥感估测模型,分析了遥感估算样地高山松Pinus densata地上生物量的总不确定性,以期为提高森林地上生物量估算精度提供参考。
1 研究地区与数据
1.1 研究区概况
研究区位于云南省西北部的迪庆藏族自治州香格里拉市 (26°52′~28°52′N,99°20′~100°19′E),该区总面积为11 613 km2,平均海拔为3 459 m。香格里拉市的森林覆盖率较高,达75%,主要植被类型为寒温性针叶林,优势树种有云杉Picea asperata、冷杉Abies fabri、高山松、云南松Pinus yunnanensis、高山栎Quercus semicarpifolia等。
1.2 数据来源
于2015年11月和2016年3月,在每个乡镇高山松纯林区域随机布设60块样地,大小为30 m×30 m,且每块样地间隔3 km以上。样地高山松林分的龄级主要为近熟林和成熟林。外业调查记录胸径大于5 cm样木的树高、胸径,同时对每块样地的林分密度进行了计算(表1)。
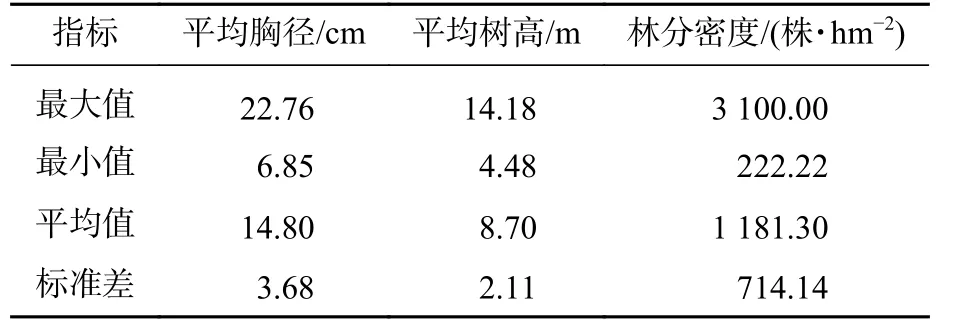
表1 外业调查样地统计Table 1 Basic statistics of the sample plots of field surveys
Landsat 8影像数据来源于美国地质调查局(http://glovis.usgs.gov/),3景数据为2015年11—12月的成像,云量均小于2%。
2 研究方法
2.1 样地地上生物量计算
采用每木检尺,通过胸径、树高和单株生物量模型[15]计算每株地上生物量,进而得出每块样地的地上生物量。单株生物量模型()如下:

2.2 Landsat数据处理
对影像数据进行辐射定标,采用FLAASH方法进行大气校正。以研究区SPOT-5影像为参考数据,选取100个地面控制点,采用二项式方法对影像进行几何校正,使用双线性内插法将影像重新采样为30 m×30 m,误差控制在1个像元内[8];接着,采用坡度匹配模型[16]进行地形校正。具体操作方法参考文献[17]。
对预处理后的影像数据提取4类遥感因子:①原始波段因子,分别为C、B1、B2、B3、B4、B5、B7;②植被指数因子,分别为B43、B42、B53、B54、B57、B73、B74、B3Albedo、 B473、NDVI、ND32、ND53、ND54、ND57、ND452、DVI;③信息增强因子,分别为VIS123、Albedo、MID57;④纹理信息因子[18],分别为均值(ME)、方差(VA)、同质性(HO)、反差(CO)、相异(DI)、熵(EN)、角二阶矩(SM)、相关性(CR)、偏度(SK)。用R5和R9分别代表5×5和9×9窗口,如R9B5CR代表9窗口第5波段的相关性纹理。
岳彩荣[19]提取了5×5、7×7、9×9、15×15的4种窗口纹理信息因子对香格里拉森林生物量进行了遥感估测;张加龙等[20]基于遥感影像和连续清查固定样地对高山松地上生物量进行估算时,使用了5×5、9×9的2种窗口纹理信息因子。以上研究表明:高山松地上生物量与5×5和9×9窗口的纹理信息因子相关性更强。因此,本研究的纹理信息因子选用5×5和9×9窗口。4类遥感因子的提取和计算参考文献 [8, 17, 20]。
2.3 因子筛选方法
基于SPSS软件对样地地上生物量与备选遥感因子进行相关性分析,结果按照Pearson系数进行排序。本研究因子入选的显著性水平为P≤0.05,因子剔除的水平为P≥0.10,得到相关性排前14个的遥感因子。为避免解释变量之间的多重共线性问题,将各个遥感因子之间相关性较高的因子剔除,最终筛选9个因子参与模型建立,分别为B74、9窗口第5波段相关性(R9B5CR)、9窗口第6波段方差(R9B6VA)、9窗口第3波段熵(R9B3EN)、5窗口第5波段角二阶矩(R5B5SM)、9窗口第6波段相关性(R9B6CR)、5窗口第3波段熵(R5B3EN)、5窗口第4波段熵(R5B4EN)、5窗口第4波段偏度(R5B4SK)。
2.4 建模方法
遥感估算高山松地上生物量的3种方法分别为多元线性回归(MLR)、梯度提升回归树(GBRT)以及随机森林(RF)。从60个样地调查数据中随机选取48个(80%)进行建模,剩余12个(20%)用来独立性检验。建模和检验样本的地上生物量统计结果见表2。
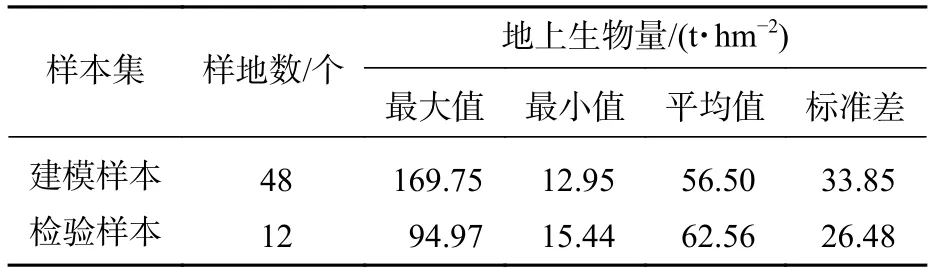
表2 建模和检验样本的地上生物量实测值Table 2 Measured values of aboveground biomass of modeling and testing samples
2.4.1 多元线性回归 多元线性回归模型可以同时对多个解释变量与1个因变量进行拟合,用回归方程来表示拟合关系。本研究多元线性回归建模在SPSS软件中实现。
2.4.2 梯度提升回归树 该方法通过构建多个弱分类器,经过多次迭代最终组合成1个强分类器[21]。本研究基于Python语言的Sklearn工具包提供的梯度提升回归树算法,对高山松地上生物量与遥感因子进行建模分析。在建模分析前需要对弱分类器的最大迭代次数(n_estimators)、学习速率(learning_rate)、最大深度(max_depth)等参数进行确定。参数确定往往需要遵循一定的经验,比如最大迭代次数通常在预测值收敛的情况下越小越好,学习速率通常小于0.10,最大深度通常不应大于15。依据这些经验设置上述参数的范围,比较参数值组合下模型学习的效果,以得到模型输出预测结果[8]。本研究根据拟合优度的最佳参数组合,选取最大迭代次数为60,子采样为0.5,学习速率为0.05,决策树最大深度为7,叶子节点最少样本数为3。
2.4.3 随机森林 随机森林适用于多数分类与回归的问题,由一系列决策树组成。基于Python语言的Sklearn工具包进行高山松地上生物量建模分析。程序使用bootstrap重采样方法从样本集中提取多个重采样样本进行建模,之后对多个模型值进行预测并组合,再通过投票得出最终值,一般会在随机产生的分类树中选出重叠次数最多的决策树作为最终模型[6]。本研究初步设置决策树数量为50,逐步增加决策树数量模拟建模过程,回归误差趋于稳定,最终确定决策树数量为300。
2.5 精度评价方法
采用的评价指标包括决定系数(R2)、均方根误差(RMSE)、预测精度(F)。各指标计算如下:
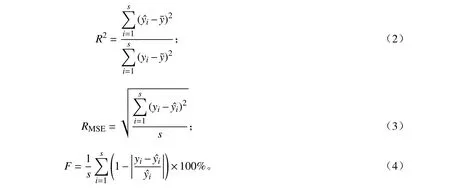
2.6 不确定性分析与度量
2.6.1 模型残差变异不确定性计算 样地尺度上单株生物量模型的残差不确定性()计算如下:

采用六步法[23]计算地上生物量遥感估算模型残差变异产生的误差:第1步升序排列样地地上生物量实测值(y);第2步利用生物量模型的预测值()计算残差(),残差为样地地上生物量实测值与预测值的差值;第3步将60块样地进行分组,每6块地为1组,共10组;第4步计算每组样地地上生物量预测值的平均值()及残差标准差。每组样地地上生物量预测值的平均值、残差、残差标准差计算如下:
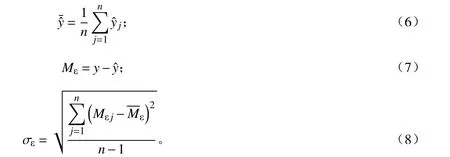

第6步将各块样地地上生物量预测值代入拟合后得出的公式,对所有样地的残差标准差求和,除以地上生物量实测值的和,从而得到模型残差的不确定性。
2.6.2 模型参数不确定性计算 对于单株生物量模型和多元线性回归模型的参数不确定性,可通过泰勒级数一阶展开式进行量化计算。生物量模型经泰勒级数一阶展开如下:

3 结果与分析
3.1 样地尺度上地上生物量的不确定性
单株生物量模型参数的协方差矩阵为:

将协方差矩阵带入式(11),得出单株生物量模型的参数不确定性为12.42%。
由式(5)计算模型的残差不确定性为10.76%。对残差不确定性与参数不确定性合成,可得出单株生物量模型的不确定性为16.43%。当模型误差叠加到样地尺度时,通过式(14)得出样地尺度不确定性为7.07%。
3.2 多元线性回归 (MLR)模型的不确定性
本研究建立了地上生物量与遥感因子的多元线性回归模型,根据拟合优度选出最佳模型为:
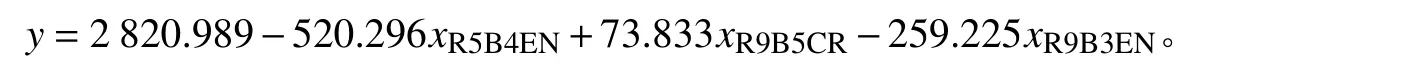
其中:y表示地上生物量预测值,xR5B4EN为5窗口第4波段熵,xR9B5CR为9窗口第5波段相关性,xR9B3EN为9窗口第3波段熵。模型R2为0.326,RMSE为26.12,预测精度为64.91%(图1A)。
对残差标准差(y)和样地地上生物量预测分组平均值(x)进行相关性拟合发现:拟合效果最佳的模型为计算后得出多元线性回归模型的残差不确定性为34.86%(图 1B)。

图1 基于多元线性回归的地上生物量预测效果和残差不确定性Figure 1 Prediction effect and the residual uncertainty of aboveground biomass based on multiple linear regression
多元线性回归模型参数的协方差矩阵为:
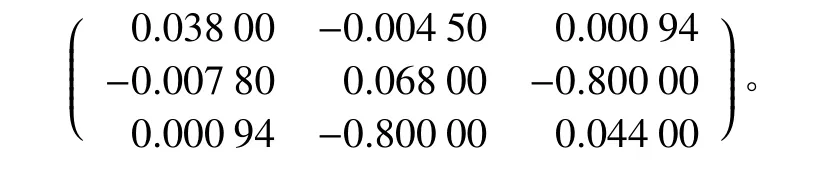
将协方差矩阵代入式(11)可得出参数变异不确定性为21.30%。对残差不确定性和参数不确定性合成,通过多元线性回归模型,估算出高山松地上生物量的不确定性为40.84%。
3.3 梯度提升回归树 (GBRT)模型的不确定性
图2A所示:梯度提升回归树模型的R2为0.815,RMSE为14.24,预测精度为74.72%。图2B所示:残差标准差(y)与样地地上生物量预测分组平均值(x)拟合的最佳模型为 y =0.260 1x-2.127 3,R2=0.670,得出残差不确定性为22.01%。非参数建模方法不考虑参数不确定性,基于梯度提升回归树模型估算得出高山松地上生物量的不确定性为22.01%。
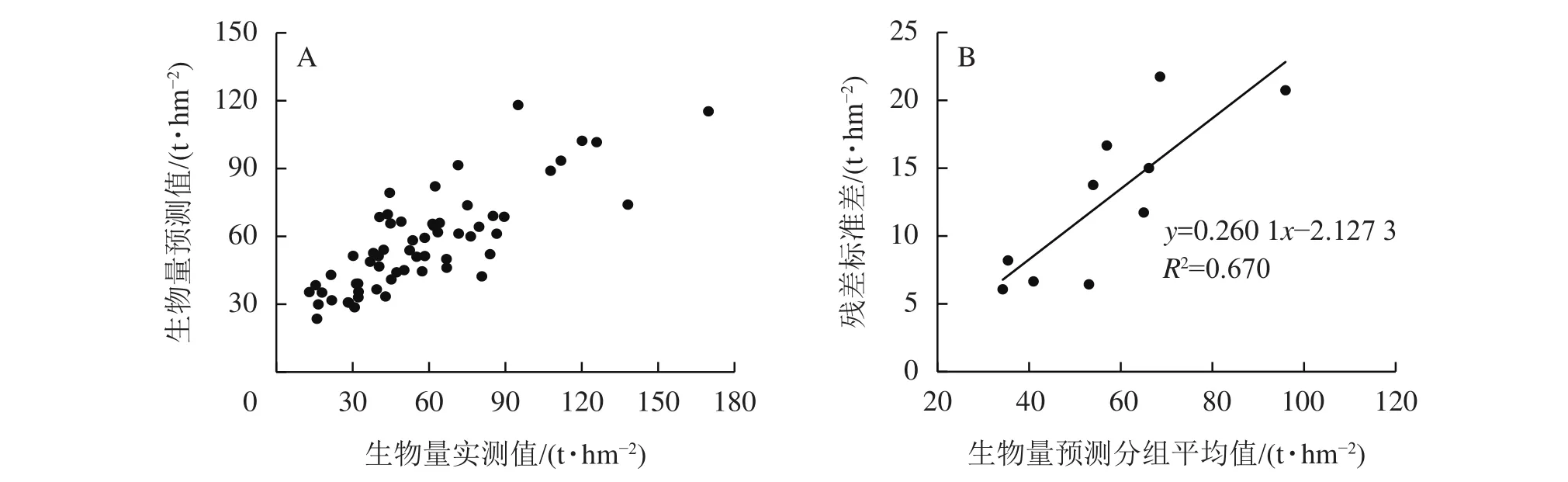
图2 基于梯度提升回归树的地上生物量预测效果和残差不确定性Figure 2 Prediction effect and the residual uncertainty of aboveground biomass based on gradient boost regression tree
3.4 随机森林 (RF)模型的不确定性
图3 A所示:随机森林模型的R2为0.889,RMSE为11.02,预测精度为76.47%。图3B所示:残差标准差(y)与样地生物量预测分组平均值(x)拟合后的最佳模型为线性关系,模型为 y =0.214 3x-1.475 7,R2=0.863。与多元线性回归和梯度提升回归树模型的残差不确定性计算方法相同,在不考虑参数不确定性的情况下,得出随机森林估算高山松地上生物量的不确定性为18.09%。
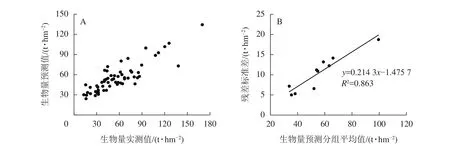
图3 基于随机森林的地上生物量预测效果和残差不确定性Figure 3 Prediction effect and the residual uncertainty of aboveground biomass based on random forest
3.5 遥感估测生物量总不确定性
由表3可见:在样地尺度上,单株生物量模型的不确定性为16.43%,总不确定性为7.07%;在3种遥感估算模型中,多元线性回归模型的不确定性为40.84%,梯度提升回归树模型的不确定性为22.01%,随机森林模型的不确定性为18.09%。基于多元线性回归、梯度提升回归树、随机森林模型,估算出高山松地上生物量的总不确定性分别为41.45%、23.12%和19.42%。

表33 种地上生物量估测模型的不确定性Table 3 Uncertainty results of the three aboveground biomass estimation models
4 结论与讨论
本研究结合误差传递的方法,量化了3种生物量遥感估测模型与样地尺度单株生物量模型产生的误差,并将各个来源的不确定性合成,得出遥感估算高山松地上生物量的总不确定性。遥感估算地上生物量的不确定性主要来源于遥感估测模型的不确定性,其中多元线性回归为40.84%,梯度提升回归树为22.01%,随机森林为18.09%。样地尺度不确定性相对较小,为7.07%。从预测效果来看,非参数的随机森林和梯度提升回归树模型的不确定性要优于参数的多元线性回归模型,随机森林模型的不确定性低于梯度提升回归树模型。单株生物量模型参数变异为12.42%,残差不确定性为10.76%,模型的不确定性主要来源于参数变异,残差不确定性相对较低。
3种遥感估算模型的不确定性研究表明:多元线性回归通过回归方程直观反映遥感因子和地上生物量的关系,但不能有效描述它们之间复杂的非线性关系,要提高参数模型的预测精度则需要大量样地观测数据。张加龙等[8]基于外业调查和Landsat 8 OLI影像,建立了多元线性回归、偏最小二乘、随机森林和梯度提升回归树4种模型,估测了香格里拉市高山松地上生物量,结果表明:随机森林和梯度提升回归树2种非参数模型的预估精度优于其他2种参数模型;陈蜀蓉等[25]运用4种模型对浙江缙云县公益林生物量进行建模估算的结果同样表明:随机森林和Erf-BP神经网络2种非参数模型优于参数模型。本研究建立的梯度提升回归树和随机森林2种非参数模型有较好的拟合优度和预测精度,不确定性优于参数的多元线性回归模型。遥感估测模型的不确定性对森林地上生物量的估算精度占主要的影响,因此,提升模型精度仍是今后生物量估算研究的重要方向。
已有不少学者进行了单株生物量模型不确定性的研究。秦立厚等[10]分别用30、42、48株杉木Cunninghamia lanceolata进行单株生物量建模时发现:一元生物量模型的残差变异引起的不确定性分别为15.2%、12.3%、11.7%,二元生物量模型的不确定性为13.3%、9.4%、8.7%,表明二元单株生物量模型的不确定性要低于一元模型,且建模样本的增加可以显著降低模型残差变异不确定性。与其相比,本研究在对样地尺度生物量不确定性进行度量时,引用了基于幂函数形式的二元单株生物量估算模型[15]。由于缺少实际伐倒的样木生物量数据,运用模型预测精度和均方根误差间接计算模型不确定性,结果可能存在偏差。CHAVE等[26]计算的热带雨林地区生物量模型残差变异误差为31.3%,而本研究模型残差变异误差为10.76%。这可能是热带树种之间的特征差异大,导致产生的残差变异较大,而本研究建模对象仅为亚热带高山松单一树种,未对抽样、测量误差以及遥感影像坐标校正误差进行分析。这在后续研究中还需进一步完善。

