猴樟叶片转录组测序及盐碱胁迫响应基因表达模式研究
韩浩章 张丽华 李素华 赵 荣 王晓立 王 芳
(宿迁学院建筑工程学院,江苏 宿迁,223800)
猴樟(Cinnamomun bodinieri)为樟科、樟属常绿乔木,树形美观,材质优良,具有生长迅速、适应性强等优点,多分布于我国湘西、湖北、云南、贵州、四川等省,在城镇荒山绿化中有较大面积栽培,是重要的造林绿化树种和经济树种[1]。目前,国内外关于猴樟的报道主要集中于精油资源[2]、材用特性[3−4]、光合特性[5]和育苗技术[6]等方面。河南农业大学2012 年将猴樟引入北方地区栽培,认为猴樟的抗寒性要优于香樟[7−8],其盆栽幼苗在−7 ℃低温持续过程中可表现出一定的抗寒性[9],使猴樟在我国郑州地区栽培成为可能。樟属植物在盐碱栽培环境条件下常表现为生长缓慢、叶色黄化、光合速率下降、抗性下降等现象[1],课题组从2016 年开始将猴樟优系引种至江苏地区进行栽培,发现猴樟在苏北地区盐碱栽培条件下表现比樟(C.camphora)更好的抗性[1,10]。植物响应碱胁迫的机制十分复杂,已发现甜菜碱醛脱氢酶基因(BADH)等渗透调节相关基因、抗坏血酸酶基因(APX)等抗氧化相关基因、Na+/K+转运蛋白基因(HKT)等离子区隔转运基因、质子泵基因(VHA)等生长调节相关基因、苯丙烷类次生代谢途径因子(MYB)等转录调控因子均与盐碱胁迫密切相关[11]。在榉树(Zelkova schneideriana)[12]、银杏(Ginkgo biloba)[13]、白沙蒿(Artemisia sphaerocephala)[14]上的研究结果表明,盐碱胁迫条件下植物体内的脯氨酸含量会大大提高,抗性强的树种中含有更高的脯氨酸含量。外源施用脯氨酸能够促进植物光合能力和水分利用能力[15−16],还能通过诱导紫花苜蓿(Medicago sativa)抗氧化酶活性提高抗盐碱胁迫能力[17],脯氨酸在猴樟耐碱胁迫过程中起重要作用。基于此,课题组在前期研究基础上[10],以2 个耐盐碱猴樟品系和1 个盐碱敏感品系猴樟3 年生种苗为材料,通过转录组测序技术,分析猴樟耐盐碱胁迫相关代谢通路,筛选出差异表达基因,以期为猴樟耐盐碱胁迫机理研究提供依据。
1 材料与方法
1.1 试验材料
试验在宿迁学院樟属植物栽培基地盐碱土壤环境试验区进行。试验区土壤pH 值8.17,电导率1 153 μS/cm,含有机质13.85 g/kg、有效钾15.94 μg/g、有效铁12.07 μg/g、铵态氮61.75 μg/g、硝态氮266.75 μg/g、有效磷36.55 μg/g,可溶性盐含量15.89 mg/g。
试验材料取自樟属植物栽培基地耐盐碱性表现较好的同一株优树,于2017 年10 月采种,2018年3 月上旬播种,2020 年3 月上旬进行种苗表型鉴定与标记,2 个耐盐碱猴樟品系(标注为YYHZ和HJHZ)和1 个盐碱敏感猴樟品系(标注为HDHZ)3 年生种苗,其中HJHZ 为普通猴樟,YYHZ 和HDHZ 为猴樟变种,YYHZ 和HJHZ 盐碱耐受能力优于HDHZ[10]。同时移入盐碱土壤环境试验区进行栽培,株行距均为1.5 m。2020 年8 月20 日取3 个表型猴樟作为材料,同一高度枝条顶部幼嫩叶片进行液氮速冻后,于−80 ℃冰柜保存,各设3 个生物学重复。
1.2 研究方法
1.2.1 总RNA 提取、文库构建及测序
用RNAperp Pure Plant Kit(天根,北京)试剂盒提取叶片材料总RNA,利用1.2%的琼脂糖凝胶电泳检测总RNA 的完整性,利用艾本德Bio-Photometer D30 核酸蛋白测定仪测定OD260/280的值,检测总RNA 的纯度和浓度,委托南京绿正生物科技有限公司进行cDNA 文库构建,采用qRT−PCR 对文库有效浓度进行准确定量,库检合格后进行Illumina 测序。
1.2.2 基因功能注释
对猴樟采用无参考基因组的转录组分析,经过原始数据过滤、测序错误率检查、GC 含量分布检查,获得后续分析使用的过滤后数据(clean reads),对聚类得到的基因序列(Unigene)进行基因功能注释。基因功能注释所用到的数据库包括 NR(NCBI non-redundant protein sequences)、NT(NCBI nucleotide sequences)、PFAM(Protein family)、KOG(euKaryotic Ortholog Groups)、SwissProt(A manually annotated and reviewed protein sequence database)、KEGG(Kyoto Encyclopedia of Genesand Genome)、GO(Gene−Ontology)。
1.2.3 差异表达基因筛选
采用 DESeq 对基因表达进行差异分析,首先对原始读数(read count)进行标准化,然后通过统计学模型进行假设检验概率(Pvalue)的计算,最后进行多重假设检验校正(BH),得到错误发现率(FDR 值)。差异基因(DEGs)的筛选条件 为Pvalue≤ 0.01,| log2Fold Changes | ≥ 1。其中,Fold Changes 代表不同处理之间基因表达量的比值。该值大于1 时,则该基因认定为上调表达;反之,则为下调表达。
1.2.4 差异表达基因功能注释和富集分析。
采用GOseq 和KOBAS 软件对每个差异比较组合的上调差异基因集、下调差异基因集进行GO 功能富集分析和KEGG 通路富集分析,以判定差异 Unigene 主要影响的生物学功能或通路。
1.3 差异表达基因验证
为了验证猴樟耐盐碱胁迫转录组结果的准确性,从差异基因数据库中挑选6 个参与精氨酸与脯氨酸代谢的差异表达基因进行分析。用RNAperp Pure Plant Kit(天根,北京)试剂盒提取材料的总RNA,按照PrimeScriptTM RT reagent Kit with gDNA Eraser(TaKaRa,大连)方法去除基因组DNA 后,用TR Prime Mix(random primer)进行反转录反应,获得cDNA 产物。以课题组前期筛选出的Actin1 为内参基因,利用 Primer 5.0 设计引物,由南京天擎科技有限公司合成引物,引物序列已列出(表1)。按照AceQ qPCR SYBR Green Master Mix (without ROX)试剂盒方法配制10 μL反应体系。通过Bio−Rad CF96X(伯乐,美国)进行实时荧光定量 PCR 分析。具体反应体系如下:5 μL 2×SYBR Green Mix,各 0.5 μL 上/下游引物(10 μmol/L),2 μL cDNA 模 板,2 μL dd H2O,反应条件如下:预变性 95 ℃ 5 min;扩增95 ℃ 10 s,60 ℃退火延伸 30 s,共 40 个循环;熔解曲线95 ℃ 15 s,65 ℃ 1 min,95 ℃ 30 s,95 ℃15 s。采用 2−ΔΔCT法计算相对表达量,每个样品 3次重复。
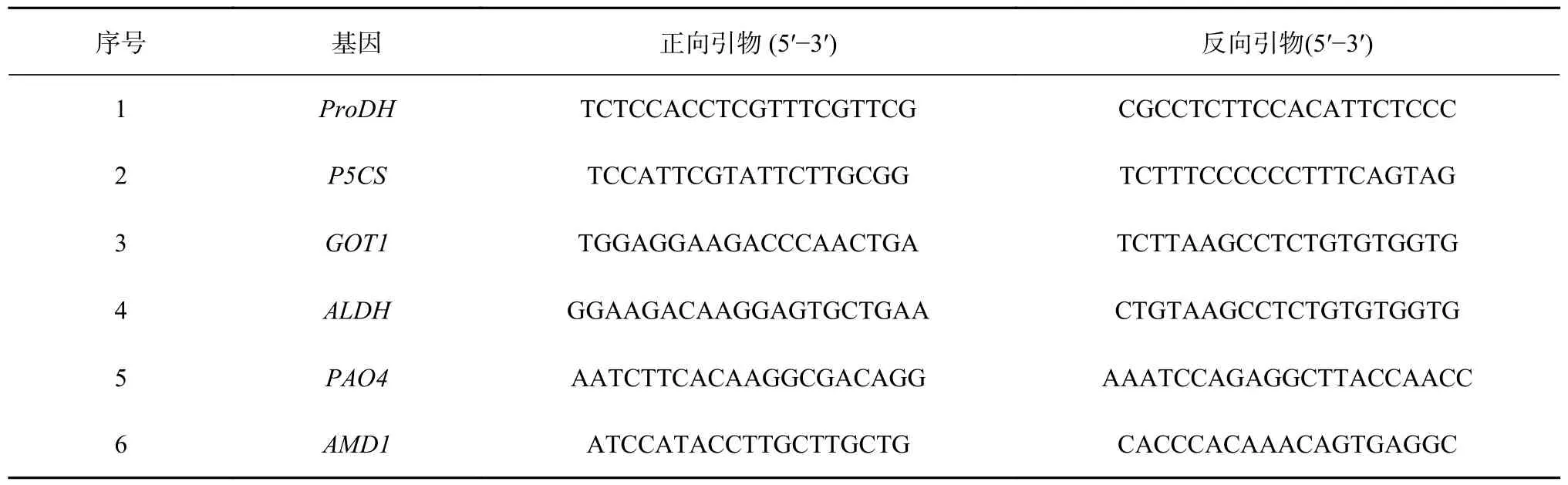
表1 验证基因的引物序列Table 1 The primer sequence of the validated gene
2 结果与分析
2.1 猴樟叶片总RNA 质量检测结果分析
由表2 可以看出,3 个不同表型的猴樟共构建了9 个cDNA 文库用于转录组测序。9 个样品最后得到21 634 177~23 235 930 条clean reads,碱基数6.49~6.97 G,数据整体测序错误率均为0.03%,Q20 为97.62%~97.99%,Q30 为93.1%~93.9%,GC含量为46.87%~47.49%。表明猴樟转录组测序的质量控制良好,得到的过滤后数据库(clean data)质量合格,数据可用于后续基因注释和功能分类等要求。
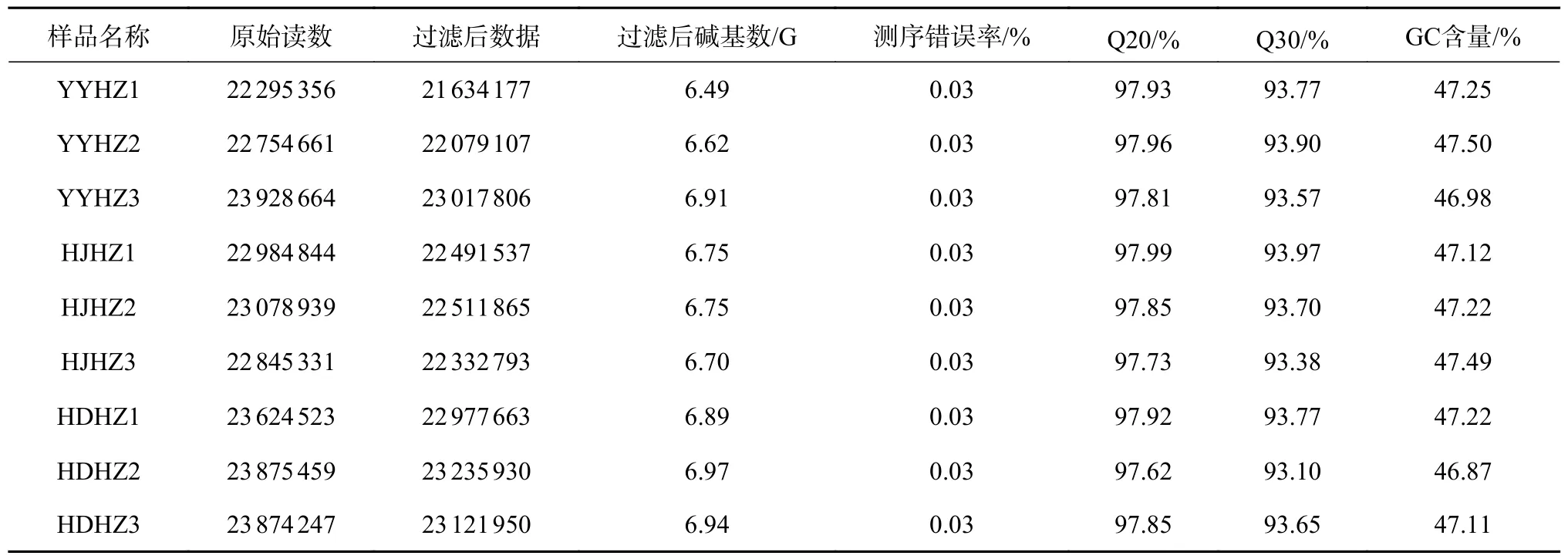
表2 转录组测序数据质量Table 2 Transcriptome sequencing quality
2.2 Unigenes 的基因功能注释
以NR、NT、KEGG、SwissProt、PFAM、GO、KOG 这7 个数据库为参考,利用Blast 将de novo 组装得到的72 369 条Unigenes 与之进行比对和注释(见表3)。注释到NR、NT、KEGG、Swiss-Prot、PFAM、GO、KOG 数据库中的Unigenes 分别有33 769、20 048、11 186、22 952、24 988、24 986和6 513 条,其中注释到NR 的Unigenes 最多,占比46.66%,而注释到KOG 数据库中的Unigenes最少,占比8.99%。在7 个数据库中至少1 个数据库注释成功的Unigenes 有39 156 条,占比54.1%,而与7 个数据库都能匹配成功的Unigenes 有3 884条,占比5.36%,另有33 213 条Unigenes 尚未成功注释,占比45.9%。
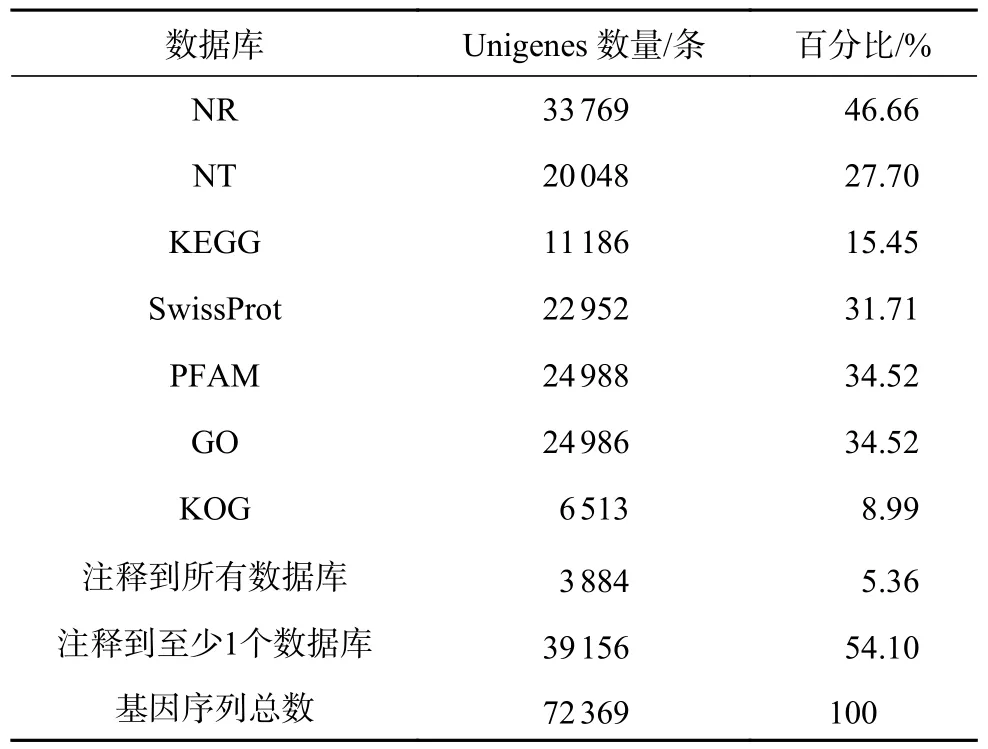
表3 基因注释结果统计Table 3 Statistics of annotation results
2.3 Unigenes 的NR 功能注释
利用Blast 将已组装所获得的Unigenes 序列与NR 数据库进行信息比对,得到属于同源序列的物种和各物种的同源序列数目。由图1 可知,共有5 个同源性较高的物种,成功注释33 769条Unigenes,其中猴樟与沉水樟(C.micranthum)有26 036 条同源Unigenes,占 比77.1%;与莲(Nelumbo nucifera)有1 114 条同源Unigenes,占比3.3%;与葡萄(Vitis vinifera)有743 条同源Unigenes,占比2.2%;与博落回(Macleaya cordata)有642 条同源Unigenes,占比1.9%;与茶(Camellia sinensis)有203 条同源Unigenes,占比0.6%;另外,仍有14.9% 的Unigenes 未能得到NR 数据库比对结果。

图1 猴樟Unigenes 在NR 数据库比对统计Fig.1 Statistical chart of C.bodinieri Unigenes in NR database
2.4 差异基因表达分析
如图2、图3 所示,以HJHZ 为对照,统计分析差异基因,结果发现:在 HDHZ vs.HJHZ 组中共有5 415 个DEGs,包含3 204 个上调基因和2 211 个下调基因,在 YYHZ vs.HJHZ 组中共有4 665 个DEGs,包含2 546 个上调基因和2 119 个下调基因。HDHZ vs.HJHZ 组和 YYHZ vs.HJHZ 组共有的上调基因数量为701 个,共有的下调基因为773 个。从 DEGs 数量可以看出,盐碱土壤条件下,HDHZ vs.HJHZ 上调基因相对较多,说明HDHZ 对盐碱土壤环境更敏感。
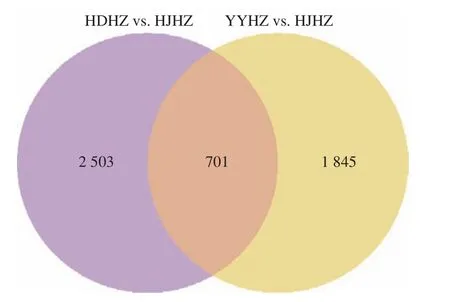
图2 HDHZ vs.HJHZ 和YYHZ vs.HJHZ 上调差异基因表达韦恩图Fig.2 Wayne map of HDHZ vs.HJHZ and YYHZ vs.HJHZ up regulating differential gene expression
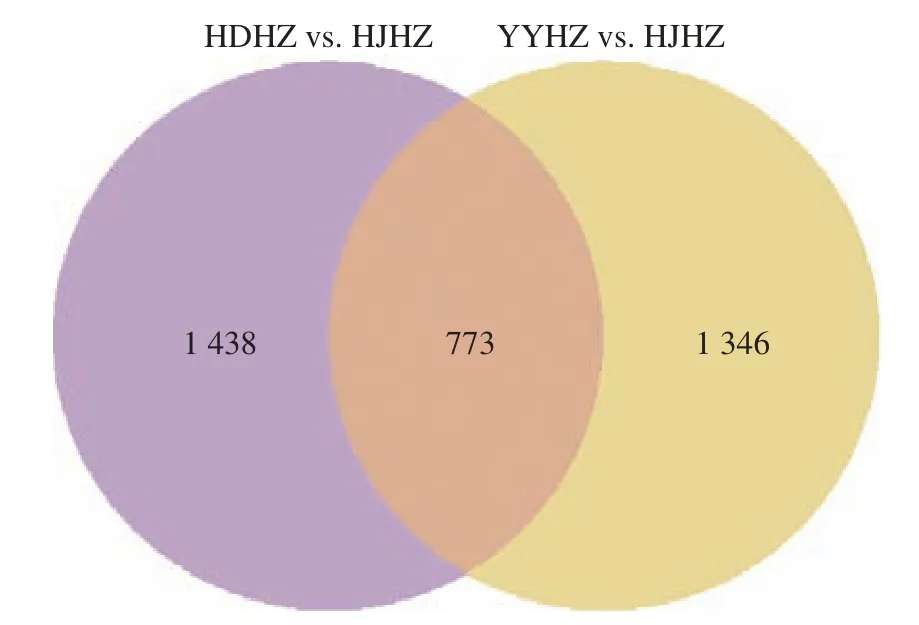
图3 HDHZ vs.HJHZ 和YYHZ vs.HJHZ 下调差异基因表达韦恩图Fig.3 Wayne map of HDHZ vs.HJHZ and YYHZ vs.HJHZ down-regulating differential gene expression
2.5 差异表达基因的GO 功能分析
GO 是描述基因功能的综合性数据库(http://www.geneontology.org/),可分为生物过程(Biological Process,BP)和细胞组成(Cellular Component,CC )分子功能(Molecular Function,MF)3 个本体(ontology)。GO 功能显著性富集分析基于基因组背景,以Padj(通过Benjamini-Hochberg 校正的Pvalue值)小于0.05 为显著富集,给出差异表达基因与哪些生物学功能显著相关,并统计被显著富集的各个GO term 中的基因数。由表4 可知,HDHZ vs.HJHZ 组的上调基因中共有3 834 个DEGs 显著富集到GO 注释的42个小分类中,包括20 个BP 子分类、2 个CC 子分类和20 个MF 子分类。在BP 分类中,含有DEGs 数目最多的前3 个小分类为“蛋白质磷酸化”、“磷酸化”和“磷代谢过程”。在CC 分类中主要是“转录调控复合物”。在MF 分类中富集的 DEGs 总数量最多,其中最多的为“转移酶活性”、其次是“作用于蛋白质的催化活性”和“核苷酸结合”,再次是“腺嘌呤核苷酸结合”、“腺苷酸结合”、“含磷基团转移酶活性”,说明磷素代谢、核苷酸代谢、能量代谢和转录调控参与HDHZ 响应盐碱胁迫过程。HDHZ vs.HJHZ 组的下调基因中共有169 个DEGs 显著富集到GO 注释的8 个小分类中,包括2 个 BP 子分类、6 个MF 子分类。在BP 分类中主要是“DNA 整合”,在MF分类中富集的 DEGs 总数量最多的为“ADP 结合”、其次是“作用于成对供体上、与分子氧结合或还原的氧化还原酶活性”,再者是“碳氧裂解酶活性”及“镁离子结合”,说明盐碱胁迫可能影响了HDHZ 的能量代谢和镁离子结合能力,进一步影响叶绿素合成。
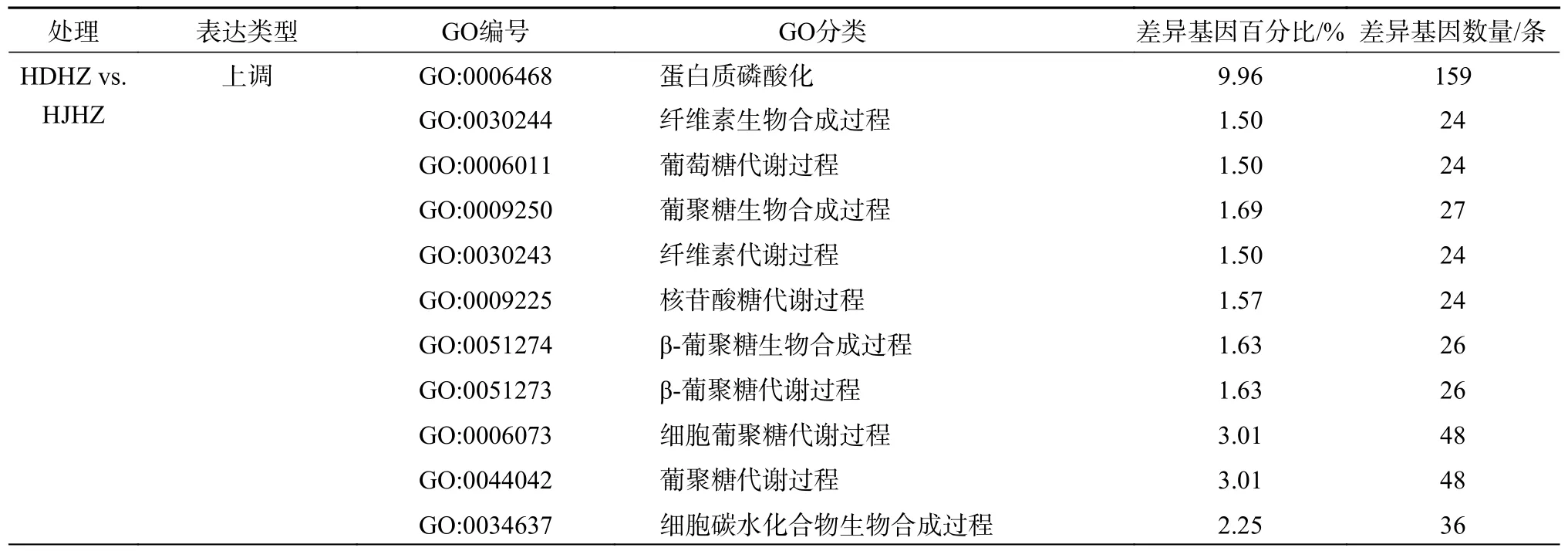
表4 猴樟转录组DEGs 的GO 功能分类统计Table 4 GO functional classification and statistics of C.bodinieri transcriptome Unigenes

续表4

续表4
YYHZ vs.HJHZ 组的上调基因中共有188 个DEGs 显著富集到 GO 注释的7 个小分类中,包括1 个 BP 子分类、6 个 MF 子分类。在 BP 分类中,主要是“DNA 整合”,在 MF 分类中富集的DEGs 总数量最多的为“ADP 结合”、其次是“转移己糖基的转移酶活性”,说明糖基化在YYHZ 响应盐碱胁迫中起重要作用。YYHZ vs.HJHZ 组的下调基因中共有143 个DEGs 显著富集到 GO 注释的4 个MF 小分类中。主要为“ADP结合”、“四吡咯结合”、“血红素结合”、“作用于成对供体上、与分子氧结合或还原的氧化还原酶活性”,说明色素合成、呼吸代谢与HJHZ响应盐碱胁迫有关。
2.6 差异表达基因的KEGG 代谢通路分析
DEGs 富集的 KEGG 通路分析是指 DEGs 在某一通路上是否发生显著差异(over-presentation)通常用富集因子(Enrichment Factor,DEGs 注释到某通路的基因比例与所有基因中注释到该通路的基因比例的比值)和qvalue(多重假设检验校正之后的Pvalue)来表示,一般富集因子越大,表示DEGs 在该通路中的富集水平越显著,qvalue越小,表示差异表达基因在该通路中的富集显著性越可靠,因此,在 DEGs 注释的 KEGG 通路散点图中越靠近右下角的图形代表的通路,参考价值越大。由图4~5 可知,在HDHZ vs.HJHZ 组中,上调差异表达基因主要富集于酪氨酸代谢、植物−病原菌互作、苯丙氨酸、酪氨酸和色氨酸的生物合成、异喹啉生物碱生物合成、类胡萝卜素生物合成、精氨酸和脯氨酸代谢。由此可以看出,生物碱类代谢、氨基酸代谢在HDHZ 响应盐碱胁迫中起重要作用。下调差异表达基因主要富集于倍半萜和三萜的生物合成、内质网蛋白质加工、卟啉和叶绿素代谢、植物−病原菌互作、光合作用−天线蛋白、角蛋白、枯草碱和蜡质生物合成、光合生物的碳固定、柠檬酸循环(TCA 循环)。说明HDHZ 的光合作用、呼吸作用及生长发育受到盐碱胁迫的抑制。如图6、图7 所示,在YYHZ vs.HJHZ 组中,上调差异表达基因主要富集于倍半萜和三萜的生物合成、内质网蛋白质加工、植物−病原菌互作、类黄酮生物合成;下调差异表达基因主要富集于酮体的合成与降解、二苯乙烯、二芳基庚烷和姜辣素的生物合成、非同源末端连接、鞘糖脂生物合成−globo 系列、丁酸代谢。说明YYHZ 和 HJHZ 响应盐碱胁迫的机制不尽相同。
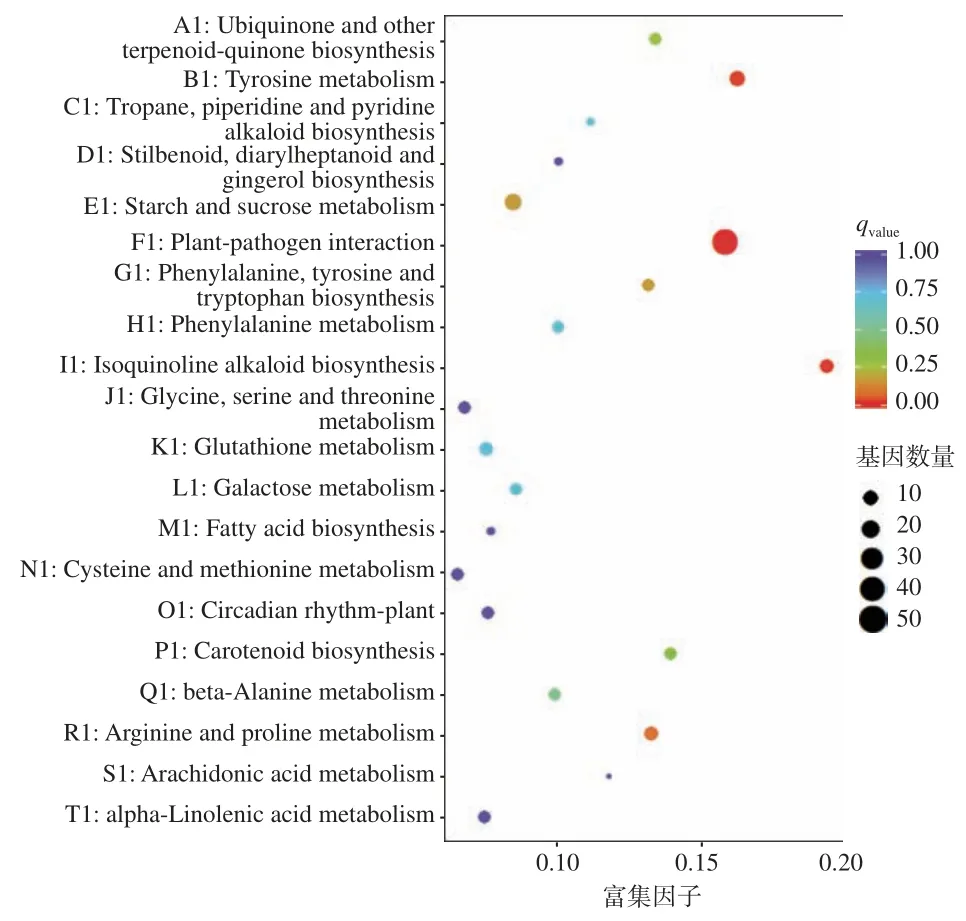
图4 HDHZ vs.HJHZ 组上调差异表达基因KEGG 富集散点图Fig.4 HDHZ vs.HJHZ group upregulated differential expression gene KEGG enrichment distribution map
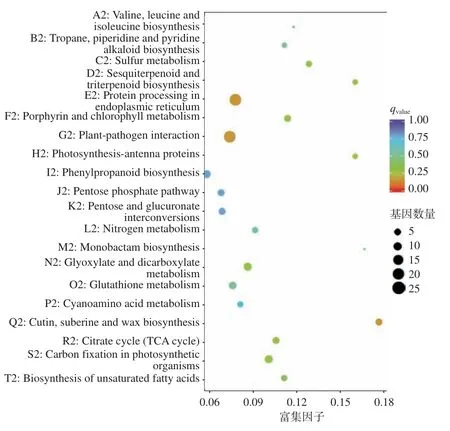
图5 HDHZ vs.HJHZ 组下调差异表达基因KEGG 富集散点图Fig.5 HDHZ vs.HJHZ group down-regulated differential expression gene KEGG enrichment distribution map
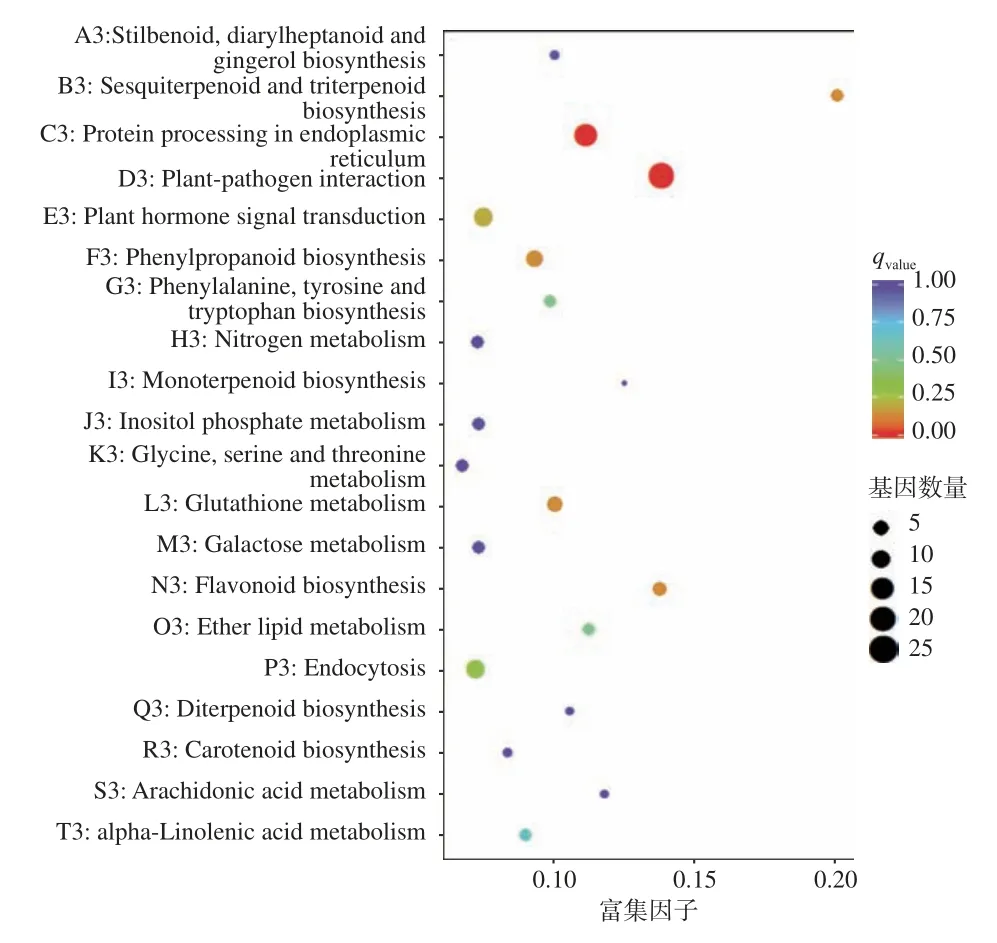
图6 YYHZ vs.HJHZ 组上调差异表达基因KEGG 富集散点图Fig.6 YYHZ vs.HJHZ group upregulated differential expression gene KEGG enrichment distribution map
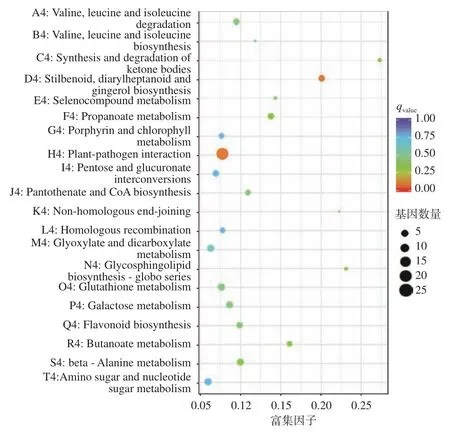
图7 YYHZ vs.HJHZ 组下调差异表达基因KEGG 富集散点图Fig.7 YYHZ vs.HJHZ group down-regulated differential expression gene KEGG enrichment distribution map
2.7 实时荧光定量验证
为了验证转录组测序结果的可靠性,随机选取与猴樟耐盐碱胁迫相关的精氨酸和脯氨酸代谢通路中6 个相关基因Cluster−10916.3837(脯氨酸脱氢酶,ProDH)、Cluster−10916.26309(δ−1−吡咯啉−5−羧酸合成酶,P5CS)、Cluster−10916.42256(天冬氨酸转氨酶,GOT1)、Cluster−10916.29904(NAD+)醛脱氢酶,ALDH)、Cluster−10916.33165(多胺氧化酶,PAO4)、Cluster−10916.8872(S−腺苷甲硫氨酸脱氢酶,AMD1),以Actin1 为内参基因,进行实时荧光定量PCR 验证。将qRT−PCR数据和转录组数据进行比较,结果如图8 所示,通过pearson 相关性分析表明,qRT−PCR 结果与转录组测序结果在 0.05 水平下相关性显著,所选择的基因的表达趋势基本一致,从而说明本研究RNA 测序结果较为可靠,同时也表明精氨酸和脯氨酸代谢与猴樟耐盐碱胁迫有关。
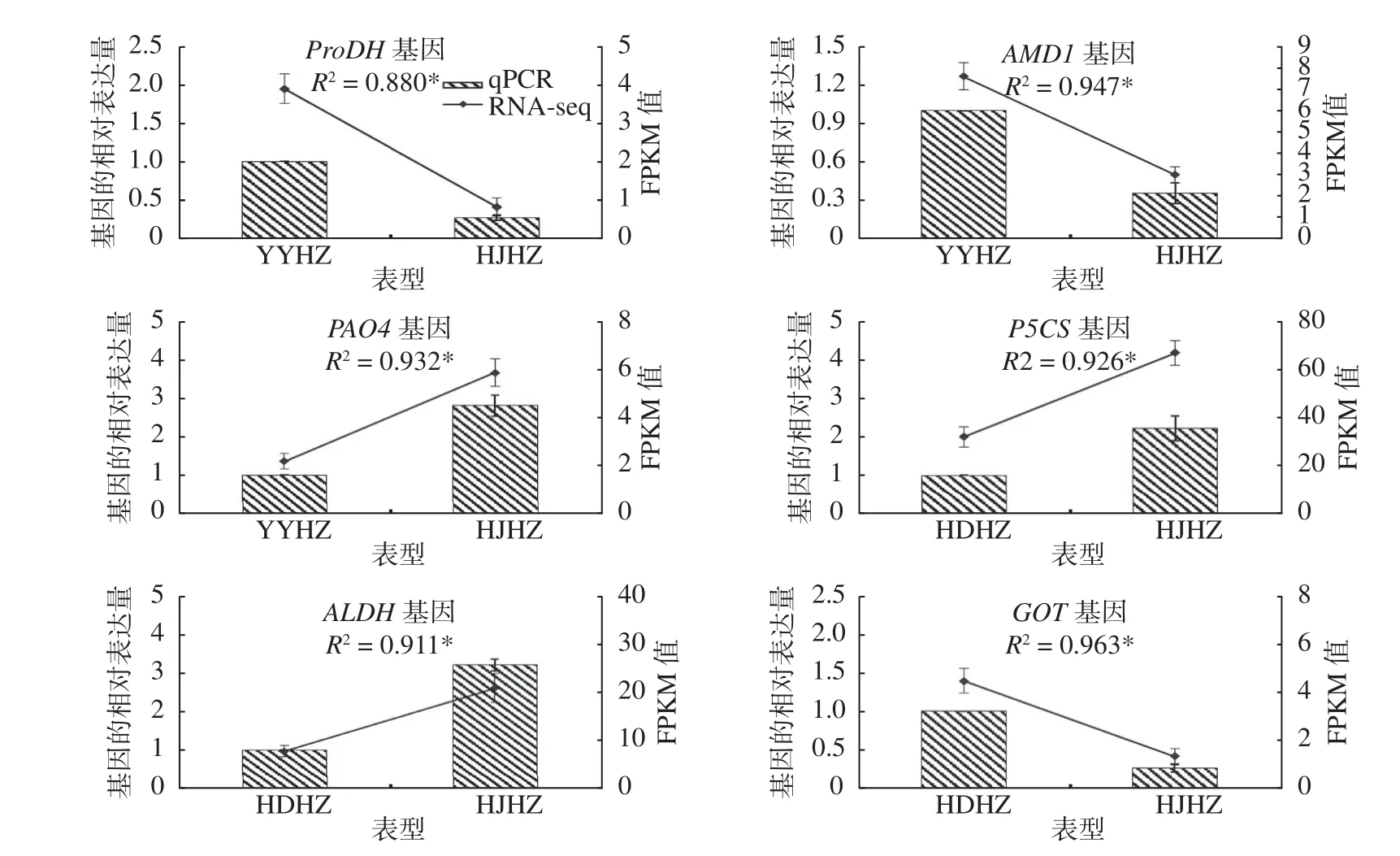
图8 猴樟叶片转录组qPCR 验证结果Fig.8 The qPCR verification results of the transcriptome of C.bodinieri leaves
3 结论与讨论
叶片是植物与环境因子信息交换的媒介,直观反映植物与环境因子的关系,能从信号传导、渗透调节、离子区化、活性氧清除、次生代谢、光合作用、呼吸作用等多个方面应对盐碱胁迫[18−19]。本研究利用Illumina HiseqTM测序平台对3 个不同表型猴樟叶片在盐碱栽培环境条件下的转录本信息进行分析,根据差异基因(DEGs)Pvalue≤ 0.01,| log2Fold Changes | ≥ 1 的条件,在HDHZ vs.HJHZ 组中共筛选出5 415 个DEGs,包含3 204 个上调基因和2 211 个下调基因,在 YYHZ vs.HJHZ 组中共筛选出4 665 个DEGs,包含2 546 个上调基因和2 119 个下调基因。HDHZ vs.HJHZ 组中上调基因最多,HDHZ 对盐碱环境条件更为敏感,与课题组前期研究结论一致[10]。通过GO 功能分析来看,HDHZ vs.HJHZ 组的上调基因中共有3 834 个DEGs 得到注释,下调基因中仅有169 个DEGs 得到GO 注释,主要富集于能量代谢、氧化还原能力、镁离子结合能力和磷素代谢等方面。盐碱胁迫会引起植物叶绿素含量下降[20],抗氧化酶系统先升后降[21−22],呼吸作用改变[23],氧化还原能力受到影响[24];盐碱胁迫还会引起根际土壤缺磷,从而诱导有机酸的产生[23],土壤供磷能有效提高植物的盐碱适应能力[25],磷素代谢与猴樟响应盐碱胁迫密切相关,与多田琦[26]在柳枝稷(Panicum virgatum)上、李慧姬[18]在辣椒(Capsicum annuum)上的研究结论基本一致。YYHZ 和HJHZ 对盐碱胁迫耐受能力均高于HDHZ,YYHZ vs.HJHZ 组的上调基因中仅有188 个DEGs 得到GO 注释,主要富集于糖基化作用,而植物体内的糖基化作用与激素合成、黄酮类物质合成、非生物防御密切相关[27],与多田琦[26]在柳枝稷上的研究结果类似。YYHZ vs.HJHZ 组的下调基因中仅有143 个DEGs 得到GO 注释,主要富集于色素代谢和氧化还原能力方面,与He 等[24]的结论类似。由此可见,由于遗传背景和胁迫程度的差异,植物盐碱胁迫响应受多基因表达的调控。
植物体内存在多条通路响应盐碱胁迫,如碱胁迫下番茄(Lycopersicon esculentum)中SAM 合成酶基因SISAM1 可以通过多胺代谢调控提高耐碱性[27],星星草(Puccinellia tenuiflora)中很多与H+转运及柠檬酸合成相关的基因上调表达[28],生长素及茉莉酸途径在柑橘适应碱胁迫中发挥重要作用[29],野生大豆(Glycine max)通过改变光合途径使其保持盐碱胁迫下较高的光合作用和根系活力[30]。本实验中对DEGs 注释的KEGG 通路分析结果表明,HDHZ vs.HJHZ 组中的上调基因多富集于生物碱类代谢和氨基酸代谢通路,在YYHZ vs.HJHZ 组中上调差异表达基因主要富集于倍半萜和三萜的生物合成和类黄酮生物合成通路,而下调控基因主要富集于色素代谢、酚类物质代谢相关通路。因而,不同植物响应盐碱胁迫的代谢通路不尽相同,HDHZ 的耐盐碱能力较弱,结合研究中选取的6 个差异表达基因的qRT−PCR 结果来看,生物碱类、精氨酸和脯氨酸代谢可能在猴樟响应碱胁迫过程中起主要作用,与贾旭梅等[31]在海棠(Malus spectabilis)上、曹盈[32]在辽宁碱蓬(Suaeda liaotungensis)上的研究结论一致。而HJHZ 和YYHZ 的耐盐碱能力均优于HDHZ,但两者参与盐碱胁迫响应的代谢通路不尽相同,其原因还有待于进一步研究。

