淀粉酶链霉菌几丁质酶克隆表达及催化功能分析
李 芹 王立梅 齐 斌,
(1. 苏州大学医学部药学院,江苏 苏州 215123;2. 常熟理工学院苏州市食品生物技术重点实验室,江苏 常熟 215500)
几丁质是一种天然碱性线性多糖,其结构由多个N-乙酰-D葡萄糖胺通过β-1,4糖苷键连接组成,是来自甲壳动物的外骨骼以及真菌和昆虫细胞壁的第二个最丰富的天然生物聚合物[1]。其年生物合成量约为1 000亿t,降解产物中的几丁寡糖、壳低聚糖和N-乙酰氨基葡萄糖被广泛应用于食品、纺织、农业等领域,具有普遍的生物活性和广阔的市场开发前景[2]。几丁寡糖是由几丁质或壳聚糖降解获得的水溶性物质,聚合度为2~20,是目前自然界中唯一带正电的碱性氨基酸寡糖[3],具有良好的水溶性、抗氧化和抗肿瘤活性[4]。目前,制备几丁寡糖的方法包括化学和物理降解、酶催化水解[5]和转糖基化介导的合成[6]。大多数几丁寡糖是通过化学方法生产的,主要使用酸或氧化剂来降解几丁质[7],该方法会造成严重的环境污染和资源浪费[8]。转糖基化介导的几丁寡糖合成是几丁寡糖酶促生产的新方法,该反应取决于在供体和受体糖之间引入新的糖苷键[9]。据报道[10-12],只有少数GH18几丁质酶具有转糖基化(TG)活性。酶催化因具有反应条件温和、酶促副反应少、产品安全性高、无环境污染等优点,将逐步取代传统的酸性或氧化降解方法[13]。因此,利用几丁质酶降解法降解几丁质生产几丁寡糖的研究较多[14-15]。
几丁质酶是一类可以催化几丁质并将其专一性降解为几丁寡糖、壳聚糖和N-乙酰葡萄糖胺的糖苷键水解酶[16]。然而,目前产几丁质酶活性高、用于工业化生产几丁寡糖的菌株极少,只有少数黏质沙雷氏菌和木霉菌等。张博阳等[17]对桑氏链霉菌几丁质酶基因ChiKJ40进行克隆表达,得到几丁质酶活性为0.080 U/mL。Li等[18]采用PCR方法对Streptomycessampsonii(Millard&Burr)Waksman KJ40的几丁质酶基因ChiKJ406136进行克隆并在大肠杆菌BL21(DE3)中表达,其粗蛋白质和纯化蛋白质溶液中的几丁质酶活性仅为0.045,0.033 U/mL。研究拟对淀粉酶链霉菌CS1801几丁质酶PROKKA01070基因在大肠杆菌中进行克隆,通过原核表达系统获得可溶性几丁质酶,对其活性进行探究,结合同源建模、催化域氨基酸比对及其催化域关键氨基酸位点突变,验证其活性口袋内部关键氨基酸的功能,揭示这些氨基酸残基在酶催化中的作用,为该菌的后续深入研究及工业生产几丁寡糖提供依据。
1 材料与方法
1.1 材料与仪器
1.1.1 材料与试剂
淀粉酶链霉菌(Streptomycesdiastaticus)CS1801:由实验室从虾酱中筛选得到;
大肠杆菌Top10感受态、大肠杆菌BL21(DE3)、细菌DNA抽提试剂盒、高GC含量PCR扩增试剂盒、DNA凝胶回收试剂盒、质粒小提取试剂盒、定点突变试剂盒、氨苄青霉素钠:生工生物工程(上海)股份有限公司;
PMD-19T载体基因克隆试剂盒以及限制性内切酶(EcoRⅠ和XhoⅠ):宝日医生物技术(北京)有限公司;
pET-32a(+)载体:南通柯侎克生物科技有限公司。
1.1.2 仪器与设备
全温振荡培养箱:ZQZY-CF型,上海知楚仪器有限公司;
微孔板分光光度计:XMARK型,美国Bio-Rad公司;
恒温培养箱:GHP-9270型,上海索普仪器有限公司;
电子天平:XS105DU型,瑞士Mettler Toledo公司;
高速台式离心机:Legend Micro 17R型,德国Thermo公司;
高速冷冻离心机:CR22GⅡ型,日本Hitachi公司;
垂直电泳系统:proteanⅡ型,美国Bio-Rad公司;
PCR基因扩增仪:My Cycler型,美国Bio-Rad公司;
琼脂糖水平电泳槽:DYCP-31BN型,美国Bio-Rad公司;
凝胶成像系统:Gel Doc X型,美国Bio-Rad公司;
核酸浓度测定仪:NanoDrop 2000型,德国Thermo公司。
1.2 方法
1.2.1 胶体几丁质制备 称取10 g几丁质粉末,加至200 mL浓盐酸中,均匀搅拌至全部溶解,过滤,加入1 000 mL 蒸馏水,离心,用蒸馏水洗沉淀至中性,4 ℃保存备用。
1.2.2 几丁质酶ChiA基因扩增和克隆 根据细菌DNA提取试剂盒提取淀粉酶链霉菌CS1801(StreptomycesdiastaticusCS1801)基因组DNA。根据已获得的淀粉酶链霉菌全基因组测序数据库信息中,几丁质酶PROKKA01070基因序列用Primer Premier 5.0生物软件设计两条引物,在两条引物的5’端分别引入酶切位点,如表1所示。

表1 引物序列Table 1 Primer sequences
去除几丁质酶PROKKA01070基因序列的信号肽,PCR扩增目的基因片段,PCR反应条件:95 ℃ 3 min,95 ℃ 30 s,55 ℃ 30 s,72 ℃ 1 min,共30个循环,72 ℃ 30 min,4 ℃保存。琼脂糖凝胶电泳检测PCR结果,将PCR产物与pMD19-T载体通过T4连接酶16 ℃连接过夜后转化至Top10感受态细胞中并进行蓝白斑筛选,挑选白色单克隆菌落进行菌液PCR验证,将鉴定为阳性的重组菌株送生工生物工程(上海)股份有限公司进行测序。
1.2.3 表达载体的构建 将测序正确的ChiA/pMD19-T质粒与表达载体pET-32a(+)分别用EcoRⅠ酶和XhoⅠ酶双酶切。验证酶切产物后,切胶回收质粒和基因片段。酶切的ChiA基因片段用T4连接酶与酶切的pET-32a(+)连接。转化Top10感受态细胞,培养过夜。菌落PCR和质粒酶切鉴定后,获得的阳性重组菌ChiA/pET-32a(+)/Top10送至生工生物工程(上海)股份有限公司进行测序。
1.2.4 重组几丁质酶SDS-PAGE分析 将测序正确的ChiA/pET-32a(+)转化至大肠杆菌BL21(DE3)感受态细胞,涂布于含有氨苄青霉素的LB平板上,挑取阳性克隆于50 mL含氨苄青霉素的LB液体培养基中,37 ℃、200 r/min培养过夜。按2%接种量接种至新鲜的LB液体培养基中,37 ℃、200 r/min震荡培养至OD600 nm为0.6~0.8,用终浓度为0.5 mmol/L的IPTG,16 ℃诱导24 h。收集菌体,按1 g/5 mL加入裂解液,加入溶菌酶1 mg/mL,冰育45 min,分别于-20 ℃、室温下进行冻融循环。破壁后,10 000 r/min离心20 min,取菌体、上清和沉淀进行SDS-PAGE分析[19]。
1.2.5 酶活测定 采用DNS(3,5二硝基水杨酸)法[20-21]。N-乙酰-D-氨基葡萄糖标准曲线为y=3.310 6x-0.077 4,R2=0.995 9。
1.2.6 重组几丁质酶的表征
(1) 反应温度:采用标准几丁质酶活性测定法测定20~70 ℃下几丁质酶活性。
(2) pH值:分别在柠檬酸—柠檬酸钠缓冲液(pH 3.0~5.0)、磷酸氢二钠—磷酸二氢钠缓冲液(pH 6.0~8.0)、Tris-HCl缓冲液(pH 9.0)和碳酸氢钠—氢氧化钠缓冲液(pH 10.0~11.0)中进行标准酶活性测定,确定ChiA的最佳反应pH,所有缓冲液浓度均为50 mmol/L。
(3) 金属离子及化学试剂:分别用1,5,10 mmol/L的MgCl2、FeCl3、ZnCl2、CaCl2、KCl、NaCl、C2H3O2Li、EDTA、巯基乙醇、SDS,在标准几丁质酶测定条件下探究几丁质酶活性。
(4) 底物:用1%的胶体几丁质、粉状几丁质、虾壳粉、蟹壳粉、壳聚糖、羧甲基纤维素钠,于磷酸氢二钠—磷酸二氢钠盐缓冲液(pH 7.0)中,50 ℃反应30 min,测定ChiA的底物特异性。
1.2.7 同源建模 在NCBI的PDB结构数据库中,将几丁质酶ChiA的氨基酸序列进行BLAST,将检索出的同源性高的蛋白作为模板,利用同源模建的方法构建几丁质酶ChiA的三维结构。几丁质酶同源建模选用SWISS-MODEL在线软件[22],建模后用PROCHECK作拉氏图评估预测三维结构的可信度[23]。
1.2.8 氨基酸多序列比对 根据几丁质酶ChiA同源建模模型,利用Clustal Omega在线软件,比对分析几丁质酶ChiA编码的催化域氨基酸序列与其他同源性高并且晶体结构确定的几丁质酶催化域的氨基酸序列,推测几丁质酶ChiA的催化反应机制。
1.2.9 催化域关键氨基酸的突变 以ChiA/pET32a(+)/Top10作为出发菌株,采用定点突变试剂盒[24],根据18家族几丁质酶ChiA催化域氨基酸比对结果,选择催化结构域中的关键氨基酸位点进行定点突变,探究其催化活性。为了确保所需的突变是序列中唯一的突变,在生工生物工程(上海)股份有限公司中对插入的DNA的整个区域进行测序。
1.2.10 突变体在大肠杆菌BL21(DE3)中的表达 将序列突变正确的重组质粒转化至大肠杆菌BL21(DE3)中,挑取单菌落至50 mL含有Amp(100 μg/mL)的LB培养基中,37 ℃、200 r/min培养过夜。按2%接种量接种至新鲜的LB液体培养基中,37 ℃、200 r/min震荡培养至OD600 nm为0.6~0.8,用终浓度为0.5 mmol/L的IPTG,16 ℃ 诱导24 h。收集菌体,按1 g/5 mL加入裂解液,加入溶菌酶1 mg/mL,冰育45 min,于-20 ℃、室温下进行冻融循环。破壁后,10 000 r/min离心20 min,去沉淀留上清酶液。
1.2.11 突变体酶活力和动力学参数测定 用DNS法测定突变体酶活。动力学参数测定:在反应管中加入不同质量浓度(1.25,2.50,5.00,7.50,10.00,12.50 mg/mL)的底物胶体几丁质,加入相同量的适量酶液,50 ℃、200 r/min 摇床反应30 min。加入1.5 mL DNS,煮沸5 min 显色,冰水中冷却至室温,10 000 r/min离心5 min,取上清液于540 nm处测定吸光值。通过制作Michaelis-Menten动力学曲线测定Km、Vmax和kcat值。
2 结果与分析
2.1 基因克隆及鉴定
由图1可知,几丁质酶PROKKA01070基因扩增结果与实验室前期全基因组测序结果一致,大小为1 065 bp;菌液PCR验证结果在约1 065 bp处有目的条带,与测序结果一致,说明阳性克隆ChiA/pMD19-T/Top10构建成功。

表2 用于定点突变的引物序列Table 2 The primer sequences used in site-directed mutation
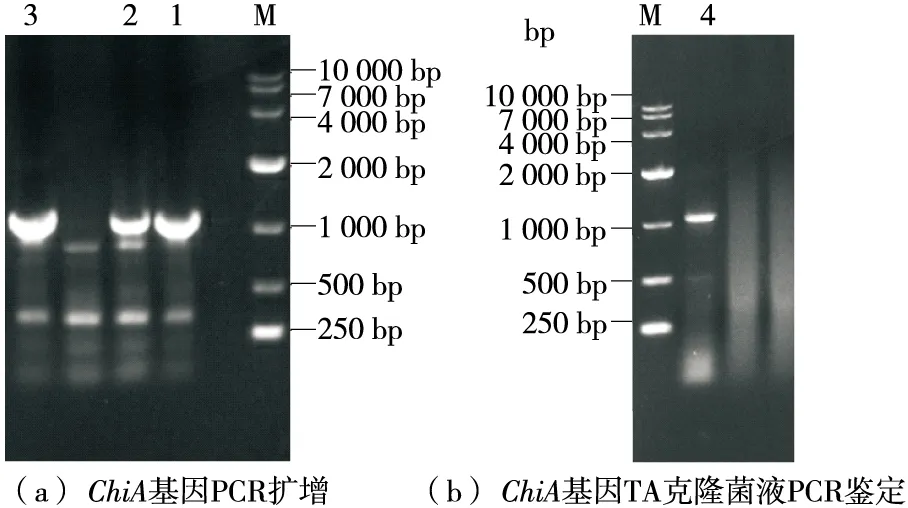
M.10 kb DNA Marker 1~3.ChiA基因PCR扩增产物 4.ChiA/pMD19-T PCR产物图1 ChiA基因PCR扩增及克隆鉴定Figure 1 PCR amplification and clone identification of ChiA gene
2.2 ChiA/pET32a(+)表达载体的构建与鉴定
由图2可知,菌落PCR电泳显示在约1 065 bp处有一目的条带。双酶切验证结果表明,约在1 065,6 000 bp有目的条带,大小与预期结果相符,表明目的基因和表达载体连接方向正确,证明原核表达载体ChiA/pET32a(+)构建成功。
2.3 表达产物的鉴定及酶活检测
由图3可知,45 kDa附近有目的条带,与预测蛋白大小相差无几,说明该重组菌成功表达蛋白。将构建好的重组菌株诱导表达后,在pH 7.0,50 ℃下,诱导后的酶活为132 U/L,而未诱导的检测不到酶活性,说明重组几丁质酶ChiA在大肠杆菌中成功表达且重组几丁质酶活性比原始酶活性(100 U/L)提高了32%。
2.4 重组几丁质酶的表征
2.4.1 pH和反应温度 由图4可知,重组酶在pH 7.0下显示出最高的活性,最佳反应温度为50 ℃。
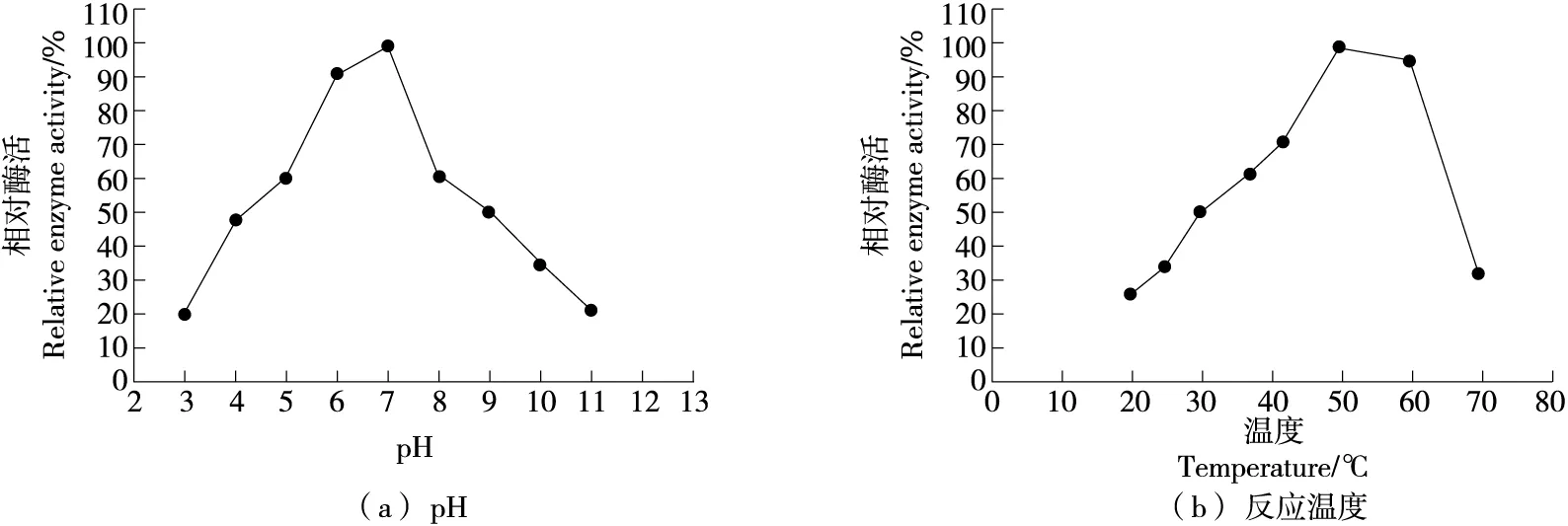
图4 pH和反应温度对ChiA活性的影响Figure 4 Effects of pH and temperature on ChiA activity
2.4.2 金属离子和化学试剂 由表3可知,10 mmol/L浓度下,Zn2+和Fe3+对该重组酶有较强烈的抑制作用,相对酶活力只有37.7%和35.3%;Mg2+、Ca2+、SDS、巯基乙醇、EDTA对该酶也有不同程度的抑制,但K+、Na+、Li+对该酶分别有6.3%,10.6%,8.6%的促进作用。1,5 mmol/L 浓度下,Zn2+和SDS对重组酶有较强的抑制作用,其他离子和试剂对该酶无明显影响。1 mmol/L浓度下,Ca2+、Fe3+和Mg2+对该酶有一定的促进作用。试验浓度下,Na+和Li+对该酶均有促进作用。
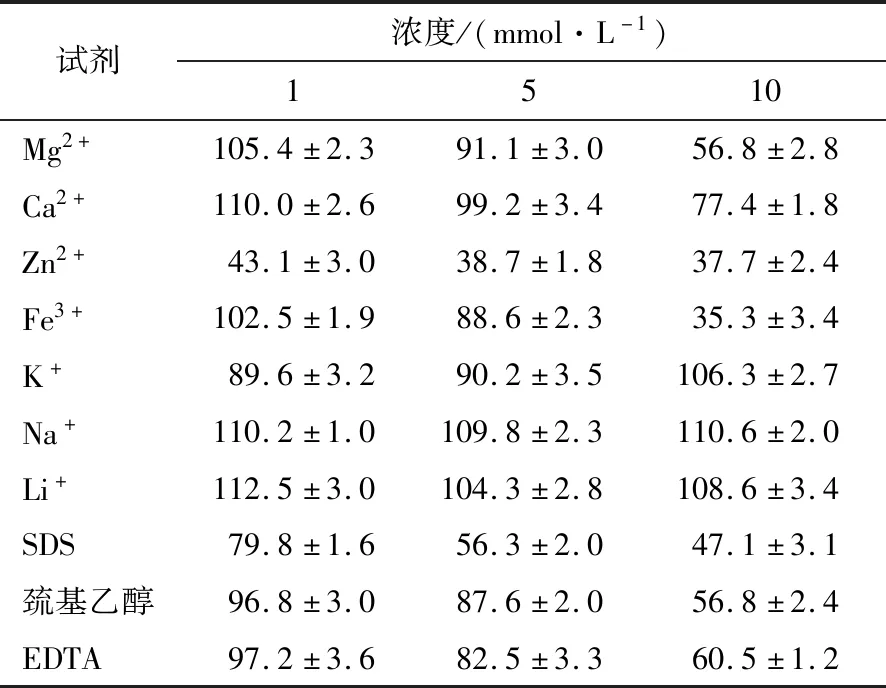
表3 离子和化学试剂对重组酶ChiA酶活力的影响Table 3 Effects of different metal ions and chemicals on ChiA activity %
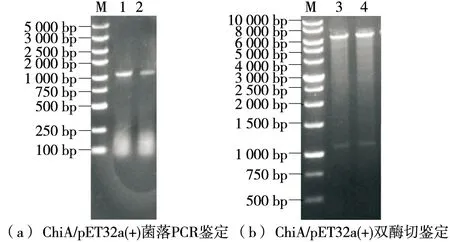
M.5 000 bp DNA marker 1~2.ChiA/pET32a(+) PCR产物 3~4.ChiA/pET32a(+) 双酶切条带图2 重组质粒ChiA/pET32a(+)鉴定Figure 2 Identification of recombinant plasmid ChiA/pET32a(+)
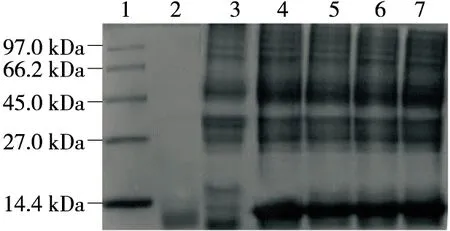
1.蛋白Marker 2.诱导前全菌体 3.诱导后全菌体 4~5.试剂盒提取酶蛋白 6~7.诱导后破壁上清图3 重组蛋白SDS-PAGE分析Figure 3 SDS-PAGE analysis of recombinant protein
2.4.3 底物 由表4可知,当以胶体几丁质为底物时,重组酶的相对酶活性最高;当以粉末为底物时,重组酶的相对酶活性为9.8%。当以1%虾壳粉和蟹壳粉为底物时,只检测到微弱酶活;当以壳聚糖和羧甲基纤维素钠为底物时,检测不到酶活。综上,重组酶可以有效地降解胶体几丁质,对几丁质具有底物特异性,而壳聚糖、羧甲基纤维素钠等几丁质结构类似物不能与重组酶特异性结合。
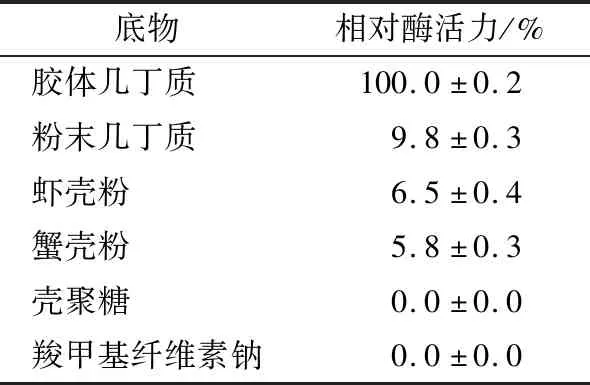
表4 ChiA的底物特异性测定结果Table 4 Determination of substrate specificity of ChiA
2.5 同源建模
经比对分析,选择与几丁质酶ChiA催化结构域中的结构基序(PDBID:4w5u)在相应的密码子区域具有69.03% 的氨基酸序列相似性的嗜热链霉菌(StChi40)的几丁质酶作为ChiA同源建模的模板。由图5可知,ChiA属于18家族糖苷水解酶,其结构由8个α-螺旋内桶和8个β-折叠外桶组成,它们紧密地挤在一起,并由一个不规则的卷曲段连接。18家族中对应β-3和β-4链的两个高度保守的氨基酸片段具有相似的结构,底物结合位点位于β-3和β-4链的保守序列形成的环缝中。
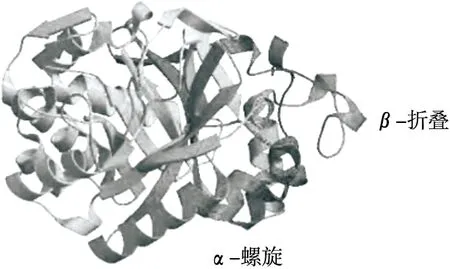
图5 几丁质酶ChiA的三级结构模型Figure 5 Three-dimensional structure model of chitinase ChiA
由图6可知,90.4%,8.6%,1.0%和0%的氨基酸分别落入核心区、偏好允许区、一般允许区和不允许区。总的来说,90%以上的氨基酸在其偏好区,说明预测的几丁质酶三维结构可信。
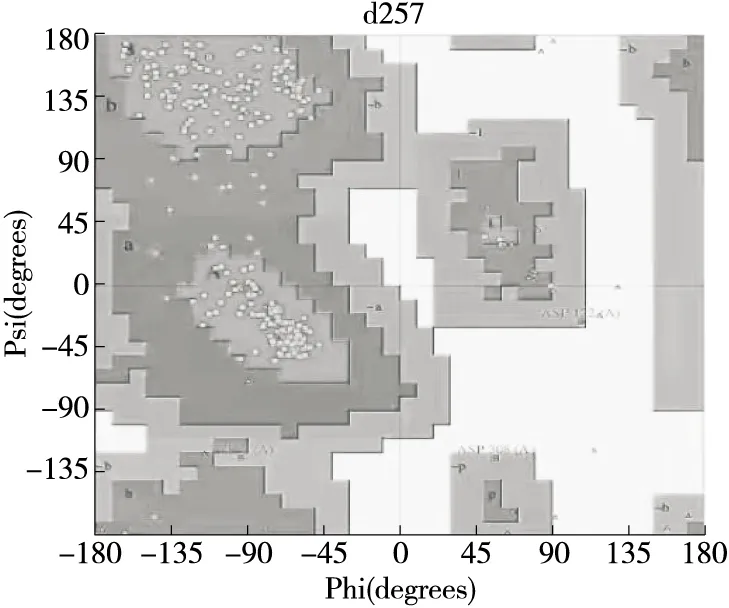
图6 几丁质酶三维结构拉氏图Figure 6 Ramachandran plot of the modeled 3D structure of chitinase
2.6 ChiA催化氨基酸序列分析
序列比对分析所选序列,包括环状芽孢杆菌(BacilluscirculansWl-12)菌株Chitinase A1的A链(PDB:1ITX_A)、嗜热链霉菌(StreptomycesthermoviolaceusStChi40)菌株几丁质酶的A链(PDB:4W5U)、关节杆菌(Arthrobactersp.TAD20)菌株Chitinase B的A链(PDB:1KFW)、黏质沙雷氏菌(Serratiamarcescens)菌株Chitinase A的A链(PDB:1X6L)、黏质沙雷氏菌(Serratiamarcescens)菌株Chitinase A的A链(PDB:2WK2)、黏质沙雷氏菌(Serratiamarcescens)菌株Chitinase A的A链(PDB:1RD6)和哈氏弧菌(VibrioHarveyi)菌株几丁质酶的A链(PDB:3B8S_A)。由图7可知,8种菌的氨基酸序列和功能有差异性,但都具有高度保守的催化域的关键氨基酸。ChiA具有两个典型的18家族糖苷水解酶的保守域,分别为87~90位的“SxGG”结构和125~132位的“DxxDxDxE”结构,在催化域的环缝部位。有研究[25-27]表明,几丁质酶“SxGG”保守模块的活性中心负责酶与底物结合,“DxxDxDxE”保守域的催化活性中心具有催化降解底物功能。125DxxDxDxE132形成即(βα)8桶的β-4链,构成催化域的核心部位。
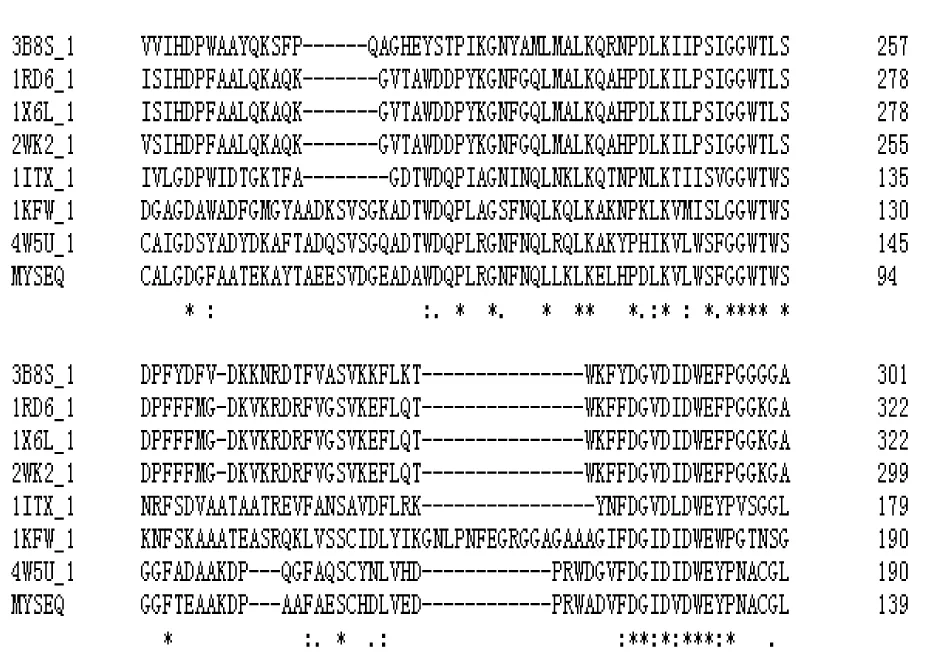
图7 几丁质酶ChiA催化域与其他生物体的催化域多重序列比对Figure 7 Multiple sequence alignment of the catalytic domain of chitinase ChiA with that of other organisms
2.7 重组菌株中活性位点氨基酸的定点突变
为了证实ChiA中保守氨基酸的功能,将D128、D130和E132残基用Asn、Ala或Gln取代,产生6个突变体:D128N、D128A、D130N、D130A、E132Q和E132A。
由图8可知,突变体的比活性大大降低,特别是E132A、E132Q几乎完全失活(12.7,13.2 U/L),证实了Glu-132在酶催化中起重要作用;在突变体中,D128N、D128A、D130N和D130A也表现出较低的比活性,证实了D128、D130、E132氨基酸在18家族的保守区域中起重要作用。有研究[28]报道,Glu-132在催化反应中作为质子供体起着关键作用。突变体D130N的比活性在阴性突变体中最高,因为天冬酰胺提供的氢键可以取代天冬氨酸提供的氢键,而丙氨酸不能产生氢键[25]。天冬氨酸在130位的催化作用主要由氢键支持,因此氢键的存在极大地稳定了ChiA的催化活性[29]。第128位的天冬氨酸被认为是D130的辅助因子,增加了D130的pKa[30]。此外,DXXDXDXE保守结构域中的中间Asp可以增加几丁质酶的转糖基活性[31]。
2.8 重组几丁质酶突变体的动力学常数
由表5可知,6个突变体的催化效率与重组酶相比显著降低,其Km值明显升高,可能是突变体与底物胶体几丁质之间的亲和力减小,证明了D128、D130、E132是催化几丁质酶降解几丁质生产几丁寡糖的关键氨基酸活性位点。
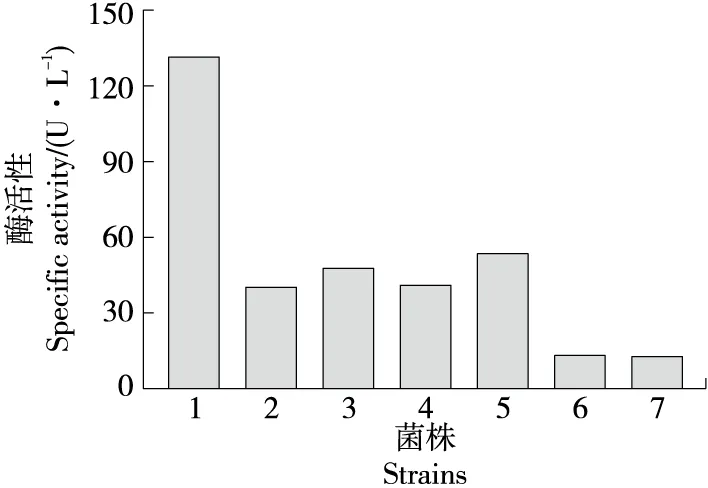
1~7分别为ChiA、D128A、D128N、D130A、D130N、E132Q和E132A菌株图8 各个突变体酶活Figure 8 The specific activities of the mutants
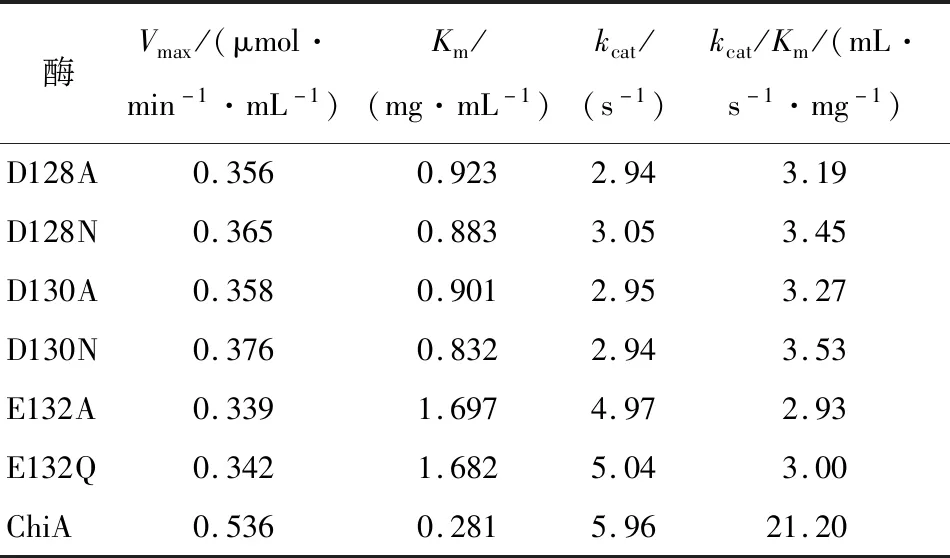
表5 ChiA及其突变体的动力学参数Table 5 Kinetic parameters of ChiA and its mutants
3 结论
以淀粉酶链霉菌CS1801几丁质酶基因序列为研究对象,以大肠杆菌BL21(DE3)为表达宿主,成功表达了几丁质酶ChiA。结果表明,几丁质酶重组工程菌培养周期缩短至24 h,酶活为132 U/L,较原始酶提高了32%。重组几丁质酶的最适反应温度为50 ℃,pH为7.0,且该酶具有胶体几丁质底物特异性。第128,130位的天冬氨酸和132位的谷氨酸是几丁质酶ChiA催化结构域内的催化活性氨基酸。为进一步提高几丁质酶的活性及酶法生产几丁寡糖的效率,可通过分离纯化、酶固定化等技术对几丁质酶进行研究,获得催化生产几丁寡糖效率得到提高的几丁质酶。

