全景式多路径知识图谱构建研究*
——以水稻粒型基因领域为例
曹雨晴 鲜国建,2 黄永文,3 陈博立 李娇,3 罗婷婷,3 孙坦
(1. 中国农业科学院农业信息研究所,北京 100081;2. 农业农村部农业大数据重点实验室,北京 100081;3. 国家新闻出版署农业融合出版知识挖掘与知识服务重点实验室,北京 100081;4. 中国农业科学院,北京 100081)
作为驱动人工智能和推进认知智能的核心技术,知识图谱(Knowledge Graph)采用基于图模型的关联知识表达方式,具有强大的语义处理能力和开放关联能力。知识图谱根据应用场景范围分为通用知识图谱和领域知识图谱,其中领域知识图谱可最大限度地挖掘领域知识之间的潜在关联以及相关科技资源的多维特征,在金融、医学等领域都具有广泛的应用,为促进领域突破性研究奠定基础。利用知识图谱对海量领域资源进行有效融合,不仅能够全面揭示领域内部的知识组织体系,还可以帮助用户发现潜在的知识关联、提供新的研究视角。如何通过细分领域专业知识图谱的构建与应用,有效解决垂直领域多源异构资源的知识组织与融合问题,是当前领域知识组织研究的重点和热点。
以作物领域为例,随着高通量生物技术的不断发展,各种描述作物基因调控、蛋白质编码以及表型等多源异构作物组学大数据呈指数级增长,为科研人员从多维度研究作物基因调控机制带来了丰富的数据来源以及先验知识。然而,这些数据的组织和利用还面临诸多挑战:①多组学研究之间耦合性较低;②海量研究数据语义化组织与深度挖掘亟待加强;③研究成果间关联度不强,难以进行有效分析与关联发现等问题[1]。只有将基因组、转录组、蛋白组、表观组等多组学数据进行系统整合关联,才有利于科研人员全面挖掘和利用多组学大数据中潜在规律和知识,促进向数据密集型计算科研范式转变。
本文以水稻粒型基因研究涉及的多源异构多组学数据为例,从全景式、多路径两个维度,研究探索具备较强普适性和通用性的领域知识图谱构建方案,并对知识图谱驱动的启发式知识关联发现等应用场景进行展望,将水稻粒型基因等专业领域各类数据信息表达为更贴近人类认知的形式[2],形成一种更好地组织、管理和理解领域海量信息资源的能力,促进科技文献等通用信息与领域知识的深度融合关联。
1 国内外研究进展
知识图谱作为一种结构化的语义知识库,因其灵活的组成结构和丰富的语义表示能力,已成为人工智能应用的重要基础。知识图谱根据存储方式一般分为基于RDF数据结构的语义知识图谱和基于属性图数据结构的广义知识图谱[3]。近年来,国内外学者在知识图谱构建与应用领域开展了广泛研究与实践,如侧重通用性知识的百科知识图谱[4-5],以及面向细分领域或服务场景的领域知识图谱建设应用实践。
目前学者研究主要聚焦于对非结构化数据的知识抽取。面向海量的非结构化文本,基于词典和规则匹配的传统抽取方法具有较高的准确性,但是人工成本较高。随着深度学习技术不断发展,学术界逐渐倾向利用小样本标注结合深度学习技术来实现自动化知识抽取,研究成果注重对模型算法的优化与提升,尤其是随着自然语言处理领域预训练模型的提出,促进对领域非结构化文本的知识抽取,在金融、医学、电商等领域都取得了显著的实践应用成果[6-9]。在知识图谱实际建设过程中,仅依赖于非结构化知识抽取的方式,难以保证知识图谱的数据质量,更无法为用户提供较为宏观和全面的知识掌握。
具体到作物领域知识图谱的构建方面,法国农业国际合作研究发展中心(French Agricultural Research Centre for International Development,CIRAD)搭建的AgroLD知识图谱[10],基于关联数据技术对多种植物的领域数据集进行集成并构建知识图谱。英国洛桑研究所(Rothamsted Research)面向作物领域的开放关联数据模型提出一种应用型领域本体BioKNO并基于该本体构建大型领域知识图谱KnetMiner,为追溯作物复杂性状的基因调控网络信息提供借鉴[11]。作为作物领域知识图谱的典型范例,两类图谱都在一定程度上实现了面向作物领域的多组学数据融合与集成,为本研究提供了高质量数据基础与参考。
当前,面向细分垂直领域的知识图谱构建研究与实践越来越多,但也面临诸多挑战。一方面,领域知识表示是一个非常复杂的系统工程,要求参与人员具有较高专业素养;另一方面,领域数据来源分散、数据结构复杂多样,导致知识表示、知识融合工程都较为复杂,构建过程中需要领域专家多方求证。领域知识图谱的模式层构建策略以自底向上或自顶向下的方式为主,本体的维度设计局限于领域知识体系范围,并未完全解决海量科技资源与领域知识之间的分散割裂问题。如何提出一套较为全面、完善的领域本体构建策略,是实现全景式知识图谱建设的核心。在知识图谱数据层构建方面,侧重于对非结构化文本的知识抽取,缺乏多路径的知识抽取的流程实践,无法实现多类型知识抽取融合的有机统一。本文针对这些挑战,拟分别在图谱的模式层全景式设计和数据层多路径构建策略上进行一定的集成应用与实践探索。
2 水稻粒型基因领域知识图谱构建
中国作为世界上第二大水稻种植国家,水稻总产量占全球30%以上。培育优良水稻品种,提升水稻亩产量,有助于打赢种业翻身仗,保障国家粮食安全,是我国农业发展的重要战略目标。水稻粒型作为影响水稻产量和品质的重要因素之一,其相关研究一直是作物领域的重要分支。水稻粒型基因领域研究涉及多组学知识,不同数据源之间的关联性较低,知识分散程度较高。本文接下来将集成应用现有相关理论方法,开展水稻粒型基因领域知识图谱的构建实践。
在模式层构建方面,本文提出领域知识图谱模式层“全景式”构建策略,基于专家先验知识,结合对领域通用本体的复用与融合,自顶向下构建图谱模式层。同时利用文本挖掘技术自底向上对本体模型进行迭代完善。“全景式”旨在从纵向维度对领域知识组织体系的深度挖掘,同时从横向维度对领域科技文献等其他类型信息资源的关联汇聚,更加系统全面地揭示知识图谱模式层中各类实体、概念及语义关联关系。在数据层构建方面,为充分整合现有多种形态科技资源、提升多源异构数据之间语义互操作性,提出了综合现有图谱数据剪切、结构化/半结构化知识转化映射以及非结构化知识抽取等方式的“多路径”知识抽取和自底向上补充完善知识图谱本体模型策略,实现全景式深层次知识关联融合。
2.1 全景式本体模型设计
2.1.1 多层次领域本体模型概要设计
领域本体模型是揭示领域知识组织体系的核心,为后续整合海量多源异构资源,实现深层次语义挖掘与关联奠定基础。本文基于自顶向下构建多层次领域本体模型的思路,参考多层次领域本体模型框架[12],结合水稻粒型基因领域专家先验知识,对多种领域本体资源进行复用与融合,实现对细分领域知识体系的多层次概念体系描述。
领域顶层本体作为构建领域本体的基本框架,须适应不同专题领域的应用需求变化,同时可增强各专题领域之间的互操作性。基本形式化本体[13](Basic Formal Ontology,BFO)作为顶层本体在OBO本体库中的成功应用,显示了其在生物学领域强大的可扩展性。本文选取BFO作为顶层本体框架,基于语义科学集成本体(Semanticscience Integrated Ontology,SIO)的主要概念分类体系,将实体(Entity)分为属性(Attribute)、对象(Object)和过程(Process),并梳理领域权威受控词表等资源,参考多种领域本体资源如基因本体(Gene Ontology)、植物本体(Plant Ontology)、植物性状本体(Plant Trait Ontology)、关系本体(Relation Ontology)等,从描述水稻基因组学、表型组学信息、蛋白质组学、代谢组学等多组学领域知识维度,构建描述水稻粒型基因调控的核心概念体系;同时结合2.2节多路径知识抽取转换过程中发现的新实体和实体关系类型,自底向上对领域本体模型进行优化迭代,使其尽可能满足多维度定义领域知识体系的本体构建需求。以水稻“粒型基因-性状调控网络”的概念体系为例,对本研究中涉及的多层次领域本体中的核心概念进行详细阐述,如图1所示。
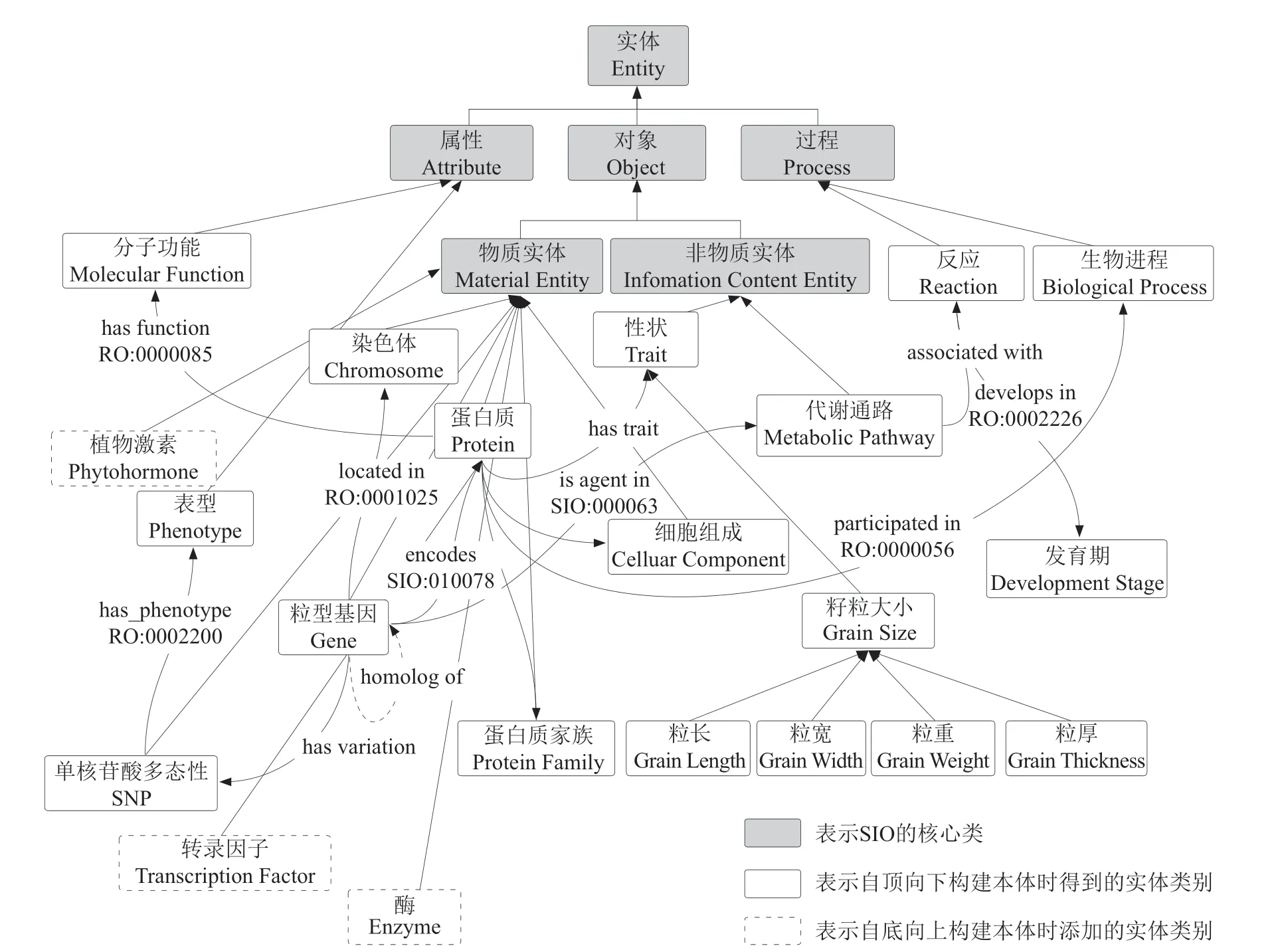
图1 多层次领域本体模型概要
2.1.2 融合科技资源的全景式领域本体模型
数据密集型科研时代背景下,科学数据、科技文献等资源呈现出多来源、跨媒体、多模态等特点,面对海量的多源异构科技资源,基于科技资源聚合的视角,提出融合科技资源的全景式领域本体模型,旨在有效解决领域知识与科技资源的割裂问题,为领域知识关联与发现服务提供有价值的数据支撑。本文通过引入以科技论文及相关资源为主要研究对象的科研通用本体,复用BIBO、DCMI、FOAF、DoCO等通用性较强的文献数据模型,围绕水稻粒型基因领域科技资源,从文献元数据、主题词、外部关联信息、文献登录号、作者信息、出版信息、参考文献信息等多维度对实体对象进行高细粒度描述,有效揭示水稻粒型基因领域科研活动主体与各科研对象之间的关联,添加包含期刊论文、科技报告、科研机构、科研人员、多媒体资源、科技图书、基金项目、科学数据集8类实体。以各类型科研实体的主题词属性作为衔接领域知识组织体系和科技文献资源的核心关联点,使单维度的领域知识组织体系转化为多维立体的知识聚合网络(见图2),有效实现领域知识和科技文献资源的语义互通,为构造全景式领域知识图谱提供更多研究视角。
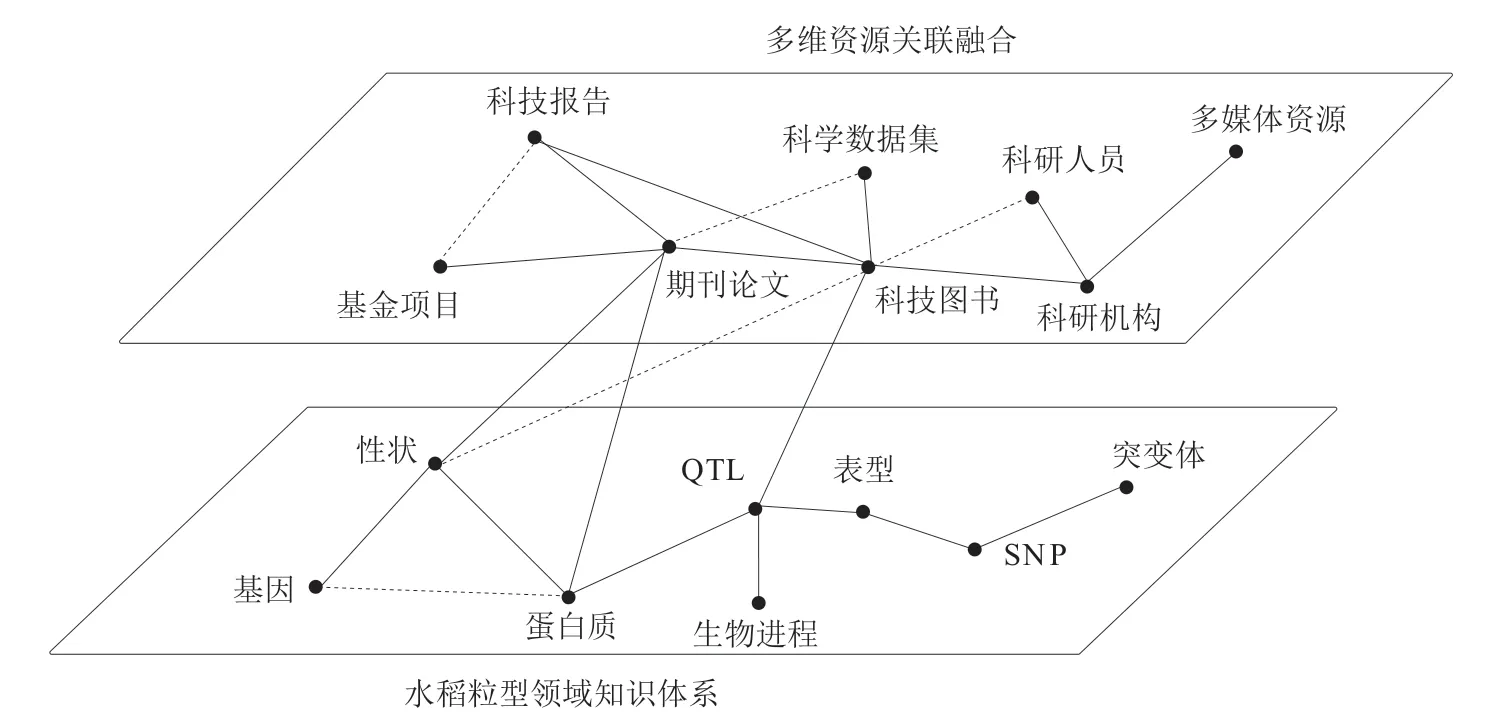
图2 全景式领域本体模型概要
2.1.3 基于Protégé的本体构建与管理
为完成全景式领域本体的概念体系和相关属性的设计,更好地对实体及实体间关系进行梳理,本文选择Protégé本体建模编辑器对水稻粒型基因领域实现面向本体的知识建模。在实体类方面,最终定义了11个一级类和17个二级类,其中核心类可根据描述性质的不同分为科研实体类和领域实体类:科研实体类包括期刊论文类(Article)、研究人员类(Researcher)、基金项目类(Project)、科研机构(Organization)、科技图书(Book)、科学数据集(Dataset)、多媒体资源(DataSource);领域实体类包括基因(Gene)、数量性状基因座(QTL)、蛋白质(Protein)、蛋白质家族(Protein Family)、过程类(Process)、本体注释(Annotation)、表型(Phenotype)等。在对象属性方面,主要定义为三类:第一类是对领域实体间关系进行界定的18个对象属性,如编码(encodes)、正调控(positively regulates)、负调控(negatively regulates)等;第二类是描述科研实体之间相互关联关系的6个对象属性,主要包括被引关系(cited_by)、发表于(published in)等;第三类是衔接领域实体与科研实体的2个对象属性,即与…关联(associated_with)、具有…主题词(has_topic)。最后通过基因符号(GeneSymbol)、资源标识符(IRI)、术语名称(TermName)等36个数据属性对实体类的特征实现具体化描述,便于后续多路径知识抽取过程中的映射转化。
2.2 多路径知识抽取转换
领域知识图谱的构建核心是基于既有的本体模型,针对领域数据的特点,结合图谱构建需求实现对不同数据类型的知识抽取。以水稻粒型研究为代表的作物领域包含基因组学、代谢组学、蛋白质组学等丰富类型的多组学数据及以科技文献为代表的科技资源,具有数据量庞大、多源异构等特点。如何将这些多类型数据转化为具有应用价值的知识单元并建立关联关系,成为当前领域知识组织创新中亟待解决的难题。利用多路径知识抽取转换的方式,是对多类型数据资源实现多维聚合和知识关联的有效手段。
本文基于水稻粒型基因领域专家团队针对水稻粒型基因调控的综述论文[14-15],整理归纳出65个已克隆的水稻粒型基因基本信息,其中包括基因名称、分子功能描述、染色体位置信息、突变体信息、表型信息、MSU登录号等,形成以专家先验知识为主的“种子”术语知识库(以下简称“种子知识库”)。在此基础上,面向AgroLD、Ensemble Plants等多个高质量数据源,利用对现有图谱数据剪切、结构化/半结构化知识转化映射以及非结构化知识抽取等多路径,基于2.1节构建的领域本体模型,对数据进行知识抽取、转换与融合,挖掘知识间潜在的关联关系,实现对领域知识的深层次挖掘和理解,如图3所示。
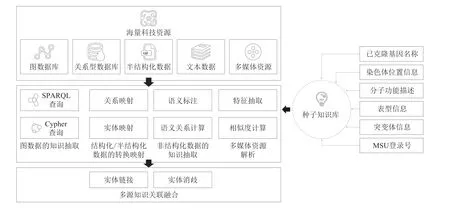
图3 多路径知识抽取总体思路
2.2.1 基于图数据的知识抽取
近年来,以关联数据发布的不同研究领域的数据集增长迅速,为促进各领域资源的开放、互操作、共享、复用奠定了基础。采用RDF数据模型的关联数据技术在生物信息领域和作物领域的实践应用探索一直较为活跃,为作物领域的多组学研究积累了大量宝贵的图数据资源。图数据通过三元组的方式将知识结构化保存,是一种基于事务关联关系的模型表达,具有数据天然可解释性[16],相较于传统关系型数据库,图数据库对语义关系表达、存储和高效复杂查询提供了良好支撑。目前图数据模型主要分为RDF图模型和属性图模型(Property Graph)。RDF图模型的超图本质,较强的理论性,以及语义Web多年标准化工作的推进,都促进RDF图模型在学术研究社区的主流地位[17],但随着以Neo4j为代表的图数据库在各个领域的广泛应用,基于属性图模型的图存储凭借其强大的可操作性逐渐获得更高认可。因此,本节主要探索如何解决两类图数据之间的互操作以实现基于图数据的知识抽取。
就图数据之间的互操作,笔者将其分为以下4种方式。①RDF图之间的互操作。随着RDF数据的不断丰富与扩展,不同的RDF格式文件之间的相互映射转换以及RDF数据库之间的互操作成为亟待解决的问题。目前主流的RDF数据格式有RDF/XML、N-Triples、Turtle、JSON-LD等,而数据库有Virtuoso、GraphDB等,笔者团队基于开源工具Kettle,提出了大规模RDF一体化治理流程和工具RDFAdaptor[18],显著增强了RDF三元组数据在不同格式和RDF数据库间的互操作性。②属性图之间的互操作。目前关于属性图之间的互操作研究较少,主要是通过不同属性图之间的模式层建立映射,再实现实例层的转换。③RDF图向属性图的转化。Neo4j官方推出的neosemantics插件[19]支持将不同格式的RDF数据较低损耗地导入Neo4j图数据库,但模式层的映射规则不支持自定义,不利于满足用户个性化的应用需求。为此,关于自定义实现RDF图向属性图的转化研究越来越多,其中rdf2neo[20]基于既有的领域本体结合自定义SPARQL查询实现大规模RDF图数据面向Neo4j图数据库的存储。rdf2pg[21]面向不同格式三元组支持用户自定义映射规则实现RDF图向属性图直接转化。④属性图向RDF图的转化。PREC[22]提出一种转化方法:设计统一的RDF规范化数据模型对属性图的节点、边、标签等进行描述,再依次利用MATCH查询语句获取节点信息、边信息、标签信息,实现三元组的重构。
本文以种子知识库中的65个水稻粒型基因的唯一登录号(identifier)作为筛选条件,针对CIRAD搭建的AgroLD联邦型RDF图数据库,采用作者团队研发的RDFAdaptor工具,基于SPARQL的CONSTRUCT图查询,批量得到描述水稻粒型基因的RDF子图并导入Virtuoso中,便于后续管理操作。针对RDF图数据,一般可通过两大步骤实现向属性图的转化处理。首先,实现主语及其标识和所属对象类的转换。RDF三元组的主语是具有URI属性的资源类型节点(Resource Nodes),根据主语确定属性图中的实体(Entity),并将URI属性转化为属性图中实体的URI属性;同时,根据特定谓语标签rdf:type判断资源类型节点的所属类别(ClassType),并基于本体设计,将所属类别转化为属性图的实体类别(Label)。其次,完成RDF谓语(数据属性、对象属性)和宾语的转换。三元组中的宾语可分为两类节点,一类是具有URI属性的资源类型节点,另一类是不具有URI属性的字面量类型节点(Literal Nodes)。当宾语是资源类型节点时,则谓语(对象属性)映射为属性图中的关系(Relationship);当宾语是字面量类型节点时,则谓语(数据属性)映射为属性图中的属性(Property)的键(Key),字面量映射为值(Value),从而实现RDF图向属性图的完整转化。
2.2.2 基于结构化/半结构化数据的映射转换
当前,大量有价值、高质量的数据资源仍以关系型数据库(结构化)或文件(半结构化)形式存储,为知识图谱实例层扩充奠定重要的数据基础。在作物领域,面向结构化/半结构化的数据源主要有以下类型。①结构化数据:关系型数据、Excel文件、CSV文件等。②半结构化数据:OBO格式的本体数据、基于JATS标准的XML格式PubMed文献数据、描述蛋白质信息的XML格式数据等。面向多来源的结构化数据的抽取与转化是填充知识图谱数据层的重要环节。半结构化数据可通过XML解析器(SAX、DOM等)、OBO解析器等成熟工具便捷地转化为结构化数据。因此本节将重点叙述如何实现结构化数据向图数据的转化,主要根据图数据模型的不同分为两项子任务:①结构化数据向RDF数据的转化;②结构化数据向属性图数据的转化。
本文基于全景式本体模型,通过自定义映射规则的方法,基于Kettle转化工具并结合插件RDFAdaptor和Neo4j GraphOutput[23]分别实现面向RDF图的转化和面向属性图的转化。两种方法本质上都是在已知输入关系表设计的情况下抽取既定实体类型,并基于既有本体模型设定相应的配置参数,实现映射转化。以实现文献元数据关系表面向两种不同数据模型的图数据的转化为例,在面向RDF图的转化中,本文基于既定本体模型依次对命名空间(NameSpace)和映射规则进行设定,实现关系表向RDF数据中主谓宾的映射转化。在面向Neo4j图数据库的转化中,本文基于既定本体模型,确定结构化数据表中各字段与本体模型(Graph Model)的映射关系,将字段间的关联关系划分为节点属性(Properties)和关系(Relationships)两种类型。此外,在实际映射过程中须考虑同名字段的异义、异名字段的匹配映射等问题。基于此映射原则,依次对属性图的节点、关系、属性、类型等关键要素以及输入表字段与各要素之间的映射关系进行设定,最终实现面向Neo4j的转化存储。
2.2.3 基于非结构化数据的知识抽取
当前,越来越多的数据以非结构化文本形式存在。以科技文献全文为代表的非结构化文本中包含大量具有丰富语义价值的知识。面向非结构化文本抽取领域知识实体及其语义关系,是当前知识图谱构建知识抽取环节的主要任务,也是最大挑战。早期研究主要集中在基于模式匹配的实体关系抽取,即结合领域知识特点通过人工构造规则实现知识抽取,其优点是知识的权威度和精准度得到了有效保证。但随着数据量的增加,与日俱增的时间与人力成本也成为其明显的缺点。随着自然语言理解技术的不断发展,自动化知识抽取方法逐步占据研究的主流地位,例如面向开放域的实体关系抽取、基于远程监督的实体关系抽取以及基于深度学习的实体关系联合抽取等[24-26]。相较于传统知识抽取方法,自动化抽取对语料库本身质量要求较高。因此,以领域知识库作为先验增强自动化抽取,是实现非结构化知识抽取质量与效率并重的重要手段。
本文结合领域知识特点,在兼顾领域知识抽取精准度和效率的基础上,对面向领域文本的知识抽取方法进行综合改进:①笔者基于PubMed构建检索式“(rice OR oryza sativa)AND粒型基因名称”批量获得239篇领域英文文献,通过去标签化等数据预处理工作,构建句子级别的纯文本文献数据集,便于后续语义标注;②利用农业领域自动化标注工具AgroPortal Annotator[27]对数据集进行较高效的自动化实体识别,并基于语义计算结果得出最优候选实体;③将自动标注获得的实体集合与种子知识库中既有的实体集合进行去重合并,构建“特征词词典”,在既有实体集合基础上新增258个领域实体,其中包括与水稻粒型基因调控密切相关的激素实例Auxin(生长激素)、BR(油菜素内酯)、ABA(脱落酸)等;④对文献数据集进行过滤筛选,仅保留包含特征词的语句28 399条,有效提高数据集质量;⑤基于开放域知识抽取工具OpenIE[28]对数据集进行自动化抽取获得57 407个三元组,仅保留特征词作为头尾实体的三元组7 268条;⑥OpenIE的优势是基于自动化抽取三元组帮助研究者发现潜在的关系类型,但是易造成同种关系多种表达的问题,如“is homologous to”和“are homologs of”都指向关系类型“homolog of”。因此,为了确保标注结果的精准度和权威性,本文邀请学科领域专家进行审核,并对数据进行规范化处理,最终对图谱进行三元组有效补充848条,新增3种实体类型Phytohormone(植物激素)、Transcription Factor(转录因子)、Enzyme(酶)以及3种关系类型homolog of(是…同源物)、ortholog of(是…垂直同源物)、paralog of(是…并行同源物)。
2.3 多来源知识关联融合
知识融合是知识图谱构建的重要环节,基于多来源的知识关联融合是对知识抽取结果进行深度加工处理和整合的过程,需要挖掘隐性知识及潜在知识关联,实现对知识的深层次语义挖掘,为后续知识发现等服务奠定良好的数据基础。多源知识关联融合主要解决多个来源的有关同一实体或概念的描述信息实现低冗余、高准确率的合并,包括概念对齐、实体对齐、属性对齐以及冲突检测与解决。
本文实现多来源知识关联融合主要包括四大任务。一是对多路径知识抽取得到的知识实体进行知识融合,其中领域实体主要依赖于不同数据库之间既有的登录号映射进行融合,文献实体可根据DOI和PMID等唯一登录号进行实体对齐。二是基于既有图谱对实体数据进行消歧处理,例如水稻粒型基因GS3、LK3、异三聚体G蛋白γ亚基均指向同一实体,需对其合并处理。主要方法是基于上述知识抽取工作,结合相似度计算和人工校验进行实体对齐处理。三是对文献中所涉及的图表信息进行抽取,基于图表在文献中的位置关系、图表标签文本的相似度计算,建立图表与基因等实体的关联。四是将百科中多媒体资源与既有图谱中的部分实体进行实体链接处理,实现多维度、细粒度的全景式知识图谱(见图4)。最终形成图谱实体类型16种、关系类型31种,实体总量42 862个以及关系数量61 014条。
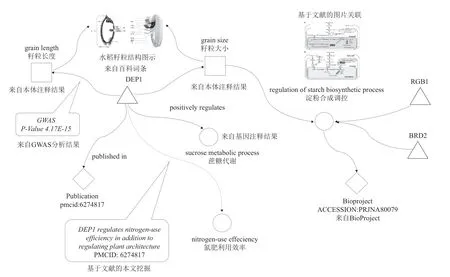
图4 多资源知识融合示意图
2.4 相关知识图谱对比分析
本节主要根据知识图谱的模式层和数据层构建逻辑,将上述所构建水稻粒型基因知识图谱与当前两大作物领域知识图谱KnetMiner和AgroLD进行了对比分析。
在图谱模式层方面,AgroLD旨在利用关联数据技术实现作物领域多类型数据的聚合,具有较强的可扩展性和通用性,但其本体模型并不涉及领域文献等科技资源,仅关注于对领域知识组织体系的描述,无法有效解决领域知识与科技资源之间的割裂问题;KnetMiner旨在利用UniprotKB自带的参考文献构建引文网络,与领域知识组织体系进行有效关联。同时,二者的本体建模主要采用自顶向下的构建模式,并未考虑利用文本挖掘技术发现新的实体和关系,不支持对既有模型进行更新迭代,缺乏一定灵活性。本文所构建全景式领域本体模型聚焦水稻粒型基因领域,针对领域应用需求进行了更多维度、更高细粒度的描述设计,不仅通过文献外部特征去实现与领域知识的关联,还通过面向全文的知识抽取发现潜在的新实体类型和关系类型,实现对领域知识的全景式描述。
在图谱数据层构建方面,AgroLD的知识抽取是对既有关联数据的集成,KnetMiner则是将知识抽取的重心放在对多种半结构化数据的知识抽取。本文立足全景式领域本体模型,采用多路径知识抽取的方法,不仅对多个主流领域知识库进行知识融合,还利用优化后的自动化三元组抽取技术将领域知识组织体系与科技文献的全文内容特征建立关联,挖掘潜在知识,既实现了对图谱数据层的填充,又支撑后续对本体模型的迭代完善。
3 知识图谱驱动下知识关联发现的应用展望
数据驱动创新的智能时代背景下,知识图谱作为实现数字资源重构和智能化应用的技术支撑,广泛应用于推荐系统、智能问答、实体检索、知识发现等多种场景,也为推进细分专业领域知识服务的个性化、智能化转型提供了重要参考。领域知识图谱的应用研究方向主要是利用知识图谱天然的图结构构建关联拓扑网络,挖掘知识单元之间的显性和隐性关联,为实现图谱驱动下的知识关联与发现服务奠定数据基础。当前,图谱驱动下的典型新型知识关联与发现服务实践包括:以Yewno[29]作为基于概念实体的知识关联服务的范例,通过关联大量数据源中的概念促进用户挖掘知识内在的深层次关联;以Open Knowledge Maps[30]为典型代表的基于聚类的知识关联服务,利用知识地图代替传统列表浏览进行文献检索,从而有效提高科学知识的可发现性;在生物医学领域面向药物间相互作用的基于推理的知识关联服务[31]也逐渐兴起。以上这些应用范例为本文探索以水稻粒型基因知识图谱所驱动下的新型知识服务场景设计提供了新的视角。
图谱驱动下的实体检索服务主要是利用知识图谱的复杂网络结构对实体与实体间的丰富语义关系进行有效的表达,不仅能够帮助用户检索和发现目标实体的相关信息,还可以深度挖掘知识实体间的潜在关联,实现复杂关联查询,有效提升了检索过程中的知识表示层次。图谱驱动下的实体检索可分为三个层次:匹配检索、扩展检索和推荐检索[32]。以水稻粒型基因GS3进行实体检索,可通过既有图谱构建的实体及实体关系的关联路径(如基因→encodes→蛋白质→published_in→文献→has_project→基金项目)实现基于GS3的多实体复杂关联检索。区别于传统的关键词检索仅能获得GS3的单一实体信息,图谱驱动下的多维度立体检索可获得与基因GS3密切关联的基因(如PGL2)、性状(如粒宽、粒重、籽粒大小)、生物进程(如粒重负调控)等实体信息以及相应的多媒体资源,彰显了全景式领域知识图谱的应用价值。
随着“数据驱动创新”理念的提出,当前知识服务系统创新的重点是基于既有知识图谱或知识库挖掘潜在的语义关系以及发现“新”的知识实体。知识发现服务主要分为两类。一类是基于实体概念之间共现关系的显性知识发现。例如,研究人员可根据图谱中的实体共现关系挖掘水稻粒型性状(粒宽,grain width)、基因(DEP1)以及生物进程(淀粉合成调控,regulation of starch biosynthetic process)之间的语义关系,促进领域科研人员对新研究方向的思考。另一类是基于语义模型的隐性知识发现,基于自动化知识抽取技术可获取与水稻粒型基因调控密切相关的“新”基因包括GGC2、RGG1等,供科研人员进一步分析思考。
4 结语
数据密集型科研时代,构建全景式领域知识图谱在深层语义揭示和关联组织领域知识、解决领域知识与科技信息等资源割裂问题等方面具有重要作用。本文以水稻粒型基因领域为例,在综合集成主流技术方法的基础上,进一步研究提出了全景式、多路径的领域知识图谱构建方案,在尽可能兼顾知识图谱中各类知识覆盖的广度和深度的同时,较充分继承整合现有的各类多源异构数据和知识,具有一定集成性和通用性,可为其他领域知识图谱的快速高效构建提供参考。
本研究也存在不足:在全景式领域本体模型构建方面,自顶向下的建模比例仍然较大,需要对专业领域知识结构有较深入理解,基于大量数据自底向上自动抽取或形成知识结构的研究不够深入;在多路径知识抽取策略方面,本文主要针对多形态数据分别提出不同的技术路线,离形成无缝集成和一体化的解决案例还有一定差距。同时,还需要重点结合深度学习和专业知识组织体系,加强知识抽取对知识图谱结构丰富和实例动态补全,基于知识图谱迭代的思想,不断实现领域知识图谱的扩充、更新、融合与增强。此外,本文目前只针对水稻粒型基因领域开展小规模实验研究,后续可通过开展大规模多组学、多物种数据之间的关联与对比,面向专业细分领域的知识服务需求,构建全景式领域知识图谱,打造图谱驱动下的新型领域知识服务系统,为研究人员提供精细化、智能化、个性化知识服务。

