乙酸胁迫条件下酿酒酵母全基因组启动子区组蛋白H4 K16 乙酰化修饰分析
马 田,王玉婧,程 爽,张小华,刘向勇
(滨州医学院药学院,山东 烟台 264003)
随着全球经济的快速发展和世界人口的不断增加,化石能源的消耗与日俱增。但化石能源为不可再生资源,同时在大量使用过程中产生空气污染、温室效应等环境问题,寻求可再生的环保型新能源成为亟待解决的社会问题。
高效、清洁、可再生能源燃料乙醇被认为是化石能源最有潜力的替代品。木质纤维素具有成本低、可再生、原料丰富等优点,其中农业废弃物(秸秆、玉米芯、蔗渣等)是木质纤维素材料的主要来源。近年来,利用酿酒酵母代谢木质纤维素生产燃料乙醇成为生物质资源开发利用的研究热点[1-2]。但木质纤维素预处理过程中会伴随产生大量有毒抑制物,严重影响微生物的发酵性能,是制约生物燃料乙醇规模化生产的重要因素[3-4]。
乙酸是木质纤维素水解液中主要抑制物之一,可导致酵母细胞内酸化,抑制细胞正常代谢与生长,甚至引发细胞死亡[5-8]。因此,深入解析酵母乙酸耐受机制,选育高乙酸耐受菌株,对推动纤维素乙醇发酵的规模化生产具有重要意义。
组蛋白是构成真核生物染色体的基本结构蛋白,其可与DNA 紧密结合组成核小体。组蛋白中某些特定的氨基酸位点在相关酶(如组蛋白乙酰转移酶、组蛋白甲基转移酶等)作用下可发生乙酰化、甲基化、ADP 核糖基化等表观遗传学修饰[9]。已有研究表明,这些组蛋白修饰可通过改变染色质的结构、影响基因转录因子结合等调节基因的表达,在多种生物学过程中发挥重要作用[9-14]。
酿酒酵母组蛋白H4 中的16 位赖氨酸(K16)是已知的重要乙酰化修饰位点[15-19]。前期研究发现,酿酒酵母组蛋白H4 K16 位点突变(赖氨酸突变为谷氨酰胺)可导致酵母乙酸耐受性升高,表明组蛋白H4 K16 位点与酵母乙酸耐受性相关[20]。为深入揭示组蛋白H4 K16 位点在酵母乙酸胁迫应答的作用,采用染色质免疫共沉淀-芯片(Chromatin immunoprecipitation-chip,ChIP-chip)技术分析乙酸胁迫条件下酿酒酵母全基因组启动子区组蛋白H4 K16乙酰化修饰的变化,进一步解析酵母乙酸耐受机制,为推进酿酒酵母代谢木质纤维素类农业废弃物,生产燃料乙醇提供理论基础。
1 材料和方法
1.1 试验材料
野生型酿酒酵母菌株BY4741(MATa his3Δ1 leu2Δ0 met15Δ0 ura3Δ0)由日本酵母遗传中心惠赠。酿酒酵母培养采用液体酵母浸出粉胨葡萄糖(Yeast extract peptone dextrose,YPD)培养基:葡萄糖20 g/L、蛋白胨20 g/L、酵母粉10 g/L,pH值调整为4.5或6.8,115 ℃高温灭菌30 min。
1.2 试验方法
1.2.1 酿酒酵母乙酸胁迫处理 接种酿酒酵母BY4741 单菌落于10 mL YPD(pH 值6.8)液体培养基中,30 ℃、100 r/min 摇床过夜培养。然后转接至100 mL 新鲜YPD(pH 值4.5)液体培养基中,酵母菌体起始量为OD600=0.1。摇床培养至酵母菌体进入对数生长阶段(OD600=0.6~0.7),将酵母培养物平均分为2份(每份50 mL),分别标记为对照组和乙酸胁迫处理组。在乙酸胁迫处理组中,添加347 μL乙酸(8.7 mol/L,pH 值4.5),至乙酸终浓度为60 mmol/L,对照组添加等体积无菌水。30 ℃、100 r/min 摇床继续培养30 min,离心收集酵母细胞。乙酸胁迫处理组和对照组均设置2个生物学重复。
1.2.2 酿酒酵母全基因组ChIP-chip 分析 酵母细胞经甲醛(终质量体积比为1%)化学交联后,采用Diagenode bioruptor UCD-200 非接触式超声波破碎仪(Diagenode公司,比利时)随机切割酵母细胞染色质DNA,并通过琼脂糖凝胶电泳检测超声破碎效果。在经超声处理的酵母染色质DNA 提取物中取出一部分作为Input DNA(ChIP-chip 的内参对照),另一部分加入组蛋白H4 K16 乙酰化特异性抗体(Active motif 公司,美国)和Protein A/G 免疫沉淀磁珠(Santa cruz 公司,美国)进行交联染色质的免疫沉淀。免疫沉淀复合物经NaCl(终浓度为0.2 mol/L)处理解交联后,采用QIAGEN DNA 纯化试剂盒(Qiagen 公司,德国)回收免疫沉淀DNA。免疫沉淀DNA 经 GenomePlex®complete whole genome amplification kit(Sigma-aldrich 公司,美国)扩增后,采用QIAquick PCR purification kit(Qiagen 公司,德国)进行DNA 纯化。纯化的DNA 经NanoDrop ND-1000(Thermo scientific™公司,美国)定量后,使用NimbleGen dual-color DNA labeling kit 进行荧光标记。荧光标记DNA 与Agilent yeast whole genome array芯片(型号:G48104-14810,Agilent公司,美国)杂交之后,采用Agilent scanner G2505C 进行芯片扫描。芯片数据经软件Agilent feature extraction software(version 11.0.1.1)和Bioconductor packages分析后,使用NimbleScan v2.5 软件(Roche-NimbleGen 公司,美国)筛选位于酿酒酵母基因组启动子区域的组蛋白H4 K16 位点乙酰化修饰改变(升高或降低)富集峰。组蛋白H4 K16 乙酰化修饰差异位点最终选取在2个生物学重复试验结果中呈现相同变化趋势的富集峰区域。采用在线基因富集分析工具WebGestalt(http://www.webgestalt.org/)[21]和 Saccharomyces genome database(http://www.yeastgenome.org)数据库对启动子区组蛋白H4 K16乙酰化修饰发生差异变化的基因进行功能聚类分析。
1.2.3 染色质免疫沉淀-实时定量PCR 以免疫沉淀DNA 和Input DNA 为模板,采用适用于SYBR GreenⅠ荧光法检测的实时定量PCR 预混液SYBR Green Real-Time PCR Master Mix(Arraystar 公司,美国),在QuantStudio ™5 Real-Time PCR System(Applied biosystems 公司,美国)中进行实时荧光定量PCR 扩增。染色质免疫沉淀-实时定量PCR 所用引物见表1。PCR 反应条件:95 ℃,10 min;40 个PCR 循环[95 ℃,10 s;60 ℃,60 s(收集荧光)]。染色质免疫沉淀-实时定量PCR 数据采用Percent input(%input)=2(CtInputDNA-Ct免疫沉淀DNA)×稀释倍数×100%法[22]分析。染色质免疫沉淀-实时定量PCR试验结果以2个独立生物学重复测定结果的算术平均值表示。
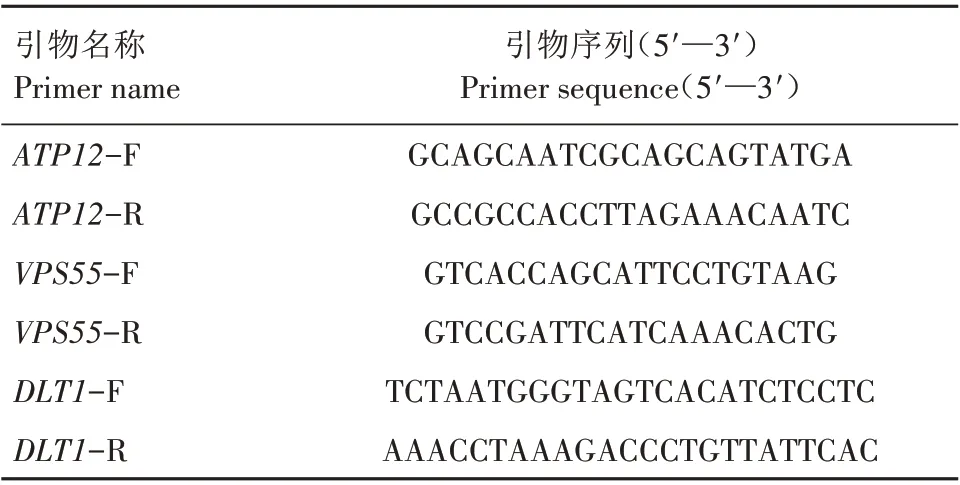
表1 染色质免疫沉淀-实时定量PCR所用引物信息Tab.1 Primer sequences used for chromatin immunoprecipitation with quantitative real-time PCR
2 结果与分析
2.1 酵母染色质DNA片段化分析
染色质DNA 片段化的效果是影响染色质免疫沉淀试验质量的重要因素。通过琼脂糖凝胶电泳分别检测酿酒酵母乙酸胁迫处理组和对照组中经超声波片段化的酵母染色质DNA。结果(图1)显示,乙酸胁迫处理组和对照组中的酵母染色质DNA片段介于250~1 000 bp,能够满足后续染色质免疫沉淀试验的要求。
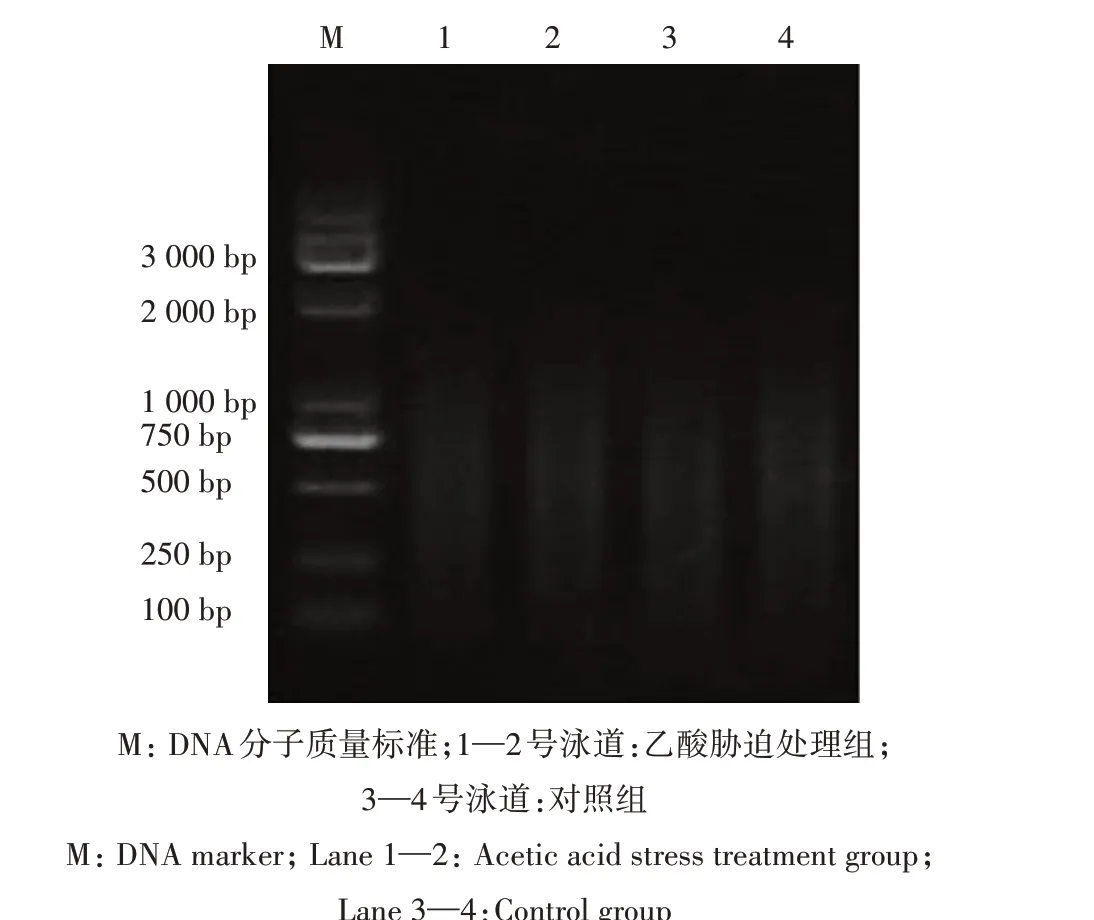
图1 超声切割酵母细胞染色质DNA大小的琼脂糖凝胶电泳检测Fig.1 Determination of sheared yeast chromatin DNA size through agarose gel electrophoresis
2.2 酿酒酵母全基因组ChIP-chip分析
ChIP-chip 分析结果显示,与对照组相比,酵母细胞经60 mmol/L 乙酸胁迫处理30 min 后,73 个酵母基因的启动子区域组蛋白H4 K16 位点乙酰化修饰发生了改变(表2)。其中,组蛋白H4 K16 位点乙酰化修饰上升基因19 个,组蛋白H4 K16 位点乙酰化修饰下降基因54个。

表2 乙酸胁迫条件下启动子区组蛋白H4 K16乙酰化发生差异变化的酵母基因Tab.2 Yeast genes with differentially altered acetylation levels of histone H4 K16 in the promoter regions under acetic acid stress
进一步对组蛋白H4 K16 乙酰化修饰差异位点的染色体分布情况进行分析,结果见图2。组蛋白H4 K16 乙酰化修饰上升基因主要分布在2、4、5、7、13、15、16 号染色体上,其中7 号染色体上基因数量最多(12 个);组蛋白H4 K16 乙酰化修饰下降基因主要分布在1、2、4、5、6、7、8、10、11、12、13、14、15、16 号染色体上,其中15 号染色体上基因数量最多(10个)。

图2 乙酸胁迫条件下启动子区组蛋白H4 K16乙酰化发生差异变化基因的染色体分布Fig.2 Chromosome distribution of genes with differentially altered acetylation levels of histone H4 K16 in the promoter regions under acetic acid stress
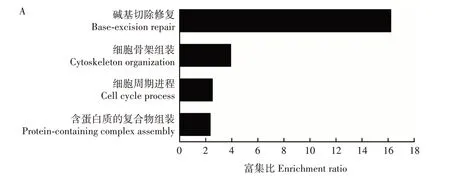
利用在线基因富集分析网站WebGestalt(http://www.webgestalt.org/)和酿酒酵母全基因组数据库(Saccharomyces genome database)(http: www.yeastgenome.org),对乙酸胁迫条件下启动子区组蛋白H4 K16 乙酰化发生差异变化基因的功能进行分析。结果(图3)显示,按生物学过程分类基因功能主要富集在碱基切除修复、细胞骨架组装、细胞周期进程和含蛋白质的复合物组装;按细胞组分分类基因功能主要富集在DNA聚合酶复合体、液泡质子转运V-型ATP 酶、质子转运双扇区ATP 酶复合物/质子转运域、有丝分裂纺锤体和核染色体;按分子功能分类基因功能主要富集在单链DNA 3′—5′外脱氧核糖核酸酶活性、蛋白酶体结合、ATP 酶活性/耦联离子跨膜运动。
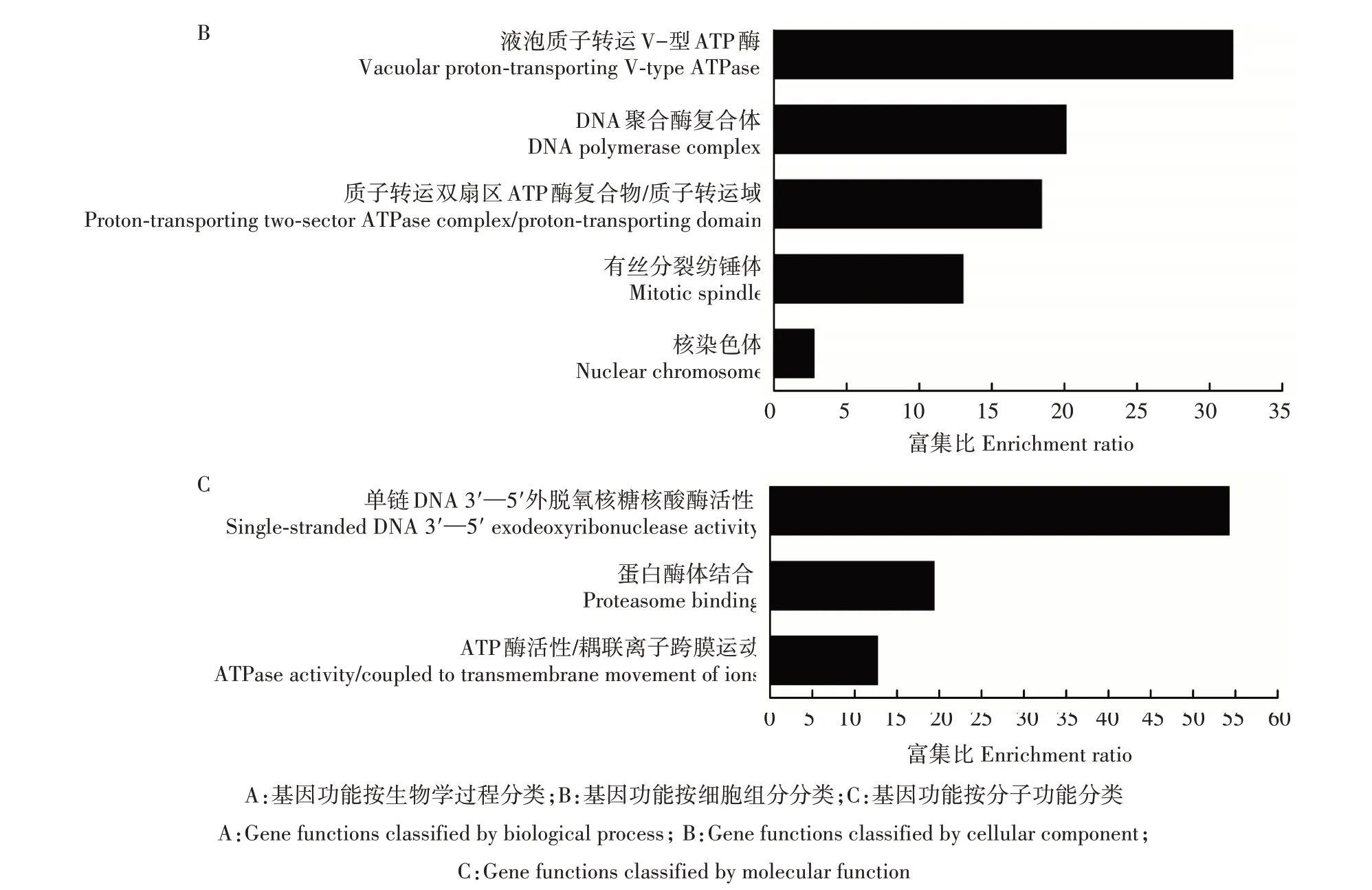
图3 乙酸胁迫条件下启动子区组蛋白H4 K16乙酰化修饰发生差异变化基因的功能分类Fig.3 Functional classification of genes with differentially altered acetylation levels of histone H4 K16 in the promoter regions under acetic acid stress
2.3 染色质免疫沉淀-实时定量PCR验证
为进一步验证ChIP-chip 试验结果,随机选取3个启动子区组蛋白H4 K16 乙酰化修饰发生差异变化的基因(ATP12、VPS55、DLT1),进行染色质免疫沉淀-实时定量PCR 检测。试验结果(表3)表明,染色质免疫沉淀-实时定量PCR 结果与ChIP-chip试验结果(表2)一致,酵母细胞经乙酸胁迫处理后ATP12和VPS55基因启动子区域组蛋白H4 K16 乙酰化修饰下降,而DLT1基因启动子区域组蛋白H4 K16乙酰化修饰上升。
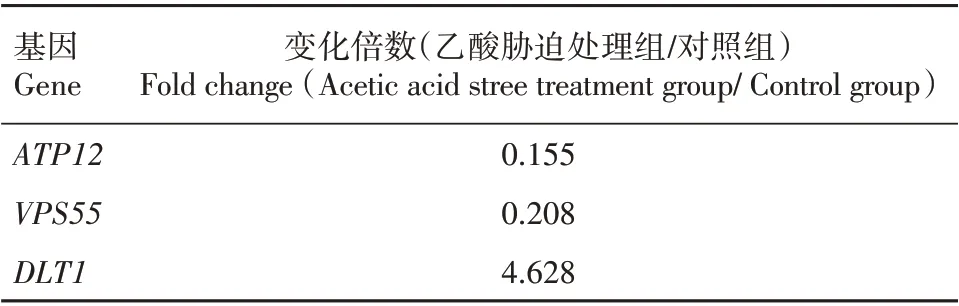
表3 染色质免疫沉淀-实时定量PCR结果Tab.3 Result of chromatin immunoprecipitation with quantitative real-time PCR
3 结论与讨论
木质纤维素预处理过程中产生的乙酸会对酵母细胞的生长代谢及发酵能力产生抑制作用[23-25],深入揭示酵母乙酸耐受机制对提高酵母乙酸耐受性和乙醇发酵产量具有重要意义。已有研究表明,酵母细胞会启动多种机制应答乙酸胁迫环境,如通过细胞壁结构重构和细胞膜成分调整来减少乙酸分子向胞内扩散、增加细胞膜和液泡膜上质子泵对细胞质中过量H+的外排,加速细胞质pH 值恢复到正常水平、调整碳水化合物代谢途径以应对乙酸胁迫引发的能量限制,以及促进大量乙酸胁迫相关转录因子活化(例如Haa1p)并激活下游靶基因等[5-8]。
虽然人们对酵母乙酸耐受机制的认识不断深入,但是对其可能涉及的表观遗传调控机制至今却知之甚少。表观遗传学是一种不涉及DNA 序列变化的可逆的基因表达调控方式,组蛋白乙酰化修饰是最普遍的表观遗传调控机制之一。研究表明,酵母组蛋白H4 K16 位点的乙酰化修饰作用与细胞寿命、染色质组装、DNA 损伤修复、基因沉默、DNA 复制等密切相关[15-19]。在前期研究发现组蛋白H4 K16位点突变可导致酵母乙酸耐受性升高的基础上[20],进一步采用染色质免疫共沉淀-芯片技术研究发现,酵母细胞经乙酸胁迫处理(60 mmol/L、30 min)后,与对照组相比,73 个酵母基因的启动子区域组蛋白H4 K16 位点乙酰化修饰水平发生了改变(乙酰化修饰上升基因19 个、下降基因54 个),并且广泛分布于多条染色体上。后续基因功能分析结果显示,在生物学过程、细胞组分和分子功能3个不同层面均有基因的显著富集并涉及多种功能类别。以上73 个酵母基因中有10 个基因已报道与酵母乙酸耐受性密切相关,具体包括液泡膜H+-ATP 酶VMA3和VMA6、分离酶ESP1、蛋白激酶GCN2、硫胺素生物合成基因的转录激活因子THI2、细胞分裂周期蛋白CDC37、转醛酶TAL1、线粒体内膜蛋白MDM32、磷酸泛酰巯基乙胺基转移酶PPT2 基因及絮凝基因FLO1,其中VMA3、VMA6、ESP1、GCN2、THI2、CDC37、MDM32、PPT2基因缺失可导致酵母乙酸耐受性显著下降[26-30],TAL1和FLO1基因过表达可提高酵母乙酸耐受性[31-32]。大量乙酸耐受性密切相关基因启动子区域组蛋白H4 K16 位点乙酰化修饰水平的改变,表明组蛋白H4 K16 位点乙酰化修饰在调控酵母乙酸胁迫应答过程中发挥重要作用。此外,已有研究表明,乙酸会抑制线粒体呼吸作用,引发能量危机[33]。本研究发现,线粒体合成酶ATP12和ATP20基因启动子区域组蛋白H4 K16 位点乙酰化修饰水平发生显著改变,提示组蛋白H4 K16位点乙酰化修饰参与了乙酸胁迫条件下酵母细胞的能量代谢调节。同时,本研究还发现,大量功能未知基因(YBR141W-A、YAR047C、YEL020C-B、YJL070C、YKL202W、YNL050C、YOL118C、YOR034CA、YOR385W)的启动子区域组蛋白H4 K16 位点乙酰化修饰水平发生了改变,后续深入解析这些基因的功能以及组蛋白H4 K16 位点乙酰化修饰对其功能的影响,将有助于进一步揭示酵母乙酸耐受机制。总之,以上结果可增加从表观遗传学角度(组蛋白乙酰化修饰)对酵母乙酸耐受机制的全新认识,为选育适用于代谢木质纤维素类农业废弃物的高乙酸耐受菌株提供理论基础。
组蛋白赖氨酸的乙酰化/去乙酰水平处于动态变化中,主要由组蛋白乙酰转移酶和组蛋白去乙酰化酶的共同调节完成[34]。最新研究发现,NAD 依赖的组蛋白去乙酰化酶Sir2中与自身谷胱甘肽化修饰相关的关键氨基酸(363 位半胱氨酸、469 位半胱氨酸和513位半胱氨酸)突变,会引起酵母细胞乙酰辅酶A 合成酶活性升高,进而促进乙酸向乙酰辅酶A转化,最终导致酵母乙酸耐受性升高[35]。此外,CHENG 等[36]的研究发现,酿酒酵母组蛋白H3 乙酰化转移酶Rtt109 缺失可提高酵母乙酸耐受性和乙酸胁迫条件下的乙醇发酵性能,其主要机制可能涉及HSP12等胁迫环境应答基因表达水平增加和过氧化氢酶等抗氧化酶活性的升高。本研究结果与以上结果一致,进一步强调了组蛋白乙酰化修饰过程与酵母乙酸耐受性的相关性,表明通过基因工程手段改变组蛋白乙酰化修饰过程是提高酵母乙酸耐受性的新途径。
此外,除以乙酸为代表的有机酸类抑制物外,木质纤维素预处理过程中还会产生呋喃类和酚类等抑制物[37-39]。本研究可为开展酵母耐受其他木质纤维素水解抑制物的高通量表观遗传学分析提供借鉴,加速木质纤维素水解抑制物毒性机制研究,进而促进以酿酒酵母代谢木质纤维素生产燃料乙醇为方向的农业废弃物资源化利用。

