高寒湿地冻融退化下微生物群落特征变化
章妮, 陈克龙, 王恒生, 杨阳
高寒湿地冻融退化下微生物群落特征变化
章妮1,2, 陈克龙2,3,*, 王恒生4, 杨阳1,2
1. 青海师范大学生命科学学院, 西宁 810008 2. 青海省青海师范大学青藏高原地表过程与生态保育教育部重点实验室, 西宁 810008 3. 青海师范大学科技处, 西宁 810008 4. 合肥师范学院生命科学学院, 合肥 230061
全球变暖大背景下, 大量冻土将面临进一步退化, 而冻融过程遍布整个青藏高原, 土壤微生物对环境变化更有着高度的敏感性。以青海湖河源湿地湿地冻融退化下的土壤微生物为研究对象, 基于16s rRNA及18s rDNA测序探究冻融退化对土壤微生物群落结构及多样性的影响。结果显示: 青海湖高寒湿地有效序列注释得到1583个细菌操作分类单元以及80个真菌操作分类单元。冻融退化并未改变土壤微生物的优势菌群: 细菌群落中的优势菌群均为变形菌门 (Proteobacteria) 及酸杆菌门 (Acidobacteria), 已知的真菌菌群中相对丰度最高的均为子囊菌门 (Ascomycota)。冻融退化使0—10 cm、10—20 cm的细菌群落结构趋于相似, 真菌群落中德福里斯孢属 ()、担孢酵母属 () 的相对丰度显著减少。冻融退化显著影响土壤微生物的多样性, 降低了细菌群落的ACE指数、Chao1指数、Simpson指数以及真菌群落的ACE指数、Chao1指数、Shannon指数, 细菌群落的Shannon指数及真菌群落的Simpson指数有所增加。本研究表明, 冻融退化对土壤微生物的群落结构、多样性均有影响, 为冻土退化下生态系统作用机制的探究提供参考。
湿地; 冻融退化; 土壤微生物; 高通量测序
0 前言
微生物因其具有类群丰富且多样的特征, 成为土壤生物体不可或缺的一部分。它主要作用于土壤生态系统的功能和调控方面, 如植物营养的释放与再吸收、分解植物凋落物、碳氮循环等[1]。在湿地生态循环系统中, 微生物更是作用于生态系统内外的诸多方面, 对生态系统内部而言,它作用于物质循环、能量流动和维持系统稳定性等方面, 对外部而言, 它在废物处理、生态修复、气体调节和生物多样性保护方面也发挥着重要作用[2-3]。现有研究表明, 土壤微生物对微环境的高敏感性可令其作为优于土壤总有机质的环境指示因子, 而其对外界环境的早期响应指标即生物量和群落结构的变化[4-6]。青藏高原的湿地面积高达13.19万平方千米[7], 青海湖位于青藏高原东北部, 其高寒湿地因环境条件的复杂性、地理位置的独特性导致生态系统脆弱而敏感[8-9], 因而对湿地的微生物特征的研究极其重要。
冻融作用对土壤微生物的生命活动、群落结构、多样性以及生物量都有影响[10-15], 青藏高原作为平均海拔超过4000米的高海拔地区, 冻融现象分布广泛[16]。多年冻土面积为106万平方千米, 季节冻土面积更是达到了146万平方千米[17-18]。IPCC最新报道指出, 截止到本世纪末, 全球地表平均温度上升了约1.5-2.0 ℃, 从2018-2100年全球气温将上升0.8—4.3 ℃[19]。全球变暖的大背景下, 青藏高原冻融交替将更加频繁, 大量冻土可能面临进一步退化。但冻融退化下的微生物群落结构以及多样性的变化还鲜有报道。本次研究旨在探究高寒湿地冻融退化下的土壤微生物群落结构以及多样性的变化, 以期进一步了解湿地生态系统作用机制, 更深层次的剖析湿地生态系统中生物与环境因子之间的偶联关系。
1 研究区概况
瓦颜山观测站 (37°43′38.03′′—37°46′20′′N、100°01′58.87′′—100°05′04. 17′′E) 位于青海湖北岸距刚察县52 km的沙柳河上游支流源头处, 为典型的河源湿地。该区的平均海拔为3720—3850 m, 属高原大陆性气候, 多年平均温度为-3.31 ℃, 日均最高气温为11.87 ℃, 最低为-19.73 ℃, 年降水量为420.37 mm, 且该区为高寒草甸, 植被以小嵩草 () 为主。
2 材料与方法
2.1 土壤样品的采集
于2019年8月进行土壤样品的采集, 本次实验设置三个重复, 在自然状态 (Wck) 未退化土壤以及冻融退化 (WD) 区分别每隔0.5 m围三个样方, 即Wck1、Wck2、Wck3、WD1、WD2、WD3。用直径为4.5 cm土钻在每个样方采用五点法取样, 取0—20 cm土层土样(0—10 cm、10—20 cm), 同层土壤混匀、过筛, 装到10ml的EP管中。最终标记为Wck1-1、Wck1-2、Wck2-1、Wck2-2、Wck3-1、Wck3-2、WD1-1、WD1-2、WD2-1、WD2-2、WD3-1、WD3-2, 置于液氮罐用于北京组学生物科技有限公司的高通量测序。以ck1、ck2、D1、D2表示不同分组的不同土层。
2.2 研究方法
2.2.1 土壤微生物群落结构及多样性测定
提取样品的总基因组DNA后, 选择细菌的两个高变区(V3+V4区)以及真菌的ITS区(ITS1, ITS2)通过保守区设计引物进行PCR扩增, 再对V3、V4区以及ITS区进行高通量测序分析。首先采用Mio-bio公司的土壤微生物总DNA提取试剂盒 (PowerSoil DNA Isolation Kit) 对DNA进行抽提, 琼脂糖凝胶电泳检测DNA质量, -80℃保存合格DNA, 用于后续的PCR扩增过程。再对细菌16s RNA的V3+V4区以及真菌的ITS区域进行扩增, 正向引物分别为5’-CCTAYGGGRBGCASCAG-3’和5’-CTTGGTCATTTAGAGGAAGTAA-3’, 反向引物分别为5’- GGACTACNNGGGTATCTAAT-3’和5’-GCTGCGTTCTTCATCGATGC-3’。采用50 μL反应体系进行PCR扩增, 扩增程序为: 预变性1 min(98 ℃), 进行(98 ℃, 10 s; 50 ℃, 30 s; 72 ℃, 30 s)的30个循环; 72 ℃, 5 min, 10 ℃保温。PCR扩增后再次用琼脂糖凝胶电泳对产物质量进行检测。
2.2.2 数据分析处理
PCR扩增产物经纯化、定量、均一化形成测序文库, 质检后Illumina 测序平台进行文库测序, 原始文件由碱基识别(Base Calling)转化得到原始测序序列(Sequenced Reads)。FLASH v1. 2. 7软件[20]进行PE reads 拼接得到原始Tags数据(Raw Tags), Trimmomatic v0. 33软件[21]、UCHIME v4. 2软件[22]对Raw Tags过滤并去除嵌合体得到Effective Tags, QIIME(Version 1.9.1)[23]在序列的相似性高于97%的程度上分别基于Silva(细菌)[24]和 UNITE(真菌)[25]聚类得到OTU数目以及韦恩图, 同时生成不同分类水平的物种丰度表。利用 R 语言工具绘制群落结构图并进行Heatmap聚类分析, 采用Mothur软件[26]评估α-多样性, Qiime软件进行β-多样性分析, Circos绘制物种丰度与样品相互关系图。SPSS 21.0进行ANOVA单因素分析, 对组间及组内数据作显著性检验。Excel 2018进行统计图的绘制。
3 结果与分析
3.1 土壤微生物样品稀释曲线
稀释曲线是基于OTU对样品进行质控过滤, 验证样品覆盖度的合理性。绝大多数样品的稀疏曲线为平缓趋势(图1), 表明细菌和真菌的测序数量使得OTU已经达到了饱和, 即再次增加测序深度后, OTU的数量不会产生波动。
3.2 冻融退化下土壤微生物群落结构变化
高通量测序得到24个不同门的细菌类群, 可明确所测样品中中变形菌门(Proteobacteria)、酸杆菌门(Acidobacteria)均为优势种群, 两菌群丰度占比分别为51%和20%, 细菌Circos图(图2a)中, WD与Wck的优势菌群具有一致性。不同组在绿弯菌门 (Chloroflexi)、芽单胞菌门 (Gemmatimonadetes)、匿杆菌门 (Latescibacteria) 及硝棘菌门 (Nitrospinae) 四个菌群具有极显著差异(<0.01), Nitrospirae门和Patescibacteria门具有显著差异(<0.05)。分别对0—10 cm和10—20 cm同层细菌群落结构进行ANOVA分析, ck1和D1的细菌群落结构在五个菌群中具有极显著差异(<0.01), 即Chloroflexi门、Entotheonellaeota门、Gemmatimonadetes门、Latescibacteria门和Nitrospinae门, 在Acidobacteria门也存在显著差异(<0.05)。ck2和D2的细菌群落中, 六大菌群具有极显著差异(<0.01), 包括Gemmatimonadetes门、Latescibacteria门、Nitrospirae门、Planctomycetes门、Tenericutes门及Zixibacteria门, 而Firmicutes门、Modulibacteria门、Nitrospinae门、WPS-2门和WS2门差异性显著(<0.05)。不同土层的细菌群落结构分析中, ck1的细菌群落以变形菌门为最具优势菌群, 次优势菌群为酸杆菌门, ck2的优势菌群与其一致, Wck的不同土层ANOVA分析表明, Wck中的Entotheonellaeota门、Planctomycetes门、Tenericutes门、WPS-2门、Zixibacteria门具有极显著差异(<0.01), Elusimicrobia门、Firmicutes门及Modulibacteria门具有显著差异(<0.05)。WD不同土壤层次的ANOVA分析显示, 两者仅在Actinobacteria门和Bacteroidetes门差异显著(<0.05)。细菌门水平相对丰度大于0.1%的有13个菌群, 百分比堆积条形图显示(图3a), 自然与冻融退化下不同土层的细菌群落结构中各菌群所占的比例各异, ck1在Verrucomicrobia门占比最高(56.56%), ck2在Zixibacteria门(99.74%)及Firmicutes门(95.91%)有绝对的优势, D1与D2皆在Latescibacteria门占比较高, 分别占比34.44%、45.61%。一定程度上反应了Wck组内不同层的差异以及WD组内的相似性, 表明自然状态下不同土层的细菌群落结构差异较大。

图1 细菌 (a) 及真菌 (b) 稀释性曲线
Figure 1 Dilution curves of bacteria (a) and fungi (b)
真菌门水平绝大部分都为未知类群, 占比达到了79%, 已知菌群为子囊菌门(Ascomycota)和担子菌门(Basidiomycota), 显著性检验表明门水平下的真菌菌群群落结构差异不显著。真菌属除未知菌群外测得25个菌群, 其属水平Circos图(图2b)显示, Wck中克劳氏菌属()、丝盖伞属()丰度值相对较高, 占比分别为6%和4%, 而WD中链格孢属()及球旋霉属()相对丰度值较高。ANOVA分析结果显示, Wck与WD的属有极显著差异(<0.01),属及未知菌群具有显著差异(< 0.05)。分别对0-10cm、10-20cm的WD和Wck的真菌群落结构进行同层ANOVA显著性分析, ck1与D1的真菌群落结构没有显著性差异, ck2与D2在属有显著性差异(<0.05)。不同土层的真菌群落结构中, ck1与ck2、D1与D2间的真菌菌群结构差异不显著。真菌属相对丰度大于0.1%除未知菌群的真菌菌群为19个, 其百分比堆积条图(图3b)清晰展示了ck1、ck2、D1、D2在不同真菌属所占的比例大小, ck1在属占比最高(93.65%), ck2则在属(94.42%)、属(86.92%)占比较高, D1的属(82.00%)及属(81.04%)占比最高, D2占比最高的属则为属(99.32%), 各组真菌菌群结构存在差异, 但差异并不显著。以上结果表明, 冻融退化对真菌群落结构的影响较小,属、属所占比例明显减少, 未知菌群的比例增大。

图2 细菌 (a) 及真菌 (b) Circos图
Figure 2 Circos diagram of bacteria (a) and fungus (b)

图3 细菌 (a) 及真菌 (b) 百分比条图
Figure 3 Bar chart of percentage of bacteria (a) and fungus (b)
3.3 冻融退化下土壤微生物群落多样性变化
3.3.1 土壤微生物多样性分析
高通量测序结果显示, 细菌OTU数为1583个, 真菌OTU数为80个。韦恩图结果显示, 冻融(WD)退化的微生物种类完全从属于自然状态(Wck)的微生物种类。其中Wck的细菌OTU数量比WD多121个, 真菌OTU则多6个。表明冻融对微生物种类影响较小, 可能对微生物均匀度影响较大。细菌群落的多样性指数中, 丰富度指数即ACE指数 (1371—1518) 和Chao1指数 (1387—1537), 均匀度指数为Simpson指数 (0.0056—0.1724) 以及Shannon多样性指数 (3.8134—6.2080), 真菌群落多样性指数中, ACE指数范围为32—72, Chao1指数在36—74之间, Simpson指数在0.0947—0.8781范围, Shannon多样性指数范围为0.3791—2.9244。与WD比较, Wck土壤细菌群落平均ACE指数(1446.391), 平均Chao1指数 (1457.942), 平均Simpson指数(0.034733)相对较高, 平均Shannon指数 (5.672415) 较低。Wck土壤真菌群落平均ACE指数 (67.88167), 平均Chao1指数 (68.325), 平均Shannon指数 (2.338095) 相对较高, 平均Simpson指数(0.229947)较低。单因素分析结果显示, Wck与WD的土壤细菌群落多样性没有显著差异, 且土壤真菌的均匀度指数也没有显著差异, 但其丰富度指数具有极显著差异 (0.01)。分别对比同层Wck、WD的土壤微生物多样性, ck1与D1细菌的丰富度指数及均匀度指数均存在显著差异, 除ACE指数(<0.05)外, 其余指数显示差异极显著 (<0.01); 但ck1与D1真菌仅丰富度指数差异显著 (<0.05), 表明0—10 cm的真菌均匀度并未受到冻融退化的显著影响。ck2和D2细菌的ACE指数、Chao1指数具有极显著差异 (<0.01), 即10—20 cm的细菌丰富度受到了显著影响。不同层的微生物多样性指数的ANOVA分析表明, ck1与ck2的细菌在丰富度指数上差异极显著 (<0.01), D1与D2细菌的均匀度指数存在显著差异 (<0.05), 且Shannon指数差异极显著 (<0.01)。以上结果表明, 冻融退化显著影响了土壤真菌的丰富度指数, 对0—10 cm的土壤真菌丰富度的影响更为明显; 对不同土层细菌的多样性指数均有显著影响, 尤其对土壤细菌的丰富度指数的影响较大, 但冻融退化对土壤微生物多样性的影响并未在整体上体现出细菌多样性指数的显著变化。不同土层微生物多样性差异在一定程度上发生了中和, 使整体未能表现出统计学上的显著差异。
3.3.2 土壤微生物多样性差异分析
土壤细菌群落多样性的相似性较大(图4a), PCA分析中, 主成分1和主成分2的解释程度为95.91%, ck与D较为聚集, 即WD组内差异较小, Wck组内差异明显。差异性热图更为清晰的展示了样品间的差异性(图4c), Wck1-2、Wck2-2、Wck3-2单独聚为一支, Wck1-1、Wck2-1、Wck3-1与WD组样品聚为一支, 即ck1与WD的相似性更大, 表明冻融退化对不同土层的细菌多样性产生的影响使得10—20 cm土层的细菌多样性趋于0—10 cm土层的细菌多样性。土壤真菌群落多样性差异较为显著(图4b), 主成分1、主成分2解释程度为75.75%, 前三个主成分的解释程度则达到86.89%, ck与D离散程度较高, 表明Wck与WD的组间差异较大, 但Wck、WD组内的差异性较小。冻融对于真菌群落多样性影响可能更为显著, 这与土壤微生物多样性指数的差异分析结果一致。差异分析热图显示(图4d), 不同样品间都存在差异, 表明不同采样点以及不同土层对样品有一定程度的影响, 表层土壤和深层土壤的真菌多样性都受冻融退化的影响。Adonis/ANOSIM非参数检验方法表明差异具有显著性(<0.05), 进一步提供了土壤真菌多样性具有差异的佐证。
4 讨论
4.1 冻融退化下高寒湿地土壤微生物群落结构特征变化
冻融退化并未影响高寒湿地细菌群落结构中的优势菌群, 这与湿地土壤常见的细菌门的优势菌群差异不大[27-29], 表明各湿地生态系统中起主要作用的功能菌群有极大程度的一致性。但冻融退化使Chloroflexi门、Gemmatimonadetes门、Nitrospinae门、Nitrospirae门的丰度显著增加, 同时降低了Latescibacteria门、Patescibacteria门的丰度, 即细菌群落结构发生显著改变。Sharma等的冻融对土壤微生物群落结构的研究也表明能观察到土壤微生物群落结构的变化[14]。Wck与WD的同层细菌群落结构分析中, 冻融退化使0—10 cm及10—20 cm土层的芽单胞菌门、匿杆菌门、硝棘菌门都发生了显著变化, 表明这三个细菌门可能对冻融退化更为敏感。芽单胞菌门和匿杆菌门均为革兰氏阴性菌, 而硝棘菌门则为一类亚硝酸盐氧化细菌, 在碳固定上起着重要作用[30], 表明冻融退化下的土壤微生物可能具有更大的碳固定潜能。不同层的组内分析表明, ck1的细菌群落结构与ck2的细菌群落结构差异较大, 与Fierer等人研究的不同土层的微生物群落结构存在明显差异具有一致性[31]。而WD中不同土层的细菌结构仅放线菌门和拟杆菌门存在显著差异, 表明冻融退化下不同土层细菌群落结构的相似度较高。冻融退化改变了部分菌群的丰度, 显著影响了细菌的群落结构, 同时使不同土层的细菌结构趋于相似。
表1 土壤微生物丰富度及多样性指数
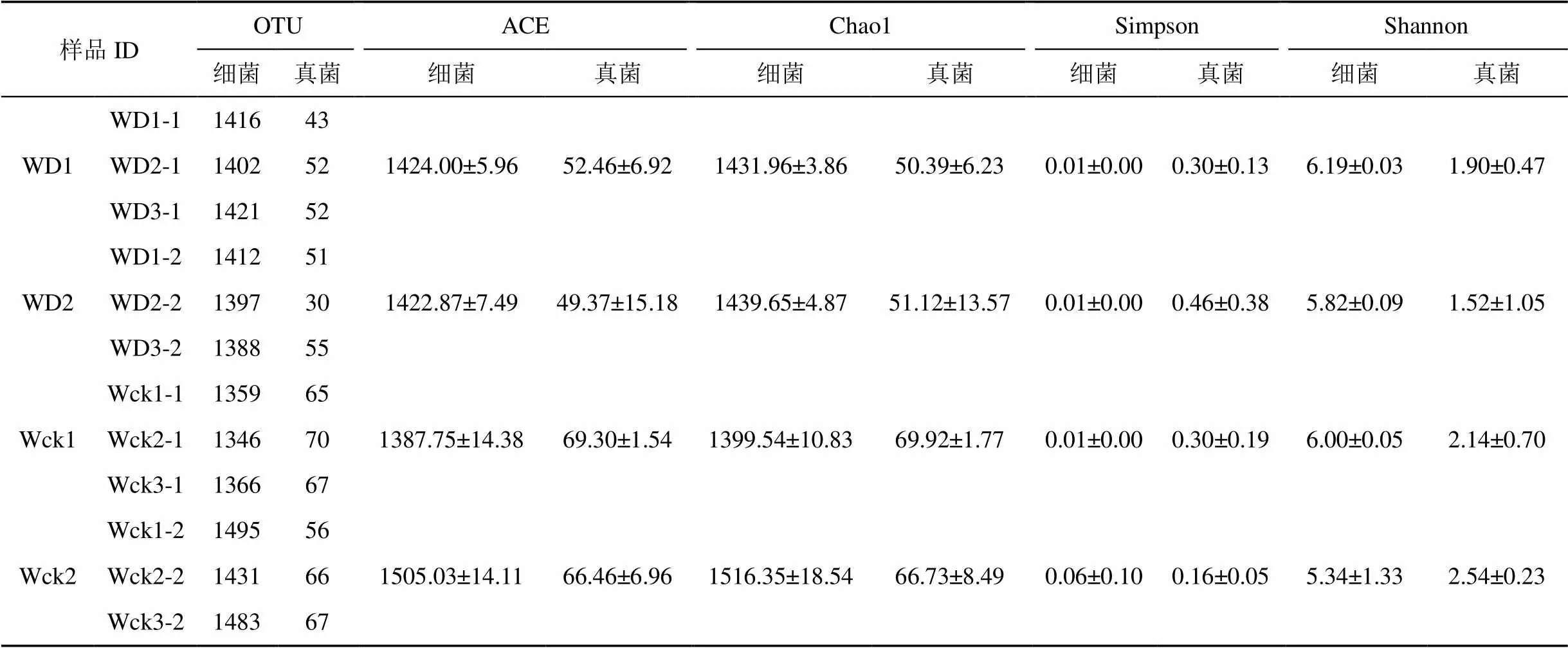
Table.1 Soil microbial richness and diversity index

图4 细菌及真菌多样性差异分析
Figure 4 Diversity analysis of bacteria and fungi
冻融退化对高寒湿地真菌群落结构存在影响, 但并不显著, 真菌门水平下的方差分析结果表明, Wck与WD不存在显著性差异。属水平下分析结果表明在德福里斯孢属)、担孢酵母属 () 的组间差异显著, 德福里斯孢属为无性型耐热真菌[32], 其在ck2与D2同层分析也表现出显著差异, 担孢酵母属则属担子菌门。而未知真菌菌群丰度的增加表明了其在冻融退化下具有一定的优势。Wck、WD的组内单因素分析没有显示出显著差异, 表明不同土层土壤真菌的群落结构相似, 真菌对冻融退化的耐受能力较强[10]。冻融对真菌群落结构的影响在于使已知菌群中的两个属丰度降低以及未知菌群丰度增大, 但对真菌群落整体结构影响不大。真菌群落结构中未知菌群的群落及功能有必要进一步剖析, 以便于对湿地生态系统机制进行深层探究。
4.2 冻融退化对高寒湿地微生物多样性的影响
冻融退化对高寒湿地土壤微生物的多样性指数影响显著。其Alpha多样性指数存在一定的差异, 且细菌的种类和数量有着绝对的优势, 赵先丽等对湿地土壤微生物的研究也表明了细菌比真菌有更强的生长繁殖能力[33]。冻融退化下真菌群落多样性的丰富度指数差异极显著, 且WD 冻融退化改变了青海湖湿地土壤微生物的群落结构以及多样性。土壤细菌群落的芽单胞菌门、硝棘菌门相对丰度显著增加, 匿杆菌门相对丰度显著减少, 优势菌群并未变化; 整体上细菌群落的多样性指数呈降低趋势, 但不同层次的细菌受到的影响相反, 在一定程度上中和了差异, 其变化并不显著。冻融退化对土壤真菌群落结构的影响较小, 德福里斯孢属和担孢酵母属的相对丰度降低, 表明部分真菌菌群可以反应环境的变化情况; 真菌群落多样性指数整体呈降低趋势, 其丰富度指数降低尤为显著。冻融退化改变了土壤微生物的群落结构, 降低了土壤微生物群落的多样性。本文阐明了高寒湿地冻融现象导致的土壤退化下土壤微生物群落特征的变化, 但“土壤-微生物-根际”存在着相互关系, 冻融退化对土壤的影响并不只是土壤微生物这一单因素, 因此冻融退化对湿地土壤的理化性质以及根际植物等的影响有待进一步的探究, 以期对冻融退化下湿地生态系统作用机制的深层次剖析。 [1] 卫云燕, 尹华军, 刘庆, 等. 气候变暖背景下森林土壤碳循环研究进展[J]. 应用与环境生物学报, 2009, 15(6): 888–894. [2] 邵鹏帅, 解宏图, 鲍雪莲, 等. 森林次生演替和土壤层次对微生物群落结构的影响[J]. 生态学杂志, 2020, 39(05): 1455–1463. [3] 刘银银, 李峰, 孙庆业, 等. 湿地生态系统土壤微生物研究进展[J]. 应用与环境生物学报, 2013, 19(03): 547–552. [4] ZELLES L, BAI Q Y, BECK T, et al. Signature fatty acids in phospholipids and lipopolysaccharides as indicators of microbial biomass and community structure in agricultural soils[J]. Soil Biology and Biochemistry, 1992, 24(4): 317– 323. [5] CHEN Jian, XIE Huijun, ZHUANG Xuliang, et al. Substrate-induced changes in microbial community-level physiological profiles and their application to discriminate soil microbial communities[J]. Journal of Environmental Sciences, 2008, 20(6): 725–731. [6] PARMAR T K, RAWTANI D, AGRAWAL Y K. Bioindicators: the natural indicator of environmental pollution[J]. Frontiers in Life Science, 2016, 9(2): 110–118. [7] 邢宇, 姜琦刚, 李文庆, 等. 青藏高原湿地景观空间格局的变化[J]. 生态环境学报, 2009, 18(3): 1010–1015. [8] 徐翠, 张林波, 杜加强, 等. 三江源区高寒草甸退化对土壤水源涵养功能的影响[J]. 生态学报, 2013, 33(8): 2388– 2399. [9] 曹生奎, 曹广超, 陈克龙, 等. 青海湖高寒湿地生态系统服务价值动态[J]. 中国沙漠, 2014, 34(5): 1402–1409. [10] 陈泓硕, 马大龙, 姜雪薇, 等. 季节性冻融对扎龙湿地土壤微生物群落结构和胞外酶活性的影响[J]. 环境科学学报, 2020, 40(4): 1443–1451. [11] 张超凡, 盛连喜, 宫超, 等. 冻融作用对我国东北湿地土壤碳排放与土壤微生物的影响[J]. 生态学杂志, 2018, 37(2): 304–311. [12] 谌亚, 贾雨桥, 王欣宇, 等. 冻融循环对亚高山森林土壤微生物生物量及群落结构的影响[J]. 生态环境学报, 2019, 28(4): 686–694. [13] 刘利. 季节性冻融对亚高山/高山森林土壤微生物多样性的影响[D]. 四川农业大学, 2010. [14] SHARMA S, SZELE Z, SCHILLING R, et al. Influence of Freeze-Thaw Stress on the Structure and Function of Microbial Communities and Denitrifying Populations in Soil[J]. Applied and Environmental Microbiology, 2006, 72(3): 2148–2154. [15] SCHIMEL J, BALSER T C, WALLENSTEIN M. Microbial stress-response physiology and ITS implications for ecosystem function[J]. Ecology. 2007, 88(6): 1386–1394. [16] 徐欢, 王芳芳, 李婷, 等. 冻融交替对土壤氮素循环关键过程的影响与机制研究进展[J]. 生态学报, 2020, 40(10): 3168-3182. [17] 戴黎聪, 柯浔, 张法伟, 等. 青藏高原季节冻土区土壤冻融过程水热耦合特征[J]. 冰川冻土, 2020, 42(2): 390-398. [18] 赵林, 胡国杰, 邹德富, 等. 青藏高原多年冻土变化对水文过程的影响[J]. 中国科学院院刊, 2019, 34(11): 1233– 1246. [19] IPCC. Climate change 2013: the physical science basis: contribution of working group I to the fifthassessment report of the intergovernmental panel on climate change [M]. Cambridge: Cambridge University Press, 2013. [20] MAGOC T, SALZBERG SL. FLASH: fast length adjustment of short reads to improve genome assemblies[J]. Bioinformatics. 2011, 27(21): 2957–2963. [21] BOLGER A M, LOHSE M, USADEL B. Trimmomatic: a flexible trimmer for Illumina sequence data[J]. Bioinformatics 2014, 30(15), 2114–2120. [22] EDGAR R C, HAAS B J, CLEMENTE J C, et al. UCHIME: improves sensitivity and speed of chimera detection[J]. Bioinformatics 2011, 27(16): 2194–2200. [23] EDGAR R C. Search and clustering orders of magnitude faster than BLAST[J]. Bioinformatics 2010, 26(19): 2460– 2461. [24] QUAST C, PRUESSE E, YILMAZ P, et al. The SILVA ribosomal RNA gene database project: improved data processing and web-based tools[J]. Nucleic acids research 2013, 41(1): 590–596. [25] KOLJALG U, NILSSON RH, ABARENKOV K, et al. Towards a unified paradigm for sequence‐based identification of fungi[J]. Molecular Ecology 2013, 22(21): 5271–5277. [26] SCHLOSS P D, WESTCOTT S L, RYABIN T, et al. Introducing mothur: open-source, platform-independent, community-supported software for describing and comparing microbial communities[J]. Applied and environmental microbiology, 2009, 75(23): 7537–7541. [27] 鲁青原. 辽河三角洲滨海湿地微生物群落组成及其环境意义[D]. 北京: 中国地质大学(北京), 2016. [28] 纪垚. 七星河湿地微生物群落结构与环境因子响应研究[D]. 哈尔滨: 哈尔滨师范大学, 2017. [29] AN Jiaxing, LIU Chi, WANG Qian, et al. Soil bacterial community structure in Chinese wetlands[J]. Geoderma, 2019, 337: 290–299. [30] PACHIADAKI M G, SINTES E, BERGAUER K, et al. Stepanauskas, R. Major role of nitrite-oxidizing bacteria in dark ocean carbon fixation[J]. Science, 2017, 358(6366): 1046–1051. [31] FIERER N, SCHIMEL J P, HOLDEN P A. Variations in microbial community composition through two soil depth profiles[J]. Soil Biology and Biochemistry, 2003, 35(1): 167–176. [32] 李长林. 青岛潮间带藻生与渤海沉积物真菌多样性研究[D]. 青岛: 中国海洋大学, 2013. [33] 赵先丽, 周广胜, 周莉, 等. 盘锦芦苇湿地土壤微生物数量研究[J]. 土壤通报, 2008, (6): 1376–1379. [34] 陆梅, 孙向阳, 田昆, 等. 纳帕海高原湿地不同退化阶段土壤真菌群落结构特征[J]. 北京林业大学学报, 2018, 40(3): 55–65. [35] YERGEAU E, KOWALCHUK G A. Responses of Antarctic soil microbial communities and associated functions to temperature and freeze-thaw cycle frequency[J]. Environmental Microbiology, 2008, 10(9): 2223–2235. [36] 李梓正, 朱立博, 林叶春, 等. 呼伦贝尔草原不同退化梯度土壤细菌多样性季节变化[J]. 生态学报, 2010, 30(11): 2883–2889. [37] 李皓, 董建华, 董建明, 等. 退化与正常山核桃林土壤细菌多样性比较研究[J]. 浙江林业科技, 2017, 37(5): 42– 47. [38] 陈末, 朱新萍, 蒋靖佰伦, 等. 冻融期巴音布鲁克高寒湿地土壤细菌群落变化及其响应机制[J]. 农业环境科学学报, 2020, 39(1): 134–142. [39] KUMAR N, GROGAN P, CHU H, et al. The Effect of Freeze-Thaw Conditions on Arctic Soil Bacterial Communities[J]. Biology, 2013, 2(1): 356–377. [40] 宋贤冲, 曹继钊, 唐健, 等. 猫儿山常绿阔叶林不同土层土壤微生物群落功能多样性[J]. 生态科学, 2015, 34(6): 93–99. Changes of microbial community characteristics in freezing-thaw degradation of alpine wetland ZHANG Ni1,2, CHEN Kelong2,3,*, WANG Hengsheng4, YANG Yang1,2 1. College of Life Science Qinghai Normal University, Xining 810008, China 2. Key Laboratory of Surface Processes and Ecological Conservation, Ministry of Education, Qinghai Normal University, Qinghai Province, Xining 810008, China 3. Science and Technology Department, Qinghai Normal University, Xining 810008, China 4. School of Life Science, Hefei Normal University, Hefei 230061, China Under the background of global warming, a large amount of permafrost will be further degraded, and the freezing-thawing process spreads throughout the Qinghai-Tibet plateau, and soil microorganisms are highly sensitive to environmental changes. So in this paper, soil microorganisms under freezing-thawing degradation in the wetland at the source of a river of Qinghai lake were taken as the research object. The effects of freezing-thawing degradation on the structure and diversity of soil microbial community were explored based on 16s rRNA and 18s rDNA sequencing. The results showed that 1583 bacterial and 80 fungal operational taxa were obtained from the effective sequence annotation of Qinghai lake alpine wetland. Freezing-thawing degradation did not change the dominant flora of soil microorganisms: the dominant flora in the bacterial community were all Proteobacteria and Acidobacteria, and Ascomycota had the highest relative abundance among the known fungal flora. Freezing-thawing degradation resulted in the similarity of the structure of the bacterial community of 0-10 cm and 10-20 cm, and the relative abundance of the fungal community of Devriesia and Erythrobasidium was significantly reduced. Moreover, freezing-thawing degradation significantly affected the turbidities of soil microorganisms, and reduced the ACE index, Chao1 index and Simpson index of bacterial community, as well as the ACE index, Chao1 index and Shannon index of fungal community, while the Shannon index of bacterial community and Simpson index of fungal community were increased. This study shows that freezing-thawing degradation has an impact on the community structure and diversity of soil microorganisms, providing a reference for the study of the mechanism of ecosystem action under the degradation of frozen soil. wetland; freezing-thawing degradation; soil microorganism; high throughput sequencing 10.14108/j.cnki.1008-8873.2022.02.003 Q938.1+1 A 1008-8873(2022)02-020-09 2020-06-11; 2020-07-03 国家自然科学基金项目(41661023); 国家科技支撑计划项目(2017YFC0403601); 第二次青藏高原综合科学考察研究(2019QZKK0405); 青海省科技厅项目(2017-ZJ-782) 章妮(1997—), 女, 湖北荆州人, 硕士研究生, 主要从事湿地生态学研究, E-mail:1581146264@qq.com 通信作者:陈克龙, 男, 二级教授, 博士生导师, 主要从事生物地理与湿地生态研究, E-mail:ckl7813@163.com 章妮, 陈克龙, 王恒生, 等. 高寒湿地冻融退化下微生物群落特征变化[J]. 生态科学, 2022, 41(2): 20–28. ZHANG Ni, CHEN Kelong, WANG Hengsheng,et al. Changes of microbial community characteristics in freezing-thaw degradation of alpine wetland [J]. Ecological Science, 2022, 41(2): 20–28.5 结论

