谷子HSP90基因家族鉴定及干旱胁迫下表达分析
顾鹏鹏 马鑫磊 姚锐 高慧 林小虎

摘要:热激蛋白90 (heat shock proteins 90,HSP90) 是广泛存在于植物中的一种高度保守的细胞伴侣蛋白家族,参与调节和维持各种蛋白质的构象,在植物的生物和非生物胁迫应激反应中起着重要作用。为探讨谷子HSP90基因家族(SiHSP90s)在干旱胁迫下的潜在抗旱功能,对SiHSP90s进行鉴定,并分析其在干旱胁迫下的表达情况。共鉴定出9个SiHSP90基因分别位于谷子5条染色体上。理化性质分析显示SiHSP90s蛋白长度为403~817个氨基酸,分子量为46.1~92.7 ku。系统进化分析显示SiHSP90s分为Ⅰ型和Ⅱ型2个亚家族。基因结构与蛋白基序分析显示,Ⅰ型和Ⅱ型亚族成员的外显子数量分别在2~3个和12~20个之间,且均含有保守的HATPase_c(PF02518)和HSP90(PF00183)结构域。顺式元件分析表明,SiHSP90s基因可以应对不同的非生物胁迫。qRT-PCR结果表明,干旱胁迫下SiHSP90.2和SiHSP90.3转录水平极显著上调。这些结果为阐明SiHSP90家族的进化关系以及进一步研究SiHSP90基因的功能特性提供了有价值的信息。
关键词:热激蛋白90;基因家族;干旱胁迫;表达分析;谷子
中图分类号: S515.01 文献标志码: A
文章编号:1002-1302(2022)06-0045-08
收稿日期:2021-07-06
基金项目:国家“十三五”重点研发计划(编号:2019YFD1001701-2);河北省现代农业产业技术体系创新团队(杂粮杂豆)项目(编号:HBCT2018070404);河北省自然科学基金(编号:C2019407001);河北省高等学校科学技术研究项目(编号:QN2020154);河北省在读研究生创新能力培养资助项目(编号:CXZZSS2021152)。
作者简介:顾鹏鹏(1994—),男,山东滨州人,硕士研究生,主要从事植物分子遗传与基因工程研究。E-mail:1006555310@qq.com。
通信作者:林小虎,博士,教授,主要从事植物遗传学研究。E-mail:xiaohulin2008@163.com。
作物经常遭受各种非生物胁迫,包括干旱[1]、盐碱[2]、低温[3]和高热[4]等,其会对作物的产量和品质产生严重影响。作物在长期进化过程中形成了完善的抵御非生物胁迫的应答机制。其中热激蛋白(heat shock protein,HSP)被证实具有保护细胞抵御有害刺激的功能[5],其按分子量大小可分为小分子HSP(sHSP)、HSP60、HSP70、HSP90和HSP100家族[6]。其中HSP90是一种腺苷三磷酸(ATP)依赖的分子伴侣蛋白,在原核和高等真核生物中均高度保守[7]。HSP90主要由3个高度保守的结构域组成:与底物结合的约25 ku的C端结构域、35 ku的中间结构域和与ATP结合的12 ku的N端结构域[8-9]。在真核生物中,HSP90的N端结构域和中间结构域之间有一个长的、高电荷的连接结构域[10]。HSP90中与ATP结合的区域通常处于闭合状态[11],N端ATP酶活性较低[12]。
HSP90具有稳定蛋白的能力;在变性条件下,HSP90能防止功能蛋白失稳聚集并促进非活性蛋白质复性[13]。在真菌和动物中,HSP90在胁迫信号转导中发挥广泛的作用,如类固醇激素受体、蛋白激酶和转录因子的折叠,以及激活底物来启动胁迫信号转导[14-15]。在植物中,HSP90的研究主要集中在进化分析和生理功能方面[16-17]。研究表明,HSP90与生长素、油菜素类固醇、细胞分裂素和茉莉酸等激素信号转导有直接的關系[18]。植物中HSP90不仅与多激素之间具有直接关系,同时与非生物胁迫响应密切相关。在拟南芥中,AtHSP90.2、AtHSP90.5和AtHSP90.7的过表达增强了植物对盐和干旱胁迫的敏感性,但对高浓度Ca2+的耐受性提高[19];在烟草中部分NtHSP90在非生物胁迫响应调控中起重要作用,例如NtHSP90-4、NtHSP90-5和NtHSP90-9在脱落酸(ABA)、干旱、盐、冷热胁迫的诱导下表达上调,而NtHSP90-6和NtHSP90-7不受其诱导[20]。最近在植物中进行的蛋白质组学和磷蛋白质组学[21-23]分析表明,在干旱和盐碱胁迫下都存在大量的HSP90蛋白,其可能参与了胁迫响应过程中的信号转导。
谷子(Setaria italica L.)是一种耐旱、耐贫瘠的粮食作物,广泛种植于世界干旱和半干旱地区[24]。因谷子具有C4光合特性;对干旱、高温、盐碱具有显著的耐性;且与多种生物能源禾本科作物的遗传关系密切,被认为是研究C4光合特性、非生物胁迫耐性和生物燃料特性的模式作物[25]。到目前为止,关于谷子HSP90基因家族成员的鉴定以及在干旱胁迫下的表达分析未见报道。本研究基于已测序的谷子基因组[26],对谷子HSP90基因家族成员进行鉴定,并分析了干旱胁迫下HSP90家族成员的基因表达情况,为进一步探究HSP90的抗旱功能提供理论基础。
1 材料与方法
1.1 试验材料与培养条件
供试谷子品种为豫谷1号,种植于智能人工气候箱的育苗盆中,营养土、蛭石混合比例为2 ∶1。培养箱条件为温度26/20 ℃,光周期16/8 h,湿度65%。
1.2 谷子HSP90基因家族鉴定
利用HSP90保守结构域的隐马尔可夫模型文件(PF00183,http://pfam.xfam.org/),从谷子全基因组(https://phytozome.jgi.doe.gov/pz/portal.html#)筛选候选的HSP90基因家族成员。利用SMART (http://smart.embl.de/smart/set_mode.cgi?GENOMIC=1)、Pfam和NCBI-CDD(https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)三大数据库对候选的谷子HSP90基因家族成员进行验证。利用UniProtKB/Swiss-Prot(https://web.expasy.org/protparam/)对谷子HSP90基因家族成员理化性质进行分析。
1.3 谷子近缘植物HSP90基因家族成员鉴定及系统发育分析
编辑 perl 脚本,提取拟南芥、谷子(豫谷1号)、二穗短柄草、水稻、玉米、高粱、莱茵衣藻的HSP90基因的氨基酸序列,利用 ClustalW 软件对保守结构域进行序列比对,根据序列比对的结果以及拟南芥中HSP90基因的分类标准,将谷子中的 HSP90基因分为 2个亚型,同时根据保守结构域比对结果,利用 MEGA7构建了拟南芥、二穗短柄草、水稻、玉米、高粱、莱茵衣藻和谷子HSP90基因的 N-J 无根树,bootstrap值设为 1000。
1.4 谷子HSP90基因结构和保守结构域分析
利用 GSDS(http://gsds.gao-lab.org/)分析谷子中每个 HSP90基因 3′UTR、 5′UTR、内含子和外显子的组成,CDS 在基因组上的位置信息来自 JGI 数据库,利用在线软件MEME分析每个HSP90基因中存在的保守模体。
1.5 谷子HSP90基因家族染色体定位
根据谷子HSP90基因家族的基因号在Phytozome网站查询谷子各HSP90家族成员位置信息,利用MapChart软件绘制谷子HSP90基因家族染色体定位图。
1.6 谷子HSP90基因启动子区顺式作用元件分析
在Phytozome中下载谷子的全基因组序列和基因结构注释信息GFF3文件,通过Tbtools软件的Gtf/Gff3 Sequences Extractor功能提取所有基因CDS序列上游2 000 bp的序列,再通过Fasta Extractor提取HSP90基因家族成员的启动子区序列,提交至PlantCare(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)网站进行顺式作用元件预测。将得到的预测信息分类汇总,并通过Tbtools软件的Simple Biosequence Viewer将结果可视化。
1.7 干旱胁迫下谷子HSP90基因实时荧光定量PCR
对照组正常浇水保持土壤湿润;干旱组5叶期时停止浇水,待土壤含水率低于20%时取材。取材后液氮速冻并于-80 ℃储存备用。将储存于 -80 ℃ 的对照组和干旱组的谷子幼苗取全植株,提取RNA(TaKaRa MiniBEST Plant RNA Extraction Kit试剂盒)并反转录为cDNA(UEIris II RT-PCR System for First-Strand cDNA Synthesis)-20 ℃保存备用。对SiHSP90家族的所有成员编码序列设计实时荧光定量PCR引物(表1),产物片段大小限定在80~200 bp 之间。实时荧光定量PCR使用 BIO-RAD CFX connect TM定量PCR仪进行扩增,以谷子 EF-1a-2 作为内参。扩增体系10 μL,其中cDNA 0.5 μL,正反方向引物各0.3 μL,AugeGreenTM qPCR Master Mix 5 μL,ddH2O 3.9 μL。程序设定为95 ℃预变性2 min;95 ℃变性5 s,60 ℃退火和延伸30 s,共45个循环。试验设置3次生物学重复且每个样品设置3次技术重复。试验数据采用2-△△CT法计算各基因表达量。
2 结果与分析
2.1 谷子HSP90家族成员鉴定及蛋白理化性质分析
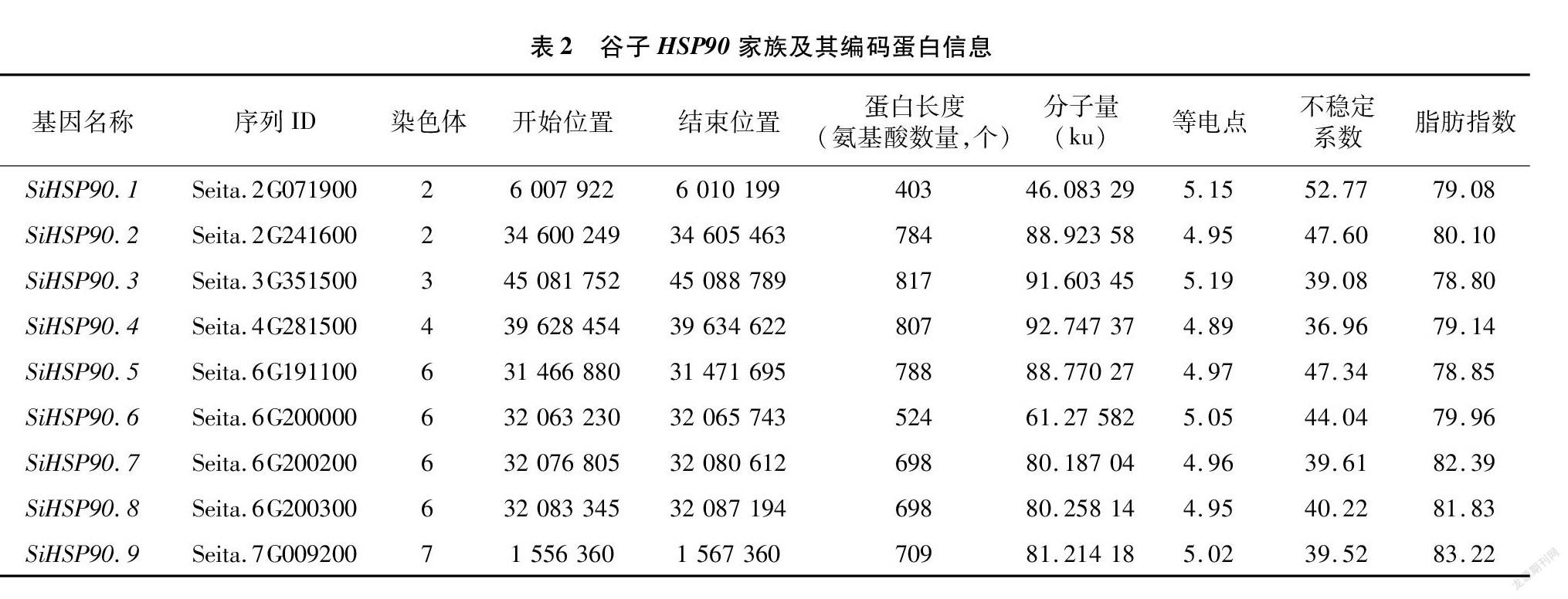
本研究利用隐马尔可夫模型(PF00183)在谷子中共鉴定得到9个编码HSP90的基因,分别命名为SiHSP90.1~SiHSP90.9(表2)。预测分析谷子HSP90蛋白序列的氨基酸数目、分子量、等电点等理化性质参数,结果显示SiHSP90家族蛋白的氨基酸残基数目在403~817个之間,其中SiHSP90.1氨基酸数目最少,SiHSP90.3氨基酸数目最多;蛋白分子量在46.083 29~92.747 37 ku之间,其中SiHSP90.4最大,SiHSP90.1最小;等电点分析显示,SiHSP90.1~SiHSP90.9均为酸性蛋白;不稳定系数分析表明SiHSP90.3、SiHSP90.4、SiHSP90.7、SiHSP90.9为稳定蛋白,SiHSP90.1、SiHSP90.2、SiHSP90.5、SiHSP90.6、SiHSP90.8为不稳定蛋白;脂肪指数分析显示SiHSP90蛋白家族成员的热稳定性均较强。
2.2 HSP90基因家族系统进化、基因结构和保守基序分析
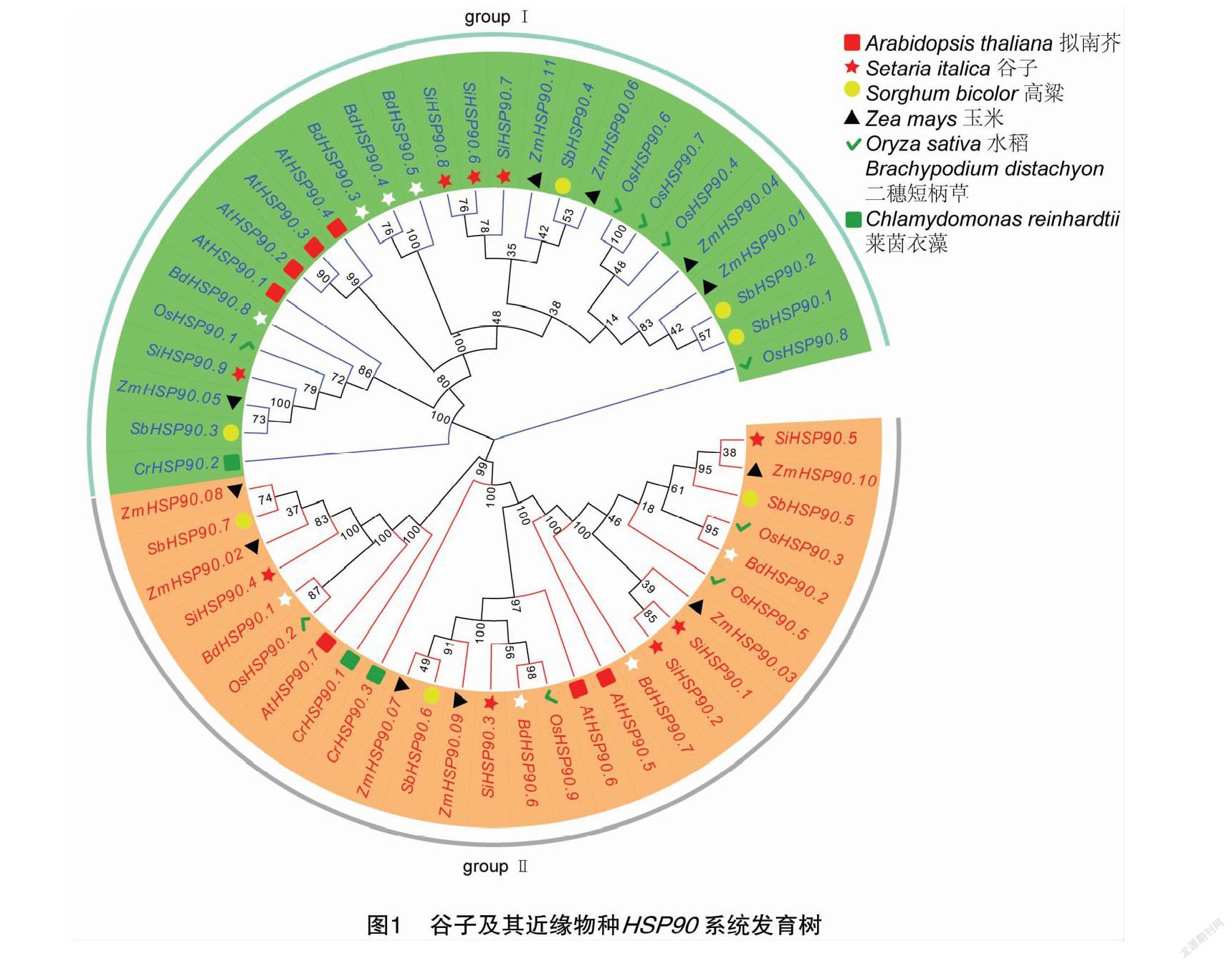
为了探究谷子HSP90基因的起源和进化,本研究分别鉴定莱茵衣藻、拟南芥、二穗短柄草、水稻、高粱、玉米和谷子7个代表物种的HSP90基因家族成员并构建系统进化树。在莱茵衣藻、拟南芥、二穗短柄草、水稻、高粱、玉米和谷子中分别鉴定出3、7、8、9、7、 11、 9个HSP90家族成员。根据进化树的分支将7个代表物种中的HSP90基因分为Ⅰ和Ⅱ 2个亚家族(图1)。在Ⅰ型亚族中莱茵衣藻、拟南芥、二穗短柄草、水稻、高粱、玉米和谷子分别鉴定出1、4、4、5、4、5、4个成员;在Ⅱ型亚族中莱茵衣藻、拟南芥、二穗短柄草、水稻、高粱、玉米和谷子分别鉴定出2、3、4、4、3、6、5个成员。本研究还发现了12个基因同源,占HSP90基因总数的22.22%。其中拟南芥1对(AtHSP90.2和AtHSP90.3),谷子2对(SiHSP90.1和SiHSP90.2,SiHSP90.6和SiHSP90.8),水稻1对(OsHSP90.6和OsHSP90.7),二穗短柄草1对(BdHSP90.3和BdHSP90.4),高粱1对(SbHSP90.1和SbHSP90.2)。
SiHSP90家族的基因结构和保守基序分析结果表明,Ⅰ型亚族成员的外显子数量较少,在2~3个之间;Ⅱ型亚族成员的外显子数量较多,在12~20个之间(图2)。在Ⅰ型亚族中,SiHSP90.9基因结构与其他成员存在较大区别,其含有一个较长的内含子序列,约占基因全长的70%。在Ⅱ型亚族中, SiHSP90.2和SiHSP90.5外显子和内含子排布最为相似;SiHSP90.1没有UTR区,SiHSP90.4的5′UTR区最长。保守基序分析显示,SiHSP90家族含有10个基序(Motif),这些Motif的长度为21~96个氨基酸。通过Pfam在线软件对10个Motif的结构域进行分析,结果显示Motif 7属于HATPase_c结构域,其余Motif均属于HSP90保守结构域。
2.3 谷子HSP90家族染色体定位分析
为了探究SiHSP90s在染色体上的位置,本研究绘制了SiHSP90s的染色体位置图(图3)。结果表明9个家族成员分别定位在谷子的第2、3、4、6和7号染色体上,其中SiHSP90.1和SiHSP90.2分别定位于第2号染色体上的6.01和34.60 Mb处;SiHSP90.3位于第3号染色体的45.08 Mb处,SiHSP90.4位于第4号染色体的39.62 Mb处;SiHSP90.5~SiHSP90.8位于第6号染色体的 31.46~32.08 Mb之间;SiHSP90.9定位在第7号染色体的1.56 Mb处。6号染色体上SiHSP90成员最多,有4个成员;3、4和7号染色体上SiHSP90成员最少,均为1个。
2.4 谷子HSP90启动子区顺式作用元件分析
为进一步探究SiHSP90在非生物胁迫中的调控机制,对SiHSP90基因启动子区(2 000 bp)进行顺式作用元件分析(图4-A),发现SiHSP90家族启动子区共存在237个顺式作用元件,可以分为3类:非生物胁迫响应类元件、激素响应类元件和光响应类元件。对于每个SiHSP90而言,光响应类作用元件分布广泛,同时每个基因都含有非生物胁迫响应类元件和激素响应类元件。非生物胁迫响应类元件主要有干旱诱导相关作用元件MBS(MYB binding site to drought-inducibitity)和低温应激相关元件LTR(low temperature response element)。其中MBS位于除SiHSP90.4外的所有基因上,SiHSP90.2、SiHSP90.3、SiHSP90.6、SiHSP90.7、SiHSP90.8和SiHSP90.9啟动子区均含有1个MBS,SiHSP90.1和SiHSP90.5启动子区分别含有3、2个MBS;LTR位于SiHSP90.5和SiHSP90.9基因上,元件的数量分别为1、3。激素响应类元件主要有脱落酸应答元件ABRE、生长素应答元件TGA-element和 AuxRR-core、茉莉酸甲酯应答元件CGTCA-motif/TGACG-motif、赤霉素应答元件GARE-motif和 P-box、水杨酸应答元件TCA-element。
对SiHSP90家族各成员上的3类顺式作用元件的数量进行统计分析, 发现除SiHSP90.4外其他基因均含有非生物胁迫响应相关作用元件,且非生物胁迫响应相关元件的数量要远低于激素响应类元件和光响应类元件(图4-B)。图4-C为各顺式作用元件在SiHSP90的2个亚家族中的分布情况,(A)、(B)、(C)分别表示激素响应类、非生物胁迫响应类和光响应类作用元件。结果显示除部分顺式作用元件外,大部分元件在谷子HSP90上的2个亚家族中都有分布,且在Ⅱ型亚族中的数量多于Ⅰ型亚族。
2.5 谷子HSP90家族干旱胁迫下表达谱
为了验证SiHSP90家族在干旱胁迫中的表达情况,通过qRT-PCR检测了此基因家族成员在干旱胁迫下的表达模式。结果表明:SiHSP90.2和SiHSP90.3在干旱胁迫下的表达水平与对照相比极显著上调;SiHSP90.1、SiHSP90.4、SiHSP90.5、SiHSP90.6、SiHSP90.7、SiHSP90.8和SiHSP90.9的表达水平与对照相比未见显著变化(图5)。
3 讨论与结论
HSP90s作为分子伴侣,可以抑制变性蛋白的不可逆聚集,从而增强植物的耐旱性[13]。目前,对HSP90家族成员鉴定及功能分析已在水稻[12]、二穗短柄草[27]、烟草[20]、大豆[28]、番茄[29]、黄瓜[30]等物种中报道,而谷子中HSP90家族成员鉴定及功能分析尚不清楚。本研究从谷子基因组中共鉴定到9个HSP90家族成员,根据理化性质分析谷子HSP90家族成员编码蛋白热稳定性均较强,推测可能与其独特的基因功能相关。本研究发现一个有趣的现象,谷子中8个HSP90家族成员蛋白的分子量均在 90 ku 左右,而SiHSP90.1蛋白分子量只有46 ku,且该蛋白氨基酸序列的长度也比其他成员的氨基酸序列长度要小,推测该基因可能在进化过程中部分序列丢失,烟草HSP90-4[20]和番茄HSP90-6[31]蛋白分子量也在40 ku左右。在莱茵衣藻、拟南芥、二穗短柄草、水稻、高粱、玉米和谷子中鉴定到的HSP90家族成员数量差别较大,在莱茵衣藻中鉴定到的基因数量较少,这可能与其属于较为低等的绿藻门植物且基因组小有关;而隶属于被子植物门的拟南芥、二穗短柄草、水稻、高粱、玉米和谷子基因数量相似,但对于同样是禾本科的玉米来说其HSP90家族成员的数量相比其他物种略高,推测这可能与玉米基因组较大有关,从总体上看高等植物HSP90家族成员数量多于低等植物。
为了进一步了解物种的进化关系,对7个代表物种HSP90氨基酸序列构建系统发育树,发现谷子与水稻、高粱和二穗短柄草等物种亲缘关系较近,与莱茵衣藻、拟南芥亲缘关系较远;推测高等植物(拟南芥、二穗短柄草、水稻、高粱、玉米和谷子)中的HSP90家族成员可能是从低等植物莱茵衣藻HSP90家族成员逐渐复制、进化而来。根据系统发育树分支,可将谷子HSP90基因分为Ⅰ和Ⅱ2个亚家族。在拟南芥中,Ⅰ型亚族蛋白AtHSP90.1~AtHSP90.4定位于细胞质中,而Ⅱ型亚族蛋白AtHSP90.5、AtHSP90.6和AtHSP90.7分别定位于叶绿体、线粒体和内质网中[32]。SiHSP90.6~SiHSP90.9与拟南芥Ⅰ型亚族蛋白同源,推测其定位于细胞质中;SiHSP90.1~SiHSP90.5与拟南芥Ⅱ型亚族蛋白同源,推测其定位于细胞器中。基因结构在多基因家族的进化中起到重要作用,其中内含子的发育是基因组进化的一个重要过程,也是物种进化的一种适应性措施[33]。谷子HSP90s的Ⅰ型亚族成员拥有1~2个内含子,而Ⅱ型亚族成员拥有11~19个内含子。内含子的数量越少,植物适应不同发育过程和环境刺激的能力就越强[34]。分析谷子HSP90家族成员的基因结构按照其差异的不同也可划分为2个亚型,这与该家族系统发育进化树结果一致。大多数SiHSP90家族成员间的保守基序分布类似,表明该家族成员间可能具有相似的功能。由Motif 7构成的HATPase_c结构域是ADP/ATP结合位点,具有ATPase活性[35],其余9个Motif共同组成的HSP90保守结构域在维持HATPase_c结构域的完整活性中具有关键作用[36]。基因家族的扩展和基因组进化机制主要依赖于基因复制事件[37],基因定位分析显示SiHSP90.5~SiHSP90.8是串联重复基因,推测基因的串联重复导致SiHSP90家族成员的扩展,促进了该家族在谷子基因组进化中新生物学功能的出现。本研究在SiHSP90s启动子区发现了脱落酸应答元件ABRE以及干旱诱导元件MBS,推测SiHSP90对干旱的响应可能是通过ABA途径实现的[38]。利用qRT-PCR验证干旱胁迫下SiHSP90家族的表达水平,结果显示SiHSP90.2和SiHSP90.3表达水平与对照相比极显著上调,推测这2个基因在参与谷子干旱胁迫的响应中发挥了一定的作用。
本研究共鑒定出9个SiHSP90基因,对其系统进化发育、基因结构、染色体位置、胁迫相关顺式元件以及在干旱胁迫中的表达模式进行分析,这些结果可为阐明SiHSP90家族的进化关系以及进一步研究SiHSP90基因的功能特性提供有价值的信息。
参考文献:
[1]Chaves M M,Maroco J P,Pereira J S. Understanding plant responses to drought-from genes to the whole plant[J]. Functional Plant Biology,2003,30(3):239-264.
[2]Fatima A,Hussain S,Hussain S,et al. Differential morphophysiological,biochemical,and molecular responses of maize hybrids to salinity and alkalinity stresses[J]. Agronomy,2021,11(6):1150.
[3]Nakashima K,Yamaguchi-Shinozaki K.Regulons involved in osmotic stress-responsive and cold stress-responsive gene expression in plants[J]. Physiologia Plantarum,2006,126(1):62-71.
[4]Gao G Z,Hu J H,Zhang X J,et al. Transcriptome analysis reveals genes expression pattern of seed response to heat stress in Brassica napus L.[J]. Oil Crop Science,2021,6(2):87-96.
[5]Sangster T A,Queitsch C.The HSP90 chaperone complex,an emerging force in plant development and phenotypic plasticity[J]. Current Opinion in Plant Biology,2005,8(1):86-92.
[6]Al-Whaibi M H.Plant heat-shock proteins:a mini review[J]. Journal of King Saud University-Science,2011,23(2):139-150.
[7]Pearl L H,Prodromou C.Structure and in vivo function of Hsp90[J]. Current Opinion in Structural Biology,2000,10(1):46-51.
[8]Terasawa K,Minami M,Minami Y.Constantly updated knowledge of Hsp90[J]. Journal of Biochemistry,2005,137(4):443-447.
[9]Pearl L H,Prodromou C.Structure and mechanism of the Hsp90 molecular chaperone machinery[J]. Annual Review of Biochemistry,2006,75:271-294.
[10]Hainzl O,Lapina M C,Buchner J,et al. The charged linker region is an important regulator of Hsp90 function[J]. Journal of Biological Chemistry,2009,284(34):22559-22567.
[11]Sung N,Lee J,Kim J H,et al. Mitochondrial Hsp90 is a ligand-activated molecular chaperone coupling ATP binding to dimer closure through a coiled-coil intermediate[J]. PNAS,2016,113(11):2952-2957.
[12]Raman S,Suguna K.Functional characterization of heat-shock protein 90 from Oryza sativa and crystal structure of its N-terminal domain[J]. Acta Crystallographica.Section F,Structural Biology Communications,2015,71(Pt 6):688-696.
[13]Morán Luengo T,Mayer M P,Rüdiger S G D.The Hsp70-Hsp90 chaperone cascade in protein folding[J]. Trends in Cell Biology,2019,29(2):164-177.
[14]Shinozaki F,Minami M,Chiba T,et al. Depletion of Hsp90 β induces multiple defects in B cell receptor signaling[J]. The Journal of Biological Chemistry,2006,281(24):16361-16369.
[15]Zuehlke A,Johnson J L.Hsp90 and co-chaperones twist the functions of diverse client proteins[J]. Biopolymers,2010,93(3):211-217.
[16]Agarwal G,Garg V,Kudapa H,et al. Genome-wide dissection of AP2/ERF and HSP90 gene families in five legumes and expression profiles in chickpea and pigeonpea[J]. Plant Biotechnology Journal,2016,14(7):1563-1577.
[17]Grigorova B,Vaseva I,Demirevska K,et al. Combined drought and heat stress in wheat:changes in some heat shock proteins[J]. Biologia Plantarum,2011,55(1):105-111.
[18]di Donato M,Geisler M.HSP90 and co-chaperones:a multitaskers view on plant hormone biology[J]. FEBS Letters,2019,593(13):1415-1430.
[19]Song H M,Zhao R M,Fan P X,et al. Overexpression of AtHsp90.2,AtHsp90.5 and AtHsp90.7 in Arabidopsis thaliana enhances plant sensitivity to salt and drought stresses[J]. Planta,2009,229(4):955-964.
[20]Song Z P,Pan F L,Yang C,et al. Genome-wide identification and expression analysis of HSP90 gene family in Nicotiana tabacum[J]. BMC Genetics,2019,20(1):35.
[21]Lv D W,Subburaj S,Cao M,et al. Proteome and phosphoproteome characterization reveals new response and defense mechanisms of Brachypodium distachyon leaves under salt stress[J]. Molecular & Cellular Proteomics,2014,13(2):632-652.
[22]Hao P C,Zhu J T,Gu A Q,et al. An integrative proteome analysis of different seedling organs in tolerant and sensitive wheat cultivars under drought stress and recovery[J]. Proteomics,2015,15(9):1544-1563.
[23]Nadeem F,Ahmad Z,Ul Hassan M,et al. Adaptation of foxtail millet (Setaria italica L.) to abiotic stresses:a special perspective of responses to nitrogen and phosphate limitations[J]. Frontiers in Plant Science,2020,11:187.
[24]Lata C R,Gupta S,Prasad M.Foxtail millet:a model crop for genetic and genomic studies in bioenergy grasses[J]. Critical Reviews in Biotechnology,2013,33(3):328-343.
[25]Muthamilarasan M,Prasad M.Advances in Setaria genomics for genetic improvement of cereals and bioenergy grasses[J]. Theoretical and Applied Genetics,2015,128(1):1-14.
[26]Bennetzen J L,Schmutz J,Wang H,et al. Reference genome sequence of the model plant Setaria[J]. Nature Biotechnology,2012,30(6):555-561.
[27]Zhang M,Shen Z W,Meng G Q,et al. Genome-wide analysis of the Brachypodium distachyon (L.) P. Beauv. Hsp90 gene family reveals molecular evolution and expression profiling under drought and salt stresses[J]. PLoS One,2017,12(12):e0189187.
[28]Xu J Y,Xue C C,Xue D,et al. Overexpression of GmHsp90s,a heat shock protein 90 (Hsp90) gene family cloning from soybean,decrease damage of abiotic stresses in Arabidopsis thaliana[J]. PLoS One,2013,8(7):e69810.
[29]Zai W S,Miao L X,Xiong Z L,et al. Comprehensive identification and expression analysis of Hsp90s gene family in Solanum lycopersicum[J]. Genetics and Molecular Research,2015,14(3):7811-7820.
[30]Zhang K J,He S S,Sui Y H,et al. Genome-wide characterization of HSP90 gene family in cucumber and their potential roles in response to abiotic and biotic stresses[J]. Frontiers in Genetics,2021,12:584886.
[31]劉云飞,万红建,杨悦俭,等. 番茄热激蛋白90的全基因组鉴定及分析[J]. 遗传,2014,36(10):1043-1052.
[32]Yamada K,Fukao Y,Hayashi M,et al. Cytosolic HSP90 regulates the heat shock response that is responsible for heat acclimation in Arabidopsis thaliana[J]. The Journal of Biological Chemistry,2007,282(52):37794-37804.
[33]Liu H,Lyu H M,Zhu K K,et al. The emergence and evolution of intron-poor and intronless genes in intron-rich plant gene families[J]. The Plant Journal,2021,105(4):1072-1082.
[34]Jin Z W,Chandrasekaran U,Liu A Z.Genome-wide analysis of the Dof transcription factors in Castor bean (Ricinus communis L.)[J]. Genes & Genomics,2014,36(4):527-537.
[35]LaPointe P,Mercier R,Wolmarans A.Aha-type co-chaperones:the alpha or the Omega of the Hsp90 ATPase cycle?[J]. Biological Chemistry,2020,401(4):423-434.
[36]Meyer P,Prodromou C,Hu B,et al. Structural and functional analysis of the middle segment of Hsp90:implications for ATP hydrolysis and client protein and cochaperone interactions[J]. Molecular Cell,2003,11(3):647-658.
[37]Yang Z F,Zhou Y,Wang X F,et al. Genomewide comparative phylogenetic and molecular evolutionary analysis of tubby-like protein family in Arabidopsis,rice,and poplar[J]. Genomics,2008,92(4):246-253.
[38]Muppala S,Gudlavalleti P K,Malireddy K R,et al. Development of stable transgenic maize plants tolerant for drought by manipulating ABA signaling through Agrobacterium-mediated transformation[J]. Journal,Genetic Engineering & Biotechnology,2021,19(1):96.

