AA野生种花生Ty1-copia类反转座子RT基因的克隆与序列分析
蔡铁城 刘俊仙 张冲 刘菁 阳太亿 蒋菁 贺梁琼 韩柱强 唐荣华 庄伟建 熊发前

摘 要: 该研究旨在克隆Ty1-copia类反转录转座子RT(reverse transcriptase)基因,为分离花生属Ty1-copia类反转录转座子全长序列和研究其功能提供序列基础。根据RT基因的保守区设计简并引物,以两份AA染色体组野生种花生Arachis duranensis为试材,利用PCR扩增其基因组DNA,回收、克隆和测序目的条带后,对所获得序列进行生物信息学分析。结果表明:(1)目的条带大小约为260 bp,分别从两份野生种花生材料中克隆到41條和27条RT基因序列,68条序列的长度变化范围为256 ~270 bp,AT所占比例范围为55.86%~68.42%,AT与GC比例范围为1.27~2.17,核苷酸序列间相似性范围为49.8%~99.2%,存在较高异质性。(2)68条序列被分为6个家族,家族Ⅰ和Ⅳ为主要家庭。(3)68条序列中的19条发生了无义突变,A. duranensis(PI219823)要比A. duranensis(PI262133)的无义突变率高。(4)氨基酸序列间相似性为4.7%~100%,呈现高度异质性。(5)各家族中代表序列的蛋白质三级结构在整体构型上一致,但在螺旋结构数、折叠结构数、转角数和氢键数上存在较大差别。(6)序列间保守基序总体一致,但也存在一定变异,呈现一定异质性;系统进化树将68条序列分为10类,大部分序列都聚在A和B两大类中。(7)另有部分AA染色体组野生种花生的RT基因序列与其他物种植物的RT基因序列亲缘关系较近,推测不同物种植物间可能发生过Ty1-copia类反转录转座子的横向传递。该研究为花生属基于Ty1-copia类反转录转座子的新分子标记开发和应用奠定基础。
关键词: 花生, Ty1-copia类反转录转座子, 反转录酶, 野生种, 异质性
中图分类号: Q943
文献标识码: A
文章编号: 1000-3142(2022)01-0100-13
收稿日期: 2021-07-15
基金项目: 国家自然科学基金(31960409,32072103,31960416);广西自然科学基金(2018GXNSFDA281027,2018GXNSFDA294004,2020GXNSFAA297081);福建省科技厅农业引导性(重点)项目(2018N0004);广西农业科学院科技发展基金(桂农科2018YM06,桂农科2017JZ13,桂农科31960409,桂农科2021YT052) [Supported by the National Natural Science Foundation of China (31960409, 32072103, 31960416); Guangxi Natural Science Foundation (2018GXNSFDA281027, 2018GXNSFDA294004, 2020GXNSFAA297081); Guiding Key Project of Department of Science and Technology of Fujian Province (2018N0004); Scientific and Technological Development Foundation of Guangxi Academy of Agricultural Sciences (GNK2018YM06, GNK2017JZ13, GNK31960409, GNK2021YT052]。
第一作者: 蔡铁城(1983-),硕士,助理研究员,主要从事花生遗传育种和分子生物学研究,(E-mail)caitiecheng1027@163.com。
*通信作者: 熊发前,博士,研究员,主要从事花生种质资源和遗传育种及分子生物学研究,(E-mail)xfq2002@126.com。
Cloning and sequence analysis of reverse transcriptase
genes of Ty1-copia-like retrotransposons in wild
peanut species with AA genome
CAI Tiecheng1, LIU Junxian3, ZHANG Chong1, LIU Jing2, YANG Taiyi2,
JIANG Jing2, HE Liangqiong2, HAN Zhuqiang2, TANG Ronghua2,
ZHUANG Weijian1, XIONG Faqian2*
( 1. College of Plant Protection, Fujian Agriculture and Forestry University, Fuzhou 350002, China; 2. Cash
Crops Research Institute, Guangxi Academy of Agricultural Sciences, Nanning 530007, China; 3. Sugarcane Research Institute, Guangxi Academy of Agricultural Sciences/Sugarcane Research Center, Chinese Academy of Agricultural Sciences/Key Laboratory of Sugarcane Biotechnology and Genetic Improvement (Guangxi), Ministry of Agriculture and Rural Affairs/Guangxi Key Laboratory of Sugarcane Genetic Improvement, Nanning 530007 )
Abstract: The purpose of this study was to clone the reverse transcriptase (RT) genes of Ty1-copia-like retrotransposons, and to provide sequences basis for isolating the full-length sequences of Ty1-copia-like retrotransposons and studying their function in genus Arachis. Degenerate primers were designed according to the conserved region of RT genes, the genomic DNA of wild peanut species Arachis duranensis with AA genome was amplified by PCR using the degenerated primers. The amplified targeted bands were recovered, cloned and sequenced, and then the sequences were analyzed through bioinformatics analysis. The results were as follows: (1) The amplified targeted bands were all about 260 bp in size. Forty-one and twenty-seven RT genes sequences were cloned from the two wild peanut species respectively. The length of sixty-eight sequences varied from 256 to 270 bp. The proportion of AT, AT/GC and the similarity between nucleotide sequences ranged from 55.86% to 68.42%, 1.27 to 2.17, and 49.8% to 99.2% respectively, showing a higher heterogeneity. (2) The sixty-eight sequences were divided into six families, Family I and Family IV were the main families. (3) Nineteen of sixty-eight sequences had nonsense mutations, and Arachis duranensis (PI219823) had a higher nonsense mutations rate than Arachis duranensis (PI262133). (4) The similarity between amino acid sequences ranged from 4.7% to 100%, showing a high heterogeneity. (5) The tertiary structures of proteins representing sequences in each family were basically similar in overall configuration, but there were great differences in the number of helix structures, folding structures, turns and hydrogen bonds. (6) The conserved motifs among sequences were generally consistent, but there were also some variations, showing a certain degree of heterogeneity. The phylogenetic tree divided sixty-eight sequences into ten classes. Most of sequences were clustered in A and B classes. (7) Some of RT genes sequences from two wild peanut species with AA genome were closely related to RT genes sequences from other plant species, which indicated that there might be transposon horizontal transmission between them. This study provides reference for the development and application of new molecular markers based on Ty1-copia-like retrotransposons in genus Arachis.
Key words: peanut, Ty1-copia-like retrotransposons, reverse transcriptase (RT), wild species, heterogeneity
花生被譽为“长生果”,全世界有106个国家种植。花生是我国主要食用油原料,我国是世界上重要的花生生产国。当前我国选育出的花生品种主要是利用传统杂交育种,但存在周期长、效率低、目的性不强等缺点,而分子育种可以加速育种进程,但是缺少简单实用高效的DNA分子标记。在花生上,传统分子标记技术想检测出丰富多态性十分困难(熊发前等,2010;王强等,2010;Xiong et al., 2011),虽然近几年在花生上也有利用SNP标记进行关联及连锁分析的研究报道(Zhang et al., 2017;Han et al., 2018;Wang et al., 2018),但当前在花生上使用最广泛的是SSR标记,然而能在任意两个栽培种花生品种间检测出DNA多态性的SSR标记引物对还比较匮乏(熊发前等,2010;Xiong et al., 2011)。
LTR反转录转座子主要包括Ty1-copia和Ty3-gypsy两大类(Kumar & Bennetzen, 1999;Feschotte et al., 2002;Bonchev & Parisod, 2013),这两类LTR反转录转座子中的RT基因序列都可以通过简并PCR技术扩增克隆(Voytas et al., 1992;Kumekawa et al., 1999)。LTR反转录转座子的普遍性、高拷贝、高度异质性和插入位点多态性等特性使其非常适合开发分子标记。
但到目前为止,在花生上尚未见到基于LTR反转录转座子开发分子标记的研究报道。而分离和鉴定LTR反转录转座子是分子标记开发利用的前提。花生LTR反转录转座子的研究报道较少,Nielen等(2010, 2012)先后分离出花生Ty3-gypsy类反转录转座子的FIDEL和Ty1-copia类反转录转座子的Matita,对FIDEL和Matita的特性和作用进行了分析。笔者曾系统对花生LTR反转录转座子和MITE转座子的分离及其应用的国内外研究现状及进展进行了归纳(熊发前等,2017)。
本研究拟从两份AA染色体组野生种花生材料Arachis duranensis中克隆Ty1-copia类反转录转座子RT基因,分析其序列特征和多样性,为分离花生属Ty1-copia类反转录转座子全长序列和研究其功能提供序列基础,为花生属基于Ty1-copia类反转录转座子的新分子标记开发和应用奠定基础。
1 材料与方法
1.1 供试材料
在广西农业科学院武鸣里建科研基地的花生野生圃里随机摘取两份AA染色体组野生种花生材料Arachis duranensis(PI262133)和Arachis duranensis(PI219823)各5株健康植株的顶端芯叶进行混合。
1.2 基因组DNA的提取
花生高质量基因组DNA的提取采用改良CTAB法(熊发前等,2019)。
1.3 RT基因的PCR扩增
上游引物为RTp1:5′-ACNGCNTTYYTNCAYGG-3′,下游引物为RTp2:5′-ARCATRTCRTCNACRTA-3′,其中,R=A/G,Y=C/T,N=A/T/C/G(Kumar et al.,1997)。PCR扩增体系和扩增程序以及PCR产物的分离检测参考熊发前等(2019)的方法。
1.4 PCR产物的回收、克隆及测序
参考阳太亿等(2019)的方法进行。
1.5 RT基因的序列分析
序列相似性检索、序列统计分析、序列图及Logo图的生成、蛋白二级结构与三级结构预测、蛋白三级结构的转角数和氢键数统计、保守基序预测等参考阳太亿等(2019)进行。运用MEGA 6.0软件的邻接法(No. of differences模型)构建系统进化树,自展值设置为1 000,所用其他物种的Ty1-copia类反转录转座子RT基因序列信息见表1。
2 结果与分析
2.1 RT基因的PCR扩增及测序
对AA染色体组野生种花生材料A. duranensis(PI262133)和A. duranensis(PI219823)的基因组DNA进行PCR扩增, 结果显示,在2份花生材料中都扩增出了大小约260 bp的特异条带(图1)。将目的条带进行回收、克隆和测序,从A. duranensis(PI262133)和A. duranensis(PI219823)中分别获得了42条和38条序列。利用DNAMAN软件去除相同序列,利用NCBI数据库对序列进行同源性分析进而去除非目标序列,最后从A. duranensis(PI262133)和A. duranensis(PI219823)中分别获得了41条和27条目标序列,并分别命名为AdRT1-X和AdRT2-X(表2)。对这些RT基因序列进行多重比对(图2),利用weblogo生成了序列logo图以展示每个位置上碱基的保守性(图3)。
2.2 RT基因序列分析
利用BioEdit软件对RT基因序列进行统计分析,结果见表2,从中可以看出,所有序列长度都在256 ~270 bp之间,存在缺失或插入突变。在A. duranensis(PI262133)的41条序列中,AdRT1-47的序列长度最短,为256 bp,AdRT1-11的序列长度最长,为267 bp,长度为266 bp的序列占总序列数的90.24%;A、T、C、G数量变化范围分别为65~88、78~94、29~61和49~67,AT所占比例范围为55.86%~68.42%,AT与GC比例为1.27~2.17;核苷酸序列间相似性范围为50%~99.2%,其中,AdRT1-26与AdRT1-25之间的相似性最高,达99.2%,AdRT1-47与AdRT1-7以及AdRT1-47与AdRT1-29之间的相似性最低,均为50%,氨基酸序列间相似性范围为4.7%~100%。在A. duranensis(PI219823)的27条序列中,AdRT2-24的序列长度最短,为257 bp,AdRT2-7的序列长度最长,为270 bp,长度为266 bp的序列占总序列数的74.07%;A、T、C、G数量变化范围分别为62~93、71~93、32~56和66~68,AT所占比例范圍为57.20%~67.29%,AT与GC比例为1.34~2.06;核苷酸序列间相似性范围为49.8%~99.2%,其中,AdRT2-3与AdRT2-4之间的相似性最高,达99.2%,AdRT2-13与AdRT2-27之间的相似性最低,为49.8%,氨基酸序列间相似性范围为14.6%~100%。
2.3 RT基因核苷酸序列聚类分析
利用MEGA 6.0软件对RT基因序列进行聚类分析(图4)。遗传进化树显示,68条序列被划分为6个家族,家族Ⅰ包含28条序列,其中,18条来自A. duranensis(PI262133),10条来自A. duranensis(PI219823),所含序列数占总序列数的41.18%;家族Ⅱ包含9条序列;家族Ⅲ只包含AdRT1-29,该序列与家族Ⅰ亲缘关系较远而单独聚为一类,造成该序列单独为一类的原因可能是碱基替换;家族Ⅳ包含23条序列,其中,有17条来自A. duranensis(PI262133),6条来自A. duranensis(PI219823),所含序列数占总序列数的33.82%;家族Ⅴ包含3条序列;家族Ⅵ包含4条序列。家族Ⅴ和家族Ⅵ与另外4个家族遗传距离较大,亲缘关系较远,因为这两个家族中的序列存在碱基缺失的现象。
2.4 RT基因氨基酸序列分析
利用MEGA 6.0软件对RT基因氨基酸序列进行分析(图5)。结果显示,68条序列中有19条发生无义突变,其中,9条序列来自A. duranensis(PI262133),占该材料序列总数的21.95%,10条序列来自A. duranensis(PI219823),占该材料序列总数的37.04%,就无义突变发生率来说,A. duranensis(PI219823)要比A. duranensis(PI262133)高。无义突变在2份花生材料中的具体表现如下:AdRT2-21发生了8个无义突变,分别在第31、第34、第65、第66、第71、第75、第76和第77个氨基酸处;AdRT2-5发生了7个无义突变,分别在第51、第58、第74、第75、第76、第77和第87个氨基酸处;AdRT1-47发生了6个无义突变,分别在第18、第26、第30、第37、第79和82个氨基酸处;AdRT1-11发生了5个无义突变,分别在第41、第71、第75、第76和第77个氨基酸处;AdRT1-16发生了5个无义突变,分别在第54、第58、第80、第81和第84个氨基酸处;AdRT2-10发生了5个无义突变,分别在第54、第58、第81、第82和第84个氨基酸处;AdRT1-4發生了2个无义突变(分别在第26和第41个氨基酸处);AdRT1-23发生了2个无义突变(分别在第8和第39个氨基酸处);AdRT1-7、AdRT1-22、AdRT1-34、AdRT1-48、AdRT2-7、AdRT2-11、AdRT2-27、AdRT2-37、AdRT2-38
和AdRT2-42都只发生了1个无义突变;部分序列存在连续无义突变的现象,无义突变会影响到反转录转座子的转录活性。
2.5 RT基因的蛋白结构预测
将获得的68条Ty1-copia类反转录转座子RT基因序列统一翻译成氨基酸后,根据核苷酸聚类结果,分别选择两份花生材料中每个家族中的代表序列,利用在线程序Phyre2预测其蛋白质的二级结构和三级结构(表3,图6,图7),代表序列蛋白三级结构匹配覆盖度最高的模板为d1hara、c4rs7R、d1sqwa1、c5xvnM,置信度均为16.5~86.2,其中只有d1hara属于逆转录酶家族,其余蛋白均未匹配到逆转录蛋白模板。二级结构包含2~3个α-螺旋和5个β-折叠; 三级结构包含2~6个转角和9~30个氢键,还存在1个明显的螺旋结构和2个不明显的折叠结构。
2.6 RT基因保守基序预测
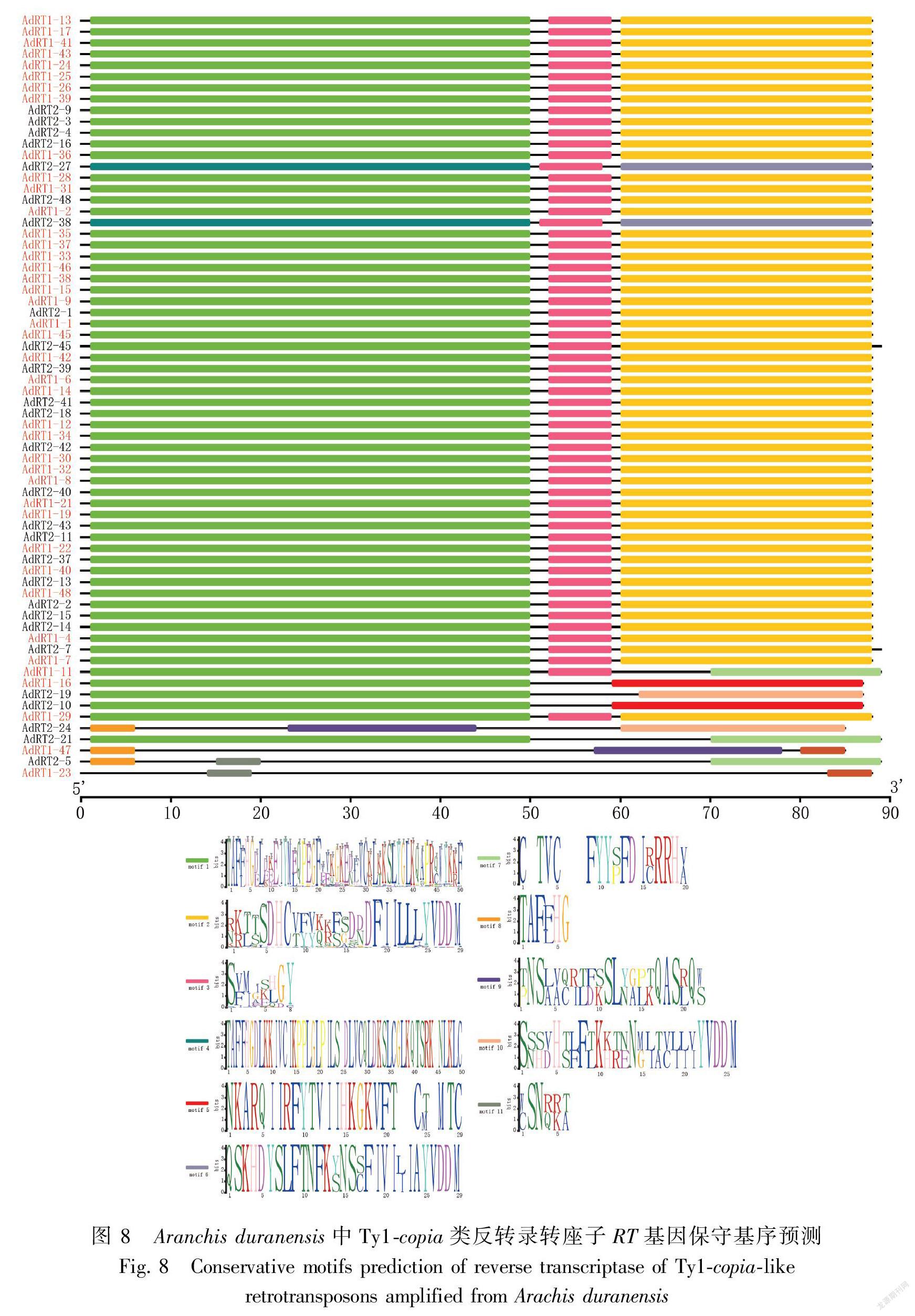
RT基因保守基序预测结果显示, 68条序列共存在11种保守基序,其中,有57条序列同时包含motif 1、motif 2和motif 3, 占总序列数的83.82%,说明这3种保守基序是AA染色体组野生种花生Ty1-copia类反转录转座子RT基因的主要保守基序,这也从保守基序角度表明两个AA染色体组野生种花生Ty1-copia类反转录转座子RT基因具有非常高的保守性与相似性。AdRT2-27和AdRT2-38同时包含motif 3、motif 4和motif 6,motif 4和motif 6这两个保守基序在位置上与motif 1和motif 2相同,但氨基酸的排列和组成并不相同;AdRT1-11包含motif 1、motif 3和motif 7;AdRT1-16和AdRT2-10同时包含motif 1和motif 5,在系统进化树中,这2条序列为同一类;AdRT2-19包含motif 1和motif 10;AdRT2-24包含motif 8、motif 9和motif 10;AdRT2-21包含motif 1和motif 7,这3条序列在系统进化树中均单独归类;AdRT1-47包含motif 8和motif 9;AdRT2-5包含motif 7、motif 8和motif 11;AdRT1-23只包含motif 11,这3条序列在系统进化树中归为同一类,其中,motif 8和motif11的长度较短,位于序列上游部分。部分保守基序在所克隆序列中出现的频率较低且长度较短,说明这些序列在进化过程中发生了突变(图8)。
2.7 RT基因序列系统进化树构建
构建系统进化树(图9),所有RT基因序列可分为10类,大部分RT基因序列都聚在A和B两大类中,表明本研究中AA染色体组野生种花生的RT基因序列具有相当高的保守性与相似性。A类包含20条来自A. duranensis(PI262133)的序列和15条来自A. duranensis(PI219823)的序列,这35条序列与来自葡萄(Vitis vinifera,CAN67451.1)、绿豆(Vigna radiata,AAT90460.1、AAT90479.1)、鹰嘴豆(Cicer arietinum,CAD59770.1)、马铃薯(Solanum tuberosum,CAA13067.1)、西洋参(Panax quinquefolius,ABU94811.1)、野茶树(Camellia sinensis,CAJ09751.1)、李子(Prunus salicina,AGX45518.1)、苹果(Malus domestica,ABS11062.1)、烟草(Nicotania tabacum,AAA03507.1)的序列之间具有较高相似性,亲缘关系较近。B类包含17条来自A. duranensis(PI262133)的序列和5条来自A. duranensis(PI219823)的序列,说明这22条序列之间具有较高相似性。C类只有AdRT1-29,该序列与来自油菜(Brassica napus,AAA32987.1)、梅花(Prunus mume,ABF57071.1)、大豆(Glycine max,E47759)、藜麦(Chenopodium quinoa,AEX61031.1)、番茄(Lycopersicon esculentum,AAC34611.1)、大狗尾草(Setaria faberi,AAL36472.1)、水稻(Oryza sativa,AAA33902.1)和玉米(Zea mays,AAK84849.1)的序列之间具有较高相似性,亲缘关系较近。D类中是来自绿豆、欧洲云杉和石蒜的3条序列,这3条序列与大多数AA染色体组野生种花生和其他物种植物的序列亲缘关系较远。E类只包含2条序列,分别是来自AA染色体组野生种花生的AdRT2-24和拟南芥的S71291,说明这2条序列之间亲缘关系最近。F-J类中的3条和7条序列分别来自Arachis duranensis(PI262133)和A. duranensis(PI219823),均不包含其他物种植物的序列。
3 讨论与结论
本研究用同一简并引物从同一种野生花生种质中克隆获得的Ty1-copia类反转录转座子RT基因的核苷酸和氨基酸序列在序列长度、序列组成及序列相似性等方面存在较大差异和多态性,表明同一种野生花生种质内同一类群反转录转座子存在较高异质性。另外,两份野生种花生材料中RT基因序列均富含AT碱基,富含AT导致呈现较高异质性;RT基因核苷酸序列间相似性呈现较高异质性; 有19条氨基酸序列发生了无义突变, 导致呈现较高异质性;RT基因氨基酸序列间相似性呈现高度异质性;68条RT基因序列间保守基序也呈现一定异质性;家族Ⅴ和家族Ⅵ中的代表序列在螺旋结构、折叠结构、转角数、氢键数上与其他家族代表序列存在较大差别,呈现较高异质性和多态性。总之,两份野生种花生材料中RT基因序列在AT碱基含量、核苷酸序列间相似性、氨基酸序列间相似性、氨基酸无义突变率、保守基序、蛋白质二级结构及三级结构上均呈现异质性。
家族Ⅰ-Ⅳ中代表序列的蛋白质三级结构在整体构型上基本类似,但在螺旋结构数、折叠结构数、转角数和氢键数上存在较大差别,家族Ⅴ中的AdRT1-16只存在2个明显的螺旋结构,其转角数和氢键数也明显少于其他序列;家族Ⅵ中的AdRT1-47的氢键数远远少于其他序列,其转角数也少于其他序列,且其螺旋结构较短;AdRT2-27相较于其他序列存在2个明显的折叠结构和1个不明显的折叠结构,其转角数和氢键数都少于其他序列,推测这些差别可能会影响到Ty1-copia类反转录转座子的拷贝数、转录活性及转座效率等。
遗传进化树显示,6个家族中的Ⅰ和Ⅳ是主要家族,表明AA染色体组野生种花生Ty1-copia类反转录转座子RT基因序列具有非常高的保守性与相似性。另外,各家族内部成員越复杂,序列相似性越高,存在有转录活性的反转录转座子的可能性也越大,其转座发生的时间可能越近(Tang et al., 2005),由此推测,家族Ⅰ和家族Ⅳ很有可能是存在具有转录活性的Ty1-copia类反转录转座子的家族,存在的历史也更为久远。
系统进化树显示,A类中的35条AA染色体组野生种花生RT基因序列与葡萄、绿豆、鹰嘴豆、马铃薯、西洋参、野茶树、李子、苹果、烟草的序列之间相似性较高;C类中的AdRT1-29与来自油菜、梅花、大豆、藜麦、番茄、大狗尾草、水稻、玉米的序列之间相似性较高;E类中的AdRT2-24与拟南芥的S71291之间的相似性较高,推测AA染色体组野生种花生Ty1-copia类反转录转座子曾可能与这些物种植物间发生过横向传递。F-J类中的RT基因序列均来自AA染色体组野生种花生,且与AA染色体组野生种花生和其他物种植物的RT基因序列遗传距离最大,亲缘关系最远,表明这几类中的RT基因序列在起源和进化上可能较为古老,特异性比较强,有可能为A. duranensis(PI262133)和A. duranensis(PI219823)所特有。
总之,本研究从AA染色体组野生种花生中成功克隆到Ty1-copia类反转录转座子RT基因序列,对花生属基于LTR反转录转座子的分子标记开发及花生分子育种具有重要意义,将为下一步分离其全长序列、研究其转录和转座活性及功能提供序列基础。
参考文献:
BONCHEV G, PARISOD C, 2013. Transposable elements and microevolutionary changes in natural populations [J]. Mol Ecol Resour, 13(5): 765-775.
FESCHOTTE C, JIANG N, WESSLER SR, 2002. Plant transposable elements:where genetics meets genomics [J]. Nat Rev Genet, 3(5): 329-341.
HAN SY, YUAN M, CLEVENGER JP, et al., 2018. A SNP-based linkage map revealed QTLs for resistance to early and late leaf spot diseases in peanut (Arachis hypogaea L.) [J]. Front Plant Sci, 9: 1012.
KUMAR A, BENNETZEN JL, 1999. Plant retrotransposons [J]. Ann Rev Genet, 33(1): 479-532.
KUMEKAWA N, OHTSUBO E, OHTSUBO H, 1999. Identification and phylogenetic analysis of gypsy-type retrotransposons in the plant kingdom [J]. Genes Genet Syst, 74(6): 299-307.
NIELEN S, CAMPOS-FONSECA F, LEAL-BERTIOLI S, et al., 2010. FIDEL—a retrovirus-like retrotransposon and its distinct evolutionary histories in the A- and B-genome components of cultivated peanut [J]. Chrom Res, 18(2): 227-246.
NIELEN S, VIDIGAL BS, LEAL-BERTIOLI SCM, et al.,2012. Matita, a new retroelement from peanut:characterization and evolutionary context in the light of the Arachis A-B genome divergence [J]. Mol Genet Genom, 287(1): 21-38.
TANG YM, MA YZ, LI LC, et al., 2005. Identification and characterization of reverse transcriptase domain of transcriptionally active retrotransposons in wheat genome [J]. J Integr Plant Biol, 47(5): 604-612.
VOYTAS DF, CUMMINGS MP, KONIECZNY A, et al., 1992. Copia-like retrotransposons are ubiquitous among plants [J]. Proc Natl Acad Sci USA, 89(15): 7124-7128.
WANG LF, ZHOU XJ, REN XP, et al., 2018. A major and stable QTL for bacterial wilt resistance on chromosome B02 identified using a high-density SNP-based genetic linkage map in cultivated peanut Yuanza 9102 derived population [J]. Front Genet, 9: 652.
WANG Q, ZHANG XY, TANG FS, et al., 2010. Construction of genetic linkage map of peanut (Arachis hypogaea L.) based on SRAP markers [J]. Chin J Oil Crop Sci, 32(3): 374-378. [王强, 张新友, 汤丰收, 等, 2010. 基于SRAP分子标记的栽培种花生遗传连锁图谱构建 [J]. 中国油料作物学报, 32(3): 374-378.]
XIONG FQ, JIANG J, ZHONG RC, et al., 2010. Application of SCoT molecular markers in genus Arachis [J]. Acta Agron Sin, 36(12): 2055-2061. [熊发前, 蒋菁, 钟瑞春, 等, 2010.目标起始密码子多态性(SCoT)分子标记技术在花生属中的应用 [J]. 作物学报, 36(12): 2055-2061.]
XIONG FQ, LIU JX, HE LQ, et al., 2017. Recent advances on the development and utilization of molecular markers based on LTR retrotransposons and MITE transposons from peanut(Arachis hypogaea L.) [J]. Mol Plant Breed, 15(2): 640-647. [熊发前, 刘俊仙, 贺梁琼, 等, 2017. 花生LTR和MITE转座子及其分子标记开发利用研究进展 [J]. 分子植物育种, 15(2): 640-647.]
XIONG FQ, LIU JX, LIU J, et al., 2019. Comparative analysis and application of five improved CTAB extraction methods for peanut DNA [J]. Mol Plant Breed,17(7): 2207-2216. [熊發前, 刘俊仙, 刘菁, 等, 2019. 花生DNA的五种改良CTAB提取方法的比较分析及其应用 [J]. 分子植物育种, 17(7): 2207-2216.]
XIONG FQ, ZHONG RC, HAN ZQ, et al., 2011. Start codon targeted polymorphism for evaluation of functional genetic variation and relationships in cultivated peanut (Arachis hypogaea L.) genotypes [J]. Mol Biol Rep, 38(5): 3487-3494.
YANG TY, LIU JX, LIU J, et al., 2019. Cloning and analysis of reverse transcriptase of Ty1-copia-like retrotransposons in Arachis monticola [J]. Shandong Agric Sci, 51(9): 9-20. [阳太亿, 刘俊仙, 刘菁, 等, 2019. 四倍体野生种花生Ty1-copia类逆转座子逆转录酶基因的克隆与分析 [J]. 山东农业科学, 51(9): 9-20.]
ZHANG XG, ZHANG JH, He XY, et al., 2017. Genome-wide association study of major agronomic traits related to domestication in peanut [J]. Front Plant Sci, 8: 1611.
(责任编辑 李 莉)
1488501186226

