长穗偃麦草LBD基因家族的鉴定与进化分析
王新华许娜丽姚明明余慧霞王彦青邱国岩
刘凤楼1,2,刘彩霞1,张晓岗1,李清峰1,2,王掌军1,2
(1.宁夏大学 农学院,银川 750021;2.宁夏优势特色作物分子育种重点实验室,银川 750021)
小麦作为世界上重要的粮食作物之一为全球粮食安全作出了巨大贡献[1-2]。由于驯化以及现代育种定向选择造成栽培小麦的遗传基础狭窄成为制约小麦发展的主要瓶颈[3-4]。利用远缘杂交将小麦野生近缘种中的高产、抗病、抗逆等优异基因导入小麦,是拓宽小麦遗传基础的有效途径[5-6]。如携带抗病、高产性状的小麦-黑麦T1BL·1RS易位系以及携带Pm21基因的小麦-簇毛麦T6VS·6AL易位系在育种和生产上被广泛应用[7-8]。因此,开展小麦近缘属优异基因的挖掘与研究对小麦的遗传改良具有重要意义。
长穗偃麦草(Thinopyrumelongatum)是禾本科小麦族偃麦草属物种,其染色体包含3种倍性:二倍体(2n=2x=14,EE)、四倍体(2n=4x=28,EEEE)和十倍体(2n=10x=70,EEEEEESt-StStSt)[9]。具有抗逆(抗寒、抗旱、耐盐)和抗病(枯萎病、锈病、白粉病)特性,是小麦外缘基因的重要来源之一[10-13]。为利用长穗偃麦草中优异基因李振声院士团队培育出‘小偃6号’‘小偃54’和‘小偃81’等一系列小麦新品种,其中‘小偃6号’已成为中国黄淮麦区的骨干亲本[14-17]。尽管长穗偃麦草在小麦遗传育种改良中得到成功应用,但对其基因定位和克隆等方面的研究相对滞后。随着二倍体长穗偃麦草基因组组装的完成,从全基因组水平研究长穗偃麦草功能基因学成为可能[18]。
LBD(Lateral organ boundaries domain)基因家族,也称AS2(Asymmetric leaves 2)基因家族,是一类植物中特有的DNA 结合转录因子,在植物生长发育过程中发挥着重要作用[19]。LBD 基因家族成员均包含LOB蛋白结构域,包括一个由高度保守的C-x(2)-C-x(6)-C-x(3)-C 半胱氨酸基序构成结合DNA 必须的锌指结构域;一个由1个保守的脯氨酸残基构成地与DNA 结合活性密切相关的GAS区域;一个参与蛋白质二聚化过程的由4个保守的亮氨酸残基L-x(6)-L-x(3)-L-x(6)-L组成的类亮氨酸拉链样基序[20-22]。LBD 基因家族可分为两个亚家族:含有上述3个完整基序的为第一个亚家族Class Ⅰ,而其余含有残留亮氨酸拉链基序的为第二个亚家族ClassⅡ[23-24]。截至目前,LBD 基因家族已在多个物种的全基因组水平上被鉴定,例如拟南芥43个[21]、小 麦75 个[25]、玉 米44 个[26]、水 稻35个[27]、大麦24 个[24]、二穗短柄草28 个[28]、烟草98个[29]等。已有的研究表明,LBD 基因家族在植物生长、发育和代谢等多个方面发挥了重要作用。如拟南芥LBD13和LBD16调控侧根的生长[30-31]、LBD19在愈伤组织形成中起负调控作用[32]、LBD29参与生长素的调控抑制纤维素合成[33];水稻OsIG1基因调控水稻的小花数和配子体结构[34];番茄SlLBD40基因参与茉莉酸信号转导是抗旱性的负调控因子[35];小立碗藓PpLBD27基因在干旱胁迫下表达且响应茉莉酸介导的对病原菌的抵抗[36];丹参LBD50基因调控茉莉酸信号转导和酚类生物合成[37];茶树Cs-LBDs基因调控类黄酮的合成[38]。尽管LBD 基因家族已经在多个物种中被鉴定和研究,但在长穗偃麦草中的鉴定和研究还未见报道。
本研究借助生物信息学方法,对长穗偃麦草LBD 基因家族进行鉴定,并对其基本特征、保守结构域、染色体分布、基因的进化和启动子的顺式作用元件进行系统的分析。同时,对长穗偃麦草LBD 基因家族与普通小麦、圆锥小麦、乌拉尔图小麦、粗山羊草和大麦进行共线性分析,揭示小麦及其近缘属物种在进化过程中染色体的重复、缺失、易位等事件的发生。本研究为长穗偃麦草LBD 基因家族中单个基因的功能分析奠定基础,同时为分析小麦及其近缘属的进化提供参考。
1 材料与方法
1.1 材料
CNCB-NGDC数据库(https://bigd.big.ac.cn/)中长穗偃麦草的全基因组数据;拟南芥数据库 (https://www.arabidopsis.org/browse/genefamily/index.jsp)中LBD 基因家族的蛋白query 序列;Ensembl(https://plants.ensembl.org/index.html)数据库中小麦[39]、圆锥小麦[40]、乌拉尔图小麦[41]、山羊草[42]、大麦[43]的基因组数据。
1.2 方法
1.2.1 长穗偃麦草LBD 基因家族的鉴定 通过长穗偃麦草的全基因组数据,利用TBtools[44]软件以拟南芥的LBD 基因家族的蛋白序列作为query序列,进行本地比对获得长穗偃麦草LBD基因家族成员的可能序列;同时利用Uniprot数据库 (https://www.uniprot.org/uniprot/?query=&sort=score)继续筛选LBD 基因家族成员,去除冗余序列。利用Pfam(https://pfam.xfam.org/search#tabview=tab1)和NCBI-CDD(https://www.ncbi.nlm.nih.gov/Structure/bwrpsb/bwrpsb.cgi)数据库对保守结构域进一步筛选,去除保守结构域中不完整的序列。利用ExPASy(https://web.expasy.org/protparam/)在线软件对LBD 蛋白的基本特性进行分析。
1.2.2 长穗偃麦草LBD 基因家族的系统发育树的构建 利用在线工具Clustal W(https://www.genome.jp/tools-bin/clustalw)进行长穗偃麦草、小麦、水稻和拟南芥LBD 蛋白的多序列比对,然后使用MEGA 7.0的最大似然法(ML)构建系统发育树,并将bootstrap 参数设置为1 000。系统发育树使用Fig Treev 1.4.4 软件(http://tree.bio.ed.ac.uk/software/figtree/)进行美化。
1.2.3 长穗偃麦草LBD 基因结构和蛋白结构域预测和亚细胞定位 利用TBtools工具分析和可视化长穗偃麦草LBD 基因的外显子/内含子结构。使用MEME在线软件(http://meme-suite.org/)预测LBD 基因家族中蛋白质的保守结构域,重复设定为One Occurrence Per Sequence,宽度为6~50 个氨基酸,最大基序为10。利用SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl? page=npsa_sopma.html)二级结构预测分析软件对LBD 基因蛋白进行二级结构的预测和分析。利用基于同源建模法的在线软件SWISS-MODEL(https://swissmodel.expasy.org/)对LBD 蛋白的三级结构进行预测。使用在线工具 PSORT Prediction(http://psort1.hgc.jp/form.html)进行亚细胞定位。
1.2.4 长穗偃麦草LBD 基因的顺式作用元件分析 利用TBtools截取长穗偃麦草全基因数据中LBD 基因上游2 kb 的基因组序列,利用Plant-CARE启动子预测数据库(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)对顺式作用元件预测并手工简化和整理保留有一定查看目的元件,使用在线网站GSDS2.0(http://gsds.gao-lab.org/)对顺式作用元件进行可视化并绘制顺式作用元件分布图,并利用Origin 2017软件对各顺式作用元件在LBD 基因家族中分布的数目绘制热图。
1.2.5 长穗偃麦草LBD 基因家族的染色体定位及与小麦族物种的共线性分析 利用长穗偃麦草的基因注释文件和LBD 基因家族的基因ID,通过TBtools工具实现染色体位置的可视化。使用MCScan X 来检测串联重复基因以及长穗偃麦草自身基因的同源关系及其进化分析。通过自身的共线性分析以及长穗偃麦草与其他5个物种的共线性分析,分析长穗偃麦草LBD 基因家族自身的进化以及与小麦族5 个物种的共线性比对分析LBD 基因在小麦及其近缘属中的进化,通过TBtools工具对共线性分析的结果可视化。
2 结果与分析
2.1 长穗偃麦草LBD基因家族的鉴定
在长穗偃麦草中共鉴定到32个LBD 基因,并根据该基因在染色体上的位置命名。长穗偃麦草LBD 基因的蛋白序列长度为124~383 aa;分子质量为13.5~41.7 ku;等电点为5.05~9.75;不稳定系数为29.53~74.45,其中只有Tel-1ELBD4和Tel-3E-LBD5两个基因的不稳定系数小于40,是稳定蛋白;脂肪族氨基酸指数为56.79~88.21;亲水指数为-0.621~0.11(表1)。
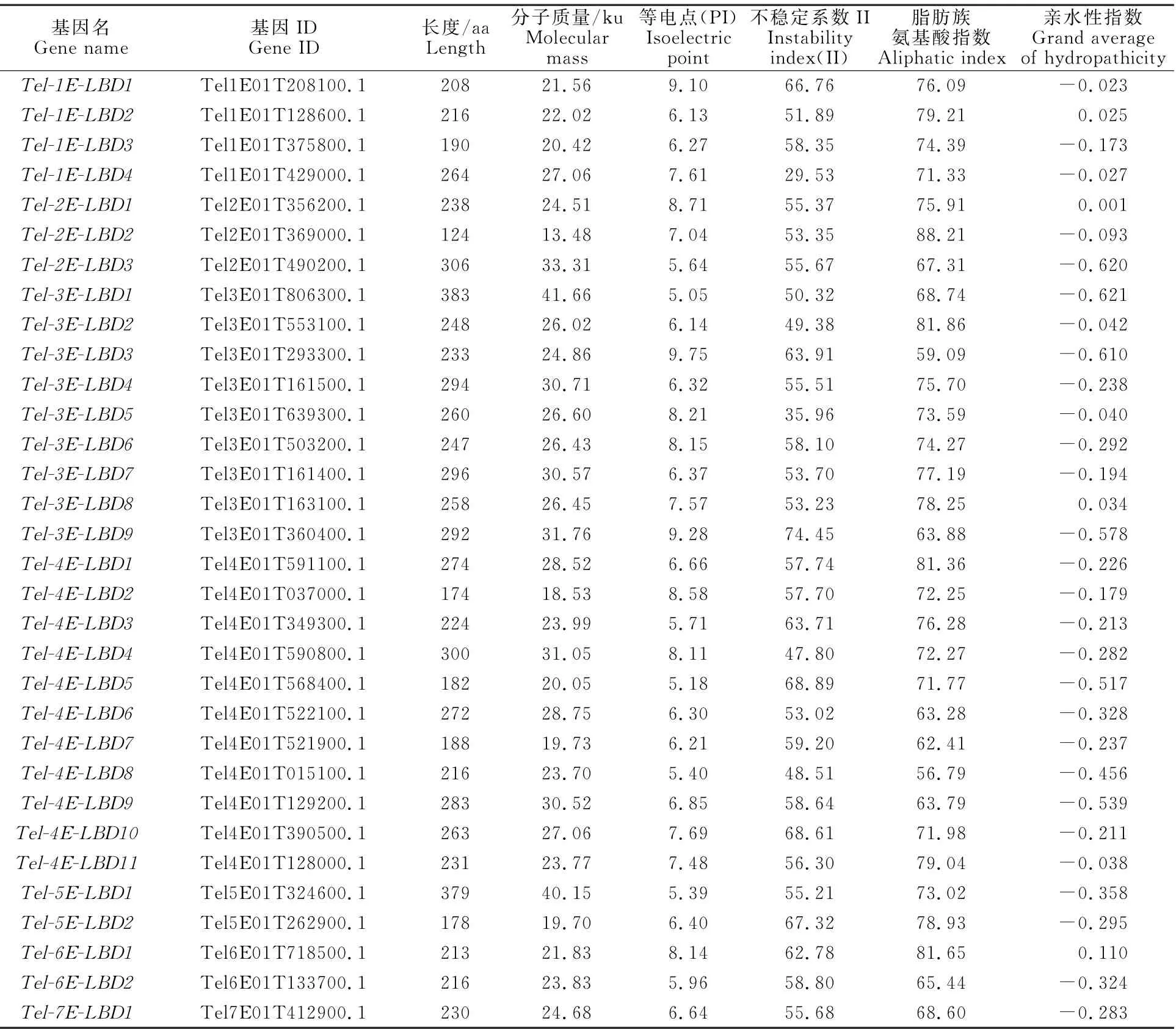
表1 长穗偃麦草LBD蛋白的理化性质Table 1 Physicochemical properties of LBD protein in Thinopyrum elongatum
2.2 长穗偃麦草LBD基因家族的系统发育分析和分类
2.2.1 长穗偃麦草LBD 基因家族系统发育树的构建 根据LBD 基因家族蛋白多序列比对构建的无根系统发育树(图1),可将所有LBD 基因分为ClassⅠ和ClassⅡ两个亚族7大类,在每大类中均包含有长穗偃麦草、小麦、水稻和拟南芥的LBD 基因,表明在双子叶和单子叶植物分化之前LBD基因已经完成了分化。在长穗偃麦草32个LBD 基因中,两个亚家族都有明显的特征。ClassⅠ亚家族包含25个成员,含有完整的高度保守的半胱氨酸结构域C-x(2)-C-x(6)-C-x(3)-C、GAS基序和类亮氨酸拉链基序L-x(6)-L-x(3)-L-x(6)-L;Class Ⅱ亚家族包含7个成员,只含有完整的、高度保守的半胱氨酸基序C-x(2)-C-x(6)-C-x(3)-C。根据保守结构域的数目和位置情况,Class Ⅰ可以进一步细分为5 个亚类:ⅠA~ⅠE,ClassⅡ可分为ⅡA~ⅡB两个亚类。
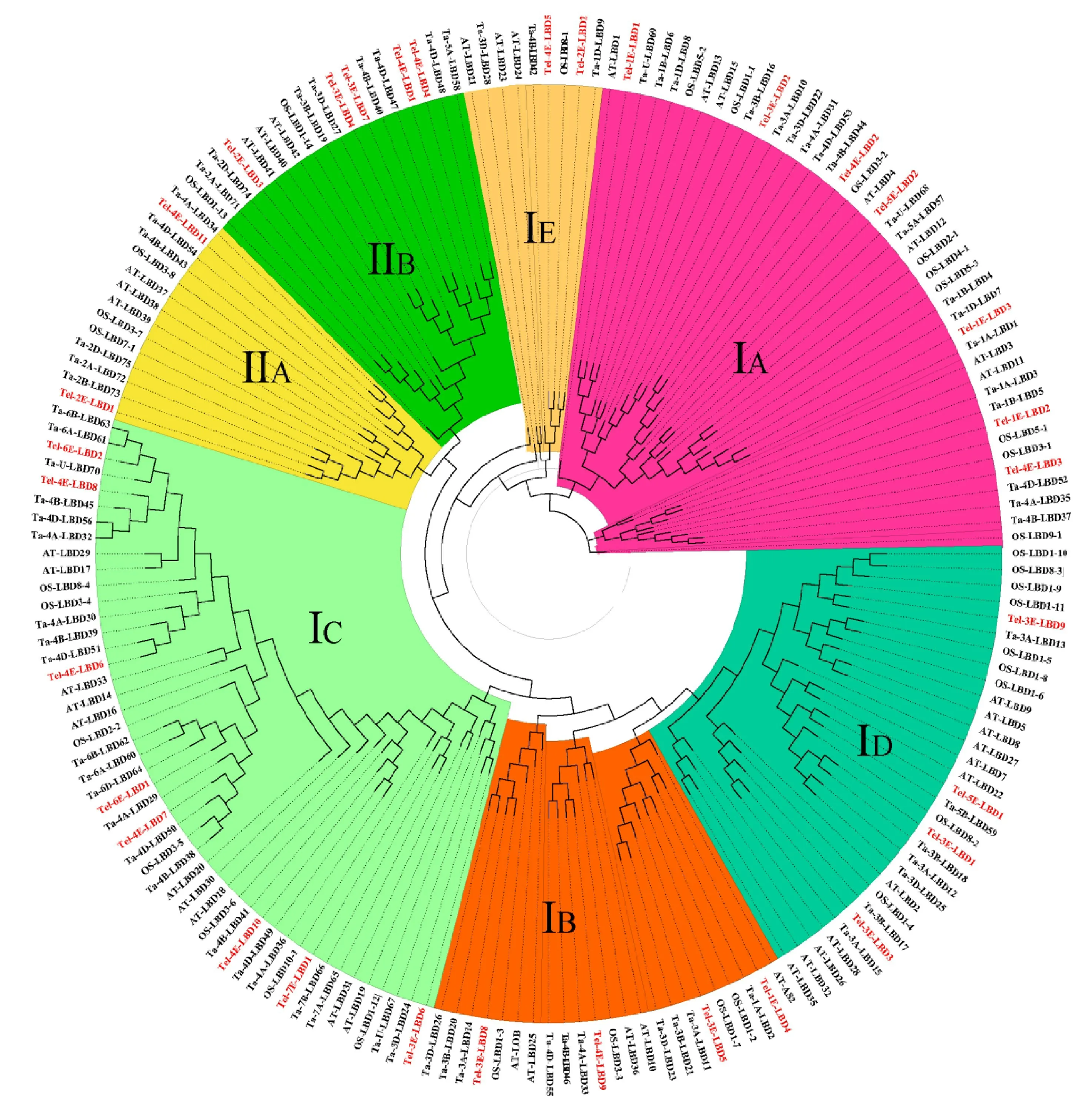
图1 长穗偃麦草、小麦、水稻、拟南芥LBD蛋白系统发育树Fig.1 Aphylogenetic tree of LBD proteins of Thinopyrum elongatum,Triticum aestivum,Oryza sativa Japonica and Arabidopsis
2.2.2 长穗偃麦草LBD 基因家族蛋白的二级结构预测及亚细胞定位 在蛋白质二级结构预测和分析的结果中发现,Tel-4E-LBD3和Tel-2ELBD2两个基因二级结构只含有α-螺旋和无规则卷曲两种构型,其余基因均含有3 种构型。在LBD 基因中α-螺旋分布由24.75%到67.23%,无规则卷曲分布由2.12%至58.42%,这两种构型在蛋白序列中均有分布且占比相对较大;β-折叠构型在含有的基因蛋白序列中分布由0.40%至11.81%占比相对较小(表2)。亚细胞定位结果表明,长穗偃麦草的LBD 基因定位到线粒体12个,叶绿体5个,细胞质10个,细胞核2个,内质网1个,其他2个(表2)。
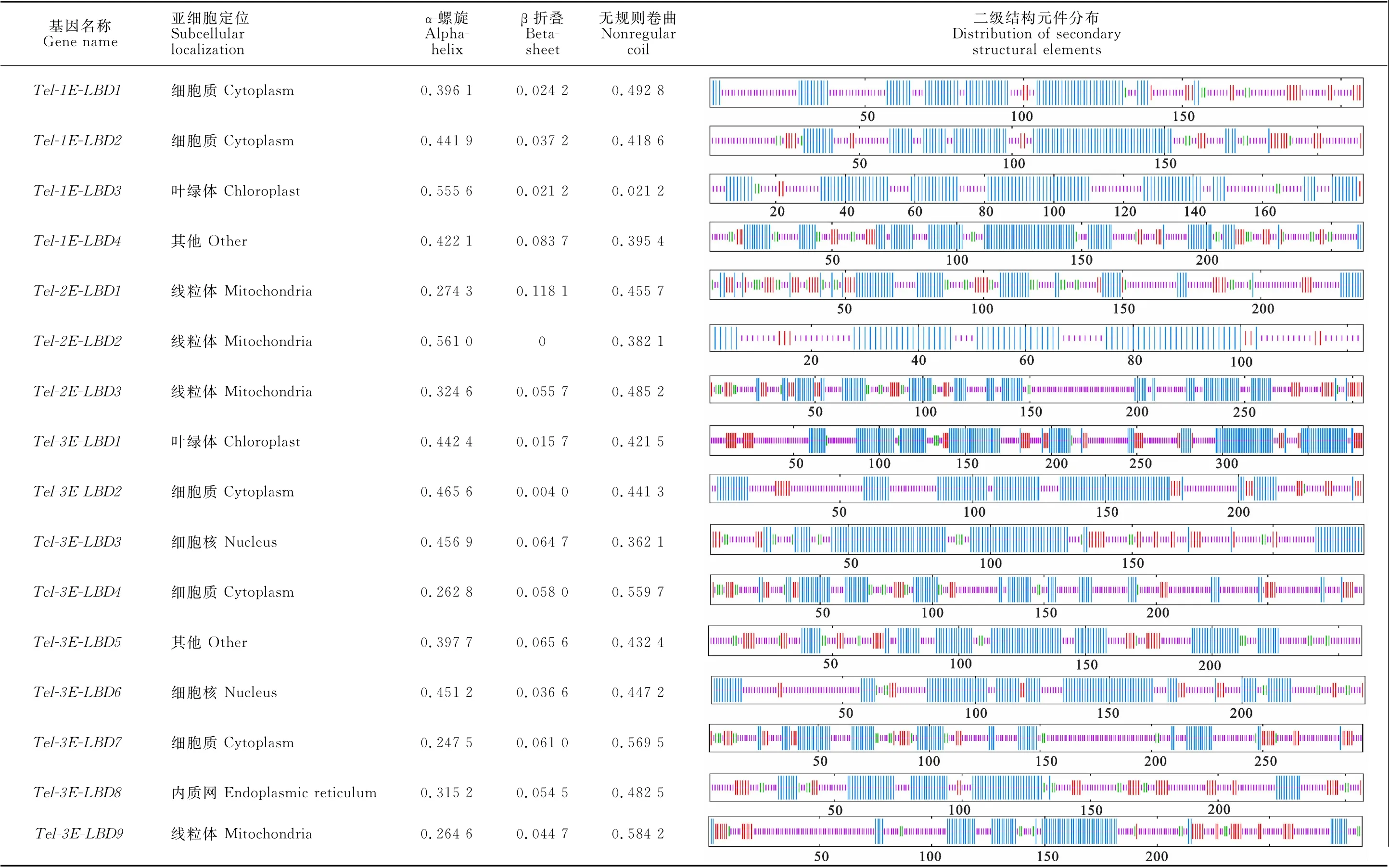
表2长穗偃麦草LBD 蛋白亚细胞定位与二级结构分析Table 2 Subcellular localization and analysis of protein secondary structure of LBD protein in Thinopyrum elongatum
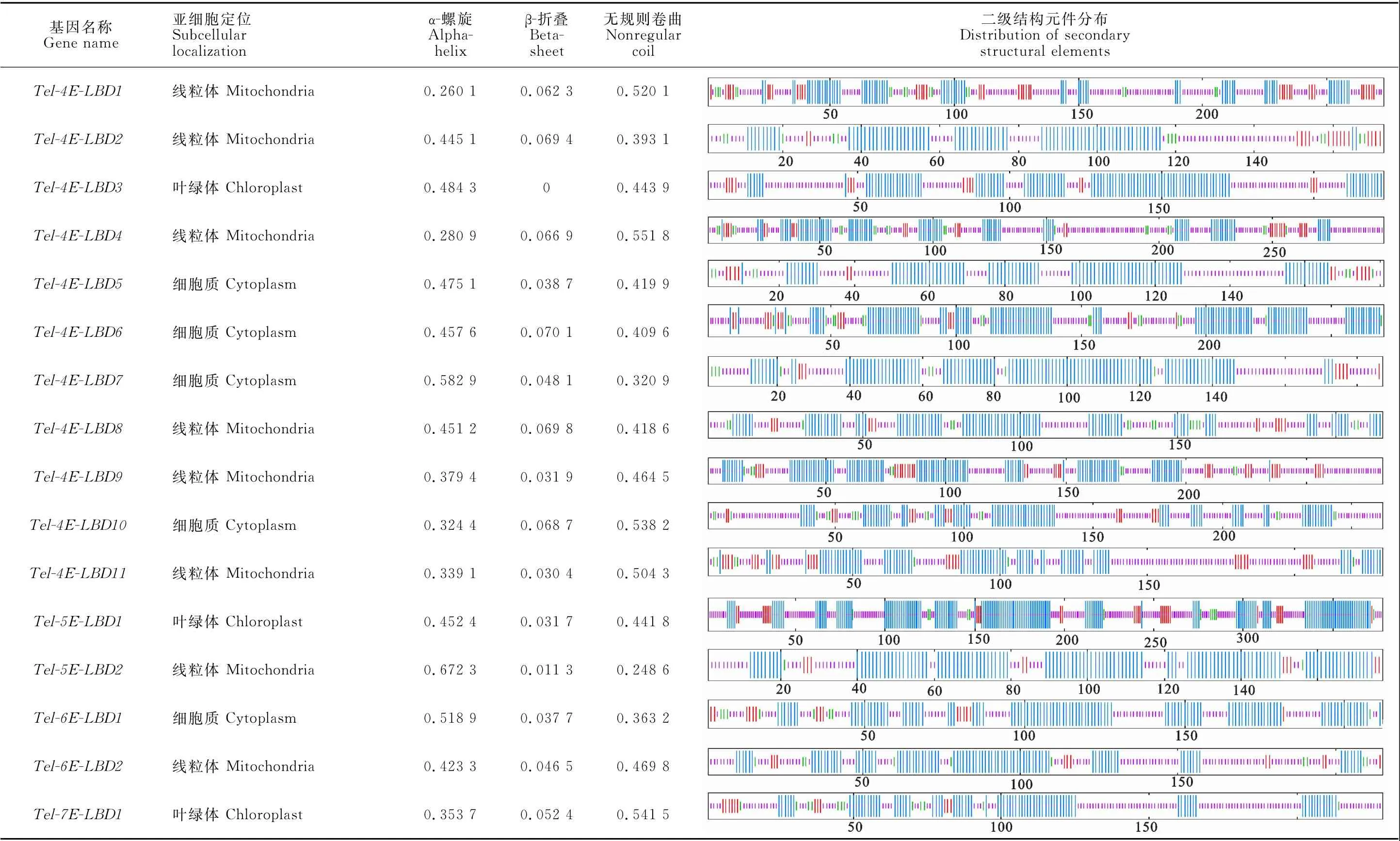
(续表2 Continued table 2)
2.2.3 长穗偃麦草LBD 基因家族蛋白的三级结构预测 蛋白质的三级结构预测表明,长穗偃麦草LBD 基因蛋白保守性强,各个蛋白存在一定的差异,但总体上LBD 基因蛋白的三级结构相似(图2)。其中Tel-3E-LBD8基因与比对到的模型蛋白一致度达到99.22%,总体上来说预测到的LBD 基因家族三级结构与模型蛋白具有较好一致度。
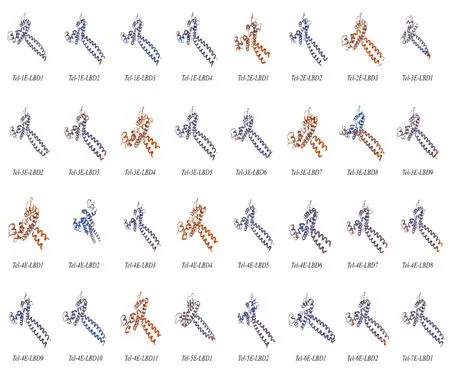
图2 长穗偃麦草LBD蛋白质三级结构预测Fig.2 Prediction of protein tertiary structure of LBD protein in Thinopyrum elongatum
2.3 长穗偃麦草LBD基因蛋白的结构分析及保守结构域分析
分析了长穗偃麦草LBD 基因的结构(图3)。结果表明长穗偃麦草LBD 基因家族的基因结构相对简单,其中3个基因Tel-5E-LBD2、Tel-4ELBD8、Tel-LBD3均有两个内含子,Tel-3ELBD7含有一个内含子,其余LBD 基因均不包含内含子。而外显子数量分布为2至4个,其中大部分基因含有4个外显子。总体来看,每个亚家族成员间基因结构基本一致,但在两个亚家族之间有所差异。

图3 长穗偃麦草LBD保守基序(左)与基因结构(右)Fig.3 Conservative motif(left)and gene structure(right)of LBD in Thinopyrum elongatum
对LBD 基因家族32个成员蛋白的特征区域其保守结构域的数目及组成位置的分析,预测了10个保守的基序,并列出特征基序的具体氨基酸序列(motif 1、motif 3、motif 5)(图4)。其中基序1和基序2在所有LBD 基因中均被鉴定到,表明基序1和基序2最为保守。在ClassⅠ亚家族中均鉴定到基序1、2、3和5,在ClassⅡ亚家族中均鉴定到基序1、2、4和6,这表明同一亚家族的成员大多具有相同的序列和共同位置的基序,在同一亚群中聚集的LBD 成员可能具有相似的生物学功能。
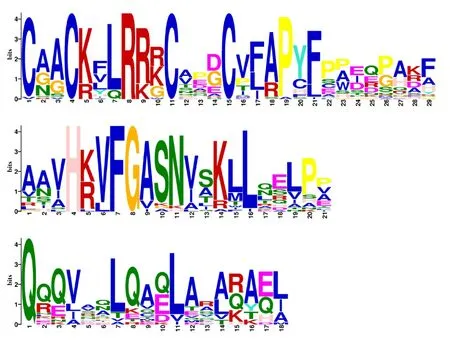
图4 长穗偃麦草LBD基因家族特征结构域Fig.4 Characteristic domain of Thinopyrum elongatum LBD gene family
2.4 长穗偃麦草LBD基因家族的顺式作用元件分析
通过对长穗偃麦草LBD 基因上游2 kb基因组顺式作用元件预测,去除启动子和增强子区域共鉴定到43 类顺式作用元件,根据其功能分为12大类(图5-A)。在所有LBD 基因上游区域中均含有多个光响应元件和MeJA(茉莉酸甲酯)响应元件,其中防御和应激响应元件分布最少,只在8个基因上游区域存在,此外还有MYB 结合位点、赤霉素响应、脱落酸响应元件、水杨酸响应元件、低温响应元件、生长素响应元件、厌氧诱导响应元件、玉米醇溶蛋白代谢调控元件和分生组织响应元件9类不同的顺式作用元件不均匀地分布在LBD 基因的上游区域(图5-B)。
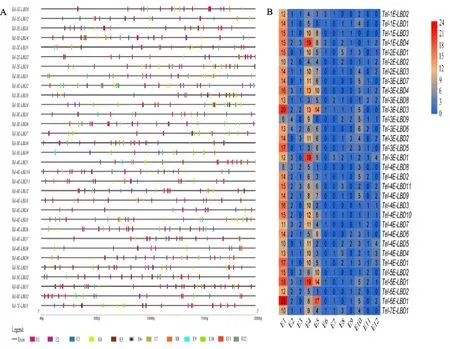
图5 长穗偃麦草顺式作用元件分布(A)与数量分布(B)Fig.5 Cis-acting elements distribution(A)and quantity distribution(B)in Thinopyrum elongatum
2.5 LBD 基因家族的染色体定位、进化分析及与小麦及其近缘属物种的共线性分析
2.5.1 长穗偃麦草LBD 基因家族的染色体定位及自身的进化分析 染色体定位结果表明,LBD基因家族中的32个成员分布在7条染色体上,在3E和4E染色体上存在基因簇(图6)。通过共线性分析,结果发现在3E 染色体上Tel-3E-LBD4基因和Tel-3E-LBD7基因存在串联重复现象,4E染色体上Tel-4E-LBD1基因和Tel-4E-LBD4基因存在串联重复现象(图6)。同时在32个LBD基因中发现了5对存在共线性的基因,其中在1E与3E染色体存在2对共线性基因、2E分别与4E和6E各存在1对共线性基因、4E与6E存在1对共线性基因。
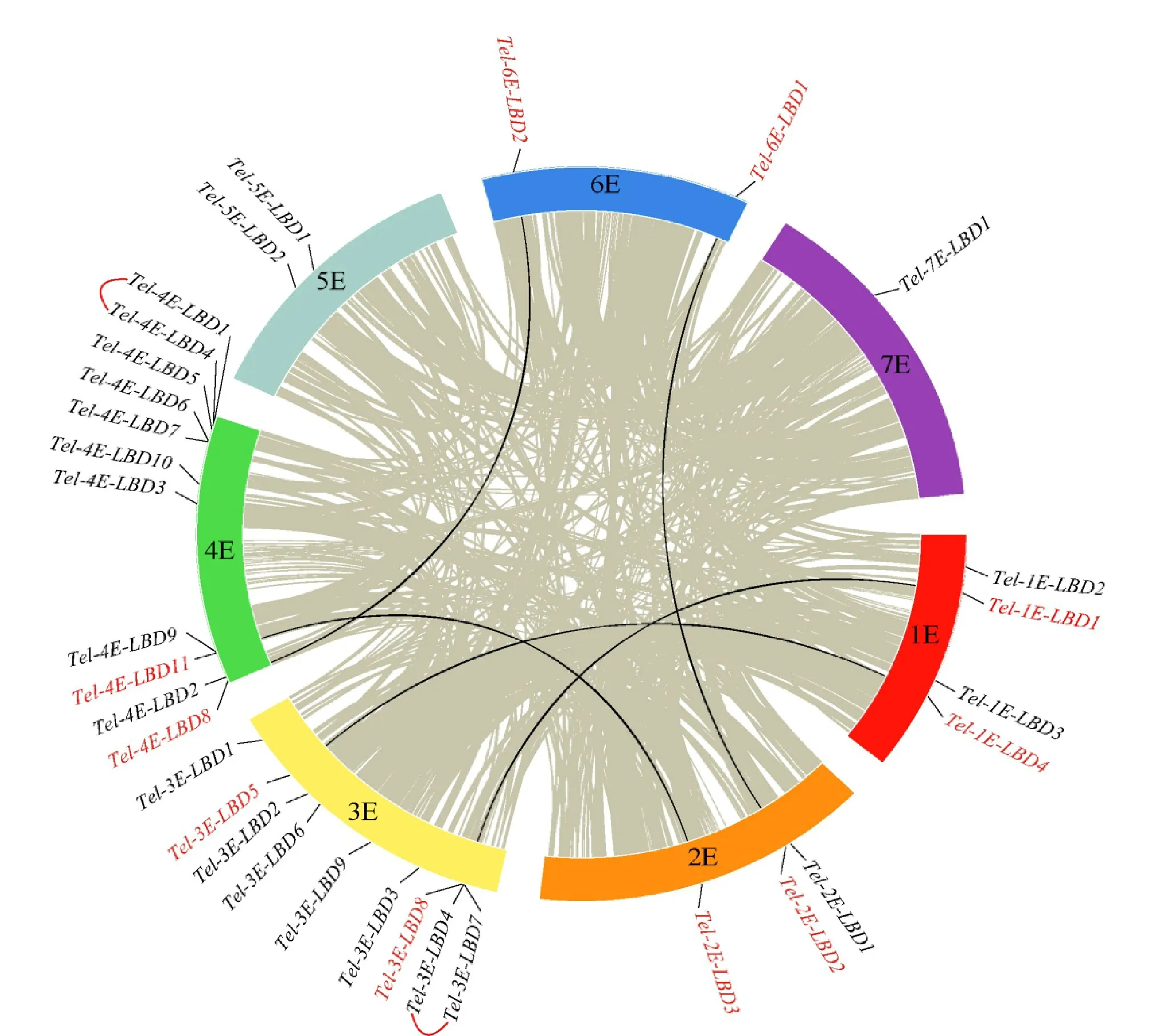
图6 长穗偃麦草LBD基因的共线性分析Fig.6 Collinearity analysis of LBD gene in Thinopyrum elongatum
2.5.2 基于长穗偃麦草LBD 基因家族与5个小麦族物种的共线性分析 长穗偃麦草与5个小麦族内物种的共线性分析发现长穗偃麦草LBD 基因在小麦族中具有较好的保守性。同时,鉴定到长穗偃麦草特有的LBD 基因Tel-2E-LBD2、Tel-3E-LBD4、Tel-4E-LBD1、Tel-4E-LBD6(图7)。在共线性分析过程中还发现LBD 基因家族中有染色体重复、易位和倒位等事件的发生。如Tel-1E-LBD4和Tel-3E-LBD5基因在A、B、D 和H 基因组中的第1、3同源群均存在1个共线性基因,表明A、B、D、H 基因组在进化过程中发生了染色体重复现象;在普通小麦的4A 染色体上与长穗偃麦草Tel-4E-LBD3和Tel-4E-LBD10存在共线性的对应的基因在4A 染色体上发生了易位现象;在长穗偃麦草中基因的排列顺序为Tel-4E-LBD2、Tel-4E-LBD11、Tel-4E-LBD9,在 普通小麦和圆锥小麦4A 对应的存在共线性的基因上发生了倒位现象,染色体位置变为Tel-4ELBD9、Tel-4E-LBD11、Tel-4E-LBD2。此外在长穗偃麦草上LBD 基因与普通小麦的A、B、D 基因组存在共线性基因数均最多,说明在进化的过程中普通小麦基因组染色体间发生了大量的易位与重复,A 基因组和B基因组重排区块明显高于D 基因组,这与普通小麦形成过程中染色体组的加入相一致。
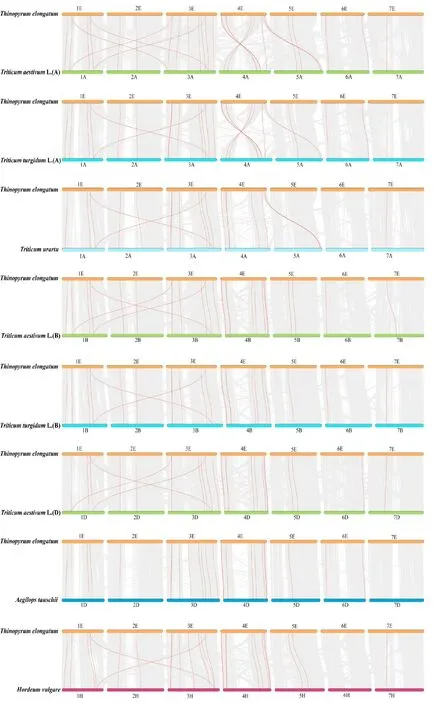
图7 长穗偃麦草与5个物种间(A、B、D、H 基因组)的共线性分析Fig.7 Collinear analysis of Thinopyrum elongatum and five species(A、B、D、H genome)
3 讨论
LBD 基因的表达与上游启动子区域的顺式作用元件有关,LBD 基因上游顺式作用元件涉及到植物生长发育调控和逆境胁迫。如拟南芥DDA1基因在光形态建成中发挥作用[45],AtLBD40基因参与了植株的赤霉素响应[46],ASL1、ASL16、ASL18、ASL20基因响应生长素应答 元 件 ARF7 和 ARF19 调 控 侧 根 的 生长[20,47],AtLBD14参与ABA 的响应调控侧根的生长[48-49]。水稻Os-LBD37基因参与了氮代谢调节[50]。大豆GmLBD12基因参与了逆境响应[51]。香蕉MaLBD5基因参与了茉莉酸甲酯介导的耐寒性的转录调控[52]。长穗偃麦草LBD 基因的启动子区域分布着43类与生长发育调控和逆境胁迫相关的顺式作用元件,如Tel-1ELBD4、Tel-3E-LBD1和Tel-6E-LBD1基因存在丰富的茉莉酸甲酯响应元件;Tel-3E-LBD3基因含有丰富的光响应元件;Tel-3E-LBD2含有生长素响应元件等。表明长穗偃麦草中的LBD基因参与了光、激素、逆境等的表达和调控,为深入研究长穗偃麦草中LBD 基因家族中具体成员的功能奠定基础。
基因重复产生功能差异,促进了新基因的生成、对环境适应以及在新物种形成中具有重要的作用[53]。不同的物种中均存在串联重复事件,这表明基因重复在进化中起着重要的推动作用[54]。例如AP2/ERF和WRKY 基因家族的扩展主要是全基因组倍增、片段复制和串联重复[55-56]。长穗偃麦草LBD 基因家族内共检测到5对共线性基因和2对串联重复基因,说明长穗偃麦草在长期的进化过程中出现了基因重复,而基因重复事件的出现促进了基因新功能产生。
尽管小麦近缘种属的优异性状已经在小麦生产中得到了大量的应用,但由于外源染色体片段和小麦染色体片段在减数分裂过程中不进行联会,以至于通过正向遗传学的方法很难对外源基因进行定位和克隆。因此,近缘种中绝大多数基因的克隆及作用机理的研究相对滞后。本研究从基因组水平完成了对长穗偃麦草LBD 基因家族的鉴定与生物信息学分析,与传统的基因鉴定方法相比,可以快速完成对单个基因的初步鉴定,为后期对这些基因的深入研究和利用奠定基础。
——以多重共线性内容为例

