基于宏基因组分析酸马奶的微生物多样性及功能基因
夏亚男, 刘 皓, 双 全
(内蒙古农业大学食品科学与工程学院 呼和浩特010000)
传统酸马奶(Koumiss)制作和食用历史悠久,是最受中亚和东欧地区人们喜爱的传统发酵乳饮料,它不仅营养丰富,而且对某些疾病有治疗效果[1-2]。在我国,酸马奶是一种具有民族特色的发酵乳饮料, 我国内蒙古和新疆地区人民对其十分偏爱。新鲜的马奶在乳酸菌、酵母菌和其它微生物的共同作用下,经特定温度和时间发酵形成酸马奶。酸马奶具有良好的、 特殊的风味口感和一定的医疗保健作用,如有助于人体消化吸收、预防各种肠胃道疾病,还可以降低血脂和胆固醇[3-4]。
酸马奶独特风味及功能特性的形成与其复杂的微生物群落密切相关。 微生物将鲜马奶中的各类营养物质(碳水化合物、蛋白质、脂肪酸等)不断分解、代谢,生成相应的风味物质以及具有特定功能的生物活性成分。 近年来,采用16s rRNA 技术[5-6]检测酸马奶的微生物多样性时发现传统方法发酵得到的酸马奶中, 乳酸菌和酵母菌占菌群的绝大部分,不同微生物之间存在相互作用,其微生物菌群体系与开菲尔非常相似[7-8]。采用16s rRNA技术检测酸马奶的微生物多样性只能确定到属水平,无法精确到种水平,对功能基因的挖掘存在一定的局限性。
宏基因组学(Metagenonics)是基于提取特定环境中全部微生物的DNA, 构建宏基因组文库,来探索微生物群落功能, 挖掘功能基因的一门技术[5-8]。传统宏基因组学研究技术包括芯片、克隆测序、变性梯度凝胶电泳等,这些技术已在许多领域取得重大突破, 例如在土壤和海洋等环境样品的研究中[9-11]。 Li 等[12]通过宏基因组学技术研究3 种牛乳(新鲜牛乳、含抗生素的牛乳以及经巴氏杀菌且含有抗生素的牛乳) 对牛犊瘤胃中微生物的影响,结果表明,饮用含有抗生素的牛乳影响瘤胃中微生物群落结构及丰度, 同时与细胞周期相关的酶的数量也发生变化。 通过对功能基因及关键酶的挖掘,揭示牛乳中抗生素残留的安全问题。
目前,已有对酸马奶微生物多样性的研究,然而,微生物在种水平上尚不明确,功能基因也几乎处于空白阶段,需要开展深入研究。本试验利用宏基因组学和生物信息学分析手段, 在分类学地位“种”水平上对酸马奶中核心微生物类群进行甄别,分析酸马奶的微生物多样性,并挖掘群体微生物中的功能基因, 以期为我国酸马奶产业的发展及乳酸菌资源开发提供理论依据。
1 材料与方法
1.1 试验材料
酸马奶于2019年6月采自于内蒙古自治区锡林郭勒盟阿巴嘎旗。发酵方法:经前处理后的新鲜马奶倒入酸马奶发酵桶中,按照6%的接种量添加酸马奶自然发酵剂并在22~26 ℃进行发酵,定时搅拌确保混合均匀且快速发酵。 整个发酵过程持续30 h, 发酵结束后将酸马奶样品密封并在干冰中冷却,转移至实验室-80 ℃冷冻保存用于后续试验。
1.2 试剂与仪器
NanoDrop2000 超微量分光光度计, 美国Thermo Fisher Scientific 公司;DYY-6C电泳仪,北京市六一仪器厂;GeneAmp9700 型PCR 仪,美国ABI 公司;5424R 高速台式冷冻离心机, 美国Eppendorf 公司;Miseq PE300 测序仪, 美国Illumina 公司。
1.3 试验方法
1.3.1 样品DNA 抽提 酸马奶经过离心处理后,采用E.Z.N.A土壤DNA 提取试剂盒提取酸马奶中全部微生物的DNA, 再利用1%琼脂糖凝胶进行电泳检测抽提微生物基因组的DNA。
1.3.2 宏基因组检测
1) 构建PE 文库 连接“Y”字形接头;通过磁珠筛选去除接头自连片段;利用PCR 扩增进行文库模板的富集;氢氧化钠变性,产生单链DNA片段。
2) 桥式PCR 引物碱基与DNA 片段的一端互补, 固定在芯片上;DNA 片段的另一端随机与附近的另外一个引物碱基互补, 也被固定在芯片上,形成“桥(Bridge)”;利用PCR 扩增,产生DNA簇;DNA 扩增子线性化成为单链。
3) Illumina Hiseq 测序 加入改造过的DNA聚合酶和带有4 种荧光标记的dNTP,每次循环只合成一个碱基;用激光扫描反应板表面,读取每条模板序列第一轮反应所聚合上去的核苷酸种类;将“荧光基团”和“终止基团”化学切割,恢复3' 端黏性,继续聚合第二个核苷酸;统计每轮收集到的荧光信号结果,获知模板DNA 片段的序列。
1.3.3 数据分析
1.3.3.1 基因序列拼接组装及预测 使用Megahit拼接软件对clean 序列进行拼接组装,根据不同的kmer 大小进行组装, 从中选择最优结果。 使用MetaGeneMark 对拼接结果中的conting 进行基因预测,并将其翻译为氨基酸序列。
1.3.3.2 物种注释及丰度分析 通过使用Kraken软件对测序酸马奶数据进行物种注释, 获得物种分类信息。
1.3.3.3 功能基因注释分析
1) COG 注释 通过与COG 数据库进行blastp 比对, 酸马奶基因可以获得对应的COG 注释,再根据COG 的功能编号进行分类;
2) KEGG 注释 运用FMAP 软件对KEGG注释和差异Module 分析;
3) CAZy 注释 通过与CAZy 数据库进行比对分析, 碳水化合物酶类的物种来源、 酶功能分类、基因序列、蛋白质序列及其结构等信息就可以清楚地展现出来。
2 结果与分析
2.1 酸马奶微生物多样性分析
经检测,酸马奶一共鉴定到72 364 条组装序列,总碱基(15 932 809 200)中碱基G 和C 的含量为45.29%, 碱基质量大于或等于20 的碱基可达97.09%。 基于酸马奶内的物种系统进化关系及相对信息, 使用KRONA 软件对酸马奶样物种进行标注, 酸马奶中微生物分类在门水平上有30 个,在科水平上有331 个,在属水平上有913 个,在种水平有2 692 个。 其中,相对含量大于1%的优势菌群在门水平上有2 个,在科水平上有3 个,在属水平上有6 个,在种水平有8 个(图1)。
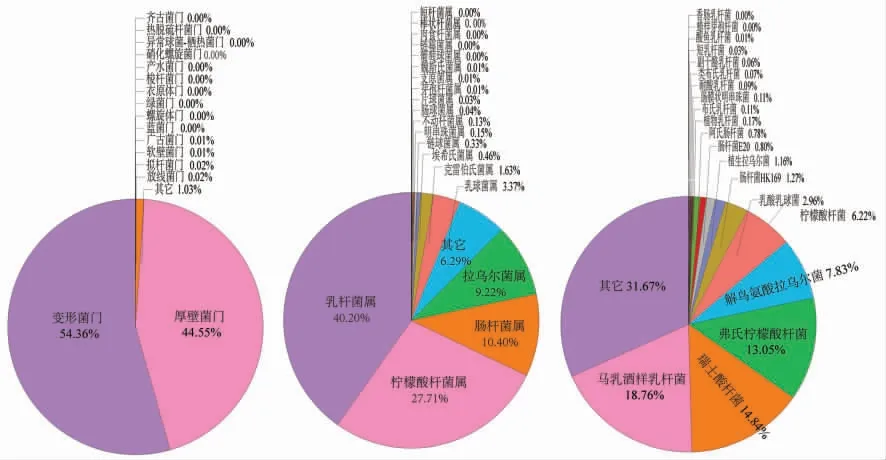
图1 酸马奶样品微生物群落饼图Fig.1 Pie chart of microbial community of koumiss
在门水平上,排名前6 位的分别是变形菌门、厚壁菌门、放线菌门、拟杆菌门、软壁菌门和广古菌门。优势菌群是变形菌门和厚壁菌门,相对含量分别为54.36%和44.55%,占据总含量的98.91%。在属水平上,排名前5 位的优势菌属是乳杆菌属、柠檬酸杆菌属、 肠杆菌属、 拉乌尔菌属和乳球菌属, 相对含量分别为40.20%,27.71%,10.40%,9.22%和3.37%,占据总含量的90.90%。 此外,在酸马奶中发现了埃希氏菌属(0.46%)、克雷伯氏菌属(1.63%)及微量的芽孢杆菌属(0.01%)和支原菌属(0.01%)。由于传统酸马奶的制作过程中缺少杀菌工艺, 这导致酸马奶中微生物的群落信息复杂多样,所以酸马奶中除了有益菌乳酸菌等外,还存在着少量的腐败菌和有害微生物。近些年来,大量文献报道称酸马奶中发现较多的抑菌微生物,能产生抗菌肽等抗菌成分[13-15],这也解释了未经杀菌操作的酸马奶未产生安全问题的原因。尽管如此,在充分研究酸马奶微生物及保健功能的基础上,仍需要进一步研究适合于鲜马奶的杀菌技术,以保证酸马奶稳定的品质及更加突出的保健功效。
在种水平上,相对含量大于2.0%的优势菌种主要包含马乳酒样乳杆菌、瑞士酸杆菌、弗氏柠檬酸杆菌、解鸟氨酸拉乌尔菌、柠檬酸杆菌和乳酸乳球菌, 其平均相对含量分别为18.76%,14.84%,13.05%,7.83%,6.22%和2.96%, 其余细菌相对含量较少。 其中,马乳酒样乳杆菌、瑞士乳杆菌和乳酸乳球菌为酸马奶常见优势菌种,本试验中,弗氏柠檬酸杆菌、 解鸟氨酸拉乌尔菌和柠檬酸杆菌平均相对含量也较高, 且与文献报道的酸马奶中常见优势菌种含量相近[16-18],由此可以判断弗氏柠檬酸杆菌、 解鸟氨酸拉乌尔菌和柠檬酸杆菌也属于酸马奶的优势菌种。 由于研究酸马奶微生物多样性确定到细菌种水平的报道较少, 该结果丰富了酸马奶微生物多样性的具体类别信息。
2.2 常用数据库注释分析
2.2.1 COG 数据库注释分析 经COG 注释分析,酸马奶的10 849 个基因根据其功能大致可分为23 类,其中,去除功能未知的R 和S 两部分,其中功能基因数目在800 个以上, 主要有氨基酸转运和代谢类基因(1 208 个)、翻译、核糖体结构及合成基因(1 207 个)、碳水化合物转运与代谢类基因(947 个)、转录类基因(931 个)和复制、重组和修复类基因(807 个);其它类的功能基因统计结果见图2。
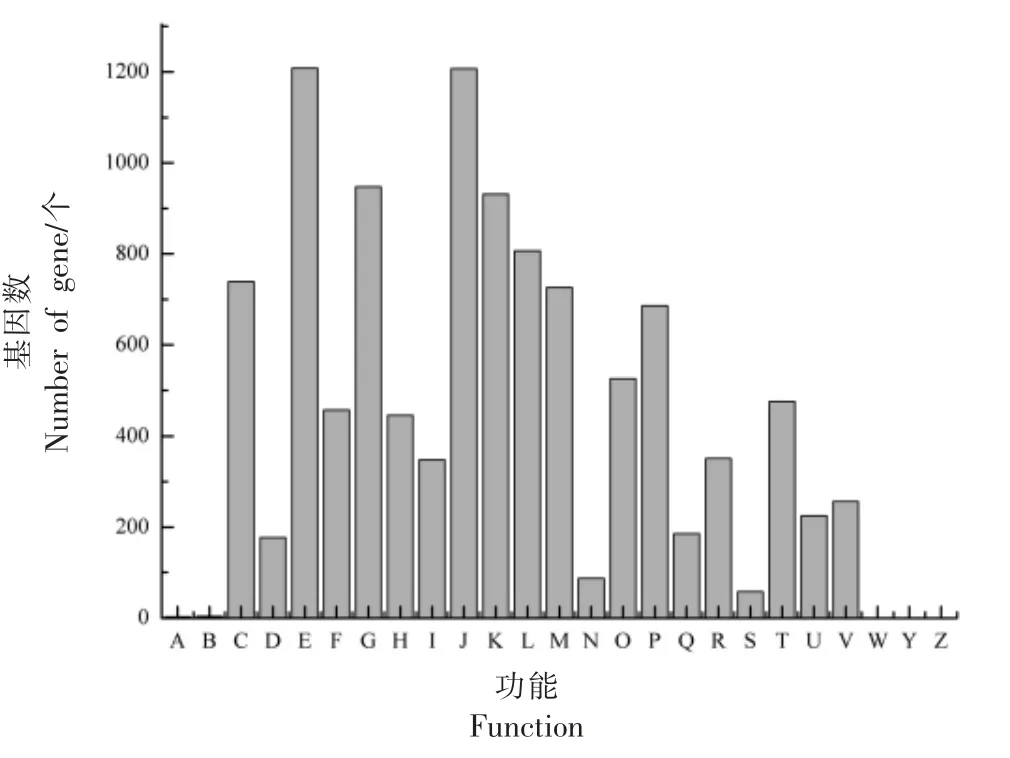
图2 酸马奶中COG 主要功能分类的基因数目Fig.2 The number of genes in COG primary functional classification in koumiss
2.2.2 KEGG 数据库注释分析 运用FMAP 软件对酸马奶的214 338 个基因进行KEGG 注释和差异Module 分析,统计其可能参与或涉及的代谢途径。 酸马奶功能注释基因主要可归属为6 大类的代谢通路, 主要功能基因二级代谢途径有42 类。KEGG 注释结果显示, 酸马奶功能基因主要位于细胞群体——原核生物、膜运转、信号转导、氨基酸代谢、碳水化合物代谢、辅酶因子和维生素的新陈代谢和核苷酸代谢等代谢途径(图3)。
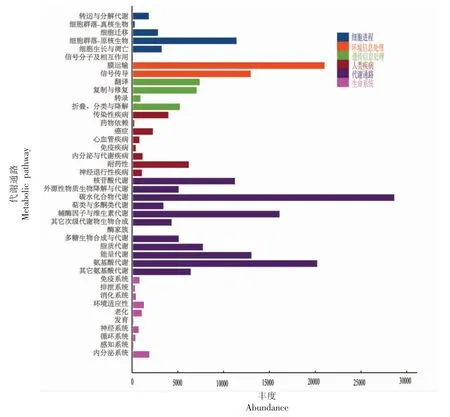
图3 酸马奶中KEGG 主要功能分类的基因数目Fig.3 The number of genes in KEGG primary functional classification in koumiss
2.2.3 CAZy 数据库注释分析 将酸马奶基因序列与CAZy 数据库进行比对,酸马奶共鉴定出3 495 个碳水化合物活性酶, 其中糖基转移酶(1 238 个)和糖苷水解酶(1 430 个)类最多,占据酸马奶碳水化合物活性酶的76%, 其次是碳水化合物结合模块 (235 个) 和碳水化合物酯酶(435个),而辅助活动酶(109 个)和多糖裂合酶(48 个)类最少, 仅占据酸马奶样品检测到酶总数的4.49%。
2.3 蛋白质水解系统基因分析
2.3.1 蛋白酶 在乳制品研究中,芳香族氨基酸、支链氧基酸以及含硫氨基酸被认为是奶酪风味形成的主要前体物质, 乳中酪蛋白被细胞壁黏附蛋白水解为寡肽, 主要控制基因为CEPs 族基因prtB, prtP, prtR 等。 马奶与牛奶在蛋白质组成上有较大差别,经分析,酸马奶中未发现与奶酪风味形成相关的CEPs 族基因,可见酸马奶与常见奶酪在风味形成中的蛋白酶种类也有一定差异。 经检索,酸马奶中发现3 种RRT12 蛋白酶[EC:3.4.21.-]和2 种serralysin 金属蛋白酶基因。RRT12 蛋白酶与枯草溶菌素的形成有关,而serralysin 金属蛋白酶的活性依赖于二价金属离子锌同HEXXH 组件的结合水平(表1)。 尽管检索到的蛋白酶并不丰富, 但在酸马奶中发现第六型蛋白分泌系统(T6SS)基因较丰富,分属于21 种类型。 第六型蛋白分泌系统(T6SS)是一种广泛分布于革兰氏阴性细菌中、 既能作用于真核细胞也能作用于细菌细胞的蛋白分泌系统, 它通过细胞间直接接触和物理性穿透转运有细胞毒性或抗菌的分泌蛋白[19-20]。然而,其分泌蛋白的机理尚不清楚。此一系列基因的发现将有助于马奶样品中蛋白分解及分泌蛋白的深入解析。

表1 酸马奶蛋白酶基因分析Table 1 Gene analysis of protease in koumiss
2.3.2 肽转运系统 蛋白质水解系统的第2 阶段为胞外蛋白降解形成的短肽及寡肽通过不同的肽转运系统转运至细胞内。目前,已知乳酸菌的3 个肽转运系统为寡肽Opp 系统、二/三肽Dpp 系统和DtpT 系统。 如表2 所示,酸马奶中存在丰富寡肽Opp 系统(158 个)和二/三肽Dpp 系统(74 个)基因, 具有非常强的肽转运能力。 在寡肽Opp 系统中, 寡肽Opp 系统寡肽连接蛋白基因数有75 个;在二/三肽Dpp 系统中,DppA/P 二/三肽寡肽联合蛋白透性酶基因数有41 个,表明酸马奶在寡肽连接、寡肽联合蛋白通透性方面具有较好的潜力,而具体功效需进一步转录表达验证。另外,酸马奶中不含有DtpT 基因,因此不能对连接有离子的二肽或三肽进行转运。
2.3.3 肽酶 肽酶是蛋白质水解系统中至关重要的组成部分, 不仅可以产生大量维持乳酸菌生长所必需的氨基酸, 同时还可以降低乳制品的不良风味,如干酪的苦味。 α-酪蛋白和β-酪蛋白水解产生的肽是干酪苦味的主要来源, 利用肽链内切酶对其苦味肽进行水解已成为该领域的研究热点。 如表2 所示,酸马奶中含有17 个pepO、19 个pepF 和11 个PepE 肽链内切酶的控制基因,说明在发酵过程中具有潜在的降低苦味肽的能力。 同时, 酸马奶中发现含有PepB (18 个)、pepN(28个)、PepM(2 个)和PepA(9 个)的编码基因,这些氨肽酶与抑制血管紧张素转化酶活性的ACE 抑制肽的形成有关[21-22],表明酸马奶具有潜在的降血压功能。 另外,酸马奶中还具有丰富的二/三肽酶(67 个)和脯氨酸肽酶(60 个)。 由此可见,酸马奶中可能蕴含着丰富的风味肽及功能肽, 有待后续进一步纯化鉴定。

表2 酸马奶肽转运系统及肽酶基因分析Table 2 Peptide transport system and gene analysis of peptidase in koumiss
2.4 氨基酸风味形成途径
氨基酸风味形成途径是指氨基酸经转氨酶作用,转化为相应的酮酸,酮酸较不稳定,进一步转化成相应的醛类、醇类和酯类,这些都是食品中重要的风味成分。
2.4.1 转氨酶 支链氨基酸、 芳香族氨基酸以及甲硫氨酸的转氨作用可以通过不同的转氨酶进行催化。 在乳酸菌菌株中,支链转氨酶(BcAT)显示了对支链氨基酸和蛋氨酸的催化活力, 而芳香转氨酶(ArAT)对芳香族氨基酸、亮氨酸和蛋氨酸具有催化活力。 如表3 所示,酸马奶中编码了26 个ArAT 基因,包括ARO8、tyrB、ybdl 3 类,未发现支链转氨酶(BcAT)基因,说明酸马奶发酵过程中可对芳香族氨基酸、 亮氨酸和蛋氨酸进行催化。 例如, 亮氨酸可以通过转氨酶转化为α-酮异己酸,有报道称α-酮异己酸是发酵香肠中的主要风味物质[23]。
2.4.2 酮酸转化酶 酮酸以3 种途径进行进一步转化: 第1 种途径是酮酸经过氧化脱羧反应可被直接的转化成羧酸。 在这个途径中, 酮酸脱氢酶(KaDH)、磷酸转乙酰酶(PTA)和酰基激酶(ACK)对支链氨基酸的基质表现出活性。如表3 所示,酸马奶基因组存在编码PTA(8 个)和ACK(32 个)的基因。 酸马奶发酵过程中可能会通过该途径将上述氨基酸的酮酸转化为相应的羧酸类物质。 第2 种途径是酮酸经羧酸脱氢酶(HycDH)分解为羧酸。 HycDH 有D-羧酸脱氢酶(D-HycDH)和L-羧酸脱氢酶(L-HycDH)两种立体专一性酶。目前,虽然并不能证明此途径可以直接导致风味物质的形成, 但是却可以通过将所形成的风味物质前体分流到非风味产品内造成风味物质合成的减少。 酸马奶基因组中未发现这2 种羧酸脱氢酶, 不会因为以上途径造成整体风味的减少。 第3 种途径是α-酮酸经α-酮酸脱羧酶(KdcA)的催化转化成相应的醛类。截至目前,乳制品微生物组中并未发现KdcA 活性,这与乳制品发酵中只有很少暈的氨基酸转化为醛类的观察结果相一致。 酸马奶基因中也未发现编码α-酮酸脱羧酶(KdcA)的基因,表明酸马奶发酵中不会通过此途径对支链氨基酸酮酸进行脱羧反应,生成相应的醛类物质。

表3 酸马奶氨基酸风味形成相关基因分析Table 3 Analysis of amino acid flavor formation gene in koumiss
2.4.3 醇和醛脱氢酶 醛脱氢酶(AldDH)和醇脱氢酶(AlcDH)可以分别催化醛(醇)类物质转化成相应的醇类和羧酸类物质, 控制基因主要是AlcDH 总科和1 个双官能团的AlcDH/AldDH 总科。酸马奶基因中发现51 个编码醇脱氢酶(AlcDH)和68 个编码醛脱氢酶(AldDH)的基因,醇脱氢酶(AlcDH)包括Adh1、Adh2、adhP、AKR1A1、yjgB 和yghD 6 类,醛脱氢酶(AldDH)包括adhE、aldB、aldh、mhpF、aldA 和feaB 6 类。 因此酸马奶发酵时具有催化醛类物质转化成相应的醇类和羧酸类物质的潜能。如可以催化异戊醛、活性戊醛和异丁醛等生成相应的异戊醇、活性戊醇和异丁醇,并进一步生成相应的羧酸类物质(如具有甜味和奶酪味的二甲基丁酸,具有汗味的2-异戊酸以及酸甜味的异丁酸)。
2.4.4 乙酰酯酶 带有短链脂肪酸的酯类物质大多具有水果风味,对特色乳制品的开发非常重要。酸马奶基因中编码了34 个可以催化乙酸酯类合成的乙酰酯酶的基因(aes),可以合成具有水果香味的乙酸乙酯、乙酸异丁酯、乙酸正丁酯、乙酸异丁酯等, 为酸马奶系列产品的风味提升及品质优化提供了思路。
3 结论
酸马奶独特的风味及功能特性的形成与其复杂的微生物菌群结构密不可分。 本试验基于宏基因组技术分析酸马奶的微生物多样性, 挖掘蛋白质分解系统及氨基酸风味形成系统的功能基因。酸马奶中鉴定出微生物30 个门,331 个科,913 个属,2 692 个种。 其中,相对含量大于1%的优势菌群在门、科、属、种水平上分别有2,3,6,8 个。优势菌种为乳酒样乳杆菌、瑞士乳杆菌、弗氏柠檬酸杆菌、解鸟氨酸拉乌尔菌、柠檬酸杆菌属和乳酸乳球菌。 COG、KEGG 数据库分别注释到10 849 和214 338 个基因。 代谢通路中,碳水化合物代谢和氨基酸代谢功能突出, 其次为辅酶因子和维生素的新陈代谢和核苷酸代谢等代谢活动。 经CAZy数据库注释分析,糖基转移酶(1 238 个)和糖苷水解酶(1 430 个)的数量最多,占据酸马奶碳水化合物活性酶的76%。 同时,在蛋白质分解系统中,酸马奶基因中发现3 种RRT12 蛋白酶、2 种serralysin 金属蛋白酶、 第六型蛋白分泌系统(T6SS)基因、232 个肽转运系统及231 个肽酶控制基因,表明其具有较强的蛋白质分解转运潜力。 酸马奶中编码了26 个(ArAT)基因、40 个酮酸转化酶、51个编码醇脱氢酶 (AlcDH)、68 个编码醛脱氢酶(AldDH)基因和34 个乙酰酯酶(aes)基因,说明酸马奶发酵时具有从氨基酸形成浓郁风味物质的基因基础, 本试验结果可为酸马奶品质的提升及乳酸菌功能基因库的挖掘提供理论依据。

