基于线粒体全基因组结构的鲳属鱼类分子分类研究
闫永斌,程起群
(1.中国水产科学研究院东海水产研究所,上海 200090;2.上海海洋大学海洋科学学院,上海 201306)
鲳属(Pampus)鱼类隶属于硬骨鱼纲(Osteichthyes),鲈 形 目(Perciformes),鲳 科(Stromateidae),是世界上,特别是印度太平洋海域 的 重 要 海 洋 经 济 鱼 类[1]。1758年,LINNAEUS[2]建立鲳属(原属名Stromateus),将鲳鱼定名为Stromateusfiatola,于是在最初阶段,是以Stromateus作为鲳属的属名;后经FOWLER[3]更正,将鲳属改名为Pampus,此后,Pampus作为鲳属属名一直被沿用至今[3-4]
自鲳属建立以来,其分类问题一直存在不同的观点,至今尚未统一。研究初期主要依据形态特征进行分类,依次经历了“两种说”、“三种说”、“五种说”、“六种说”和“八种说”。例如REGAN[5]依据尾鳍形态特征将鲳属分为两个种:即尾鳍为截形或浅叉形的中国鲳(P.chinensis)和尾鳍深叉形、下叶延长的灰鲳(P.cinereus)。HAEDRICH[6]根据鳍的形态、鳍条鳍棘数目、脊椎骨数目、鳃耙数目等特征,将鲳属分为3个种,即:中国鲳、银鲳(P.argenteus)和高丽鲳(P.echinogaster),并视灰鲳为银鲳的同物异名。成庆泰和郑葆珊[7]根据鳍和吻的形态特征,将中国沿海鲳属鱼类分为3个种:燕尾鲳(P.nozawae)、银鲳和中国鲳;邓思明等[8]依据侧线管系统、骨骼系统、耳石结构及食道侧囊特征,朱元鼎[9]基于鳍的形态和颜色、头部后上方侧线管的形态特征以及脊椎骨数目,也都将鲳属分为3种,即银鲳、灰鲳和中国鲳。后LIU和LI[10]根据最大体长、鳍的形状、耳石和脊椎骨数等形态特征发表了鲳属鱼类一新种——珍鲳(P.minor),并根据鳃耙、幽门盲囊、耳石、侧线管、头颅、脊椎骨等形态特征,进一步将中国沿海鲳属鱼类分为5个种,即银鲳、翎鲳(P.punctatissimus)、灰鲳、中国鲳和珍鲳[4]。2013年,LIU和LI[11]又报道了分布于中国东南沿海鲳科鲳属的一新种——刘氏鲳(P.liuorum),该鱼从吻的形状、鳃耙的数量和形状、脊椎骨数、胸鳍比例和眼径大小等方面与其他鲳鱼区别开来,认为这是鲳属的第6个种,但其有效性存疑[12-13]。而JAWAD和JIG[1]通过研究鲳属鱼类轴向骨架的7个骨骼特征,将鲳属分为8个种:银鲳、镰鲳、翎鲳、灰鲳、中国鲳、珍鲳、刘氏鲳和燕尾鲳。
随着分子生物学技术的发展,分子标记也被用于物种鉴别,CUI等[14]利用线粒体16S rRNA和COI基因的部分序列,将鲳属分为5个物种:中国鲳、灰鲳、珍鲳、翎鲳和未定名新种(Pampussp.)。YIN等[12]根据核基因标记将鲳属分为中国鲳、灰鲳、银鲳、翎鲳和珍鲳。李渊[15]基于形态和DNA条形码的综合分析,认为鲳属鱼类有6种,即银鲳、镰鲳、翎鲳、灰鲳、中国鲳和珍鲳;并认为前人所研究的分布于黄海、东海和渤海的“银鲳”实际上是镰鲳,而真正的银鲳分布于台湾海峡以南。
总体而言,鲳属鱼类分类存在以下问题:1)鲳属鱼类存在几个有效物种?2)银鲳与镰鲳的分类关系如何?3)灰鲳和翎鲳是否为同一物种?4)缺乏有效的鲳属鱼类种间的分子鉴别标记。
线粒体基因组目前已被广泛应用于系统发育及物种间鉴别研究,因为它比核DNA进化速率快,导致其在近缘种间的差异较大,易于识别[16]。线粒体基因组的大小通常在15~20 kb,一般包含2个核糖体RNA基因(ribosomal RNAs,rRNAs),13个蛋白质编码基因(protein-coding genes,PCGs),22个转移RNA基因(transfer RNAs,tRNAs)和非编码控制区(control region,CR或D-loop)[17]。其中,RNA基因进化速率最慢,D-loop区最快,而细胞色素b(cytochrome b,Cytb)和NADH脱氢酶亚基(NADH dehydrogenase subunit,ND)基因进化速率适中[18-20],16S核糖体RNA基因(16S rRNA)和细胞色素C氧化酶第一亚基(cytochrome c oxidase subunit I,COI)基因标记常被用作DNA条形码,用于鉴定形态相似的近缘种。DNA条形码(DNA barcoding)是一段短的、标准化的DNA序列,于2003年被HEBERT首次提出,由于其既具有稳定的种内保守性,又包含足够的种间遗传变异而被用于物种的分子鉴定[21]。
本研究测定了鲳属鱼类5个物种的线粒体全基因组序列,并结合NCBI数据库中已有的鲳属鱼类线粒体全基因组序列,进行综合分析,以期为解决鲳属鱼类分类学、进化遗传学及种间鉴别上的问题提供参考。
1 材料与方法
1.1 样品采集及组织DNA提取
采集5种鲳属鱼类样本(银鲳、灰鲳、翎鲳、中国鲳和珍鲳),样本信息见表1。所有样本均按照形态和解剖学特征进行分类[4,7,22]。每个样本分别取其肌肉组织,采用常规的苯酚-氯仿法提取基因组DNA,并于-20℃保存备用。

表1 实验所用的样本信息Tab.1 Information of samples used in this study
1.2 引物设计及PCR扩增、测序
依据NCBI已公布的鲳属鱼类线粒体全基因组序列(序列登录号见表2),应用软件Primer Premier 5.0设计特异性引物,所有特异性引物均在生工生物工程(上海)股份有限公司合成。
除珍鲳的ZC06和ZC08引物外,其余所有引物的PCR扩增体系为:12.5μL 2×PCRTaqPCR Master Mix,10μmol·L-1的引物各1μL,20 ng·μL-1的DNA模板1μL,最后加双蒸水至混合液总体积为25μL。反应条件:在94℃时预变性5 min;然后循环35次,每一个循环包含94℃变性45 s,退火45 s,72℃延伸60 s;最后在72℃下延伸8 min。
珍鲳的引物ZC06和ZC08由于其扩增产物片段过长(分别为5 641 bp和3 292 bp),采用长PCR扩增。扩增体系为2.5μL 10×Long PCR buffer with 15mM MgCl2,2.5 mmol·L-1的dNTP Mix 2μL,5 U·μL-1的Long PCR Enzyme Mix 0.25μL,10μmol·L-1的引物各1μL,20 ng·μL-1的DNA模板1μL,最后加双蒸水直至混合液的总体积为25μL。反应程序为:在94℃时预变性2 min;然后循环35次,每一个循环包含94℃变性45 s,60℃退火50 s,68℃延伸4~8 min;最后在68℃下延伸10 min。
在1.5%的琼脂糖凝胶中电泳,并在复日FR980凝胶电泳成像系统中拍照来检测PCR扩增产物的质量。将PCR扩增产物纯化后,送上海桑尼生物技术有限公司双向测序。
1.3 数据处理
利用DNASTAR、CHROMAS、MEGA等软件,对测序结果进行拼接、注释,获得鲳属鱼类完整的线粒体全基因组序列,并提交至GenBank(序列登录号见表2);确定线粒体基因组上13个PCG、2个rRNA基因以及D-loop区的相应位置[23-24],通过tRNAscan-SE 1.21[25]确定大部分tRNA基因,其他不能确定的tRNA基因通过相关tRNA的二级结构图及反密码子来确定[26-27]。

表2 鲳属鱼类引物设计模板序列及所测序列NCBI登录号Tab.2 Accession numbers of templates and sequencing sequences
1.4 序列分析
1.4.1 线粒体全基因组特征
目前NCBI网站中只有6种鲳属鱼类的线粒体全基因组序列,即银鲳、灰鲳、翎鲳、中国鲳、珍鲳和镰鲳,本研究测定了除镰鲳外其余5种鲳鱼的线粒体全基因组序列,因此下载镰鲳线粒体全基因组序列(GenBank登录号NC_023258),用于综合分析鲳属鱼类线粒体全基因组序列特征及差异。
利用MEGA[28]软件分析每个物种线粒体基因组全序列的长度、结构和基因排布;以及每个基因位点的变异率、遗传距离和碱基含量等信息。
1.4.2 控制区结构及特征分析
对于6种鲳鱼的控制区(D-loop)序列,参考前人的方法,寻找控制区序列特有结构并进行结构描述[29]和串联重复筛选[30]。
1.4.3 置信度分析
从NCBI网站下载所有已命名的鲳属鱼类线粒体全基因组序列,分别提取其PCG序列进行置信度分析。利用MEGA中的邻接法(neighborjoining,NJ),以Kimura 2-parameter(K-2-P)模型构建系统发育树(bootstrap=1 000),评估PCG标记对鲳属鱼类系统进化分析的适用性[31]。该方法主要依据构建NJ树的过程中bootstrap获取的置信度高低判断系统树的可靠性,计算得到单一序列构建系统树的置信度和以及碱基信息量(置信度和/序列长度),比较分析基因组的不同区域在系统进化分析中的适用性。
1.4.4 DNA条形码分析
根据置信度以及线粒体每个基因位点变异率的结果,选取适合的基因标记进行DNA微型条形码(mini-barcodes)分析[32]。在NCBI网站下载鲳属鱼类相应基因的剩余序列,增加分析结果的可靠性,共获得18条序列(表3)。将所有序列进行比对,筛选合适的同源序列片段进行DNA条形码分析,并将该片段从5′端到3′端平均分成3或6个小片段,即总共9块区域,进行微型条形码分析。微型条形码的筛选参考RASMUSSEN等[32]的标准:1)原始条形码序列长度必须大于500 bp;2)微型条形码序列中不能有间断点。
表3 鲳属鱼类DNA条形码分析序列Tab.3 Sequences of Pampus fishes used for DNA barcode analysis
利用DAMBE软件[33]对DNA条形码进行替换饱和性检验,使用MEGA软件[28]进行以下分析:对序列进行比对;计算条形码的碱基组成、保守位点数、变异位点数、简约信息位点数、转换(transition)和颠换(transversion)频率的比值;基于各候选条形码,计算鲳属鱼类各物种种间和种内遗传距离,比较种间和种内遗传距离,筛选出可用于区分鲳属鱼类的最适DNA条形码。
1.4.5 系统发育分析
利用MEGA软件[28],选择刺鲳属鱼类刺鲳(Psenopsisanomala)作为外群,利用线粒体全基因组以及所筛选的DNA条形码对鲳属鱼类进行系统发育分析。
2 结果与分析
2.1 线粒体全基因组基本特征
6种鲳属鱼类的线粒体全基因组全长在16 551 bp到18 062 bp之间,都含有2个rRNA基因(16SrRNA和12SrRNA)和13个PCGs。灰鲳、翎鲳和中国鲳分别含有22个tRNA基因,而银鲳、镰鲳和珍鲳分别含有23个tRNA基因(银鲳和镰鲳多了1个tRNAMet,珍鲳多了1个tRNAPro)。同时,在线粒体全基因组组成上,珍鲳有2个D-loop区,而其余5种鱼分别只有1个Dloop区。银鲳、镰鲳、灰鲳、翎鲳和中国鲳的线粒体基因组结构和分布特征大体是一致的,基本符合硬骨鱼线粒体基因组结构和分布特征,而珍鲳在16SrRNA-tRNAIle区域发生了ND1的重排,并且出现了额外的tRNAPro和D-loop区。
由表4可以看出,6条线粒体全基因组序列(不含D-loop区)经比对获得一致序列长度为11 901 bp,其中变异位点3 950个,占所有位点数的33.2%,发生转换1 311次,颠换602次,转换颠换比为2.2。
鲳属鱼类线粒体基因组序列、PCGs序列以及tRNA拼接序列的G含量均比其他碱基低,表现出明显的碱基偏好性,但是rRNA序列中T的含量小于或等于G的含量。13个PCGs中,ATP8的变异程度和遗传距离最大(分别为43.5%、0.305),其次是ATP6(43.1%、0.303),变异程度和遗传距离最小的是ND4L(21.2%、0.119)。rRNA和tRNA均表现出较小的变异程度和遗传距离,尤其是tRNA拼接序列,变异程度(12.9%)和遗传距离(0.067)在所有基因中最低(表4)。
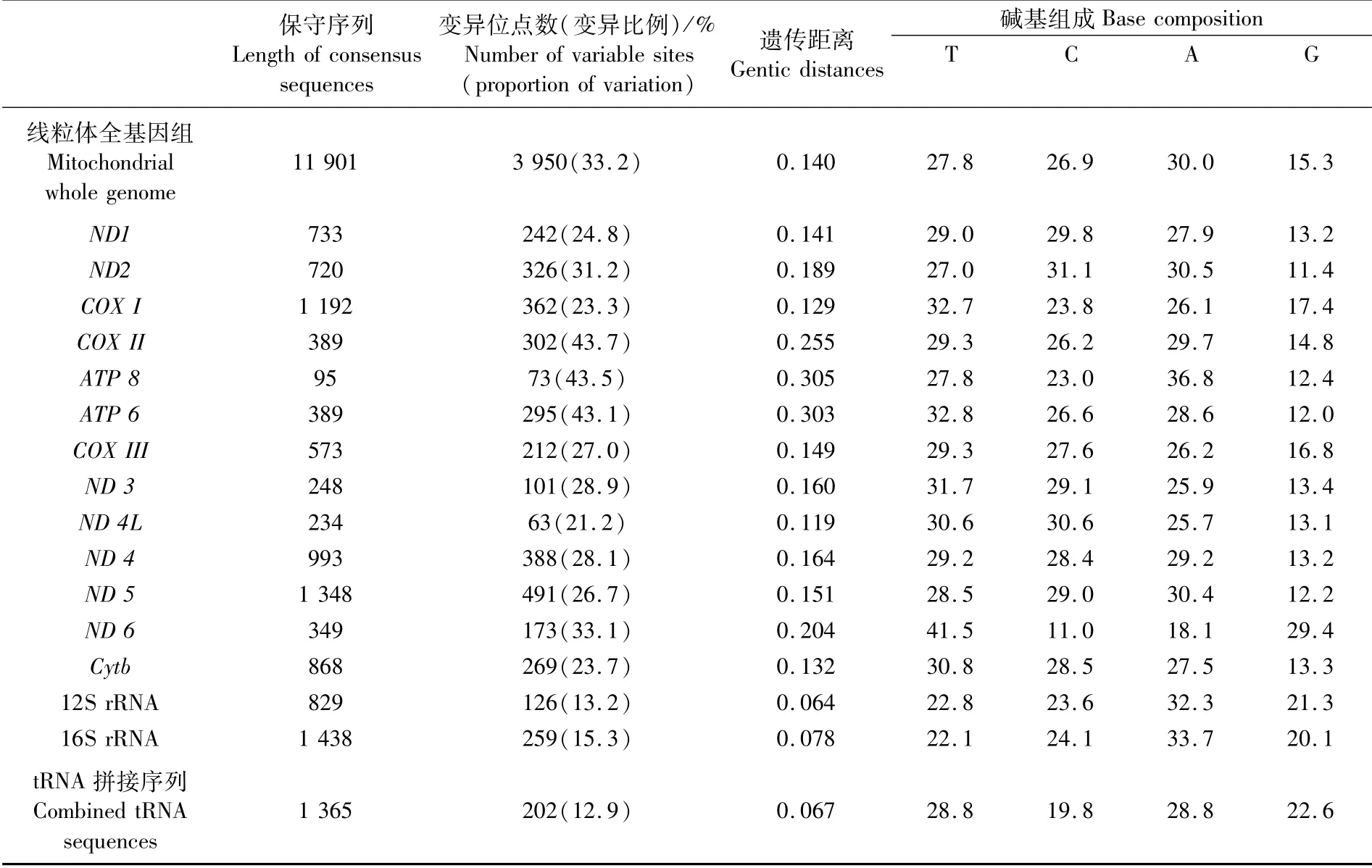
表4 鲳属鱼类线粒体基因组及不同区域的保守序列长度、变异位点数(比例)、遗传距离和碱基组成Tab.4 Length of consensus sequences,number of variable sites,genetic distances and base composition of mitochondrial genomes of different regions of 6 Pampus fishes
2.2 控制区结构及特征
鲳属鱼类的D-loop区位于tRNAPro和tRNAPhe基因序列之间(本研究珍鲳有一重复D-loop区序列在tRNAPro和tRNALeu之间),确定长度为831~1 928 bp。银鲳、镰鲳含有1个终止序列区(TAS),而中国鲳与珍鲳有2个终止序列区(TAS),翎鲳和灰鲳有3个终止序列区(TAS)。6种鲳鱼都含有1个中央保守结构域(CSB-D,E,F),除珍鲳外都具有3个保守序列块CSB1、CSB2、CSB3。
银鲳D-loop区包括5个含有CSB3和启动子的串联重复序列(715~1 328),而镰鲳D-loop区包括10个含有CSB3的串联重复序列(719~1 924),灰鲳与翎鲳D-loop区的串联重复片段完全一致,都具有两种片段,分别在位点19~59、37~78之间,重复片段的前两段完全一致,最后一段由于核苷酸的缺失而不完整。中国鲳D-loop区包含一种重复序列,在位点17~60之间。珍鲳D-loop区无重复序列。
2.3 置信度分析
在13个PCGs中,由于ND6基因的碱基组成与其他基因存在明显差异且在系统发育建树分析中的效果较差[34],因此暂不考虑,其余基因结果见表5,由表5可知,对于COXII、ATP8、ATP 6、ND4L等基因,虽然单碱基的信息量较高,但置信度和较低,无法形成可靠的系统发育树,所以也暂不考虑。除这些基因外,置信度和最高的是COXI基因(N=985),随后是ND1、ND3、COX III、ND2基因,单碱基信息量最大的是ND3基因(N/L=2.7192),随后4个是COXIII、ND1、ND2、COXI基因。综合考虑,对于鲳属鱼类的系统进化分析,线粒体蛋白质编码基因的适用情况为:最好的是ND3和COXIII,其次是ND1和ND2,较差的是COXII、ATP8、ATP6、ND4L、ND5,其他基因表现一般。

表5 鲳属线粒体蛋白质编码基因组构建的NJ树的置信度分析Tab.5 Bootstrap values analysis of NJ trees built based on different regions in mitochondrial genomes of Pampus fishes
2.4 DNA条形码分析
根据基因标记变异率和置信度分析结果,综合考虑,选取ND2基因作为DNA条形码对鲳属鱼类进行分析鉴定。将18条基因序列通过Clustal W[23]方法进行多重序列比对,最终选取了一段长为633 bp的同源序列作为完整条形码进行分析,如表6所示,这段序列中,腺嘌呤(A)、胸腺嘧啶(T)、胞嘧啶(C)、鸟嘌呤(G)的平均含量分别为30.6%、27.5%、29.1%、12.7%,A+T明显大于C+G,这符合后生动物线粒体基因碱基组成偏移性的特点[16]。3个密码子中A+T的含量差别较大,其中第三位点的A+T含量最高,达到66.1%,第一、第二位点的A+T含量分别为51.1%、57.2%。
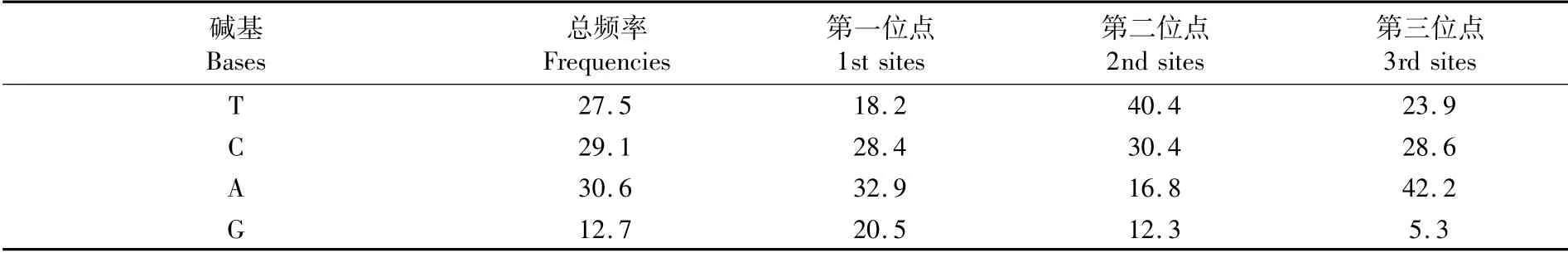
表6 鲳属鱼类ND2序列碱基分布频率Tab.6 Nucleotide frequencies of ND2 sequences in Pampus fishes
条形码序列的核苷酸变异分析见表7,如表7所示,这633个位点中分别有416个保守位点(conserved sites)、217个变异位点(variable sites)和211个简约信息位点(parsimony informative sites)。变异位点和简约信息位点主要集中在序列的第三位点上,分别占两者总和的65.9%和65.4%,而第一和第二位点较保守。

表7 鲳属鱼类ND2序列核苷酸变异情况Tab.7 Sequence variation of ND2 gene in Pampus fishes
DAMBE软件[33]对DNA条形码序列进行替换饱和性检验,结果显示,序列未饱和,可进行建树分析。建树分析结果见图1,由图1可知,鲳属鱼类系统发育树的聚类十分混乱,可能是由于NCBI网站上的序列被错误的鉴别导致,并且灰鲳没有形成明显的分支,因此本实验将混乱的序列进行重新分类,暂将其假设为其他物种,以便进行后续分析,假设的序列见表8。

表8 假设序列Tab.8 Hypothetical sequence
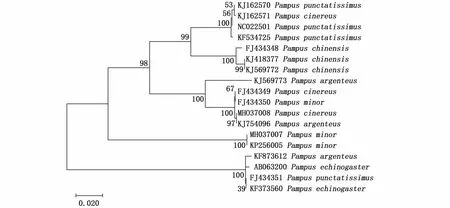
图1 基于ND2基因片段采用NJ法构建的鲳属系统进化树Fig.1 Phylogenetic relationship tree of Pampus fishes built based on partial sequences of ND2 gene by using neighbor-joining method
将完整条形码5′端到3′端平均分成3或6个小片段,每个片段长为211 bp或105 bp,总共9个微型条形码区域。通过MEGA软件,采用K-2-P模型计算各条形码对应的平均种内遗传距离和平均种间遗传距离。由表9可知,微型条形码变异位点占所有位点比为27.62%~39.05%,简约信息位点占比为26.67%~39.05%。除微型条形码105-2和105-4外,其他微型条形码序列的变异情况与完整条形码相差不大。HEBERT等[21]通过COXI条形码序列对鸟类进行鉴定,结合其他动物类群的相关研究,提出“10×”的阈值规则,即种间平均遗传距离大于种内平均遗传距离的10倍的标准用于区分物种。本研究中的10个条形码的平均种间距离是种内遗传距离的18~40倍,说明每个条形码都具有鉴定鲳属鱼类的潜力。

表9 鲳属鱼类完整条形码与微型条形码比较Tab.9 A comparison of full-length barcode and mini-barcodes of Pampus fishes
当物种间的种间最小遗传距离大于种内最大遗传距离,即可判定该物种存在条形码间隙(barcode gap)。条形码间隙是否存在决定了该条形码能否有效鉴别物种。研究通过MEGA软件,采用K-2-P模型计算了6个鲳属鱼类的种内遗传距离及它们与其他所有个体两两间的遗传距离。图2中直线表示1∶1比例线,当数据点高于比例线时,说明种间最小遗传距离大于种内最大遗传距离,即存在条形码间隙;反之,说明此条形码不能用于区分该物种与其他物种。
由图2得知,所有条形码的数据点均在1∶1比例线之上,存在条形码间隙,说明所有微型条形码均可以有效区分不同的物种。优质的条形码还应使各物种的种内遗传距离尽可能小,并使物种间的遗传距离尽可能大,以保证鉴定结果的可信度。比较得知,长度为211 bp的微型条形码中,211-2区分效果最佳;长度为105 bp的微型条形码中,105-3最佳。
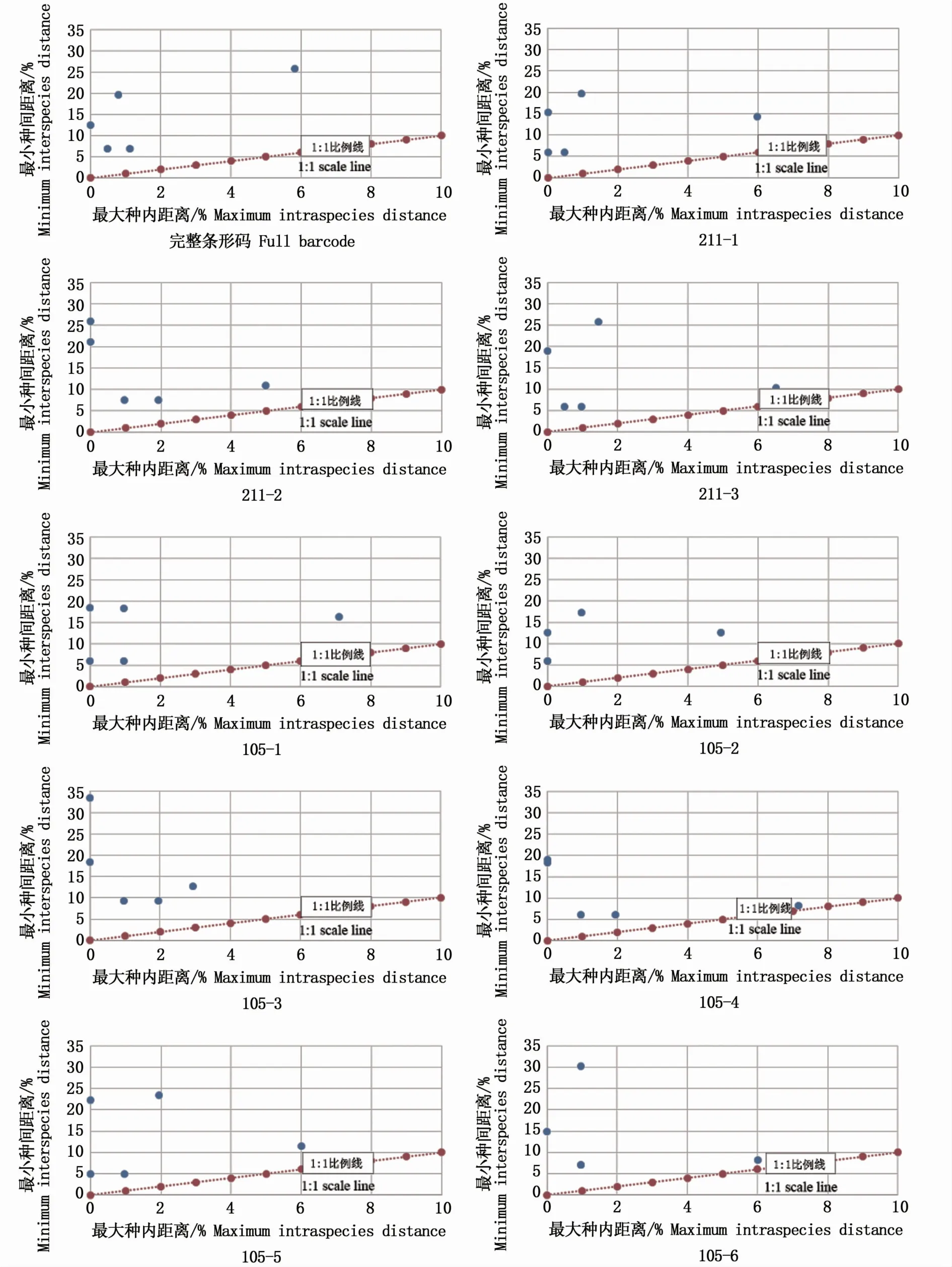
图2 鲳属鱼类ND2序列条形码间隙Fig.2 Barcode gaps of ND2 sequences in Pampus fishes
2.5 系统发育分析
线粒体全基因组的系统发育分析结果见图3,由图3可见,本实验所测得的序列中,银鲳与NCBI网站的镰鲳聚为一组,灰鲳与翎鲳聚为一组;在全部鲳属鱼类线粒体基因序列中,灰鲳的两条基因组序列一条与翎鲳分为一组,一条与银鲳聚为一组,剩余鲳属鱼类形成了较为可信的分支。

图3 基于线粒体全基因组采用ML法构建的鲳属系统进化树Fig.3 Phylogenetic relationship tree of Pampus fishes built based on whole mitochondrial genome sequences by using maximum likelihood method
DNA条形码建树结果如图4,完整条形码以刺鲳为外群进行建树分析。由图4可知,鲳属鱼类DNA条形码的聚类结果与全基因组聚类分化结果大致相同,不同点在于全基因组发育树中珍鲳与镰鲳聚为一支,而在完整条形码发育树中,镰鲳最先分化出来。

图4 基于ND2条形码采用ML法构建的鲳属系统进化树Fig.4 Phylogenetic relationship tree of Pampus fishes built based on ND2 DNA barcode sequences by using maximum likelihood method
3 讨论
鲳属鱼类线粒体基因组结构与硬骨鱼类线粒体基因组结构相似,通常情况下,包含13个PCGs、2个rRNA基因、22个tRNA基因和一个Dloop区。本实验所测得的银鲳线粒体基因组出现了一个重复的tRNAMet结构,而NCBI网站的基因数据库中镰鲳(KF373560)的线粒体基因组同样包含一个tRNAMet结构,CUI等[35]在Pampussp.的线粒体基因组中也发现了相同的结构,结合系统发育树的聚类结果,这3条序列可能属于同一物种。另外,珍鲳在16S rRNA-tRNAIle区域发生了ND1的重排,而且出现了额外的tRNAPro和Dloop区,其他鲳属鱼类线粒体基因组中没有这种现象,并且NCBI网站的基因数据库中珍鲳(MH037007)的基因组也没有这种结构,产生这种情况的原因还需进一步研究。
3.1 银鲳与镰鲳的关系
银鲳在1788年首次被EUPHRSSEN提出并进行了描述[36],由于仅有一尾样品,导致很多特征描述的并不详实,很难进行准确的物种鉴别。近年来,有许多研究从形态特征对银鲳进行了分类描述,可以将其与其他鲳属鱼类区分开来,然而根据这些特征无法将其与镰鲳区分开来,李渊[15]认为前人所研究的银鲳实际为镰鲳,而真正的银鲳分布于台湾海峡以南;DIVYA等[37]认为银鲳与镰鲳在分子鉴定中没有达到种间标准,属于同一物种;YIN等[12]通过COXI基因标记研究也认为分布于中国东海的银鲳与镰鲳为同一物种。在本研究的结果中,所测银鲳序列与已有的镰鲳序列具有相同的重复结构,两条序列在系统发育树中聚为一支,且采集地点均为东海,而其他银鲳的序列聚为另一支,采集地点为南海,这说明李渊[15]与YIN等[12]的观点可能是正确的。即分布于东海的银鲳与镰鲳为同一物种,真正的银鲳分布于台湾海峡以南。
3.2 灰鲳与翎鲳的关系
1795年,BLOCH首次对灰鲳进行描述,但没有进行详细的分类特征描述,对灰鲳标本的采样地点也并未记录,因此对原始标本的具体描述并不能详尽的给出[38]。翎鲳于1845年首次被TEMMINCK和SCHLEGEL描述,采集于日本长崎,但是所有描述是基于2尾鳍有损坏的样本,且仅对鳍式、鳞片、吻部特征和头部横枕管等进行简单描述,遗漏了很多重要的特征如鳃耙、脊椎骨数等,之后一直被学者认为是银鲳的同物异名[15]。1998年,LIU和LI[10]将翎鲳列为鲳属鱼类的一种。而在本实验中,所测得的灰鲳与翎鲳的线粒体基因组序列十分相似,DNA条形码结果表示其种间距离基本为0,可判断属于同一物种。根据系统发育树分析结果,两条灰鲳基因组序列一条与翎鲳聚为一支,一条与银鲳聚为一支,没有形成独立的分支,因此灰鲳的物种有效性有待商榷,其可能与翎鲳为同一物种,还需结合形态
特征进行综合分析判断。
3.3 鲳属的物种数及鉴定方法
鲳属的种类划分问题,国内外众多学者说法不一,且经历了多个时期[4],目前,在世界鱼类数据库(FishBase)中有5种鲳属鱼类[39]:银鲳、中国鲳、镰鲳、翎鲳和珍鲳。但是在鱼类目录中有8种鲳鱼[40],除上述5种外,新增了燕尾鲳、刘氏鲳和灰鲳。燕尾鲳在一些研究中被认为是灰鲳的同物异名[41],但JAWAD和JIG[1]根据对轴向骨7个骨骼特征的研究,认为燕尾鲳是一个独立的物种。刘氏鲳作为一新种,其研究较少,JAWAD和JIG[1]认为其为有效种,而YIN等[12]通过对其线粒体及核基因的研究,认为其为无效种,因此还需进一步的研究。本实验的结果表明,灰鲳可能为翎鲳的同物异名。因此,目前鲳属鱼类可确定的物种仅有5种,即银鲳、中国鲳、镰鲳、翎鲳和珍鲳,而其余鲳鱼的有效性存在争议。鲳属鱼类的鉴定由于其形态上的相似性,导致根据外观很难鉴别,因此较为准确的方法是通过比较基因序列进行鉴定,CUI等[14]通过对鲳属鱼类的16S rRNA和COXI基因标记进行扩增分析,结合GenBank同源序列,对鲳属鱼类进行了系统发育树分析,将鲳属鱼类分为了5个物种:中国鲳、灰鲳、珍鲳、翎鲳和Pampussp.。李渊[15]通过对6种鲳属鱼类DNA条形码进行扩增,提出了标准的DNA条形码,并对GenBank中的相关序列进行纠正。本研究以ND2基因标记作为DNA条形码对鲳属鱼类进行了分子鉴别分析,结果表明,无论是完整的条形码还是微型条形码,均可以将不同种的鲳属鱼类分辨开来。
然而,DNA条形码鉴定中阈值法的判定规则存在争议,阈值法是在物种间能够形成DNA条形码间隙的前提下,设定一个经验遗传距离作为阈值,当个体间遗传距离大于阈值时,可以鉴定为不同物种[42]。阈值法在DNA条形码物种鉴定中发挥着重要作用。然而,随着DNA条形码的深入研究,发现不同物种及不同DNA条形码区段的阈值难以统一定量[43]。因此CUI等[35]认为可通过线粒体序列中的重复片段来鉴别不同的鲳鱼,本研究中,所测东海区银鲳序列中存在重复tRNAMet结构,珍鲳存在重复D-loop结构,且在5种鲳属鱼类的控制区序列中,除珍鲳外均有重复串联序列,这些序列特征可为鲳属鱼类的鉴别提供参考。
另外研究发现,NCBI网站基因数据库中一些序列存在错配情况,例如序列号为FJ434351的翎鲳序列,其存在与本研究中东海区银鲳和镰鲳相同的重复tRNAMet结构,而其他翎鲳序列无此结构,所以此序列明显为镰鲳或东海区银鲳。其余序列也出现了类似的情况,因此本研究对一些序列进行了校正。
3.4 鲳属的系统发育分析
关于鲳属鱼类的系统发育分析,已有许多学者对其进行了研究,且结果不尽相同。刘静等[44]根据分支系统学原理和方法,利用形态特征以刺鲳为外群对中国沿海鲳属鱼类的系统发育进行了分析,认为珍鲳是最原始、最早分化出来的种;银鲳也是较原始、分化较早的种;中国鲳是较特化的种;翎鲳和灰鲳是一对姐妹种。李渊[15]通过分子学研究发现,鲳属鱼类的进化属于单系发生类群,珍鲳和镰鲳属于较为原始的种类,而剩余的4种鲳属亲缘关系较近,应属于晚分化的物种,翎鲳与中国鲳聚为一支,银鲳和灰鲳处于系统进化树的顶端,代表着最新演化的种类。YIN等[12]和CUI等[14]的系统发育树结果一致,即分布于中国东海和黄海的银鲳或镰鲳为同一物种,与珍鲳聚为一支,为最先分化出的物种;翎鲳先与中国鲳聚为一支,之后再与灰鲳聚为一支,然后所有鲳属鱼类聚为一支。本研究结果显示,中国东海的银鲳与镰鲳为同一物种,与珍鲳聚为一支,为首先分化出的物种;翎鲳与中国鲳聚为一支,之后再与南海的银鲳聚为一支。分子进化结果与形态进化结果之间不是完全吻合的,这可能是由各种类间生活习性的趋同进化所引起的。
综上可知,中国东海区的银鲳或镰鲳与珍鲳是鲳属鱼类中最先分化的物种,翎鲳和中国鲳为姐妹种,争议点在于灰鲳是否为有效种以及南海区银鲳的的系统发育地位,还需进一步的研究。
致谢:张衡博士参与部分样本采集,孙丹丹硕士参与部分实验,谨致谢忱。

