罗非鱼耐盐数量性状位点(QTL)定位研究
刘峰 余钧剑 顾楠
(上海市水产研究所,上海市水产技术推广站,上海 200433)
罗非鱼(tilapia)是原产非洲、中东的部分丽鱼科鱼类的通称,是世界重要的养殖经济鱼类,2018年全球罗非鱼养殖总产量达到603万t[1]。罗非鱼是广盐性鱼类,但耐盐性状的种间差异较大。其中,尼罗罗非鱼为主养种,其生长速度快,但耐盐性能较差[2-3],只能在淡水或低盐度水体中养殖[4]。莫桑比克罗非鱼的耐盐性较好[5],可耐受120的盐度[6],在49盐度下可正常繁殖[7],并能适应淡水、半咸水、海水、高盐水体等各种渗透压环境[4]。由于淡水资源日趋紧张,且咸水中养殖的罗非鱼品质优于淡水罗非鱼,售价更高,因此,采用莫桑比克罗非鱼等高耐盐品种与尼罗罗非鱼、红罗非鱼等生长速度快的品系进行杂交,再从杂交子代中进行耐盐罗非鱼的选育,已成为罗非鱼育种重要的研究方向。
数量性状位点(quantitative trait locus,QTL)是控制数量性状的基因或DNA片段所在的基因组区域。通过在参考群体中进行基因型鉴定及QTL定位分析,可获得重要性状的QTL,解析性状差异的成因,QTL区域内与性状相关的分子标记可用于分子标记辅助选育[8]。在QTL定位分析中,常用的分子标记主要有微卫星及单核苷酸多态性(SNP)标记[9]。采用传统选育方法进行耐盐罗非鱼的选育,选育周期长、成本高,耐盐性状相关位点难以纯合。若能筛选得到与耐盐性状相关的分子标记,再采用分子标记辅助选择技术进行选育,可加速选育进程,并有利于在维持较高遗传多样性的基础上对耐盐性状进行纯化。本研究以耐盐性好的莫桑比克罗非鱼与生长快的红罗非鱼杂交建立参考家系,进行耐盐QTL定位分析,所获得的耐盐相关QTL及耐盐相关分子标记将有利于耐盐罗非鱼分子标记辅助育种。
1 材料和方法
1.1 试验对象
以莫桑比克罗非鱼为父本、红罗非鱼为母本,繁育获得用于QTL定位分析的全同胞参考家系。从该家系中随机选取300尾水花鱼苗,放养于容量5 m3的循环水养殖系统中。养殖期间水温25~28 ℃,每日投饵2次,采用淡水饲养至140日龄,存活的282尾个体均用于试验。
1.2 试验方法
1.2.1 耐盐性状测定和采样
将所有试验鱼从淡水循环水养殖系统同时转入容量5 m3的海水循环水养殖系统。海水养殖系统中提前加注盐度为33的海水,足量充氧。观察各个体的状态,直至试验鱼死亡时将其捞出,记录每尾鱼的存活时间,作为耐盐性状表型值。根据生殖孔外观及性腺形态鉴定各个体性别,剪取每一个体的部分尾鳍,保存于95%乙醇中用于DNA提取。
1.2.2 基因型测定与遗传连锁图谱重建
采用Yue[10]的方法进行DNA提取。
筛选均匀分布于22个连锁群、在至少一方亲本中具有多态性的158个已发表的耐盐罗非鱼遗传连锁图谱分子标记[11]及13个后续开发的微卫星分子标记(见表1),对282尾参考家系个体进行第1轮基因型检测。在前期的研究中,该参考家系的性别决定系统已被确认为XY型[12],所有雌性的基因型均为XX型,所有雄性的基因型均为XY型,“性别”也被作为1个分子标记用于分析。首轮QTL定位分析后,为对第1连锁群中鉴定得到的两个QTL进行精细定位,从GenBank数据库下载罗非鱼全基因组序列,采用SiRoKo软件在第1连锁群中进行微卫星序列搜索,开发出19个微卫星分子标记(见表2),再对各参考家系个体进行第2轮基因型检测。
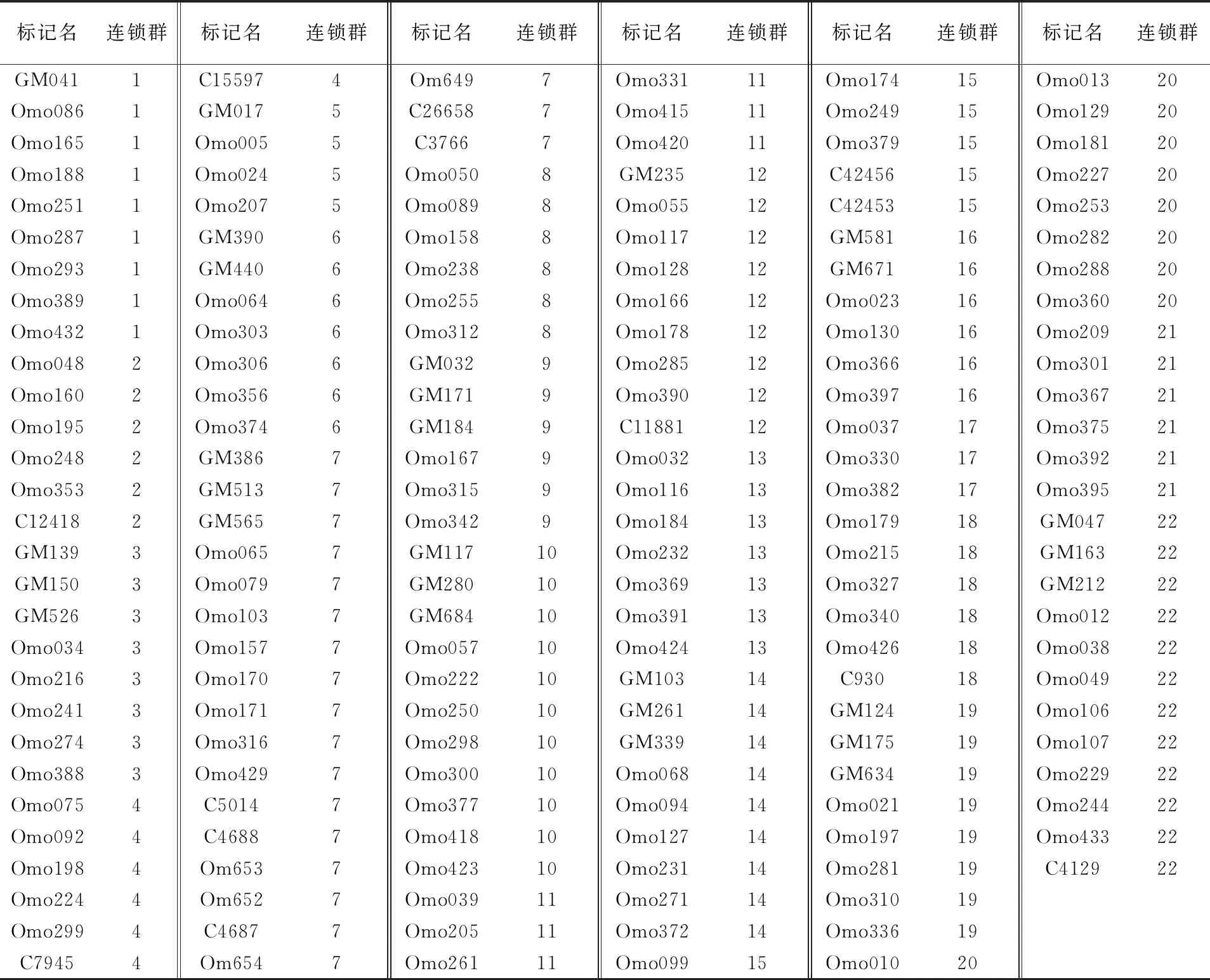
表1 首轮QTL定位用分子标记
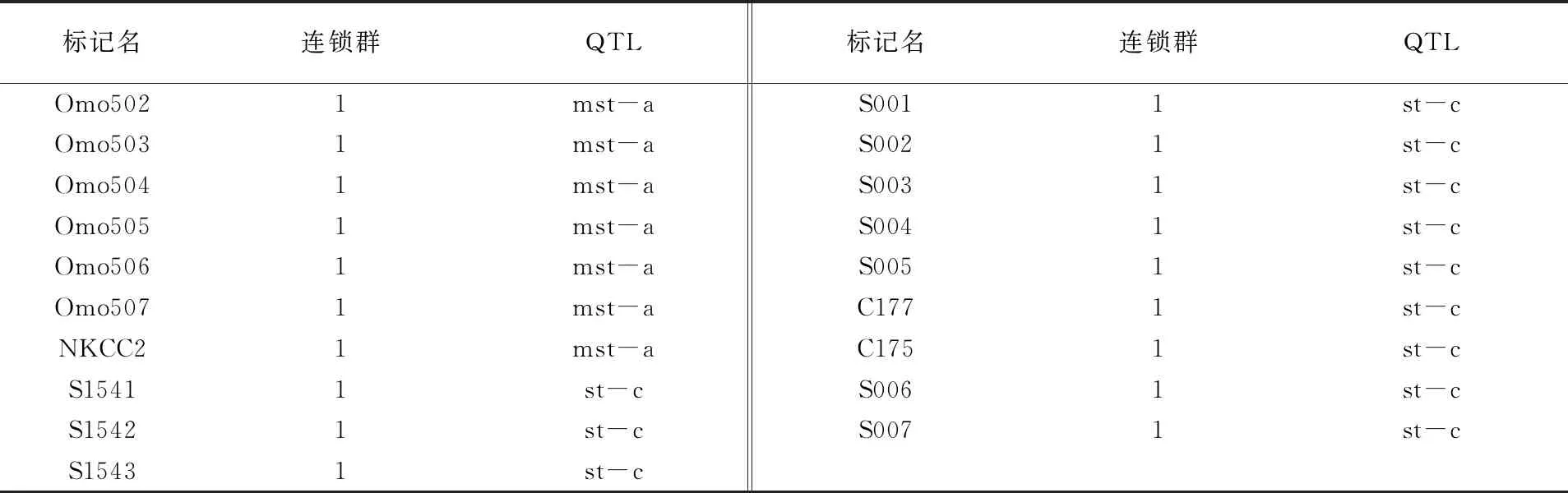
表2 QTL精细定位用分子标记
在进行基因型检测时,先采用荧光标记引物对各个体DNA进行PCR扩增,再采用ABI 3730测序仪对PCR扩增产物进行测定,内参采用GeneScan-500 ROX,基因分型软件采用GeneMaper 4.0。微卫星分子标记开发方法与已发表的文献[11]相同。
为排除不同家系重组率差异的影响,QTL定位分析前,先在参考家系中进行遗传连锁图谱的重建。基于参考家系282个全同胞个体及亲本的基因型,采用JoinMap 4.0软件进行遗传连锁图谱的构建,采用Kosambi作图,标记分群阈值设置为LOD 4.0。作图时,先根据母本、父本的基因型分别构建雌性、雄性图谱,再对雌、雄图谱进行合并,构建整合图谱。遗传连锁图谱的构建参照JoinMap 4.0使用说明进行。
1.2.3 QTL定位分析
QTL定位分析采用MapQTL 5.0软件进行,相关方法参照软件的使用说明。先将基因型数据及重建的遗传连锁图谱从JoinMap软件中导出,再与表型数据一起导入MapQTL软件,然后进行后续分析。采用IM、MQM分析方法进行QTL定位,采用P-test分析确定基因组的显著水平阈值。首轮QTL分析完成后,在选定QTL所在连锁群中开发新的分子标记进行第2轮基因型检测,基于两轮基因型测定结果,对选定QTL进行精细定位分析,方法与首轮分析相同。
1.2.4 数据处理
采用SPSS 19.0软件进行数据处理。均值结果以“平均值±标准差”表示,均值比较采用单因素方差分析(one-way ANOVA),设显著性水平为0.05。
平均存活时长=个体存活时间总和/个体总数
(1)
2 结果
2.1 参考家系耐盐性状
将参考家系转入盐度为33的海水后,经260 min出现第1尾死亡个体,之后死亡个体数逐渐增多,在410~434 min达到峰值。有小部分个体耐盐能力较强,直至711 min,最后1尾个体死亡。所有个体的平均存活时长为417.4 min。各个体在海水中的存活时长分布如图1。
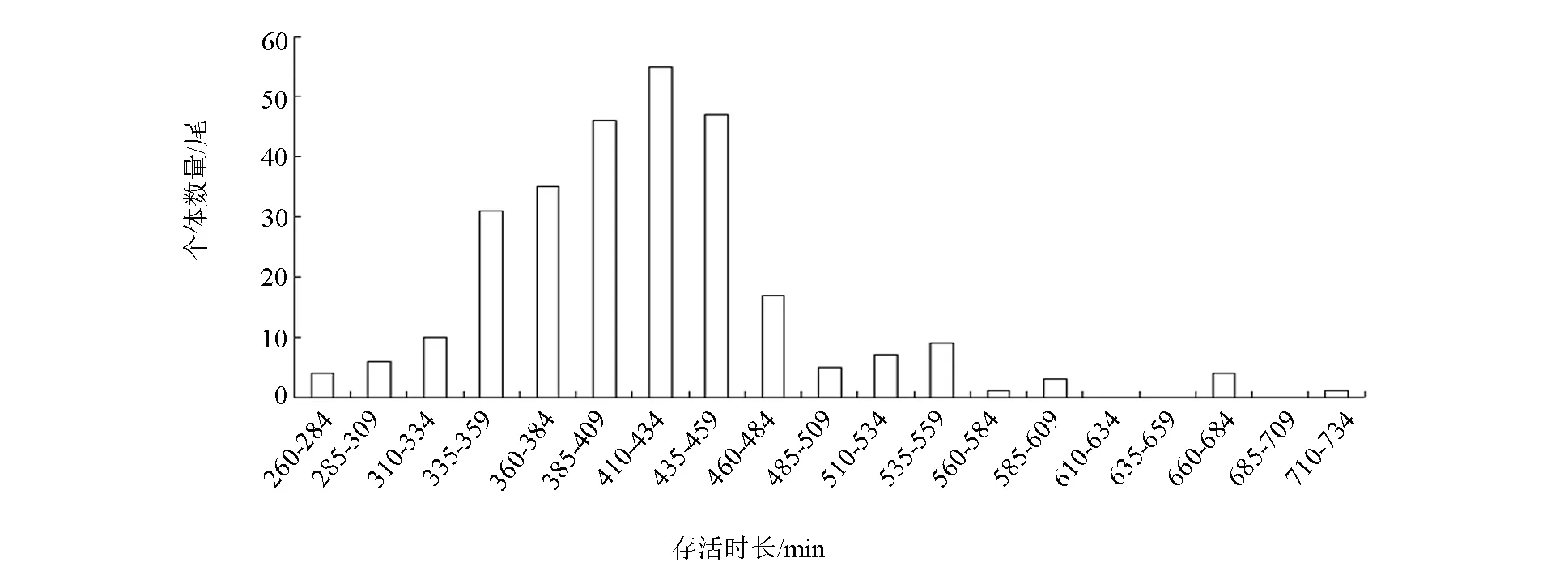
图1 各个体在海水中存活时长分布(死亡个体数量)
2.2 基因型检测与连锁图重建
采用171个标记对282个参考家系个体进行第1轮基因型检测,均在95%以上个体中获得基因型。这些标记连同“性别”标记,共172个标记用于连锁作图。采用JoinMap软件重建遗传连锁图,获得整合图谱,该图谱由22个连锁群组成,包含172个标记。进行第一轮QTL分析后,采用第1连锁群中新开发的19个分子标记,对282个参考家系个体进行了第2轮基因型检测,并基于所有标记的基因型,再次进行了遗传连锁图谱的重建。重建后的第1连锁群包含29个分子标记(见图2),相关图谱文件被用于后续的QTL精细定位分析。

图2 重建的第1连锁群图谱
2.3 QTL定位分析
以各个体在海水中的存活时长作为耐盐性状,对检测的所有参考家系个体进行QTL分析,获得6个QTL,分别位于第1、12、21、22等4个连锁群。其中,位于第1连锁群的st-c的LOD值达到8.82,可解释19.2%的表型差异(见表3)。
在本研究中发现,雌、雄鱼的平均存活时长存在显著差异(P<0.05)。为排除两性差异对分析的干扰,获得更多的QTL,对雌、雄鱼分别进行了QTL定位分析。对雌性个体进行分析后获得6个QTL,分别分布在第10、11、12、14、22等5个连锁群;对雄性个体进行分析后获得3个QTL,分别分布在第1、12、22等3个连锁群,其中mst-a的LOD值达到6.29,可解释19.1%的表型差异(见表3)。
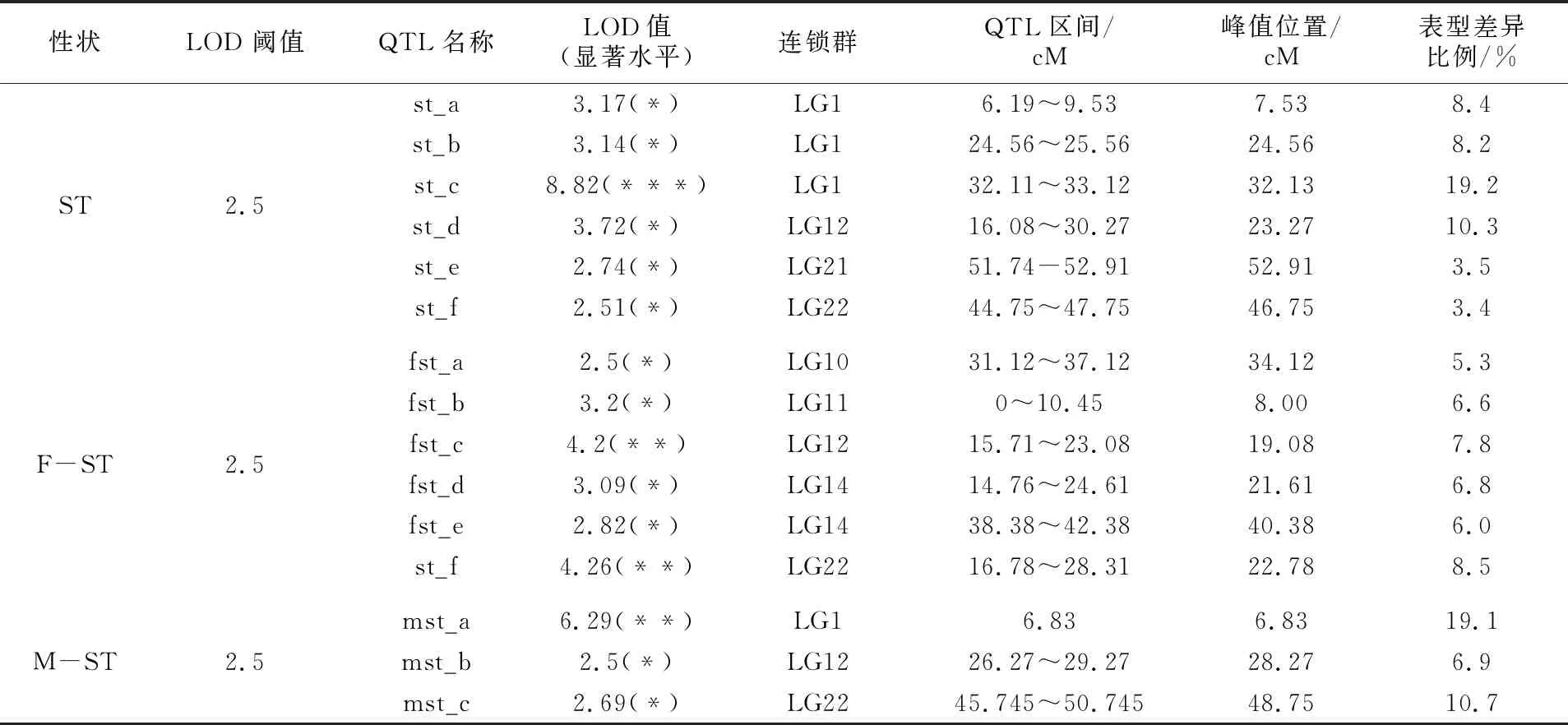
表3 耐盐QTL明细表
两性耐盐QTL st-c与雄性耐盐QTL mst-a的LOD值较高,解释的表型差异比例均大于19%,为主效QTL,因此选定这2个QTL进行进一步的精细定位。采用新开发分子标记进行第2轮基因型检测后,基于所有分子标记基因型、重建的遗传连锁图谱及各个体的表型值,采用MapQTL软件的MQM方法进行定位分析,将st-c定位于第1连锁群的32.11~33.12 cM,峰值位于32.13 cM,将mst-a定位于第1连锁群的6.83 cM处(见表3)。在st-c中,位于峰值处的标记为“性别”标记;在mst-a中,位于峰值处的标记为钠钾氯协同转运蛋白2(NKCC2)基因内含子中的1个微卫星标记(见图3)。
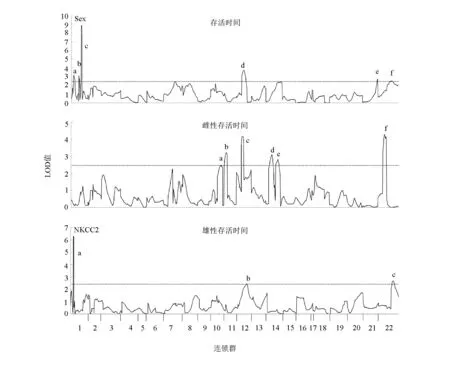
注:图中小写字母为存活时间(ST)、雌性存活时间(F-ST)、雄性存活时间(M-ST)等三个性状的QTL编号。
2.4 性别及NKCC2基因型对耐盐性状的影响
在282个参考家系个体中,雌性共156尾,雄性共126尾。对雌、雄鱼的存活时长分别进行统计,结果表明,雌、雄鱼在海水中的平均存活时长分别为394.0 min和446.7 min,二者差异极显著(P<0.001)。
NKCC2基因内标记的检测结果表明,在参考家系中存在A、B两种NKCC2基因型。对所有个体进行统计,A型的平均存活时长为420.4 min,B型的平均存活时长为414.9 min,差异不显著(P>0.05);对雌性个体进行统计,A型的平均存活时长为395.6 min,B型的平均存活时长为385.3 min,差异不显著(P>0.05);对雄性个体进行统计,A型的平均存活时长为508.8 min,B型的平均存活时为431.0 min,差异极显著(P<0.001)(见图4)。
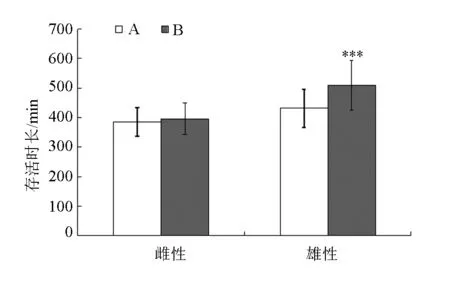
注:A、B为NKCC2的两种基因型;***表示基因型间差异极显著(P<0.001)。
3 讨论
3.1 耐盐罗非鱼的选育与耐盐QTL定位
罗非鱼是典型的广盐性鱼类,种类众多,但仅有莫桑比克罗非鱼等少数种类及佛罗里达红罗非鱼等杂交种适合在海水等高盐度水体中养殖[13]。然而,莫桑比克罗非鱼等耐盐罗非鱼生长较慢,在生产中应用的罗非鱼养殖品系主要来自于耐盐性较差的尼罗罗非鱼及部分杂交种[14]。此外,采用海水或半咸水养殖的罗非鱼没有淡水养殖罗非鱼常有的泥腥味,肉质更好[15],售价高于淡水罗非鱼。由于全球淡水资源缺乏,进行耐盐罗非鱼的选育与养殖已成为罗非鱼产业的一个重要方向[5]。采用传统方法进行罗非鱼选育周期较长,如能开发耐盐相关分子标记进行辅助选择,则可加速选育进程。Jiang等[16]、Gu等[17]对耐盐性较差的尼罗罗非鱼及星洲红罗非鱼进行了耐盐QTL定位研究,在尼罗罗非鱼的LG14及LG18中定位了2个QTL,在星洲红罗非鱼LG18中定位了1个QTL。本研究以高耐盐的莫桑比克罗非鱼与低耐盐的红罗非鱼杂交建立参考家系,并进行耐盐QTL的定位分析,从中筛选得到15个耐盐QTL,分布于LG1、LG10、LG11、LG12、LG14、LG21、LG22等7个连锁群,这些QTL中的分子标记将可用于耐盐罗非鱼分子标记辅助选育,促进罗非鱼耐盐机理的研究以及耐盐罗非鱼新品系的快速选育。
3.2 NKCC2与罗非鱼耐盐性状
钠钾氯协同转运蛋白(NKCC)是一类进行钠、钾、氯离子跨膜转运的蛋白,在脊椎动物的体液平衡中起到极为关键的作用,主要包括NKCC1、NKCC2两种[18]。NKCC1主要在鱼类的鳃部表达,与鳃的氯离子分泌有关[19]。NKCC2主要在肠、肾中表达,通过氯离子的转运促进水的吸收[20]。海水中生活的欧鳗,在黄鳗、银鳗两个时期,其肠NKCC2的表达量均显著高于淡水中生活的个体[20];海鲷(SparusaurataL.)在盐度55条件下肠NKCC2的表达量为盐度12条件下的3倍[21]。这些研究均表明,NKCC2在鱼类的渗透压调节及海水适应中起到非常重要的作用。本研究中发现,NKCC2基因位于第1连锁群的主效QTL mst-a峰值处,该QTL可解释雄性耐盐性状19.1%的表型差异;NKCC2的A、B两种基因型在雄性中的耐盐性状差异极显著,但在雌性中的差异不显著。这些结果表明,NKCC2的基因差异很可能决定罗非鱼耐盐性状的部分个体差异,且性状差异的大小与性别存在一定关系。本研究开发的NKCC2基因内标记可作为重要候选分子标记用于罗非鱼耐盐性状的分子育种。
3.3 罗非鱼耐盐性状的两性差异
本研究在第1连锁群定位了1个主效耐盐QTL st-c,可解释两性耐盐性状的19.2%,QTL的峰值处为“性别”标记,该标记代表着1个XY性别决定位点。对雌、雄罗非鱼耐盐性状的统计表明,罗非鱼具有显著的两性耐盐差异,从淡水直接转入海水后,雌性和雄性罗非鱼的存活时长分别为394.0 min和446.7 min,二者差异极显著。这些结果表明,雄性罗非鱼的耐盐性状显著优于雌性。已有研究表明,柳、青杨等植物具有显著的两性耐盐差异[22-23],但至今为止,鲜有鱼类两性耐盐差异的研究。本研究中发现的罗非鱼两性耐盐差异的形成机理还有待进一步研究。
本研究结果表明,在半咸水或海水中进行罗非鱼的养殖,选择全雄群体具有一定的耐盐优势。

