桃XTH家族基因鉴定及其在桃果实贮藏过程中的表达特性
郭绍雷,许建兰,王晓俊,2,宿子文,2,张斌斌,马瑞娟,俞明亮
桃XTH家族基因鉴定及其在桃果实贮藏过程中的表达特性
郭绍雷1,许建兰1,王晓俊1,2,宿子文1,2,张斌斌1,马瑞娟1,俞明亮1
1江苏省农业科学院果树研究所/江苏省高效园艺作物遗传改良重点实验室,南京 210014;2南京农业大学园艺学院,南京 210095
【目的】通过桃木葡聚糖内糖基转移/水解酶(xyloglucan endotransglucosylase/hydrolase,XTH)基因家族鉴定与不同肉质桃贮藏过程中相关基因的表达分析,发掘PpXTHs家族成员中参与桃果实软化的重要候选基因,为深入解析在果实采后贮藏过程中的功能研究奠定基础。【方法】根据XTH蛋白保守结构域Glyco_hydro_16 domain和XET_C domain,利用Hmmer 3.1软件对桃蛋白质数据库进行搜索,鉴定桃XTH基因家族成员;利用在线软件ProtParam预测其分子量、理论等电点等理化性质;利用在线分析工具Plant-mPLoc预测其亚细胞定位;MEGA11软件构建系统进化树;运用在线工具MEME对其保守motif进行分析,Tbtools呈现蛋白保守结构域和基因结构图谱;MapChart软件绘制基因在染色体上的分布图;利用实时荧光定量PCR(quantitative reverse transcription PCR,qRT-PCR)技术检测在不同肉质桃贮藏过程中的表达特性。【结果】在桃基因组共鉴定XTH基因家族成员27个,分布在7条染色体上。系统进化树显示,PpXTHs家族成员可分为祖先类群、Ⅰ/Ⅱ亚家族以及ⅢA和ⅢB亚家族。蛋白结构域分析显示所有PpXTHs成员均含有Glyco_hydro_16和XET_C两个蛋白保守结构域。qRT-PCR结果表明,属于ⅢB亚家族的随着溶质桃贮藏期延长,表达量上调,且表达量显著高于同期硬质桃贮藏过程的表达水平;克隆测序结果显示其CDS序列与桃参考基因组一致,长度为924 bp,编码307个氨基酸;激光共聚焦显微镜观察发现PpXTH33与GFP融合蛋白可能主要于细胞壁与细胞核上产生绿色荧光信号。【结论】桃27个PpXTHs家族成员蛋白结构均含有2个XTH蛋白保守结构域,不均匀分布在7条染色体上。在溶质桃和硬质桃贮藏过程中的表达特性显示,与桃采后果实软化密切相关。
桃;果实软化;XTH基因家族;基因表达;亚细胞定位
0 引言
【研究意义】桃[(L.) Batsch]原产于中国,根据联合国粮农组织统计(http://www.fao.org/ faostat/en/#data),2019年我国桃种植面积84.1万hm2,产量1 584万t,均居世界首位。然而,桃果实普遍具有成熟后迅速软化,常温条件难久贮藏,不方便运输,货架期较短的特点[1],致使经济损失严重,极大制约了我国桃产业的发展。果实中细胞壁成分分解和细胞壁结构的改变对果实软化起主要作用[2],果实软化涉及诸多细胞壁修饰酶的作用,其中XTH作为一类多基因家族酶类在果实软化中发挥重要作用[3-5]。在桃上开展XTH基因家族研究可丰富果实采后衰老软化分子基础及调控网络,为创制桃果实采后品质维持的精准调控提供重要理论基础。【前人研究进展】木葡聚糖是双子叶植物细胞壁半纤维素主要成分,而半纤维素通过氢键与纤维素微纤丝连接构成的网络结构能够给细胞壁提供机械支撑,对细胞壁膨胀松软起到限制作用[6-8]。同时,XTH以木葡聚糖为底物,对木葡聚糖分子糖基具有转移和水解双重作用,木葡聚糖内糖基转移酶(xyloglucan endo-transglucosylase,XET)和木葡聚糖内糖基水解酶(xyloglucan endo-hydrolase,XEH)通过特异性催化、水解木葡聚糖1,4--D-糖苷键完成对细胞壁的扩张、降解、修复和形态发生[9-11]。XTH被认为是细胞生长过程中调节细胞壁延展性的关键酶[9,12],因此,XTH基因伴随果实的生长发育和成熟软化。XTH通过调控木葡聚糖代谢,进而降解细胞壁,为其他细胞壁相关酶的进一步修饰做准备[13],从而对果实成熟软化起重要作用。XTHs已从多种植物或组织中得到鉴定,为多基因家族编码酶[14-17]。苹果[18]、柿子[19]、猕猴桃[13]、梨[20]、草莓[21]、香蕉[22]等果树均有XTH相关报道。XTHs在不同物种中的家族基因数量存在差异,拟南芥上有33个[23]、水稻29个[24]、烟草56个[17]、大麦24个[16]、番茄25个[25]。最初在拟南芥上XTHs根据分支和拓扑结构可以分为第Ⅰ亚家族、第Ⅱ亚家族以及第Ⅲ亚家族[11],之后XTH在更多物种上被鉴定,近期XTHs家族基因进化关系研究发现Ⅰ类和Ⅱ类亚家族成员没有明显差异[24],将它们统称为Ⅰ/Ⅱ亚家族,故XTH可分为Ⅰ/Ⅱ、ⅢA和ⅢB三个亚家族[10,14-17]。不同亚家族具有的XTH活性不同,柿属于Ⅰ/Ⅱ亚家族成员,编码的蛋白表现出XET活性[26];同样地,拟南芥属于ⅢB亚家族成员也表现出明显的XET活性[10,27],ⅢA亚家族成员Tm-NXG1表现出XEH活性[10]。XTH基因家族属于第16糖苷水解酶家族(glycoside hydrolase family 16,GH16)[28],通常含有Glyco_hydro_16和XET_C两个保守结构域,XET_C蛋白结构域可与GH16家族中的其他蛋白区分[9,29-30]。诸多被报道与果实软化密切相关,苹果中的与果实成熟相关,且外源乙烯可诱导表达上调,参与果实软化[31]。在番茄果实中,超表达柿子,可加快番茄果实成熟和软化速率[5]。此外,草莓中的和也被报道与果实成熟软化密切相关[4]。【本研究切入点】XTH在果实软化中发挥重要作用,而桃上的相关鉴定和功能解析还未见报道。【拟解决的关键问题】本研究拟通过生物信息学手段鉴定桃果实XTH基因家族成员并对其进行特征分析,通过其在溶质桃和硬质桃采后过程的表达分析,发掘参与桃果实软化的重要,为研究的功能和在桃果实软化中的重要作用奠定基础。
1 材料与方法
1.1 试验材料
本研究以溶质桃‘湖景蜜露’(‘HJML’)和硬质桃‘霞脆’(‘XC’)为试验材料,两品种桃均种植于国家果树种质南京桃资源圃试验园,每品种选择3棵桃树进行试验。2018年采收,采收后立即运回实验室,果实以商业采收期为标准,选取成熟度一致、大小均匀、无病虫害和机械损伤的果实用于常温贮藏。贮藏温度为(25±0.5)℃,相对湿度75%—85%。两品种桃果实常温贮藏0、3、6和9 d后,分别随机取样,每次取5个果实,3次重复,共15个果实用于试验。桃果实去皮后,切成均匀小块,经液氮速冻后,置于-80℃冰箱用于后续试验。大肠杆菌菌株DH5α、根癌农杆菌GV3101购于上海唯地生物技术有限公司。
1.2 桃XTH基因家族成员鉴定及特征分析
从Pfam数据库(https://Pfam.xfam.org/)[32]下载XTH保守蛋白结构域HMM文件Glyco_hydro_16 domain(PF00722)和XET_C domain(PF06955)[14],运用Hmmer 3.1软件对桃蛋白质数据库(https:// phytozome-next.jgi.doe.gov/)[33]进行搜索,手动删除冗余序列,所得候选序列经CDD数据库(https://www. ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)[34]验证后,最终同时包含Glyco_hydro_16和XET_C保守结构域的蛋白序列用于后续研究。分子量(molecular weight,Mw)、理论等电点(theoretical isoelectric point,pI),氨基酸数量等利用在线工具ProtParam(https://web. expasy.org/protparam/)进行分析[35]。亚细胞定位利用在线分析软件Plant-mPLoc(http://www.csbio.sjtu.edu. cn/bioinf/plant-multi/)进行预测[36]。每条蛋白序列的motif使用MEME在线工具(https://meme-suite.org/ meme/tools/meme)[37]进行预测和分析。使用TBtools对每条基因序列的内含子及外显子进行分析,最终每条PpXTHs蛋白序列保守结构域和motif使用Tbtools呈现[38]。
1.3 桃XTH蛋白系统进化树分析
桃XTH家族成员基因组序列、CDS序列及蛋白质序列均来自Phytozome数据库(https://phytozome- next.jgi.doe.gov/)[33]。拟南芥XTH蛋白序列均来自TAIR数据库(https://www.arabidopsis.org/)[39]。番茄XTH蛋白序列根据SALADIÉ等[25]的报道。其他XTH蛋白序列根据前人报道获得:Tm- NXG1[10]、MdXTH10[31]、Ptt-XET16-34[40]、DkXTH6[26]。多序列比对使用Clustal X1.8软件,利用MEGA11软件[41]最大似然法用于构建进化树,使用LG+G模型,自展重复次数设为100。
1.4 桃XTH蛋白序列结构比对分析及染色体分布
从PDB数据库中下载晶体结构TmNXG1(PDB id: 2UWA)[10]和Ptt-XET16-34(PDB id: 1UN1)[40],使用ESPript在线工具(https://espript.ibcp.fr/ESPript/ESPript/)进行二级结构预测与共有结构序列鉴定[21,42]。桃XTH基因的染色体位置信息从Phytozome数据库(https:// phytozome-next.jgi.doe.gov/)[33]获取,使用MapChart软件绘制基因在染色体上的分布图[43]。
1.5 桃XTH家族成员的表达分析
RNA提取采用多糖多酚植物总RNA提取试剂盒(天根生化科技(北京)有限公司),按试剂盒说明书步骤操作,提取的总RNA用1%的琼脂糖凝胶电泳和One DropTMOD-1000+型紫外可见分光光度计(南京五义科技有限公司,中国)分别检测提取RNA的纯度和浓度。检测合格后的总RNA,使用PrimeScriptTMRT Reagent Kit(TaKaRa,大连)反转录试剂盒,去除基因组DNA后合成第一链cDNA作为qRT-PCR的模板。通过NCBI/Primer-BLAST在线服务器设计基因的特异性表达引物,内参基因选用桃2()[44]。引物由南京擎科生物科技有限公司合成,引物序列详见表1。利用ABI7500荧光定量PCR仪(ABI,美国)进行qRT-PCR检测,qRT-PCR试剂选用TB Green® Premix Ex Taq™ II(Tli RNaseH Plus)(TaKaRa,大连),按照其说明书进行操作,反应体系为20 μL,反应程序为:95℃,30 s;95℃,5 s,60℃,34 s,40个循环;72℃,10 s。相对表达量计算公式参照2-ΔΔCT[45]。
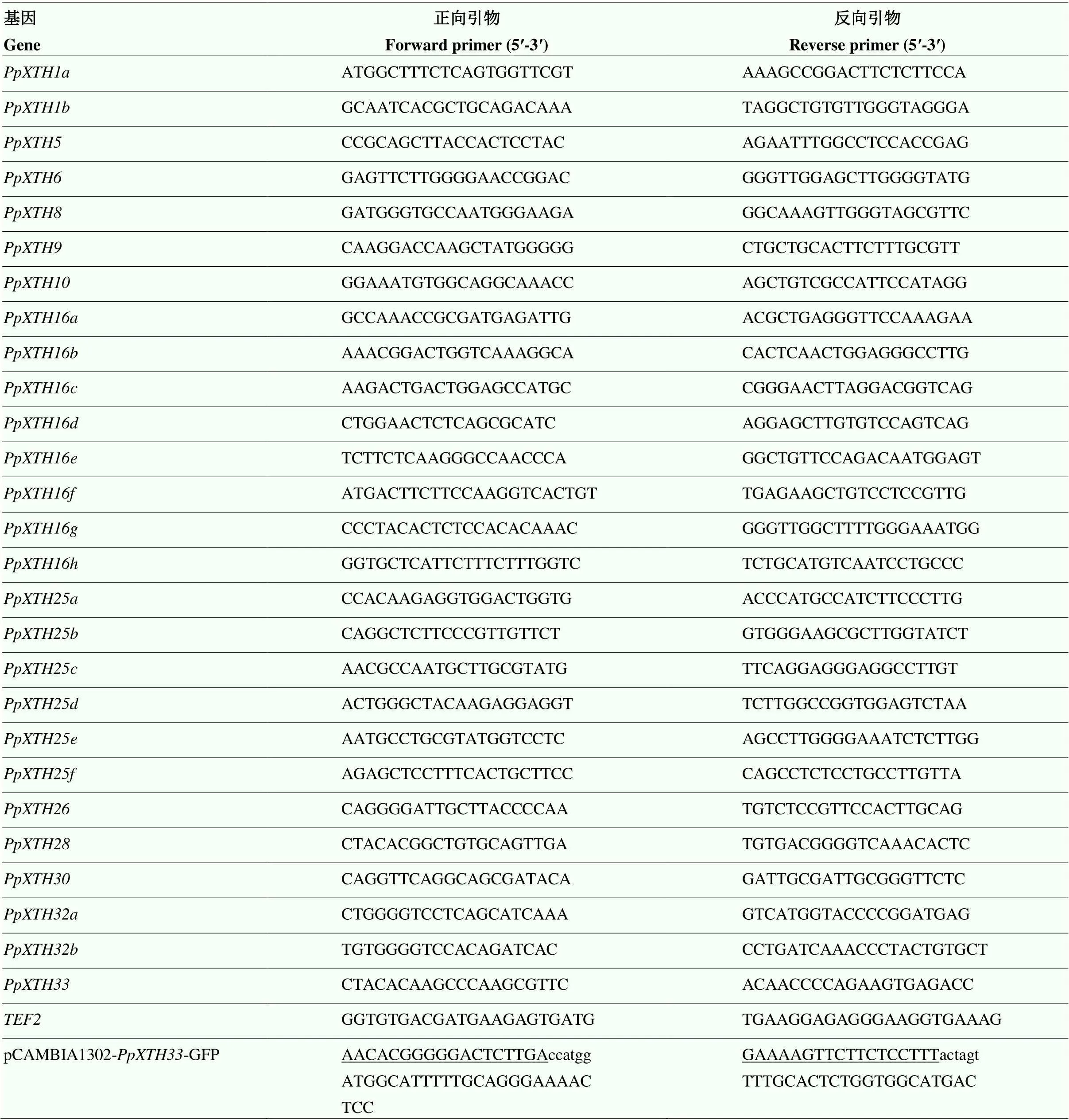
表1 用于qRT-PCR与亚细胞定位的引物
下划线为18 bp同源序列,小写字母为酶切位点
The underlined base sequences in the primers represent 18 bp extension homologous to vector ends, and lowercase letters represent corresponding restriction enzyme site
1.6 桃PpXTH33克隆与亚细胞定位
以‘HJML’和‘XC’桃果实cDNA为模板,使用PrimeSTAR® Max DNA聚合酶(TaKaRa,大连)进行扩增,PCR体系为50 μL,反应程序为:98℃,2 min;98℃,10 s,55℃,15 s,72℃,20 s,35个循环;72℃,5 min。利用ClonExpress® II One Step Cloning Kit试剂盒(诺唯赞,南京)按照试剂盒说明书使用DNA无缝克隆技术,将PCR纯化产物克隆至pCAMBIA1302载体,使用NEB限制性内切酶I和I对pCAMBIA1302载体双酶切,PCR扩增引物见表1。pCAMBIA1302-重组产物转化进大肠杆菌DH5α,过夜培养,经阳性鉴定后送南京擎科生物科技有限公司测序。将pCAMBIA1302(空载体)和pCAMBIA1302-重组质粒分别转化GV3101农杆菌,按照王昊等[46]的方法,选取1月龄烟草叶片注射后瞬时表达,注射3 d后利用激光共聚焦扫描显微镜观察在烟草叶片细胞中的定位。
1.7 数据统计分析
用Microsoft Excel 2019 进行数据整理和作图,使用SPSS 19.0 的t-检验,进行数据的差异显著性分析。
2 结果
2.1 桃XTH家族基因全基因组鉴定及特征分析
本研究去除冗余序列后共有28条XTH蛋白序列,Prupe.2G154800.1仅含有Glyco_hydro_16蛋白结构域,缺少XET_C蛋白结构域,不作为桃XTH基因家族成员进行研究。筛选后共有27条桃基因序列作为家族成员进行研究。桃根据拟南芥XTH家族成员同源序列进行命名(表2)。PpXHTs家族成员蛋白序列的氨基酸数量从269—359 ,伴随Mw从30 385.02—41 218.83 Da。pI范围从4.80—9.52(表2)。亚细胞定位预测显示桃上所有的XTHs都定位在细胞壁上。其中、、、、、、、、、、、、也能定位在细胞质中(表2)。
2.2 桃XTH蛋白系统进化树分析
本研究利用包括27个桃XTHs、33个拟南芥XTHs和13个番茄XHTs以及DkXTH6、Ptt-XET16-34、MdXTH10、Tm-NXG1在内的共77个XTH氨基酸序列构建系统进化树(图1),其中DkXTH6与MdXTH10分别在柿子和苹果上被报道与果实软化密切相关[26,31],Ptt-XET16-34具有XET活性[40],Tm-NXG1具有XEH活性[10],且Ptt-XET16-34、Tm-NXG1蛋白结构均已报道,因此根据系统进化树,分析桃XTH蛋白可能具有的活性及功能。如图1所示,构建的系统进化树中桃PpXTHs成员包含2个祖先类群(PpXTH1a和PpXTH1b),2个ⅢA亚家族成员(PpXTH32a和PpXTH32b)和3个ⅢB亚家族成员(PpXTH28、PpXTH30、PpXTH33),以及20个Ⅰ/Ⅱ亚家族成员。
2.3 桃XTH家族成员结构分析与染色体分布
如图2-A所示,PpXTHs家族成员分布在桃7条染色体上。其中第1染色体上含有的成员最多(10个),其次是第3染色体上含7个,第5染色体上仅含有,第2染色体上不包含。剩下的4条染色体上,除第8条染色体含有3个,其他染色体均只含有2个(图2-A)。为鉴定PpXTHs结构特征在进化中的保守性,对PpXTHs编码序列的外显子-内含子结构及蛋白序列含有的motif进行分析。结果表明,鉴定到的27个PpXTHs序列均包含Glyco_hydro_16和XET_C两个蛋白保守结构域(图2-C)。根据拟南芥进化树分类[11],属于第Ⅰ亚家族的PpXHTs(PpXTH5—10,除PpXTH5含5个外显子)成员含有4个外显子,第Ⅲ亚家族(ⅢA与ⅢB亚家族)和祖先类群成员同样含有4个外显子。属于第Ⅱ亚家族的桃PpXTHs成员(除PpXTH26含4个外显子),均含有3个外显子,聚集在同一亚家族中的PpXTHs成员motif分布差异不大(图2-B),表现出相似的基因结构模式(图2-D)。
根据两种完全解析的XTH蛋白结构Ptt-XET16- 34(PDB id: 1UN1)与TmNXG1(PDB id:2UWA)通过在线分析工具ESPript对PpXTHs蛋白质二级结构进行分析。(/W/R)-(/N)-E-(/L/F/V)-D-(/I/L/M)-E- (/L)-(L/M)-G(其中常见的氨基酸用下划线标示)是所有XTH中都包含的活性位点序列[9]。其中第一个谷氨酸残基(E)被认为是启动酶促反应的催化亲核体,第二个谷氨酸残基(E)激活进入的底物。如图3所示,桃上27个PpXTHs均含有活性位点序列,具有XTH的蛋白保守功能。PpXTHs蛋白序列包含保守的环序列(环1、环2、环3),环1与环3在PpXHTs蛋白序列中相对保守,变化不大,但在祖先类群PpXHT1a与PpXTH1b中环3相较于其他亚家族要短(图3-A),环2在ⅢA亚家族成员(PpXTH32a与PpXTH32b)中的长度比ⅢB亚家族及Ⅰ/Ⅱ亚家族成员要长(图3),环2在不同亚家族的差异可能导致其酶学功能的差异。
2.4 桃XTH家族成员在不同肉质桃常温贮藏过程表达分析
对21个PpXTHs家族成员在溶质桃‘HJML’和硬质桃‘XC’常温贮藏过程中的表达水平进行了分析(图4)。6个(、、、、、)因表达量太低未列图中。在Ⅰ/Ⅱ亚家族成员中有8个基因(、、、、、、、)在溶质桃‘HJML’和硬质桃‘XC’常温贮藏过程中呈现下调表达趋势,其中、和在两种肉质桃中变化不大,其他5个基因在相同贮藏期内,硬质桃的表达量高于溶质桃。在溶质桃贮藏过程中有下调趋势,在硬质桃贮藏3 d时表达量上调,随后下降,且的表达量在硬质桃贮藏过程中显著高于溶质桃。在两种肉质桃贮藏过程中贮藏6 d内表达量上升,随后下降。而在溶质桃‘HJML’和硬质桃‘XC’贮藏过程(0—6 d)表达量均上调,在贮藏期(3—9 d),硬质桃的表达量显著高于溶质桃。与的表达量在溶质桃和硬质桃贮藏过程中呈现上调趋势,贮藏9 d时的表达量在溶质桃中高于硬质桃,而的表达量在贮藏6 d时,溶质桃高于硬质桃,其他贮藏时期表达量差异不明显。在溶质桃‘HJML’贮藏期表达量一直升高,在硬质桃贮藏6 d内升高,而后表达量下降。与在两种肉质类型桃贮藏过程中的表达无明显规律。在两肉质桃贮藏3 d后表达量均上调。ⅢB亚家族成员中与的表达量在两肉质桃常温贮藏过程中均有先下降后升高的趋势,而在溶质桃常温贮藏过程中表达量显著上调,在硬质桃常温贮藏过程中只在贮藏后期(6—9 d)出现略微上调,在溶质桃常温贮藏过程中的表达量显著高于硬质桃,与两肉质桃的软化特征相符。溶质桃‘HJML’在贮藏3 d时硬度快速下降,且持续呈下降趋势,‘XC’贮藏过程中硬度变化较小,且‘XC’在贮藏0、3、6和9 d硬度显著高于‘HJML’(图5)。表明可能是编码XTH与桃果实软化相关的重要候选基因。祖先类群中在溶质桃贮藏过程中先升高后降低。
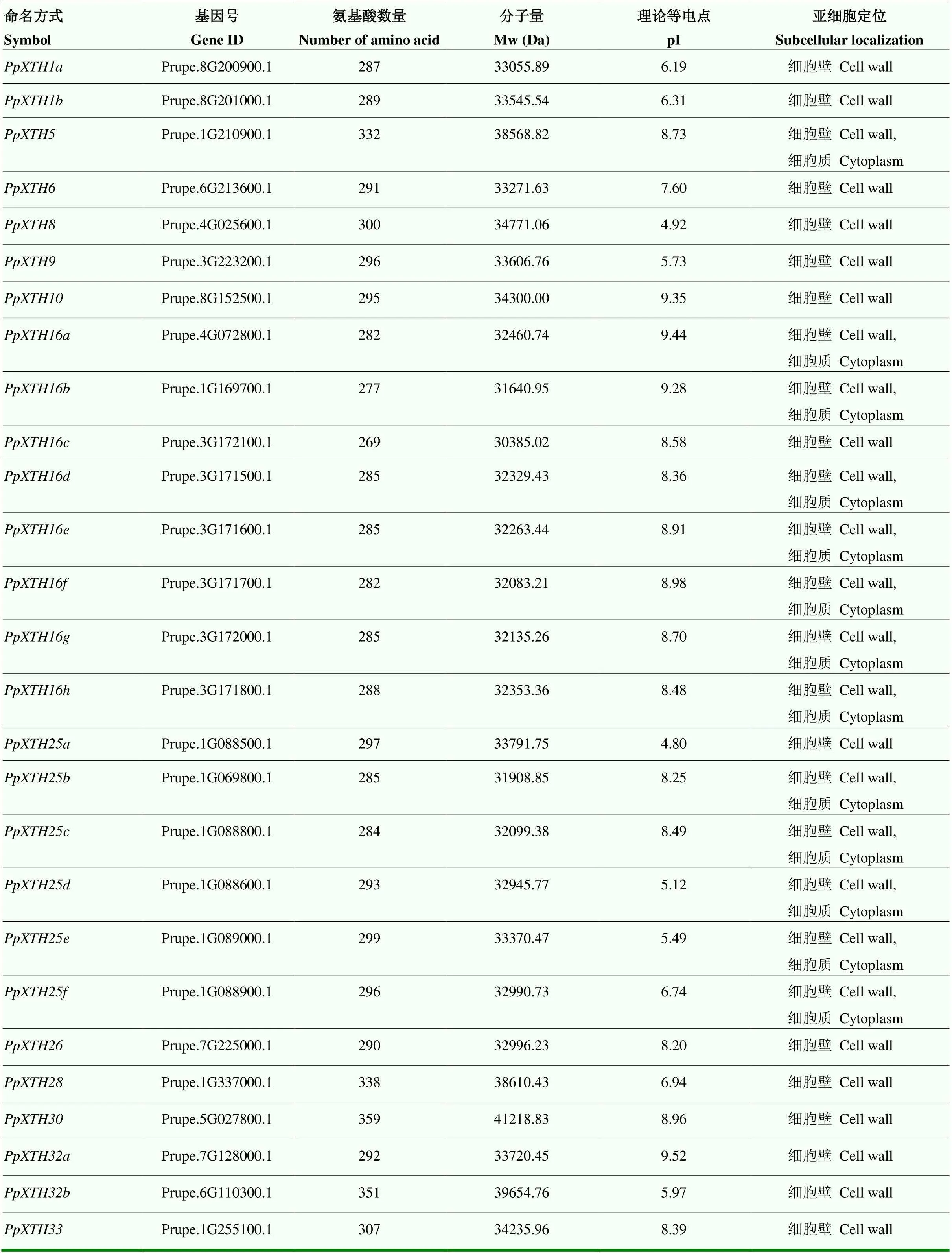
表2 桃XTH基因家族成员特征分析
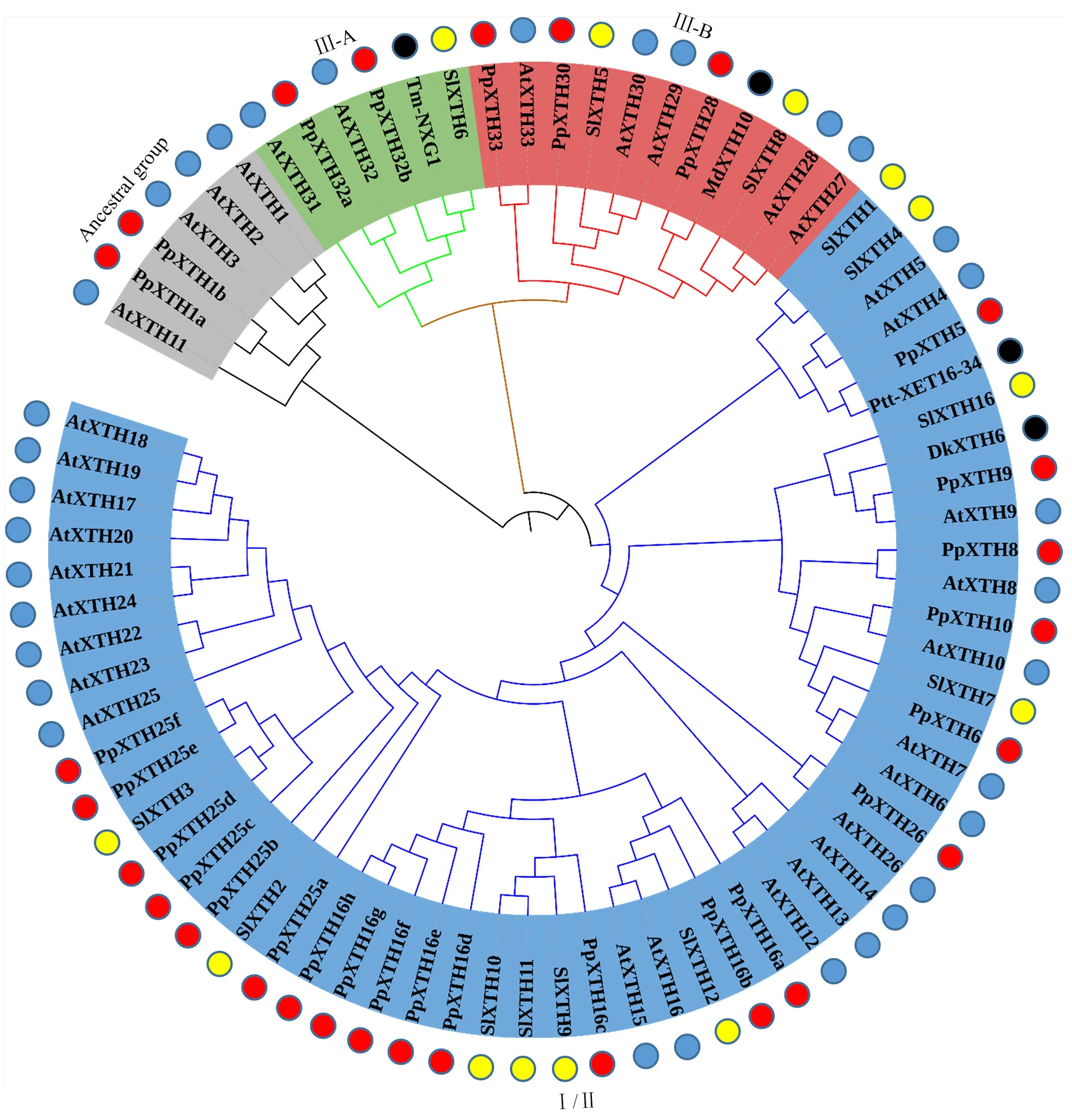
红圈代表桃XTH蛋白序列,蓝圈代表拟南芥XTH蛋白序列,黄圈代表番茄XTH蛋白序列,黑圈代表其他物种XTH蛋白序列

A:染色体分布;B:保守motif分布;C:domain分布;D:内含子与外显子结构
2.5 桃PpXTH33克隆及亚细胞定位分析
以桃果实‘HJML’和‘XC’cDNA为模板克隆,发现在溶质桃‘HJML’与硬质桃‘XC’果实中CDS序列结果一致,得到CDS序列924 bp,编码307个氨基酸,克隆得到的CDS序列与Phytozome数据库(https://phytozome-next.jgi.doe.gov/)桃参考基因组(v2.1)Prupe.1G255100.1序列一致。融合表达载体pCAMBIA1302-- GFP构建如图6-A所示。采用农杆菌介导的瞬时表达转化方法,进一步研究绿色荧光蛋白GFP标记的PpXTH33蛋白在烟草叶片表皮细胞中的定位。结合生物信息学分析结果,pCAMBIA1302-PpXTH33- GFP融合蛋白的烟草叶片可能定位在细胞壁中,同时也在细胞核发现了微弱的GFP荧光,表明PpXTH33蛋白可能主要在细胞壁中发挥作用(图6-B)。
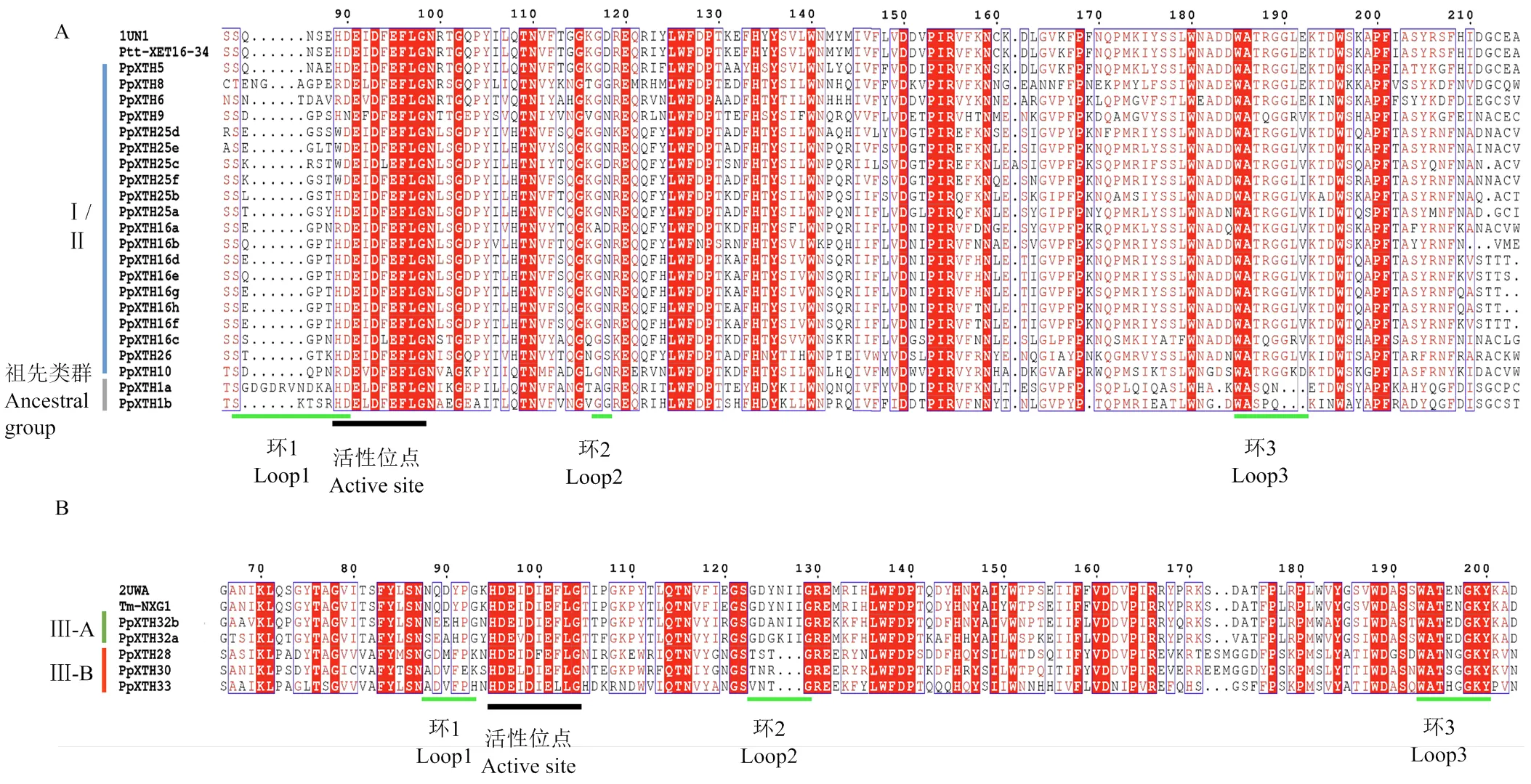
A:Ⅰ/Ⅱ亚家族、祖先类群;B:ⅢA和ⅢB亚家族,活性位点(黑色横线),环1、环2、环3(绿色横线),保守的氨基酸残基用红色字母表示
3 讨论
3.1 桃XTH家族基因鉴定及特征分析
桃果实快速衰老软化是影响品质维持的主要原因,而采后快速衰老软化的防控和相关理论的研究是降低果实采后损耗的基础。果实衰老软化是一个复杂的生理生化过程,主要包括呼吸跃变、乙烯释放、细胞壁降解及各种氧化还原酶的变化等[47-48]。果实中细胞壁成分分解和细胞壁结构的改变是引起果实软化的主要原因[2]。XTH调控木葡聚糖代谢,进而降解细胞壁,为其他细胞壁相关酶的进一步修饰做准备[13],参与果实软化。
在植物进化过程中,基因复制为基因功能多样化提供了来源,并有助于基因家族成员数量的扩增[15]。XTH家族成员在不同物种上存在的数目不尽相同[14,21,23,49-50],本研究在桃上共鉴定到27个XTH成员,与其他物种相比XTH同源基因数目不同,可能是因为在桃上基因重复事件发生的次数不同所导致。系统进化树分析PpXTHs与多数物种一样,其中ⅢA亚家族中包含的PpXTHs最少,含有2个,Ⅰ/Ⅱ亚家族成员最多,与大白菜、甘蓝、菠萝等报道结果类似[14-15]。所有PpXTHs亚细胞定位预测结果显示定位于细胞壁,与其参与细胞壁重组的功能吻合,说明PpXTHs具有XTH的保守性。尽管桃同源基因数目与其他物种不同,且PpXTHs在Mw、pI和氨基酸数量上存在显著差异,但PpXTHs在各亚家族中包含相对保守的motif和基因结构,表明同一组的XTH可能具有相似的功能。
3.2 PpXTH33为桃果实软化相关候选基因
前人研究表明属于Ⅰ/Ⅱ亚家族和ⅢB亚家族成员编码的XTH蛋白通常具有XET活性[11,26-27],而ⅢA亚家族成员的XTH蛋白具有XEH活性[6,10]。然而与果实软化相关的XTH基因通常属于Ⅰ/Ⅱ亚家族或ⅢB亚家族,如在草莓中超表达和(Ⅰ/Ⅱ亚家族成员),会导致草莓加快成熟并伴随硬度下降[4]。OPAZO等[21]研究表明属于Ⅰ/Ⅱ亚家族的与和属于ⅢB亚家族的与在果实成熟过程中表达量逐渐升高与草莓果实软化密切相关。超表达(Ⅰ/Ⅱ亚家族成员)会引起‘金冠’苹果与‘富士’苹果更快的软化速率和更早的乙烯高峰出现[3]。和在柿果实成熟期表达量升高并伴随硬度下降[19]。属于ⅢB亚家族的是成熟苹果果实表达量较高的基因[13],且受乙烯诱导表达上调[31],在番茄中超表达会导致与果实软化相关基因(、、、)的表达量上调,同时乙烯生物合成途径与信号转导途径基因(、、)表达量同样升高,表明可能激活软化相关基因或反馈调节乙烯来影响果实软化[18]。因此参与果实软化的XTHs基因可能来自Ⅰ/Ⅱ亚家族或ⅢB亚家族成员,具有XET活性,且伴随果实硬度的下降,表达量升高。‘HJML’为溶质桃果实,在常温贮藏过程中硬度迅速下降变软,‘XC’为硬质桃果实,在常温贮藏过程中长时间保持坚硬[51],这与本研究中溶质桃与硬质桃常温贮藏过程的硬度变化趋势一致。在溶质桃‘HJML’贮藏过程中随着硬度下降表达量不断升高,且随着贮藏时间延长,表达量显著高于硬质桃‘XC’(图4、图5)。因此,在转录水平上符合XTHs参与果实软化的特征,即随着硬度的下降表达量不断升高。XTH蛋白序列中Ⅰ/Ⅱ亚家族和ⅢB亚家族的环2要比ⅢA亚家族成员含有的氨基酸数量少,而环2是XTH蛋白活性偏向水解或转移酶活性的重要因素[9]。同样,环2的氨基酸数量区别于ⅢA亚家族,与Ⅰ/Ⅱ亚家族成员类似(图3),说明所编码的XTH蛋白可能具有XET活性。因此,参与果实软化可能与苹果类似,受乙烯调控,且与多数果实软化相关XTH基因类似,具有XET活性。亚细胞定位显示柿子XTH家族成员定位于细胞壁[26],说明XTH的作用部位在细胞壁,本研究中可能定位于细胞壁,同时也在细胞核中发现了GFP荧光,这可能是PpXTH33蛋白扩散所致,类似的情况也在向日葵上发现[52],因而桃XTH家族成员中的可能具有降解细胞壁的功能,也可能具有其他功能,是中参与桃果实软化的重要候选基因,在桃果实软化过程中起重要作用。但的基因功能和调控机理仍需进一步研究。
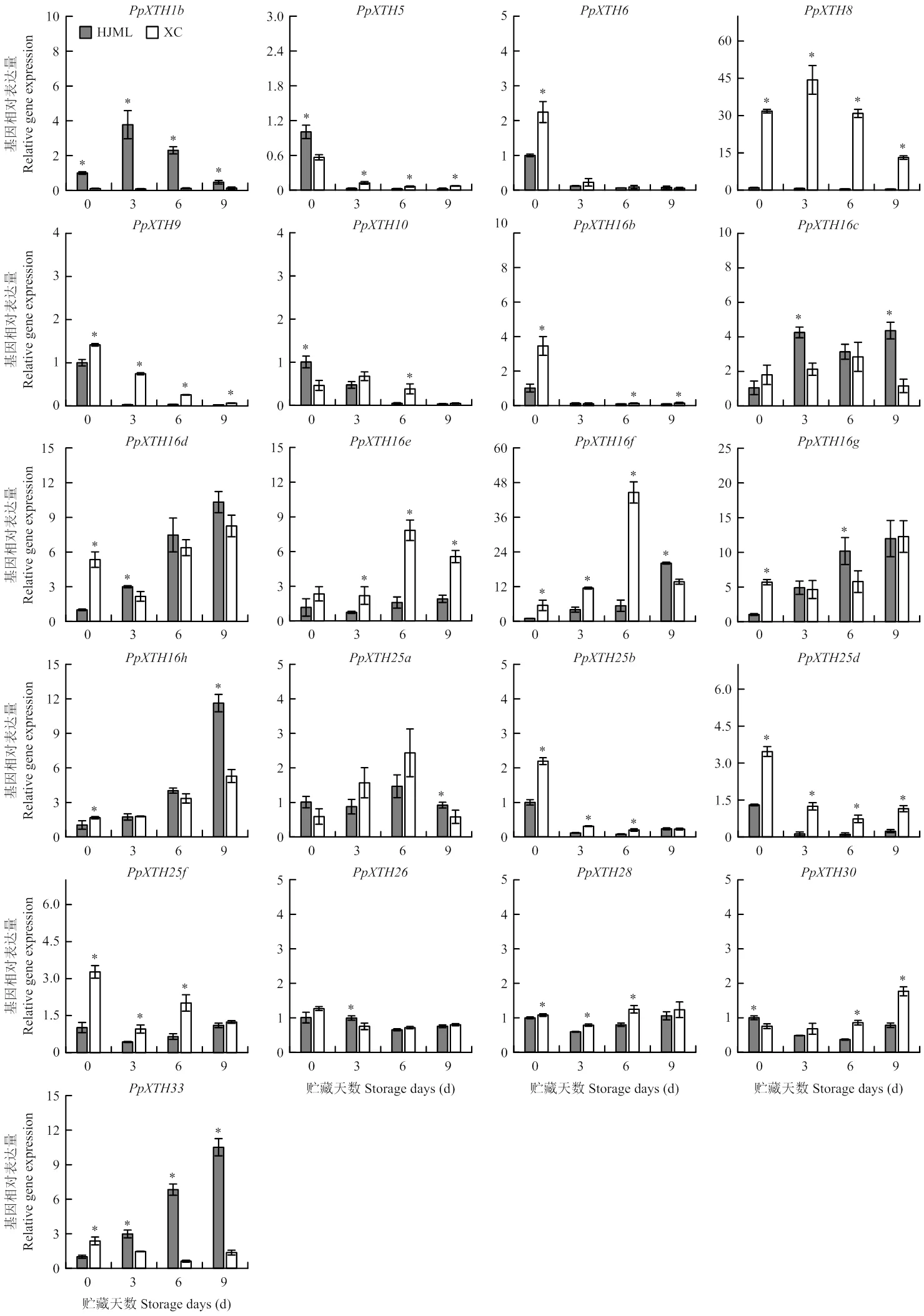
‘HJML’为溶质桃,‘XC’为硬质桃
HJML is the melting peach and XC is the stony hard peach
*表示同一时期不同肉质差异显著比较(<0.05)。下同
*indicate significant difference of different textures in the same period (<0.05). The same as below
图4 桃XTH家族成员在不同肉质桃常温贮藏过程中的表达特性
Fig. 4 Expression characteristics of XTH family members in peach fruit with different textures during room temperature storage
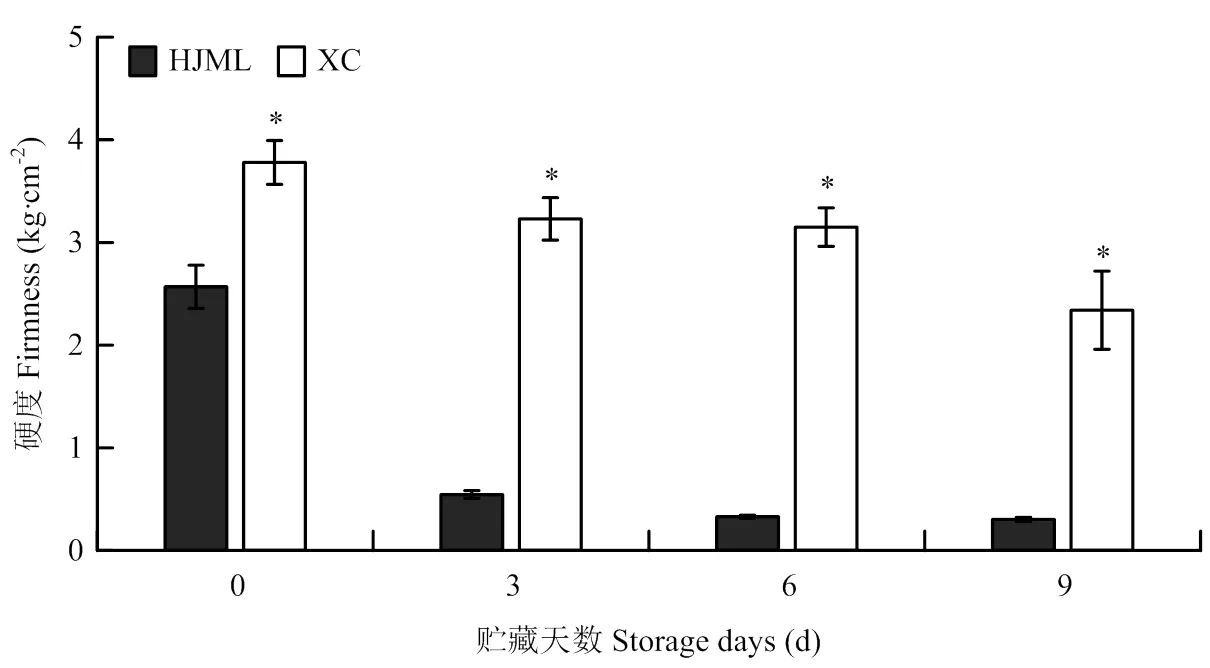
图5 不同肉质桃果实常温贮藏过程中硬度变化
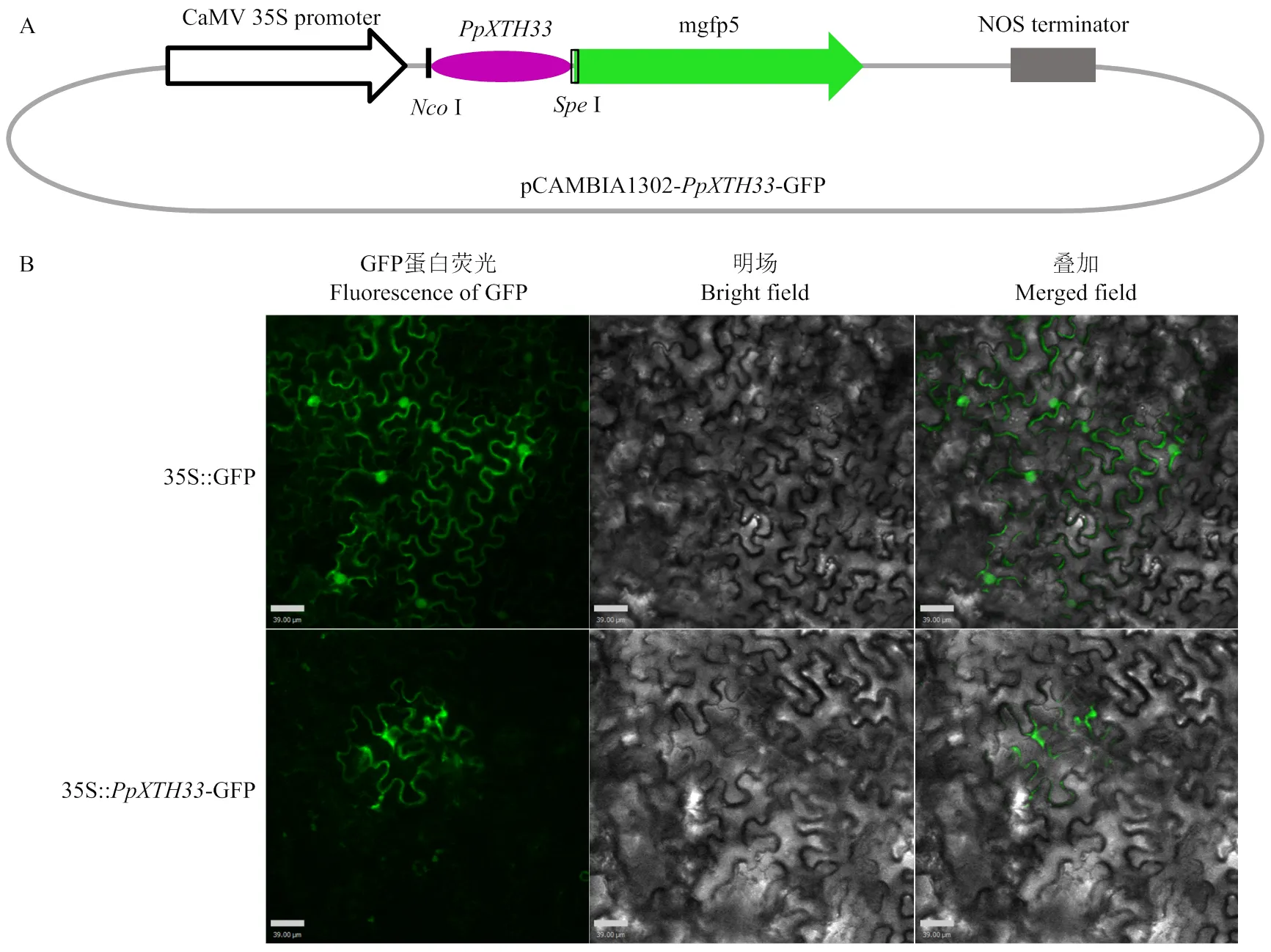
标尺长度=39.00 μm Scale bars=39.00μm
4 结论
从桃全基因组中共鉴定到27个XTH家族基因,均含有Glyco_hydro_16与XET_C保守结构域,不均匀分布在桃7条染色体上,系统进化树可将桃PpXTHs分为4个亚类。在溶质桃‘HJML’贮藏过程中随着硬度下降而表达量不断升高,且常温贮藏过程中表达量显著高于硬质桃‘XC’,为参与桃果实软化的重要候选基因。
[1] YOSHIOKA H, HAYAMA H, TATSUKI M, NAKAMURA Y. Cell wall modification during development of mealy texture in the stony-hard peach “Odoroki” treated with propylene. Postharvest Biology and Technology, 2010, 55(1): 1-7.
[2] BRUMMELL D A, HARPSTER M H. Cell wall metabolism in fruit softening and quality and its manipulation in transgenic plants. Plant Molecular Biology, 2001, 47(1/2): 311-340.
[3] MA M M, YUAN Y B, CHENG C X, ZHANG Y, YANG S L. Thegene is involved in fruit softening in ‘Golden Del. Reinders’ (). Journal of the Science of Food and Agriculture, 2021, 101(2): 564-572.
[4] WITASARI L D, HUANG F C, HOFFMANN T, ROZHON W, FRY S C, SCHWAB W. Higher expression of the strawberry xyloglucan endotransglucosylase/hydrolase genesandaccelerates fruit ripening. The Plant Journal, 2019, 100(6): 1237-1253. doi: 10.1111/tpj.14512.
[5] HAN Y, BAN Q Y, LI H, HOU Y L, JIN M J, HAN S K, RAO J P. DkXTH8, a novel xyloglucan endotransglucosylase/hydrolase in persimmon, alters cell wall structure and promotes leaf senescence and fruit postharvest softening. Scientific Reports, 2016, 6: 39155. doi: 10.1038/srep39155.
[6] 韩叶. 柿果实木葡聚糖内糖基转移/水解酶基因表达特性及功能分析[D]. 杨凌: 西北农林科技大学, 2017.
HAN Y. Expression and functional analysis of xyloglucan endotransglycosylase/hydrolase genes in persimmon fruit [D]. Yangling: Northwest A&F University, 2017. (in Chinese)
[7] ZHU Q G, ZHANG Z K, RAO J P, HUBER D J, LV J Y, HOU Y L, SONG K H. Identification of xyloglucan endotransglucosylase/ hydrolase genes (XTHs) and their expression in persimmon fruit as influenced by 1-methylcyclopropene and gibberellic acid during storage at ambient temperature. Food Chemistry, 2013, 138(1): 471-477. doi: 10.1016/j.foodchem.2012.09.141.
[8] SCHRÖDER R, ATKINSON R G, LANGENKÄMPER G, REDGWELL R J. Biochemical and molecular characterisation of xyloglucan endotransglycosylase from ripe kiwifruit. Planta, 1998, 204(2): 242-251. doi: 10.1007/s004250050253.
[9] EKLÖF J M, BRUMER H. The XTH gene family: an update on enzyme structure, function, and phylogeny in xyloglucan remodeling. Plant Physiology, 2010, 153(2): 456-466. doi: 10.1104/pp.110.156844.
[10] BAUMANN M J, EKLÖF J M, MICHEL G, KALLAS A M, TEERI T T, CZJZEK M, BRUMER H. Structural evidence for the evolution of xyloglucanase activity from xyloglucan endo-transglycosylases: biological implications for cell wall metabolism. The Plant Cell, 2007, 19(6): 1947-1963. doi: 10.1105/tpc.107.051391.
[11] ROSE J K C, BRAAM J, FRY S C, NISHITANI K. The XTH family of enzymes involved in xyloglucan endotransglucosylation and endohydrolysis: Current perspectives and a new unifying nomenclature. Plant and Cell Physiology, 2002, 43(12): 1421-1435. doi: 10.1093/ pcp/pcf171.
[12] COSGROVE D J. Growth of the plant cell wall. Nature Reviews Molecular Cell Biology, 2005, 6(11): 850-861.
[13] ATKINSON R G, JOHNSTON S L, YAUK Y K, SHARMA, N N, SCHRODER R. Analysis of xyloglucan endotransglucosylase/ hydrolase (XTH) gene families in kiwifruit and apple. Postharvest Biology and Technology, 2009, 51(2): 149-157.
[14] WU D, LIU A Q, QU X Y, LIANG J Y, SONG M. Genome-wide identification, and phylogenetic and expression profiling analyses ofgene families inL. andL.. BMC Genomics, 2020, 21(1): 782.
[15] LI Q Y, LI H Y, YIN C Y, WANG X T, JIANG Q, ZHANG R, GE F F, CHEN Y D, YANG, L. Genome-wide identification and characterization of xyloglucan endotransglycosylase/hydrolase induring development. Genes, 2019, 10(7): E537. doi: 10.3390/genes10070537.
[16] FU M M, LIU C, WU F B. Genome-wide identification, characterization and expression analysis of xyloglucan endotransglucosylase/hydrolase genes family in barley (). Molecules (Basel, Switzerland), 2019, 24(10): E1935. doi: 10.3390/molecules24101935.
[17] WANG M, XU Z C, DING A M, Kong Y Z. Genome-wide identification and expression profiling analysis of the xyloglucan endotransglucosylase/hydrolase gene family in tobacco (L.). Genes, 2018, 9(6): 273.
[18] ZHANG Z Y, WANG N, JIANG S H, XU H F, WANG Y C, WANG C Z, LI M, LIU J X, QU C Z, LIU W, WU S J, CHEN X L, CHEN X S. Analysis of the xyloglucan endotransglucosylase/hydrolase gene family during apple fruit ripening and softening. Journal of Agricultural and Food Chemistry, 2017, 65(2): 429-434. doi: 10.1021/acs.jafc. 6b04536.
[19] HAN Y, ZHU Q G, ZHANG Z K, MENG K, HOU Y L, Ban Q Y, SUO J T, RAO J P. Analysis of xyloglucan endotransglycosylase/ hydrolase () genes and diverse roles of isoenzymes during persimmon fruit development and postharvest softening. PLoS ONE, 2015, 10(4): e0123668.
[20] 丛郁, 刘洪, 李慧, 颜志梅, 俞明亮, 常有宏. 成熟砂梨果实木葡聚糖转移酶基因的克隆及其在夏季货架期的表达规律, 江苏农业学报, 2010, 26(1): 143-151.
CONG Y, LIU H, LI H, YAN Z M, YU M L, CHANG Y H. Cloning of an xyloglucan endotransglycosylase/hydrolase gene () from mature sandy pear fruit and its expression characteristics during summer shelf life. Jiangsu Journal of Agricultural Sciences, 2010, 26(1): 143-151. (in Chinese)
[21] OPAZO M C, LIZANA R, STAPPUNG Y, DAVIS T M, HERRERA R, MOYA-LEÓN M A. XTHs from: genomic structure and transcriptomic analysis in ripening fruit and other tissues. BMC Genomics, 2017, 18(1): 852. doi: 10.1186/s12864-017-4255-8.
[22] LU W J, NAKANO R, KUBO Y, INABA A, JIANG Y M. Cloning and expression analysis of an XET cDNA in the peel and pulp of banana fruit ripening and softening. Acta Botanica Sinica, 2004, 46(3): 355-362.
[23] YOKOYAMA R, NISHITANI K. A comprehensive expression analysis of all members of a gene family encoding cell-wall enzymes allowed us to predict cis-regulatory regions involved in cell-wall construction in specific organs of. Plant and Cell Physiology, 2001, 42(10): 1025-1033. doi: 10.1093/pcp/pce154.
[24] YOKOYAMA R, ROSE J K C, NISHITANI K. A surprising diversity and abundance of xyloglucan endotransglucosylase/hydrolases in rice. Classification and expression analysis. Plant Physiology, 2004, 134(3): 1088-1099.
[25] SALADIÉ M, ROSE J K, COSGROVE D J, CATALÁ C. Characterization of a new xyloglucan endotransglucosylase/hydrolase (XTH) from ripening tomato fruit and implications for the diverse modes of enzymic action. The Plant Journal, 2006, 47(2): 282-295. doi: 10.1111/j.1365-313x.2006.02784.x.
[26] HAN Y, BAN Q Y, HOU Y L, MENG K, SUO J T, RAO J P. Isolation and characterization of two persimmon xyloglucan endotransglycosylase/ hydrolase () genes that have divergent functions in cell wall modification and fruit postharvest softening. Frontiers in Plant Science, 2016, 7: 624.
[27] CAMPBELL P, BRAAM J.activities of four xyloglucan endotransglycosylases from. Plant Journal, 1999, 18(4): 371-382. doi: 10.1046/j.1365-313x.1999.00459.x.
[28] STROHMEIER M, HRMOVA M, FISCHER M, HARVEY A J, FINCHER G B, PLEISS J. Molecular modeling of family GH16 glycoside hydrolases: Potential roles for xyloglucan transglucosylases/ hydrolases in cell wall modification in the Poaceae. Protein Science, 2004, 13(12): 3200-3213. doi: 10.1110/ps.04828404.
[29] BEHAR H, GRAHAM S W, BRUMER H. Comprehensive cross- genome survey and phylogeny of glycoside hydrolase family 16 members reveals the evolutionary origin of EG16 and XTH proteins in plant lineages. Plant Journal, 2018, 95(6): 1114-1128. doi: 10.1111/tpj. 14004.
[30] MICHAILIDIS G, ARGIRIOU A, DARZENTAS N, TSAFTARIS A. Analysis of xyloglucan endotransglycosylase/hydrolase (XTH) genes from allotetraploid () cotton and its diploid progenitors expressed during fiber elongation. Journal of Plant Physiology, 2009, 166(4): 403-416. doi: 10.1016/j.jplph.2008.06.013.
[31] MUÑOZ-BERTOMEU J, MIEDES E, LORENCES E P. Expression of xyloglucan endotransglucosylase/hydrolase () genes and XET activity in ethylene treated apple and tomato fruits. Journal of Plant Physiology, 2013, 170(13): 1194-1201.
[32] FINN R D, COGGILL P, EBERHARDT R Y, EDDY S R, MISTRY J, MITCHELL A L, POTTER S C, PUNTA M, QURESHI M, SANGRADOR-VEGAS A, SALAZAR G A, TATE J, BATEMAN A. The Pfam protein families database: Towards a more sustainable future. Nucleic Acids Research, 2015, 44(D1): D279-D285. doi: 10.1093/nar/ gkv1344.
[33] GOODSTEIN D M, SHU S Q, HOWSON R, NEUPANE R, HAYES R D, FAZO J, MITROS T, DIRKS W, HELLSTEN U, PUTNAM N, ROKHSAR D S. Phytozome: A comparative platform for green plant genomics. Nucleic Acids Resesearch, 2012, 40(D1): D1178-D1186. doi: 10.1093/nar/gkr944.
[34] MARCHLER-BAUER A, BO Y, HAN L Y, HE J E, LANCZYCKI C J, LU S N, CHITSAZ F, DERBYSHIRE M K, GEER R C, GONZALES N R, GWADZ M, HURWITZ D I, LU F, MARCHLER G H, SONG J S, THANKI N, WANG Z X, YAMASHITA R A, ZHANG D C, ZHENG C J, GEER L Y, BRYANT S H. CDD/SPARCLE: Functional classification of proteins via subfamily domain architectures. Nucleic Acids Research, 2016, 45(D1): D200-D203. doi: 10.1093/nar/gkw1129.
[35] WILKINS M R, GASTEIGER E, BAIROCH A, SANCHEZ J C, WILLIAMS K L, APPEL R D, HOCHSTRASSER D F. Protein identification and analysis tools in the ExPASy server. Methods in Molecular Biology, 1999, 112: 531-552. doi: 10.1385/1-59259-584-7: 531.
[36] CHOU K C, SHEN H B. Cell-PLoc: A package of web servers for predicting subcellular localization of proteins in various organisms. Nature Protocols, 2008, 3(2): 153-162. doi: 10.1038/nprot.2007.494.
[37] BAILEY T L, JOHNSON J, GRANT C E, Noble W S. The MEME Suite. Nucleic Acids Research, 2015, 43(W1): W39-W49.
[38] CHEN C J, CHEN H, ZHANG Y, THOMAS H R, FRANK M H, HE Y H, XIA R. TBtools: An integrative toolkit developed for interactive analyses of big biological data. Molecular Plant, 2020, 13(8): 1194-1202. doi: 10.1016/j.molp.2020.06.009.
[39] LAMESCH P, BERARDINI T Z, LI D H, SWARBRECK D, WILKS C, SASIDHARAN R, MULLER R, DREHER K, ALEXANDER D L, GARCIA-HERNANDEZ M, KARTHIKEYAN A S, LEE C H, NELSON W D, PLOETZ L, SINGH S, WENSEL A, HUALA E. TheInformation Resource (TAIR): Improved gene annotation and new tools. Nucleic Acids Research, 2011, 40(D1): D1202-D1210. doi: 10.1093/nar/gkr1090.
[40] JOHANSSON P, BRUMER H, BAUMANN M J, KALLAS A M, HENRIKSSON H, DENMAN S E, TEERI T T, JONES T A. Crystal structures of a poplar xyloglucan endotransglycosylase reveal details of transglycosylation acceptor binding. The Plant Cell, 2004, 16(4): 874-886. doi:10.1105/tpc.020065.
[41] TAMURA K, STECHER G, KUMAR S. MEGA11: Molecular evolutionary genetics analysis version 11. Molecular Biology and Evolution, 2021, 38(7): 3022-3027. doi: 10.1093/molbev/msab120.
[42] ROBERT X, GOUET P. Deciphering key features in protein structures with the new ENDscript server. Nucleic Acids Research, 2014, 42(W1): W320-W324. doi: 10.1093/nar/gku316.
[43] VOORRIPS R E. MapChart: software for the graphical presentation of linkage maps and QTLs. Journal of Heredity, 2002, 93(1): 77-78. doi: 10.1093/jhered/93.1.77.
[44] TONG Z G, GAO Z H, WANG F, ZHOU J, ZHANG Z. Selection of reliable reference genes for gene expression studies in peach using real-time PCR. BMC Molecular Biology, 2009, 10: 71. doi: 10.1186/ 1471-2199-10-71.
[45] LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2(T)(-Delta Delta C) method. Methods, 2001, 25(4): 402-408.
[46] 王昊, 尹莲, 刘洁霞, 贾丽丽, 丁旭, 沈迪, 冯凯, 徐志胜, 熊爱生. 类胡萝卜素裂解双加氧酶基因AgCCD4调控芹菜不同组织的着色. 中国农业科学, 2021, 54(15): 3279-3294.
WANG H, YIN L, LIU J X, JIA L L, DING X, SHEN D, FENG K, XU Z S, XIONG A S. The carotenoid cleavage dioxygenases gene AgCCD4 regulates the pigmentation of celery tissues with different colors. Scientia Agricultura Sinica, 2021, 54(15): 3279-3294. (in Chinese)
[47] 徐小迪, 李博强, 秦国政, 陈彤, 张占全, 田世平. 果实采后品质维持的分子基础与调控技术研究进展. 园艺学报, 2020, 47(8): 1595-1609.
XU X D, LI B Q, QIN G Z, CHEN T, ZHANG Z Q, TIAN S P. Molecular basis and regulation strategies for quality maintenance of postharvest fruit. Acta Horticulturae Sinica, 2020, 47(8): 1595-1609. (in Chinese)
[48] IQBAL N, KHAN N A, FERRANTE A, TRIVELLINI A, FRANCINI A, KHAN M I R. Ethylene role in plant growth, development and senescence: interaction with other phytohormones. Frontiers in Plant Science, 2017, 8: 475. doi: 10.3389/fpls.2017.00475.
[49] ZHAI Z F, FENG C, WANG Y Y, Sun Y T, Peng X, Xiao Y Q, Zhang X, Zhou X, Jiao J L, Wang W L. Genome-wide identification of the xyloglucan endotransglucosylase/hydrolase (XTH) and polygalacturonase (PG) genes and characterization of their role in fruit softening of sweet cherry. International Journal of Molecular Sciences, 2021, 22(22): 12331. doi: 10.3390/ijms222212331.
[50] CHENG Z H, ZHANG X M, YAO W J, GAO Y, ZHAO K, GUO Q, ZHOU B R, JIANG T B. Genome-wide identification and expression analysis of the xyloglucan endotransglucosylase/hydrolase gene family in poplar. BMC Genomics, 2021, 22(1): 804. doi: 10.1186/ s12864-021-08134-8.
[51] 杨勇, 马瑞娟, 张斌斌, 宋志忠, 张春华, 郭绍雷, 俞明亮. 不同溶质桃果实的软化与乙烯合成相关基因的差异表达. 园艺学报, 2015, 42(10): 1869-1878.
YANG Y, MA R J, ZHANG B B, SONG Z Z, ZHANG C H, GUO S L, YU M L. Differential expression analysis in fruit softening and ethylene biosynthetic pathways in peaches of different flesh textures. Acta Horticulturae Sinica, 2015, 42(10): 1869-1878. (in Chinese)
[52] CHEEVARUNGNAPAKUL K, KHAKSAR G, PANPETCH P, BOONJING P, SIRIKANTARAMAS S. Identification and functional characterization of genes involved in the biosynthesis of caffeoylquinic acids in sunflower (L.). Frontiers in Plant Science, 2019, 10: 968. doi: 10.3389/fpls.2019.00968.
Genome-Wide Identification and Expression Analysis of XTH Gene Family in Peach Fruit During Storage
GUO ShaoLei1, XU JianLan1, WANG XiaoJun1,2, SU ZiWen1,2, ZHANG BinBin1, MA RuiJuan1, YU MingLiang1
1Institute of Pomology, Jiangsu Academy of Agricultural Sciences/Jiangsu Key Laboratory for Horticultural Crop Genetic Improvement, Nanjing 210014;2College of Horticulture, Nanjing Agricultural University, Nanjing 210095
【Objective】The aim of this study was to identify members of the XTH gene family from peach, and to analyze the expression ofin peach fruit with different textures during storage, which not only provided data for the research on the candidateinvolved in peach fruit softening, but also laid the foundation for further study on thefunction in peach fruit softening.【Method】The HMM profiles of the Glyco_hydro_16 domain and XET_C domain were used to search all XTH proteins with the Hmmer 3.1 software in the peach protein database. The molecular weight, theoretical isoelectric point and other physicochemical properties were then predicted by the online tool ProtParam. PpXTHs subcellular localization were predicted by the online software Plant-mPLoc. The MEGA 11 software was used to construct a phylogenetic tree. The online tool MEME was used to analyze conserved motifs, the conserved motifs, conserved protein domains and gene structure maps were draw by Tbtools.According to the PpXTH gene family location information, chromosome mapping was carried out with MapChart software. The expressions ofin peach fruit with different textures during storage were monitored by qRT-PCR. 【Result】A total of 27genes were systematically identified from peach, which were distributed on seven chromosomes. Based on the phylogenetic tree, the ancestral group, Ⅰ/Ⅱ subfamily, ⅢA subfamily and ⅢB subfamily were classified. In addition, according to the analysis of protein domains, all PpXTH gene family proteins had Glyco_hydro_16 and XET_C conserved domain. The results from qRT-PCR analysis showed thatbelonging to the ⅢB subfamily was upregulated as the storage period increased in melting peach fruit, with the expression being markedly higher than that during the storage period of stony hard peach fruit. The positive clone sequencing was consistent with the coding sequence of thereference genome with a length of 924 bp for a 307 amino acid sequence.The PpXTH33 combined with green fluorescent protein may mainly located in the cell wall and nucleus detected by confocal laser scanning microscopy. 【Conclusion】All 27protein structures contained two highly XTH conserved domains and the genes weredistributed on seven chromosomes. The expression characteristics ofwas closely associated with peach fruit softening during storage.
peach; fruit softening; XTH gene family; gene expression; subcellular localization

10.3864/j.issn.0578-1752.2022.23.011
2022-02-23;
2022-07-24
江苏省重大新品种创制(PZCZ201727)、现代农业产业技术体系建设专项资金(CARS-30)
郭绍雷,E-mail:guoshaolei0305@126.com。通信作者俞明亮,E-mail:mly1008@aliyun.com
(责任编辑 赵伶俐)

