小麦响应干旱胁迫环状RNA的鉴定
李宁,柳坤,刘彤彤,史雨刚,王曙光,杨进文,孙黛珍
小麦响应干旱胁迫环状RNA的鉴定
李宁,柳坤,刘彤彤,史雨刚,王曙光,杨进文,孙黛珍
山西农业大学农学院,山西太谷 030801
【目的】干旱是限制全球小麦生产的主要非生物胁迫之一,探索小麦应对干旱的分子调控机制对小麦分子育种具有重要意义。环状RNA(circRNA)已被证实在植物抵御外界环境胁迫的过程中扮演着重要角色。鉴定小麦响应干旱胁迫的circRNA,有助于构建小麦干旱胁迫响应的调控网络,为解析小麦的抗旱性机制奠定基础。【方法】以2个抗旱性差异显著的小麦品种(周麦13和冀麦38)为试验材料,对其在干旱及对照条件下的根部样本进行circRNA测序。鉴定小麦circRNA并对其进行特征分析,筛选与干旱胁迫响应相关的差异表达circRNA,并对其靶向microRNA(miRNA)进行预测。进一步根据miRNA及其靶基因在干旱胁迫下的表达模式,构建小麦响应干旱胁迫的潜在circRNA-miRNA-mRNA调控模块。【结果】共鉴定获得1 409个小麦circRNA,其中,多数(68.91%)为外显子circRNA,且仅有133个circRNA在2个品种中被同时鉴定获得。在干旱胁迫下共鉴定获得239个差异表达circRNA,其中138个circRNA在抗旱型品种周麦13(ZM13)中特异性差异表达,19个circRNA在2个品种中同时差异表达。共预测到34个靶向miRNA以及1 408个miRNA靶基因。根据这些差异表达circRNA、靶向miRNA以及miRNA靶基因在干旱胁迫后的表达模式,共筛选出5个分别以tae-miR9664-3p、tae-miR1122b-3p、tae-miR9662a-3p、tae-miR6197-5p和tae-miR1120c-5p为中心的小麦响应干旱胁迫的潜在circRNA-miRNA-mRNA调控模块。【结论】小麦circRNA具有明显的品种特异性,且在不同抗旱型小麦品种之间具有不同的表达模式。共鉴定到239个响应干旱胁迫的小麦circRNA以及5个潜在的circRNA-miRNA-mRNA调控模块。
小麦;circRNA;干旱胁迫;miRNA
0 引言
【研究意义】干旱是全球粮食生产所面临的严重自然灾害之一[1],提高作物的抗旱能力是广大育种工作者的主要研究方向。小麦(L.)是世界三大粮食作物之一,挖掘其响应干旱胁迫的调控网络并分析其遗传基础,对小麦耐旱性分子设计育种和耐旱性机制解析具有重要意义。【前人研究进展】环状RNA(circular RNA,circRNA)是一类闭合的非线性RNA分子,主要由pre-mRNA通过可变剪切加工产生,大量存在于真核细胞转录组中[2]。由于高通量测序和高效生物信息学分析技术的不断突破,研究人员已经在小鼠、人类、古细菌和秀丽隐杆线虫等物种中相继鉴定出了大量的circRNA[3]。对其特征分析结果表明,circRNA可以来自外显子、内含子和基因间区域,且其表达模式通常在不同组织和发育阶段存在特异性[4]。此外,有证据表明circRNA比线性RNA要更加稳定,因此,它们可能通过一些特殊的方式参与生命活动过程[4]。近年来,circRNA在动物中研究最多的功能之一是它可以与microRNA(miRNA)竞争性结合,来调节其靶基因的功能。如,Zheng等[5]研究表明,人类中的有18个潜在的miRNA结合位点,可以与9个miRNA相结合。此外,Wang等[6]研究证实,与心脏相关的circRNA()可作为竞争性内源RNA(ceRNA)与miR-223相结合并抑制其活性。此外,在人类中的研究证实,一些circRNA可以通过特定的相互作用关系来促进其宿主基因在顺式作用中的表达[7]。与动物相比,人们对植物中circRNA鉴定的研究相对较少。2014年,Wang等[8]首次在植物中进行了circRNA鉴定,并且发现了拟南芥中存在circRNA的明确证据。之后,Ye等[9]通过全基因组水平的检测,在水稻()和拟南芥中分别鉴定出12 037和6 012个circRNA,证实circRNA在单子叶植物和双子叶植物中均广泛存在。后来,研究人员相继在番茄[10]、马铃薯[11]、小麦[12]、大豆[13]、茶[14]、大麦[15]和玉米[16]中鉴定到circRNA。尽管人们已经在植物中鉴定到大量的circRNA,但迄今为止,关于circRNA在植物当中的功能研究十分有限。Tan等[17]在番茄中过表达一个来自类胡萝卜素合成关键基因()的circRNA,可导致番茄红素合成酶基因的表达降低以及番茄红素和β-胡萝卜素的积累减少。Cheng等[18]过量表达了一个来自AT5G37720第一个内含子的circRNA,可以改变约800个基因的表达并最终影响了拟南芥的发育。此外,越来越多研究表明,circRNA在调节植物对环境胁迫的响应方面发挥着重要作用。在水稻中鉴定到27个在磷酸盐充足和饥饿条件下差异表达的外显子circRNA[9]。Pan等[19]在热胁迫条件下的拟南芥中鉴定到的circRNA数量比正常条件下要多,推测其可能通过由circRNA介导的ceRNA网络参与植物对热胁迫的反应。Wang等[12]在小麦叶片中鉴定到88个circRNA,其中66个在脱水胁迫后差异表达,并且从这些差异表达circRNA中预测到26个靶向miRNA。【本研究切入点】尽管在小麦中已进行了相关circRNA的鉴定,但鉴于circRNA在不同品种间的特异性,仍需对其进行大量的鉴定工作来完善小麦circRNA信息。此外,本研究在鉴定小麦circRNA的基础上,充分利用本研究组前期在相同小麦品种中已获得的miRNA及mRNA的转录组数据,快速构建了小麦响应干旱胁迫的circRNA-miRNA-mRNA调控模块。【拟解决的关键问题】本研究利用2个不同抗旱型小麦品种为试验材料,对小麦circRNA进行鉴定和特征分析,并进一步鉴定在干旱胁迫后差异表达的circRNA,筛选小麦干旱胁迫响应相关的潜在circRNA-miRNA-mRNA模块,为探索小麦响应干旱胁迫的调控网络及其circRNA的作用机制奠定基础。
1 材料与方法
1.1 试验材料和干旱处理方法
2个小麦(L.)品种分别为周麦13(ZM13)和冀麦38(JM38)。ZM13是一个抗旱型品种,JM38是一个干旱敏感型品种[20]。小麦种子经75%乙醇溶液消毒2 min,再用2% H2O2消毒30 min,无菌水洗涤3次[21]。室温条件下,将种子置于蒸馏水中发芽3 d,然后转移至96孔塑料水培盒(13 cm×8.5 cm×11 cm),光照培养箱(22℃光照14 h/20℃黑暗10 h)中培养[22]。待幼苗培养7 d(一叶一心期)后,对其进行干旱处理,干旱溶液为20% PEG-6000的Hoagland营养液,设置对照,3次试验重复。
1.2 总RNA提取和circRNA测序
干旱胁迫4 d时,收集2个品种CK和干旱处理的幼苗根部,每个样本分别由3株幼苗全部根系混合而成,每个品种每个处理3次重复,共计12个样本,液氮冷冻,-80℃保存。按照Yin等[10]方法进行总RNA的提取和浓度的测定。利用Ribo-Zero rRNA Removal Kit(Epicentre, Madison, WI, USA)去除总RNA中的核糖体RNA,利用RNase R(Epicentre, Madison, WI, USA)去除线性RNA,构建circRNA-seq文库[10]。委托北京百迈客生物科技有限公司利用Illumina nova-seq 6000平台对文库进行双末端测序。
1.3 circRNA的鉴定和表达分析
首先去除原始数据(raw data)中未知(N)碱基超过5%、包含接头序列以及低质量碱基(Q≤20)超过50%的低质量read。使用HISAT软件[23]将高质量的read(clean read)与小麦参考基因组(IWGSC_ RefSeq_v1.1.Triticum_aestivum.)进行序列比对。利用find_circ软件[24]进行circRNA鉴定。参照Zhu等[25]方法,将每个circRNA在样本中的表达水平进行标准化。利用DESeq2[26]软件进行circRNA的差异表达分析,差异表达circRNA的筛选标准为:|log2(Fold change)|>1且<0.05。
1.4 circRNA-miRNA靶向关系预测、miRNA靶基因预测和基因的功能注释
使用Target Finder软件[27]预测差异表达circRNA的miRNA结合位点并对miRNA的靶基因进行预测。使用KEGG(Kyoto Encyclopedia of Genes and Genomes)数据库对差异表达circRNA的宿主基因进行KEGG注释。利用小麦多组学网站WheatOmics 1.0(WheatOmics sdau.edu.cn)[28]鉴定宿主基因在水稻中的同源基因,并利用国家水稻数据中心(www.ricedata. com)查询同源基因的已知功能。使用BLAST2GO软件[29]对宿主基因进行GO注释。使用Swiss-Prot数据库对miRNA的靶基因进行功能注释。
1.5 circRNA-miRNA-mRNA调控模块预测方法
利用本研究组已鉴定到的JM38和ZM13在干旱胁迫下的miRNA数据[20](NCBI,SRA数据库,BioProject PRJNA837867)分析本研究中差异表达circRNA的靶向miRNA在干旱胁迫下的表达模式,从中筛选出差异表达的靶向miRNA。之后同样利用本研究组已鉴定到的JM38和ZM13在干旱胁迫下的mRNA数据[20](NCBI,SRA数据库,BioProject PRJNA838787)分析本研究中靶向miRNA的靶基因在干旱胁迫下的表达模式,从中筛选出差异表达的靶基因。然后根据靶基因的注释功能及同源基因的已知功能对其进行筛选。最终构建以差异表达circRNA为基础,与其存在靶向关系且差异表达的miRNA为中心,以及根据注释功能选择的差异表达靶基因为目标的circRNA-miRNA-mRNA调控模块,作为小麦响应干旱胁迫的潜在circRNA-miRNA- mRNA调控模块。
1.6 差异表达circRNA的验证
利用实时荧光定量PCR(qRT-PCR)方法对随机选择的9个差异表达circRNA的表达模式进行验证。按照Trizol试剂说明书提取总RNA,按照RevertAid First Strand cDNA Synthesis Kit(Thermo Scientific, USA)反转录试剂盒说明书进行cDNA的合成。使用SYBR Green Master Mix在CFX 96实时PCR系统(Bio-Rad)上进行qRT-PCR分析。采用2−ΔΔCt方法计算每个circRNA的相对表达量。以小麦管家基因作为内参基因[12],所用引物的信息见表1。
2 结果
2.1 小麦中circ RNA的鉴定
为鉴定小麦中的circRNA,提取这两个品种在对照及干旱胁迫4 d后的根部样本进行测序。结果显示,从12个样本中共获得约210.36 Gb的高质量数据(clean data),单个样品的clean data在14.50 Gb以上,且每个样本高质量read(clean read)的Q30均能达到93.01%以上,进一步说明该测序结果的可靠性。与小麦参考基因组的比对结果显示,各样品clean read的比对效率从98.74%到99.79%不等(表2)。

表1 荧光定量PCR引物序列
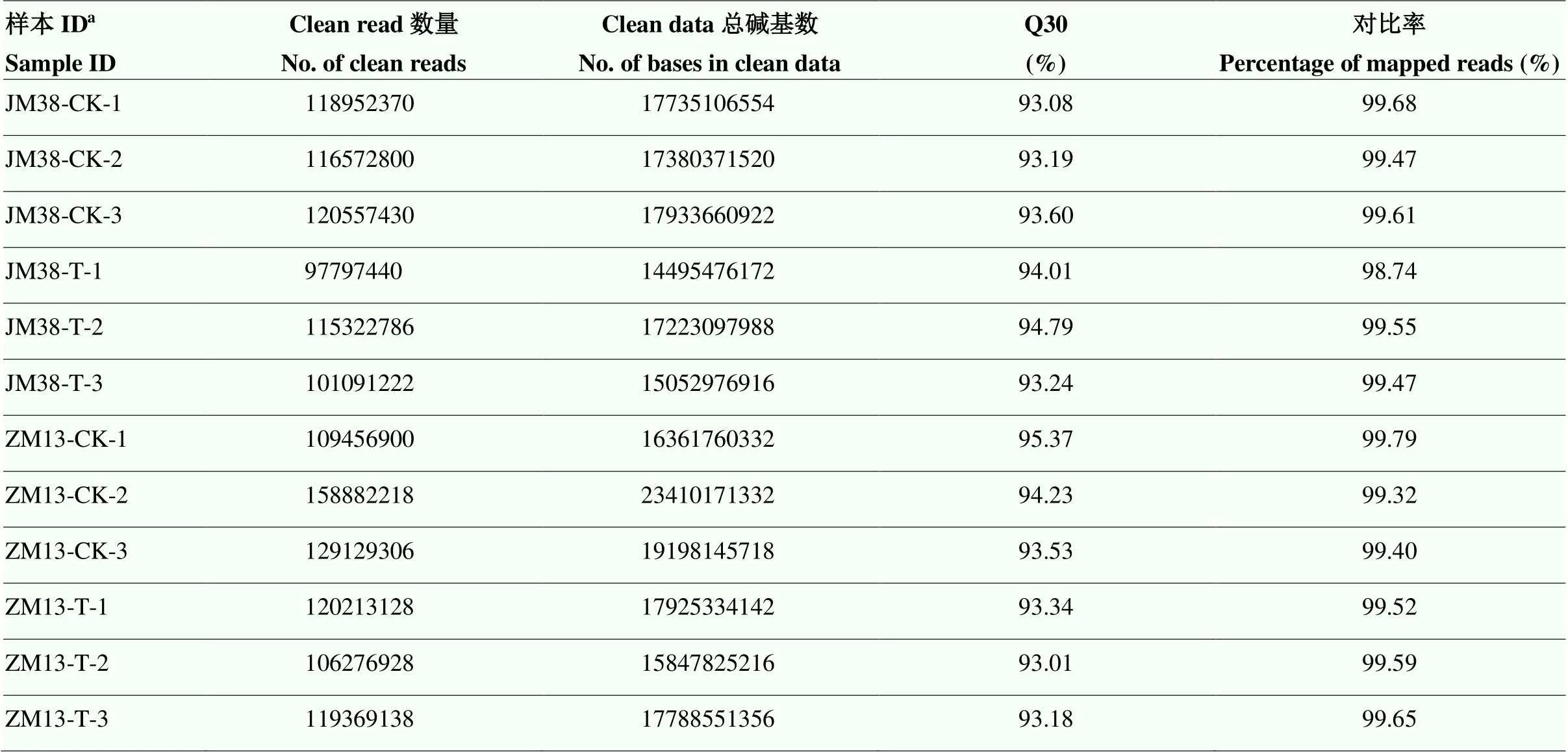
表2 circRNA测序数据结果统计
a:CK:对照条件下的样本;T:干旱条件下的样本;1—3分别代表3个不同重复
a: CK: samples under control conditions; T: samples under drought conditions; 1-3 represent three different repetitions
利用circRNA预测工具find_circ在12个测序样本中共鉴定到1 409个circRNA,其中,在干旱敏感品种JM38中鉴定到722个circRNA,在抗旱品种ZM13中鉴定到820个circRNA(图1-A)。此外,在1 409个circRNA中,有589和687个circRNA分别在JM38和ZM13中特异性表达,仅有133个circRNA在2个品种中均被鉴定到(图1-A),表明circRNA在小麦中具有较高的品种特异性。
2.2 小麦circRNA的特征分析
基因组起源分析显示,在1 409个已鉴定到的circRNA中,多数(971个,68.91%)是外显子circRNA,其次,有299个(21.22%)是基因间circRNA,其余139个(9.87%)为内含子circRNA(图1-B)。在染色体分布方面,circRNA主要分布在小麦5B、1B、2A、2B以及3B等染色体上(图1-C)。关于circRNA长度的变异情况,大多数circRNA(1 079个,76.58%)的长度低于800 bp(图1-D)。此外,大部分(78.69%)长度大于2 600 bp的长circRNA属于基因间circRNA(图1-D)。

A:2个品种中circRNA数量的韦恩图;B:由外显子、内含子和基因间区域产生的circRNA的数量和百分比;C:circRNA在小麦染色体上的分布;D:小麦circRNA的长度分布
2.3 干旱胁迫诱导的差异表达circRNA
为鉴定响应干旱胁迫的小麦circRNA,比较了2个品种的对照和干旱处理之间circRNA的表达谱。在所有鉴定到的1 409个circRNA中,有239个circRNA在干旱胁迫下存在显著差异表达,其中,在干旱敏感型品种JM38中共鉴定到91个差异表达的circRNA,在抗旱品种ZM13中共鉴定到157个差异表达的circRNA,其中138个circRNA在ZM13中特异性差异表达,19个circRNA在2个品种中同时差异表达(图2-A)。此外,值得注意的是,在JM38中更多的circRNA下调表达(55个,60.44%),而在ZM13中,有88%以上(139个,88.54%)的circRNA均为上调表达(图2-B)。说明circRNA在不同抗旱型小麦品种中存在不同的表达模式。
为验证circRNA测序结果的准确性和可靠性,利用qRT-PCR方法从239个差异表达circRNA中随机选择9个circRNA进行差异表达验证。结果显示,除了2个circRNA(chr3D:456098361|456098885和chr3B:438537516|438537814)以外,其余7个circRNA在2种试验方法中表现出相似的表达模式(图3),这可能是由于其绝对表达量较低或2种不同的表达模式计算方法所造成的结果。
2.4 差异表达circRNA宿主基因的生物学功能分析
circRNA可通过对其宿主基因的顺式调节在转录控制中发挥重要作用[7]。在2个品种中差异表达的239个circRNA中共鉴定出182个宿主基因。为了解这些差异表达circRNA的生物学功能,首先对宿主基因进行了GO注释分析。结果显示,共有114个宿主基因被注释到30个不同的GO分类上(图4-A)。分别按照生物过程、细胞成分和分子功能对这些GO类别进行分类。注释到较多宿主基因的生物过程有:代谢过程(GO:0008152)、细胞过程(GO:0009987)、单有机体过程(GO:0044699)和刺激响应(GO:0050896)。注释到较多宿主基因的细胞成分类别有:膜(GO:0016020)、细胞(GO:0005623)、膜组分(GO:0044425)、细胞器(GO:0043226)和大分子复合物(GO:0032991)。注释到较多宿主基因的分子功能类别有:结合(GO:0005488)、催化活性(GO:0003824)、核酸结合转录因子活性(GO:0001071)、转运蛋白活性(GO:0005215)和抗氧化活性(GO:0016209)(图4-A)。
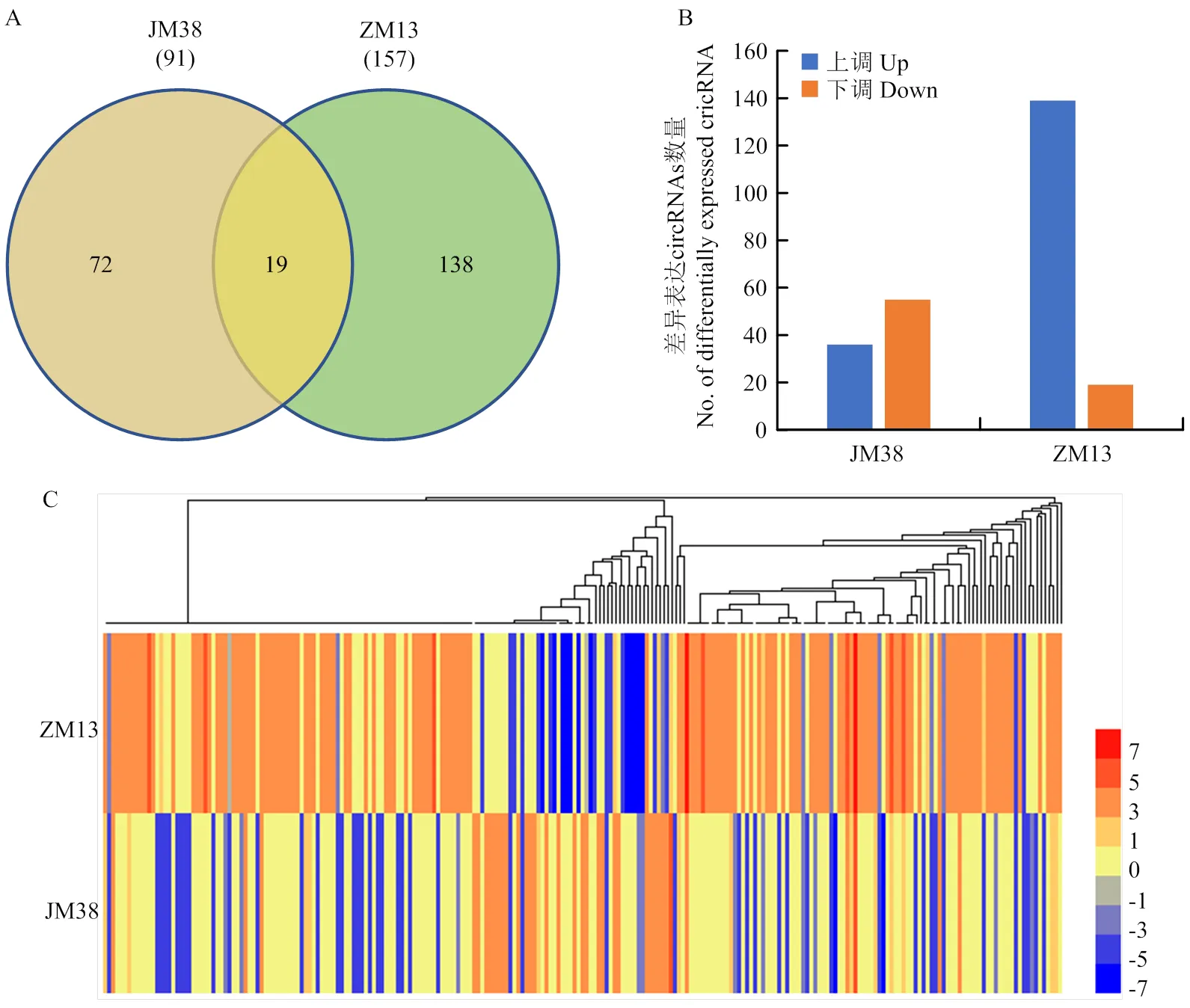
A:2个品种中差异表达circRNA数量的韦恩图;B:2个品种中上调表达circRNA和下调表达circRNA的数量;C:差异表达circRNA在2个品种中的表达热图,数字代表log2(fold change)值
其次,分析了在抗旱品种ZM13中特异性差异表达circRNA的宿主基因的GO注释结果,共鉴定出110个宿主基因,其中,共有69个宿主基因被注释到28个不同的GO类别上。和所有差异表达circRNA宿主基因的GO注释结果相似,注释到较多宿主基因的GO类别有:结合、催化活性、代谢过程、细胞过程、膜、细胞和膜组分等(图4-B)。
此外,对2个品种中共同差异表达的19个circRNA的宿主基因进行GO注释分析,共鉴定出14个宿主基因,其中有8个宿主基因被注释到20个不同的GO类别上(表3)。除了上文中提到的主要GO类别以外,注释到宿主基因数量在2个及2个以上的GO类别还有生物调节(GO:0065007)以及细胞外区域(GO:0005576)(表3)。
通过利用KEGG数据库对所有的差异表达circRNA宿主基因的功能进行进一步注释。结果显示,共有67个宿主基因被注释到36个不同的KEGG通路上。包括植物-病原菌互作(ko04626)、内吞作用(ko04144)、真核生物中的核糖体生物发生(ko03008)、植物激素信号转导(ko04075)、苯丙烷生物合成(ko00940)和过氧化物酶体(ko00190)等(表4)。通过对ZM13中特异性差异表达circRNA的宿主基因进行了KEGG注释,结果显示,共有38个宿主基因被注释到86个不同的KEGG通路上(表5)。其中,富集到较多宿主基因的通路包括:植物-病原菌互作、内吞作用和过氧化物酶体等。此外同样对2个品种中共同差异表达circRNA的宿主基因进行KEGG注释。结果显示,共有7个宿主基因被注释到7个不同的KEGG通路上(表6)。
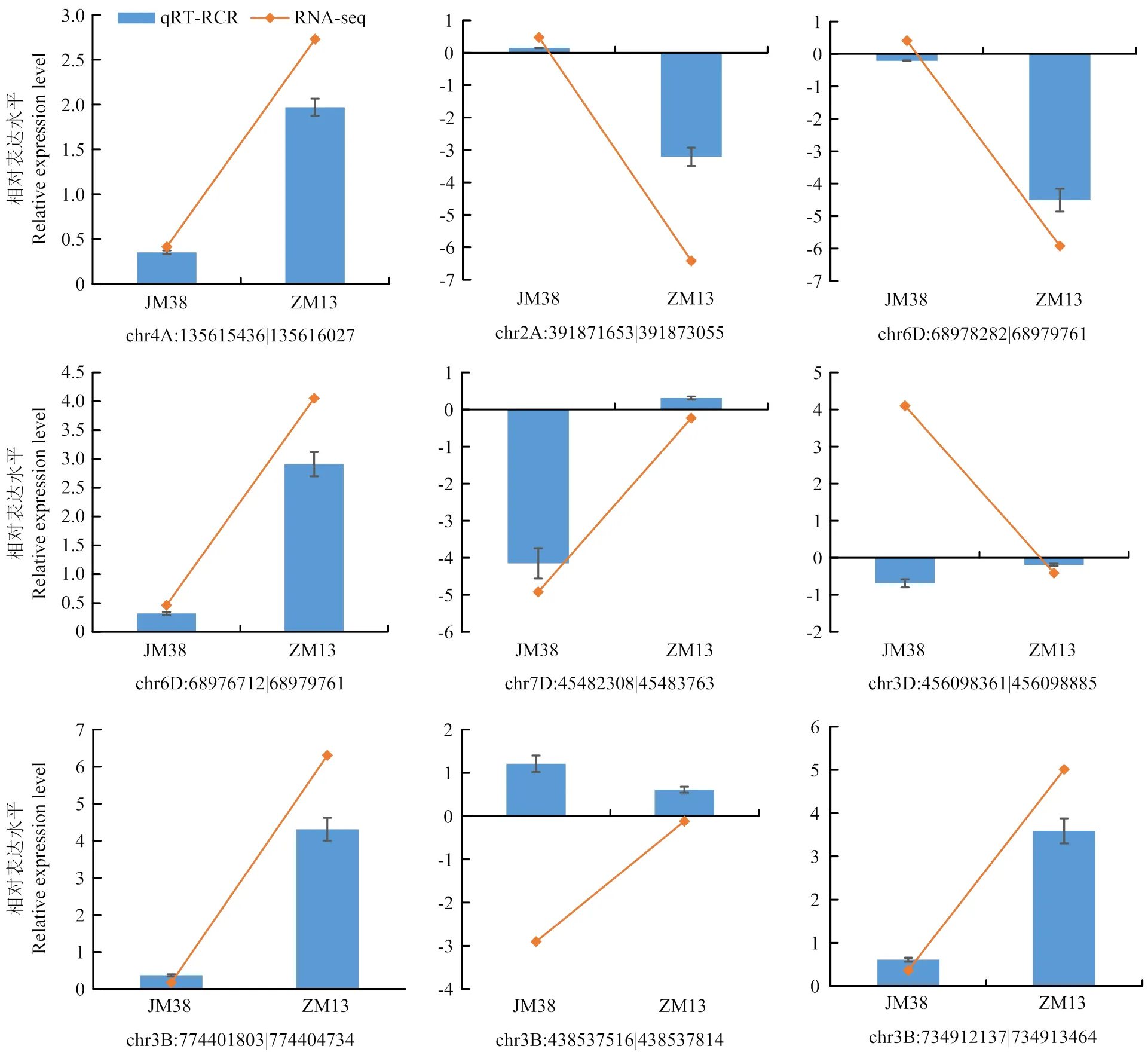
图3 差异表达circRNA的表达模式验证
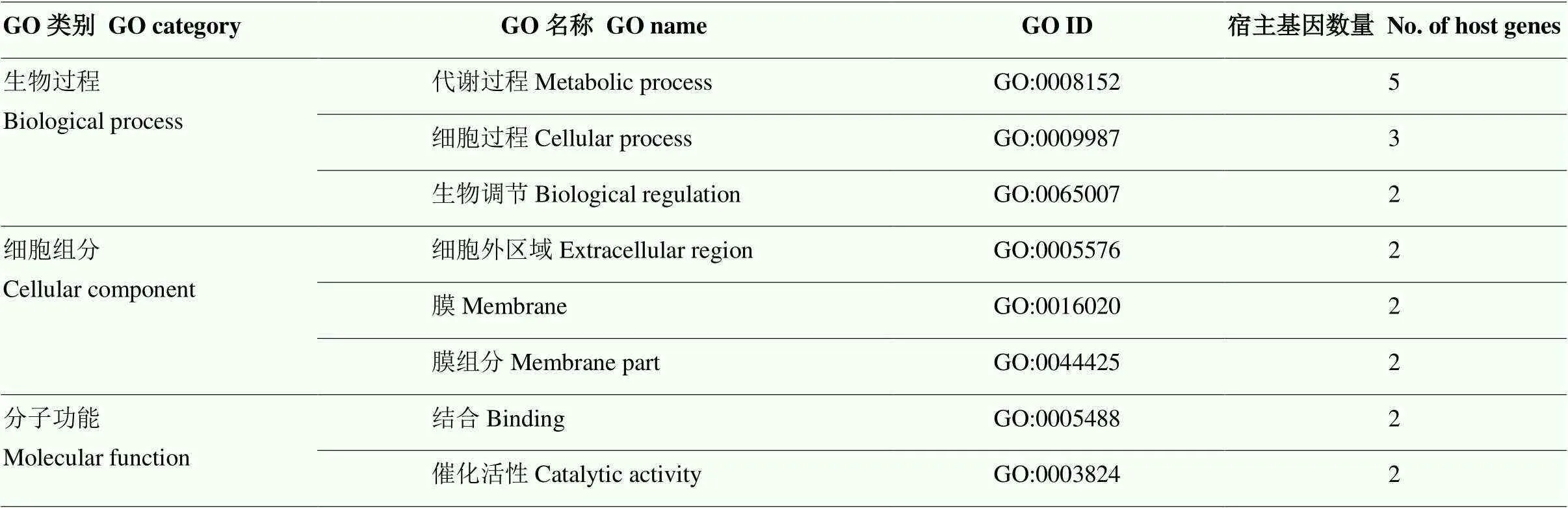
表3 2个品种中共同差异表达circRNA的宿主基因的GO注释
仅列出基因数量大于等于2的GO term Only GO terms with a No. of genes greater than or equal to 2 are listed
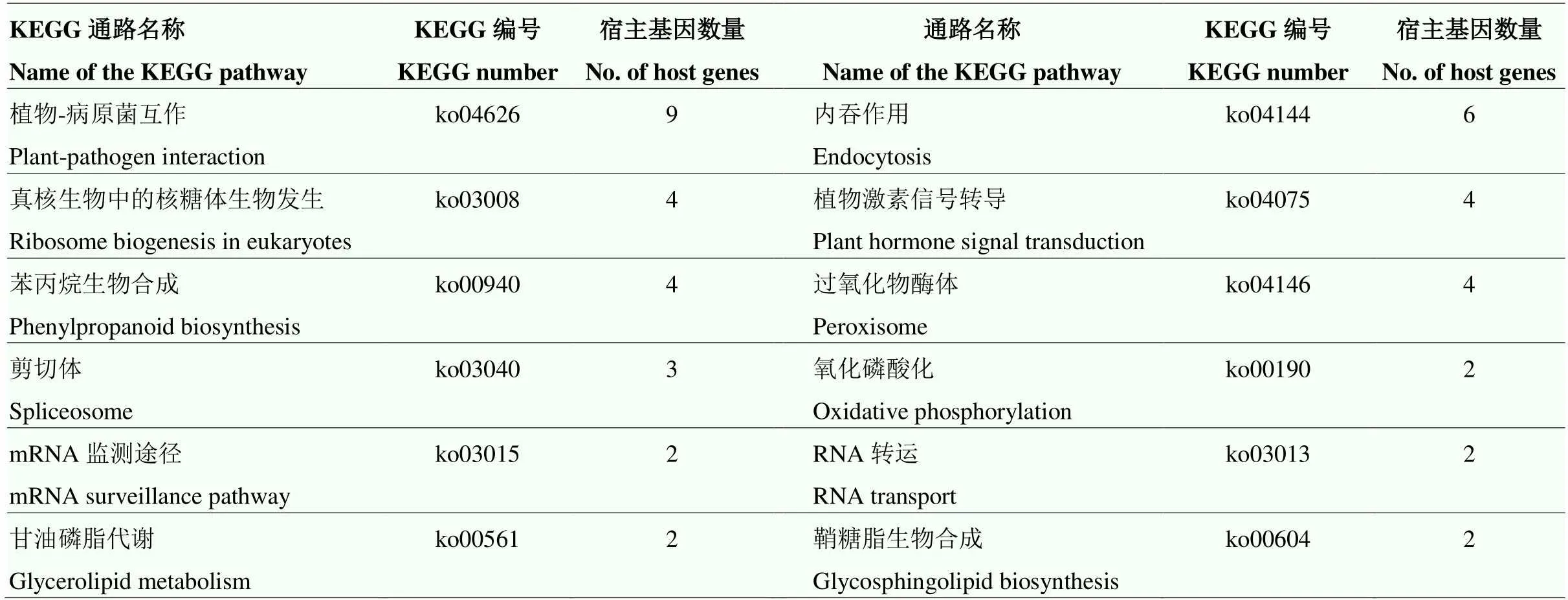
表4 全部差异表达circRNA宿主基因的KEGG注释
仅列出基因数量大于等于2的通路 Only pathways with a No. of genes greater than or equal to 2 are listed

A:两品种中所有差异表达circRNA宿主基因的GO注释结果;B:ZM13中特异性差异表达circRNA宿主基因的GO注释结果
A: GO annotation of host genes of all differentially expressed circRNAs; B: GO annotation of host genes of differentially expressed circRNAs specifically in ZM13
图4 差异表达circRNA宿主基因的GO注释
Fig. 4 GO annotation of host genes of differentially expressed circRNAs
此外,对所有宿主基因在水稻中的同源基因进行比对。通过查询这些同源基因的生物学功能发现,其中,来自9个宿主基因的10个水稻同源基因已被证实在水稻抵御多种非生物胁迫的过程中发挥着重要的作用(表7)。
2.5 差异表达circRNA的靶向miRNA预测
circRNA可以通过与miRNA竞争性结合,来阻止miRNA调节其靶基因的表达[6]。通过对circRNA的靶向miRNA进行预测,发现共有24个差异表达circRNA(其中,12个circRNA在ZM13中特异性差异表达,3个circRNA在2个品种中同时差异表达)预测到miRNA结合位点,并鉴定出34个不同的靶向miRNA(表8)。在所有差异表达的circRNA中,chr1A: 589450964|589476548具有最多的miRNA结合位点,为7个;其次是chr5B:528574134|528588868,具有6个miRNA结合位点;chr3B:778404617|778442815、chr6D: 454657953|454748495、chr7B:1308928|1352258和chr4D: 65120787|65147651均各具有5个miRNA结合位点(表8)。此外,还发现miRNA的靶向circRNA也不是唯一的,也就是说多个circRNA可能会具有同一个靶向miRNA。如tae-miR1117和tae-miR1133都具有7个靶向circRNA。在2个品种中同时差异表达的3个circRNA(chr5A:482358772|482427161、chr2B:787486469| 787538256和chr4A:668278340|668323672)共预测到5个靶向miRNA(表8)。

表5 ZM13中特异性差异表达circRNA的宿主基因的KEGG注释
仅列出基因数量大于等于2的通路 Only pathways with a No. of genes greater than or equal to 2 are listed

表6 2个品种中共同差异表达circRNA的宿主基因的KEGG注释
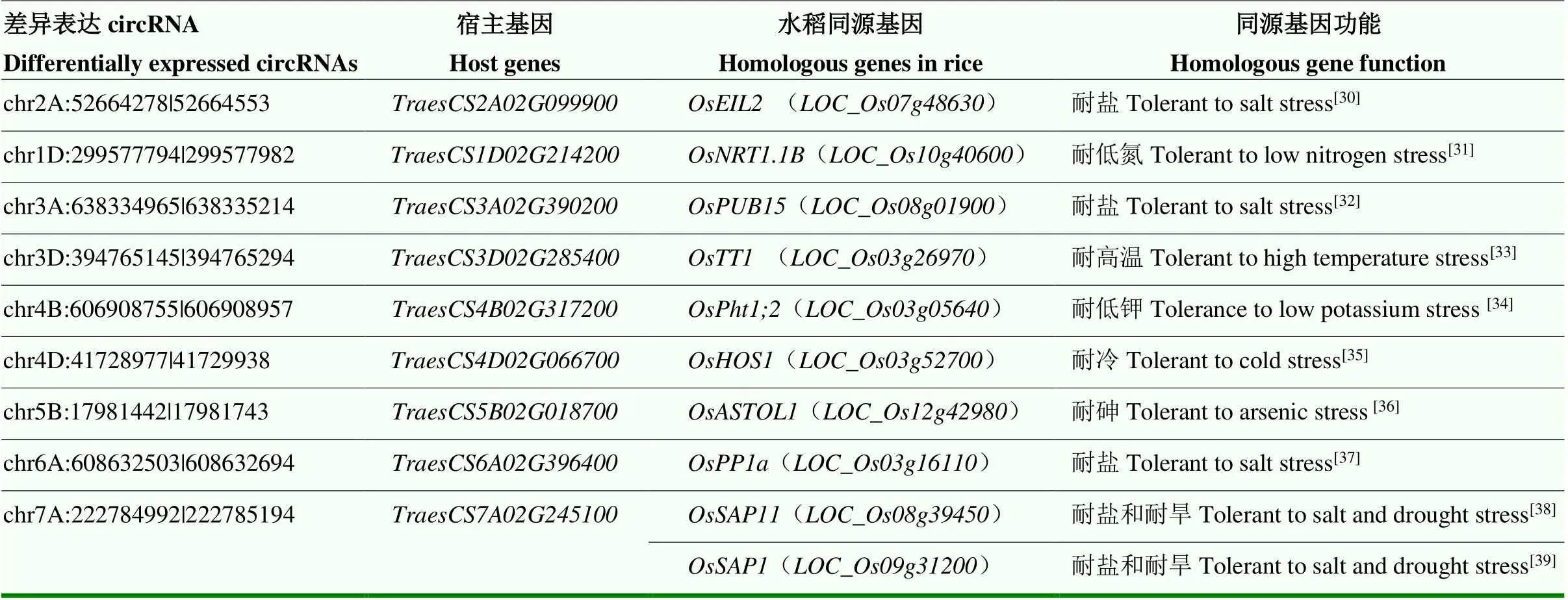
表7 差异表达circRNA宿主基因在水稻中的同源基因及其抗逆性功能

表8 差异表达circRNA及其靶向miRNA
circRNA ID斜体代表其在ZM13中特异性差异表达,circRNA ID加粗代表在2个品种中同时差异表达
CircRNA IDs in italics represent their specific differential expression in ZM13, and circRNA IDs in bold represent differentially expressed in both varieties
2.6 circRNA-miRNA-mRNA调控模块预测
为了探索小麦响应干旱胁迫的circRNA-miRNA- mRNA调控网络。首先利用本研究组已鉴定到的小麦干旱胁迫响应相关的miRNA数据分析34个靶向miRNA在干旱胁迫下的表达情况。结果表明,共有6个miRNA在干旱胁迫后差异表达,且均为上调表达(图5-A—B)。其中tae-miR9664-3p和tae-miR7757-5p在2个小麦品种均上调表达(图5-C)。然后利用Target Finder软件对这6个miRNA的靶基因进行预测,共预测到1 408个靶基因。之后,同样利用已鉴定到的小麦干旱胁迫响应相关的mRNA数据分析这1 408个靶基因干旱胁迫下的表达情况。结果表明,共有261个靶基因在干旱胁迫后差异表达(图5-D—F)。进一步查询这261个基因的注释功能,从中筛选出32个可能与小麦胁迫响应相关候选靶基因。它们的注释功能有:过氧化物酶、转录因子、细胞色素P450、钙依赖性蛋白激酶、丝氨酸/苏氨酸蛋白激酶、生长素反应蛋白和水通道蛋白等(表9)。
综上所述,最终构建了5个小麦干旱胁迫响应相关的潜在circRNA-miRNA-mRNA调控模块,其中,包括8个差异表达circRNA、5个差异表达的靶向miRNA以及32个差异表达的miRNA靶基因(表9)。其中模块3中的circRNA(chr5D:543694782| 543696753)以及其的靶向miRNA(tae-miR9662a- 3p)均在抗旱型品种ZM13中特异性差异表达(表9)。
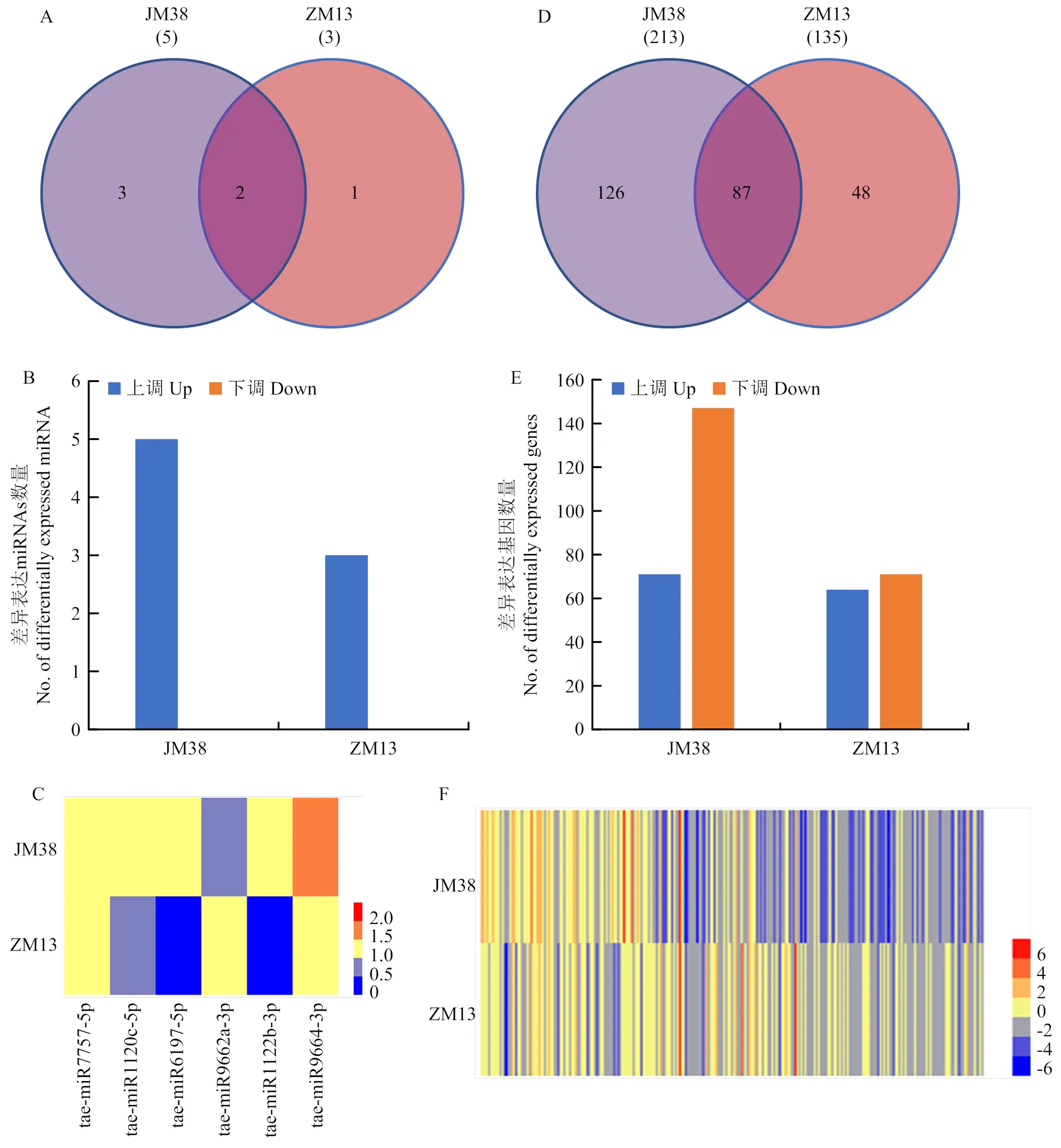
A、B:差异表达的靶向miRNA在2个品种中的分布;C:差异表达的靶向miRNA在2个品种中的表达模式;D、E:差异表达靶基因在2个品种中的分布;F:差异表达靶基因在2个品种中的表达模式
3 讨论
3.1 小麦circRNA的鉴定与特征分析
circRNA是由下游剪接供体和上游剪接受体之间的非线性反向剪接事件所产生一类特殊的RNA。近年来,随着circRNA-seq技术的发展,研究人员在动物和植物中鉴定到了大量的circRNA。这些广泛表达且高度保守的circRNA的发现增加了人们对于非编码RNA潜在功能的认识[40]。然而,与动物相比,人们对植物中circRNA的生物发生和调控功能在很大程度上都是未知的[11]。Ye等[9]对水稻和拟南芥中的circRNA进行了特征分析,并且发现植物circRNA的生物发生机制可能与动物中的有所不同。此外,最近在水稻[9]、小麦[12]和马铃薯[11]上的研究表明,circRNA在调节植株对外界胁迫的反应中发挥着重要的作用。

表9 circRNA-miRNA-mRNA调控模块
本研究分别对2个在抗旱性方面存在显著差异的小麦品种进行了circRNA测序,共鉴定到1 409个circRNA,其中,在干旱敏感品种JM38中鉴定到722个circRNA,在抗旱品种ZM13中鉴定到820个circRNA(图1-A)。通过对比2个品种中鉴定到的circRNA,发现仅有133个(9.44%)circRNA在2个品种中被同时鉴定到,说明circRNA在小麦中具有明显的品种特异性,这与前人研究的结果一致。本研究在小麦中鉴定到的circRNA数量要少于前人在水稻(26 160个)[9]、拟南芥(31 079个)[9]和大豆(5 372个)[13]中的鉴定结果,但要高于前人同样在小麦(88个)[12]以及玉米(496个)[16]、茶(342个)[14]和番茄(854个)[10]中的鉴定结果,这可能是由于不同的研究中在物种、组织(例如叶、根、果实、茎、芽等)以及circRNA预测工具(例如CIRI2、fnd_circ、CIRCexplorer)上存在的差异所导致的结果[41]。此外,前人研究结果表明,circRNA可以在外显子、内含子和基因之间的区域产生,在水稻、拟南芥、茶和番茄的研究中,大多数已鉴定的circRNA是位于外显子的circRNA[9, 14, 42]。本研究鉴定到的大多数小麦circRNA(971个,68.91%)同样是外显子circRNA(图1-B)。
3.2 小麦干旱胁迫响应相关的circRNA
植物对干旱胁迫的反应是一个非常复杂的过程,其中,涉及到许多干旱诱导基因和信号转导途径的参与。为了探索小麦circRNA在干旱胁迫响应中的作用,本研究共鉴定到239个在干旱胁迫下存在显著差异表达的circRNA。在这些差异表达的circRNA中,有超过一半(138个,57.74%)的circRNA仅来自于抗旱型品种ZM13中,还有19个circRNA同时来自于2个品种,只有72个(30.13%)circRNA在干旱敏感型品种JM38中特异性差异表达(图2-A)。这可能是由于circRNA在小麦中的品种特异性所导致的结果。此外,值得的注意的是,在JM38中更多的circRNA下调表达(55个,60.44%),而在ZM13中,有88%以上(139个,88.54%)的circRNA均为上调表达(图2-B)。这说明circRNA在不同抗旱型小麦品种中存在不同的表达模式。
本研究通过对这些差异表达circRNA的宿主基因进行了GO和KEGG注释分析。结果表明,包括刺激响应、细胞膜组分、转运蛋白活性和抗氧化活性等一些与干旱胁迫反应相关的GO term都注释到了较多的宿主基因(图4)。此外,部分宿主基因也注释到了一些重要的胁迫应答通路:植物激素信号转导、苯丙烷生物合成和过氧化物酶体等(表4)。
检索circRNA宿主基因在其他作物中的同源基因的功能,可以为预测这些差异表达circRNA的生物学功能提供一些帮助。通过查询这些差异表达circRNA的宿主基因在水稻中的同源基因及其功能发现,其中,部分水稻同源基因已被证实在抵御多种非生物胁迫的过程中发挥着重要的作用(表7)。如chr7A:222784992| 222785194的宿主基因在水稻中的同源基因和可以提高植株对干旱以及盐胁迫的耐受性[38-39]。差异表达circRNA chr2A:52664278|52664553、chr3A:638334965|638335214和chr6A:608632503|608632694宿主基因的同源基因均可提高水稻的耐盐性[30, 32, 37]。此外,其余同源基因在植株抵御高温、冷和营养胁迫等过程扮演着重要的角色[31-36](表7)。这些circRNA表达模式及功能预测结果,为挖掘小麦干旱胁迫响应相关的circRNA提供了重要依据。
3.3 小麦干旱胁迫响应相关的潜在circRNA-miRNA- mRNA调控模块
研究表明,circRNA可以通过与miRNA竞争性相合,来降低miRNA对靶基因的沉默效应,从而调节miRNA靶基因的表达[43]。为了探索与小麦干旱胁迫相关的circRNA-miRNA-mRNA调控网络。本研究构建了5个可能与小麦干旱胁迫响应相关的circRNA- miRNA-mRNA调控模块(表9)。通过分析这些差异表达靶基因注释功能,可以对于了解这些circRNA- miRNA-mRNA调控模块的潜在功能提供一定的帮助。以tae-miR9664-3p为中心的第1个circRNA-miRNA- mRNA调控模块中共筛选出10个差异表达的靶基因,其中的注释功能为过氧化物酶;的注释功能为乙烯反应转录因子;此外还包括3个细胞色素P450基因,3个钙依赖性蛋白激酶和2个ABC转运体A家族成员(表9)。众所周知,过氧化物酶可通过催化H2O2的氧化还原来调节生物体内活性氧的平衡,在植物生长发育和抵御非生物胁迫中发挥着重要作用[44]。乙烯反应转录因子与细胞发育、激素和逆境信号传递有关,在植物抗逆信号转导中具有重要的调控作用[45]。细胞色素P450同样被证实参与了植物多种生理代谢途径,其次生代谢产物在植物体信号传导以及应对生物和非生物胁迫中发挥着重要作用[46]。钙依赖性蛋白激酶是植物中重要的一类Ca2+传感器,在植物发育及多种胁迫响应中均扮演重要角色[47-48]。以tae-miR1122b-3p为中心的第2个circRNA-miRNA-mRNA调控模块中共筛选出9个差异表达的靶基因,的注释功能为丝氨酸/苏氨酸蛋白激酶,同样注释为乙烯反应转录因子;为生长素反应蛋白,是一个水通道蛋白(表9)。植物水通道蛋白是介导植物体内水分跨膜运输的主要通道,其广泛存在于植物细胞膜上,具有调节植物开花、气孔运动、生殖生长、响应多种逆境胁迫等功能[49]。此外,在水稻中的同源基因为,Cao等[50]研究证实,过量表达可以提高烟草对盐及干旱胁迫的抵抗能力。在水稻中的同源基因也被证实可以影响水稻的籽粒产量、耐盐性、根部渗透系数及种子萌发率[51]。此外,值得注意的是,以tae-miR9662a-3p为中心的模块3中的circRNA以及其靶向miRNA均只在抗旱型品种ZM13中特异性差异表达,其靶基因包括3个锌指蛋白和1个富含半胱氨酸的受体蛋白激酶(表9)。该模块对ZM13的抗旱性研究提供了一定的研究基础。
除了上述基因外,这5个模块中所涉及到的miRNA靶基因还包括1个富含半胱氨酸的受体蛋白激酶、3个转录因子、3个丝氨酸/苏氨酸蛋白激酶、2个肉桂酰辅酶A还原酶和1个普遍胁迫蛋白(表9)。这些基因均可作为小麦胁迫响应相关的候选靶基因。以上研究结果为探索小麦响应干旱胁迫的circRNA- miRNA-mRNA调控网络奠定了基础。
4 结论
共鉴定到1 409个小麦circRNA,其中多数为外显子circRNA,且表现出明显的品种特异性。共鉴定到239个受干旱胁迫诱导的差异表达circRNA,并预测到34个与之相关的靶向miRNA。根据这些差异表达circRNA、靶向miRNA以及miRNA靶基因的表达模式,共筛选出5个与小麦干旱胁迫相关的潜在circRNA-miRNA-mRNA调控模块。
[1] Akram N A, Waseem M, Ameen R, Ashraf M. Trehalose pretreatment induces drought tolerance in radish (L.) plants: some key physio-biochemical traits. Acta Physiologiae Plantarum, 2016, 38(1): 3.
[2] Jeck W R, Sharpless N E. Detecting and characterizing circular RNAs. Nature biotechnology, 2014, 32(5): 453-461.
[3] Salzman J. Circular RNA expression: its potential regulation and function. Trends in Genetics, 2016, 32(5): 309-316.
[4] Li L, Guo J, Chen Y, Chang C, Xu C. Comprehensive CircRNA expression profile and selection of key circRNAs during priming phase of rat liver regeneration. BMC Genomics, 2017, 18(1): 80.
[5] Zheng Q, Bao C, Guo W, Li S, Chen J, Chen B, Luo Y, Lyu D, Li Y, Shi G. Circular RNA profiling reveals an abundant circHIPK3 that regulates cell growth by sponging multiple miRNAs. Nature Communications, 2016, 7: 11215.
[6] Wang K, Bo L, Fang L, Wang J X, Liu C Y, Bing Z, Zhou L Y, Teng S, Man W, Tao Y.A circular RNA protects the heart from pathological hypertrophy and heart failure by targeting miR-223. European Heart Journal, 2016, 37(33): 2602-2611.
[7] Li Z, Huang C, Bao C, Chen L, Lin M, Wang X, Zhong G, Yu B, Hu W, Dai L. Exon-intron circular RNAs regulate transcription in the nucleus. Nature Structural and Molecular Biology, 2015, 22: 256.
[8] Wang P L, Bao Y, Yee M C, Barrett S P, Hogan G J, Olsen M N, Dinneny J R, Brown P O, Salzman J. Circular RNA is expressed across the eukaryotic tree of life. PLoS One, 2014, 9(3): e90859.
[9] Ye C Y, Chen L, Liu C, Zhu Q H, Fan L J.Widespread noncoding circular RNAs in plants. New Phytologist, 2015, 208(1): 88-95.
[10] Yin J L, Liu M Y, Ma D F, Wu J W, Li S L, Zhu Y X, Han B.Identification of circular RNAs and their targets during tomato fruit ripening. Postharvest Biology and Technology, 2018, 136: 90-98.
[11] Zhou R, Zhu Y X, Zhao J, Fang Z W, Wang S P, Yin J L, Chu Z H, Ma D F.Transcriptome-wide identification and characterization of potato circularRNAs in response to Pectobacterium carotovorumsubspecies Brasilienseinfection.International Journal of Molecular Sciences, 2018, 19(1): 71.
[12] Wang Y, Yang M, Wei S, Qin F, Zhao H, Suo B. Identification of circular RNAsand their targets in leaves of Triticum aestivumL. under dehydration stress.Frontiers in Plant Science, 2017, 7: 2024.
[13] Zhao W, Cheng Y, Zhang C, You Q, Shen X, Guo W, Jiao Y.Genome-wideidentification and characterization of circular RNAs by high throughputsequencing in soybean.Scientific Reports, 2017, 7(1): 5636.
[14] Wei T, Jie Y, Yan H, Li F, Zhou Q, Wei C, Bennetzen J L. Circular RNAarchitecture and differentiation during leaf bud to young leaf developmentin tea (Camellia sinensis). Planta, 2018, 248(10): 1-13.
[15] Darbani B, Noeparvar S, Borg S. Identification of circular RNAs from the parental genes involved in multiple aspects of cellular metabolism in barley. Frontiers in Plant Science, 2016, 7: 776.
[16] Chen L, Zhang P, Fan Y, Lu Q, Li Q, Yan J, Muehlbauer G J, Schnable P S, DaiM, Li L. Circular RNAs mediated by transposons are associated withtranscriptomic and phenotypic variation in maize. New Phytologist, 2018, 217(3): 3.
[17] Tan J, Zhou Z, Niu Y, Sun X, Deng Z. Identification and functional characterization of tomato circrnas derived from genes involved in fruit pigment accumulation. Scientific Reports, 2017, 7: 8594.
[18] Cheng J, Zhang Y, Li Z, Wang T, Zhang X, Zheng B.A lariat-derived circular RNA is required for plant development in. Science China Life Sciences, 2018, 61(2): 204-213.
[19] Pan T, Sun X, Liu Y, Li H, Deng G, Lin H, Wang S.Heat stress alters genome wide profiles of circular RNAs in. Plant Molecular Biology, 2018, 96(3): 217-229.
[20] LI N, LIU T T, GUO F, YANG J W, SHI Y G, WANG S G, SUN D Z. Identification of long non-coding RNA-microRNA-mRNA regulatory modules and their potential roles in drought stress response in wheat (L.). Frontiers in Plant Science, 2022, 10: 1011064.
[21] Quan X, Zeng J, Ye L, Chen G, Han Z, Shah J, Zhang G.Transcriptome profiling analysis for two Tibetan wild barley genotypes in responses to low nitrogen. BMC Plant Biology, 2016, 16(1): 30-45.
[22] Sun Y, Song K, Sun L, Qin Q, Jiang T, Jiang Q, Xue Y.Morpho-Physiological and transcriptome analysis provide insights into the effects of zinc application on nitrogen accumulation and metabolism in wheat (L.). Plant Physiology and Biochemistry, 2020, 149: 111-120.
[23] Kim D, Langmead B, Salzberg S L. HISAT: A fast spliced aligner with low memory requirements. Nature methods, 2015, 12(4): 357-360.
[24] Memczak S, Jens M, Elefsinioti A,Torti F, Krueger J, Rybak A, Maier L, Mackowiak S D, Gregersen L H, Munschauer M, Loewer A, Ziebold U, Landthaler M, Kocks C, Noble F, Rajewsky N. Circular RNAs are a large class of animal RNAs with regulatory potency. Nature, 2013, 495(7441): 333-338.
[25] Zhu Y X, Jia J H, Yang L, Xia Y C, Zhang H L, Jia J B, Zhou R, Nie P Y, Yin J L, Ma D F, Liu L C. Identification of cucumber circular RNAs responsive to salt stress. BMC Plant Biology, 2019, 19(1): 164.
[26] Love M I, Huber W, Anders S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome biology, 2014, 15(12): 550.
[27] Bo X, Wang S. Target Finder: a software for antisense oligonucleotide target site selection based on MAST and secondary structures of target mRNA. Bioinformatics, 2005, 21(8):1401.
[28] Ma S W, Wang M, Wu J H, Guo W L, Chen Y M, Li G W, Wang Y P, Shi W M, Xia G M, Fu D L, Kang Z S, Ni F. WheatOmics: A platform combining multiple omics data to accelerate functional genomics studies in wheat. Molecular Plant, 2021, 14(12): 1965-1968.
[29] Conesa A, Götz S.Blast2GO: a comprehensive suite for functional analysis in plant genomics. International Journal of Plant Genomics,2008, 2008: 619832.
[30] YANG C, LU X, MA B, CHEN S Y, ZHANG J S. Ethylene signaling in rice and: conserved and diverged aspects. Molecular Plant, 2015, 8(4): 495-505.
[31] Hu B, Jiang Z, Wang W, Qiu Y, Zhang Z, Liu Y, Li A, Gao X, Liu L, Qian Y, Huang X, Yu F, Kang S, Wang Y, Xie J, Cao S, Zhang L, Wang Y, Xie Q, Kopriva S, Chu C. Nitrate-NRT1.1B-SPX4 cascade integrates nitrogen and phosphorus signalling networks in plants. Nature Plants, 2019, 5(4): 401-413.
[32] Park J J, Yi J, Yoon J, Cho L H, Ping J, Jeong H J, Cho S K, Kim W T, An G. OsPUB15, an E3 ubiquitin ligase, functions to reduce cellular oxidative stress during seedling establishment. The Plant Journal, 2011, 65(2): 194-205.
[33] Li X M, Chao D Y, Wu Y, Huang X H, Chen K, Cui L G, Su L, Ye W W, Chen H, Chen H C, Dong N Q, Guo T, Shi M, Feng Q, Zhang P, Han B, Shan J X, Gao J P, Lin H X. Natural alleles of a proteasome α2 subunit gene contribute to thermotolerance and adaptation of African rice. Nature Genetics, 2015, 47(7): 827-833.
[34] Ai P H, Sun S B, Zhao J N, Fan X R, Xin W J, Guo Q, Yu L, Shen Q R, Wu P, Miller A J, Xu G H. Two rice phosphate transporters, OsPht1;2 and OsPht1;6, have different functions and kinetic properties in uptake and translocation. The Plant Journal, 2009, 57(5): 798-809.
[35] Lourenço T, Sapeta H, Figueiredo D D, Rodrigues M, Cordeiro A, Abreu I A, Saibo N J, Oliveira M M. Isolation and characterization of rice (L.) E3-ubiquitin ligase OsHOS1 gene in the modulation of cold stress response. Plant Molecular Biology, 2013, 83(4/5): 351-363.
[36] Sun S K, Xu X, Tang Z, Tang Z, Huang X Y, Wirtz M, Hell R, Zhao F J. A molecular switch in sulfur metabolism to reduce arsenic and enrich selenium in rice grain. Nature Communications, 2021, 12: 1392.
[37] Liao Y D, Lin K H, Chen C C, Chiang C M.protein phosphatase 1a (OsPP1a) involved in salt stress tolerance in transgenic rice. Molecular Breeding, 2016, 36: 22.
[38] Giri J, Vij S, Dansana P K, Tyagi A K. Rice A20/AN1 zinc- finger containing stress-associated proteins (SAP1/11) and a receptor- like cytoplasmic kinase (OsRLCK253) interact via A20 zinc-finger and confer abiotic stress tolerance in transgenicplants.New Phytologist, 2011, 191(3): 721-732.
[39] Mukhopadhyay A, Vij S, Tyagi A K. Overexpression of a zinc- finger protein gene from rice confers tolerance to cold, dehydration, and salt stress in transgenic tobacco. Proceedings of the National Academy of Sciences of the USA, 2004, 101(16): 6309-6314.
[40] Errichelli L, Dini M S, Laneve P, Colantoni A, Legnini I, Capauto D,Rosa A, De Santis R, Scarfo R, Peruzzi G. FUS affects circular RNAexpression in murine embryonic stem cell-derived motor neurons. Nature Communications, 2017, 8: 14741.
[41] Yin J L, Ma D F, Liu L C, Xia Y C, Zhu Y X.Biology features of circular RNAs and their research progress in plants. Acta Botanica Boreali-Occidentalia Sinica, 2017, 37(12): 2510-2518.
[42] Lu T, Cui L, Zhou Y, Zhu C, Fan D, Gong H, Zhao Q, Zhou C, Zhao Y, Lu D. Transcriptome-wide investigation of circular RNAs in rice. RNA, 2015, 21(12): 2076-2087
[43] Hansen T B, Jensen T I, Clausen B H, Bramsen J B, Finsen B, Damgaard C K. Natural RNA circles function as efficient microRNA sponges. Nature,2013, 495: 384-388.
[44] Meloni D, Oliva M, Martinez C, Cambraia J. Photosynthesis and activity of superoxide dismutase, peroxidase and glutathione reductase in cotton under salt stress. Environmental and Experimental Botany, 2003, 49: 69-76.
[45] Gutterson N, Reuber T L.Regulation of disease resistance pathways by AP2/ERF transcription factors.Current Opinion in Plant Biology, 2004, 7(4): 465-471.
[46] NELSON D R. Plant cytochrome P450s from moss to poplar.Phytochemistry Reviews, 2006, 5(2/3): 193-204.
[47] SCHULZ P, HERDE M, ROMEIS T. Calcium-dependent protein kinases: hubs in plant stress signaling and development. Plant physiology, 2013, 163(2): 523-530.
[48] DAS R, PANDEY G. Expressional analysis and role of calcium regulated kinases in abiotic stress signaling. Current genomics, 2010, 11(1): 2-13.
[49] Li M Z, Li M F, Li D D, Wang S M, Yin H J. Overexpression of theaquaporin,, promotes plant growth and stress tolerance. International Journal of Molecular Sciences, 2021, 22(4): 2112.
[50] CaoY F, Wu Y F, ZhengZ, SongF G. Overexpression of the rice EREBP-like geneenhances disease resistance and salt tolerance in transgenic tobacco. Physiological and Molecular Plant Pathology, 2006, 67(3/5): 202-211.
[51] LiuC W, FukumotoT, MatsumotoT, GenaP, FrascariaD, KanekoT, KatsuharaM, ZhongS H, SunX L, ZhuY M, IwasakiI, DingX D, CalamitaG, KitagawaY.Aquaporinpromotes rice salt resistance and seed germination. Plant Physiology and Biochemistry, 2013, 63: 151-158.
Identification of wheat circular RNAs responsive to drought stress
LI Ning, LIU Kun, LIU TongTong, SHI YuGang, WANG ShuGuang, YANG JinWen, SUN DaiZhen
College of Agriculture, Shanxi Agricultural University, Taigu 030801, Shanxi
【Objective】Drought is one of the foremost abiotic stress limiting global wheat production. Exploring the molecular mechanism of wheat response to drought stress have great significance in wheat molecular breeding. Circular RNAs (circRNAs) have been proved to play an important role in the process of plants tolerance to environmental stresses. Therefore, identifying circRNAs involved in drought stress response will help to construct a regulatory network of wheat drought tolerance, and lay a foundation for analyzing the drought resistance mechanism in wheat. 【Method】In this study, two wheat varieties (Zhoumai13 and Jimai38) with significant differences in drought resistance were used and circRNA-seq was performed on their root samples under well-watered and drought conditions. Differentially expressed circRNAs related to drought stress response were screened based on the identified circRNAs and their microRNAs (miRNAs) targets were predicted. Further, potential circRNA-miRNA-mRNA regulatory modules related to wheat drought stress response were constructed according to the expression patterns of miRNAs and their target genes under drought stress. 【Result】A total of 1 409 wheat circRNAs were identified, most of which (68.91%) were exonic circRNAs. Only 133 circRNAs were simultaneously identified in both varieties. A total of 239 differentially expressed circRNAs were identified under drought stress, of which 138 circRNAs were specifically differentially expressed in the drought-resistant variety Zhoumai 13 (ZM13), and 19 circRNAs were differentially expressed simultaneously in both varieties. Besides, 34 targeted miRNAs and 1 408 miRNA target genes were predicted. Based on the expression patterns of these differentially expressed circRNAs, targeted miRNAs, and miRNA target genes, five potential circRNA-miRNA-mRNA regulatory modules centered on tae-miR9664-3p, tae-miR1122b-3p, tae-miR9662a-3p, tae-miR6197-5p and tae-miR1120c-5p in response to drought stress were screened. 【Conclusion】Wheat circRNAs have obvious specificity in different cultivars and different expression patterns among different drought-tolerant wheat cultivars. A total of 239 wheat circRNAs and 5 potential circRNA-miRNA-mRNA regulatory modules in response to drought stress were identified in the present study.
wheat; circular RNAs; drought stress; microRNAs

10.3864/j.issn.0578-1752.2022.23.002
2022-07-25;
2022-09-05
山西省基础研究计划(20210302124148)、山西省高等学校科技创新项目(2021L124)、山西农业大学科技创新基金(2020BQ30)
李宁,E-mail:13159862006@163.cm。通信作者杨进文,E-mail:yang_jin_wen@126.com。通信作者孙黛珍,E-mail:sdz64@126.com
(责任编辑 李莉)

