侵染西番莲的东亚西番莲病毒全基因组序列特征及TC-RT-PCR检测技术
谢丽雪,张小艳,张立杰,郑姗,李韬
侵染西番莲的东亚西番莲病毒全基因组序列特征及TC-RT-PCR检测技术
谢丽雪,张小艳,张立杰,郑姗,李韬
福建省农业科学院果树研究所,福州 350013
【目的】东亚西番莲病毒(East Asian passiflora virus,EAPV)是西番莲(百香果)上危害严重的一种病毒。本研究对我国大陆地区东亚西番莲病毒福建分离物(EAPV-FJ)的全基因组序列进行测定,明确其基因组序列特征,并建立适用于EAPV特异性检测的TC-RT-PCR(tube capture RT-PCR)技术,旨在为该病毒的检测、监测及防治提供理论依据和技术支持。【方法】采用小RNA深度测序技术结合分段克隆方法和RACE技术,测定EAPV-FJ的全基因组序列,对获得的序列进行序列特征、系统发育关系和重组分析;通过反应条件及反应体系优化,建立用于EAPV快速检测的TC-RT-PCR技术,测定其特异性和灵敏度,并应用建立的TC-RT-PCR技术对福建省西番莲果园采集的样品进行检测,同时利用普通RT-PCR检测方法进行验证。【结果】测序获得的EAPV-FJ核苷酸序列全长为10 065 nt(不含polyA尾),含有一个长度为9 663 nt的开放阅读框,编码3 220 aa的多聚蛋白(polyprotein),经剪切后最终生成P1、HC-Pro、P3、6K1、CI、6K2、VPg、NIa、NIb和CP 10个蛋白。基因组序列一致性分析结果表明,EAPV-FJ全基因组与GenBank登录的4个EAPV代表分离物核苷酸序列一致性为80%—99%,其中与AO株系的越南分离物EAPV-GL1(GenBank登录号:MT450870)一致性最高,为99%;EAPV-FJ多聚蛋白核苷酸、氨基酸序列与GenBank登录的4个EAPV代表分离物一致性分别为79%—99%、82%—98%。基于多聚蛋白核苷酸序列的系统发育关系分析显示,EAPV分离物共分为两个类群(Group I为AO株系、Group Ⅱ为IB株系),未表现出明显的地理相关性,其中EAPV-FJ属于Group I,与已报道的越南分离物EAPV-GL1亲缘关系最近。重组分析结果表明,EAPV-FJ为非重组分离物。建立的TC-RT-PCR检测技术具有较好的特异性和灵敏度,仅能检测到感染EAPV的西番莲样品,而黄瓜花叶病毒(cucumber mosaic virus,CMV)、西番莲潜隐病毒(passiflora latent virus,PLV)、木槿潜隐皮尔斯堡病毒(hibiscus latent Fort Pierce virus,HLFPV)、芜菁花叶病毒(turnip mosaic virus,TuMV)、大豆花叶病毒(soybean mosaic virus,SMV)、夜来香花叶病毒(telosma mosaic virus,TeMV)等其他病毒及健康样品均无法检测到;灵敏度最低可以检测到稀释10倍的EAPV西番莲病叶提取液原液,与普通RT-PCR检测方法的灵敏度相当。应用建立的TC-RT-PCR技术从福建省西番莲果园采集的60份疑似病样中检出EAPV共13份,该结果与普通RT-PCR检测结果一致。【结论】首次报道了我国大陆地区EAPV全基因组序列,该分离物(EAPV-FJ)基因组结构与已报道的其他分离物一致,在系统发育关系上与越南分离物EAPV-GL1亲缘关系最近,且未检测到重组位点;建立的TC-RT-PCR检测技术具有操作简便、特异性强、灵敏度高和成本低的优点,能有效用于西番莲果园样品上EAPV的实际检测。
西番莲;东亚西番莲病毒;全基因组;TC-RT-PCR检测
0 引言
【研究意义】西番莲(),又称百香果,因其营养价值高、风味独特,深受国内外消费者欢迎。西番莲在栽培过程中易受病毒病的危害,对其产量和品质造成不同程度的影响。东亚西番莲病毒(East Asian Passiflora virus,EAPV)是近年来西番莲上发生较为严重的一种病毒,能够引起西番莲木质化病,给西番莲生产安全带来巨大的威胁[1]。为防控EAPV在我国的发生与危害,明确EAPV分离物全基因组序列及其分子特征,建立操作简便、特异、灵敏、经济的检测技术具有重要意义。【前人研究进展】EAPV是马铃薯Y病毒科()马铃薯Y病毒属()成员,最早在日本西番莲上被报道[2],现分布于日本、韩国、越南、马来西亚、乌干达以及我国的台湾和福建地区[1-3]。EAPV包括AO和IB两个株系,侵染西番莲均可引起叶片花叶,但在果实上引起的症状不同,AO株系主要引起果实畸形和木质化,IB株系引起果实斑驳[2,4]。此外,AO和IB两个株系的寄主范围存在一定的差异[2]。EAPV基因组为正义单链RNA,AO、IB株系的全基因组序列大小分别约10 046和9 982 nt。与其他马铃薯Y病毒属病毒一样,EAPV含有一个长的开放阅读框(open reading frame,ORF),编码多聚蛋白(polyprotein)。经病毒自身编码的蛋白酶切割后,多聚蛋白可以产生10种成熟蛋白[4],包括P1、HC-Pro、P3、6K1、CI、6K2、VPg、NIa、NIb和CP。Iwai等[3]首次报道了AO株系的全基因组序列;Fukumoto等[5]测定了IB株系的全基因组序列,并与AO株系进行比较,发现两个株系的基因组在5′端非编码区(untranslated region,UTR)和P1基因区域具有较高的多样性;Fukumoto等[1]采用RT-PCR测定2005—2010年日本鹿儿岛西番莲上EAPV的CP和多蛋白编码区,分析了EAPV群体遗传结构与变异,结果表明该地区仅存在AO株系,且在负选择压力作用下显示出较低的多样性;Chiaki等[4]以日本鹿儿岛两个不同地区的EAPV分离物为对象,进一步开展了重组、系统发育、选择压力、遗传分化和基因流等群体遗传学分析,发现两个地区的EAPV种群各自独立进化;Chong等[6]测定了我国台湾地区的EAPV全基因组序列,序列分析结果表明在台湾地区引起西番莲木质化病的病原应为EAPV,而非之前报道的西番莲木质化病毒(passionfruit woodiness virus,PWV);2018年,笔者[7]采用血清学ELISA、RT-PCR和序列测定的方法,从福建西番莲上检出EAPV,在我国大陆地区首次报道了EAPV的发生。【本研究切入点】目前,关于EAPV全基因组序列的报道较少,EAPV中国分离物仅台湾地区有报道,大陆地区分离物尚未见报道。此外,国内外针对EAPV分子检测技术的研究甚少,迄今未见TC-RT-PCR技术用于EAPV检测的报道。【拟解决的关键问题】在笔者前期报道EAPV在我国大陆地区发生的基础上,以EAPV福建分离物(EAPV-FJ)为研究对象,采用小RNA深度测序技术结合分段克隆、RACE技术,获得该分离物全基因组序列,并将其与GenBank上已报道的其他EAPV分离物进行比较,分析其基因组结构特征,揭示其分子变异情况,同时建立简便、高效的分子检测技术,以期为该病毒的防控提供科学依据。
1 材料与方法
试验于2018—2021年在福建省农业科学院果树研究所完成。
1.1 材料
EAPV-FJ采自福建省西番莲果园,新鲜样品迅速置于液氮中,-85℃超低温冰箱保存。
TRIzol 试剂为Invitrogen公司产品;RNA提取试剂盒为OMEGA公司产品;M-MuLV反转录酶、RNasin、DNA聚合酶、2×PCR mix为Promega公司产品;DNA Marker、Gel Purification Kit为天根生化科技(北京)有限公司产品;Tks GflexTMDNA Polymerase、RACE 5′/3′试剂盒、pMD-18T载体等为TaKaRa公司产品。
1.2 西番莲总RNA提取及小RNA深度测序
采用TRIzol 试剂提取西番莲病叶总RNA,参照试剂盒说明书进行。将病叶总RNA送上海生工生物工程技术服务有限公司,经质量检测合格后进行小RNA深度测序。文库构建及测序均由测序公司完成,测序平台为HiSeq X Ten(Illumina公司)。测序所得序列去除接头序列和低质量读长(reads),序列拼接成较长的重叠群(contig)后进行BLAST比对分析。
1.3 小RNA深度测序结果RT-PCR验证
利用根据EAPV基因组序列设计的特异性引物EAPV-F/EAPV-R(正向引物EAPV-F:5′-CTTGCATG TCCTAGACCTCG-3′,反向引物EAPV-R:5′-AACTGT GGTCGGTTTACCCAA-3′)[7]对小RNA深度测序结果进行RT-PCR验证。cDNA的合成采用20 μL反应体系:3 μL RNA,EAPV-R 1 μL、ddH2O 7 μL,70℃水浴10 min后迅速冰浴5 min,再加入5×M-MuLV反转录酶缓冲液5 μL、dNTPs(10 mmol·L-1)2 μL、RNasin(40 U·μL-1)1 μL、M-MuLV反转录酶(200 U·μl-1)1 μL;42℃ 1 h、70℃ 10 min。经优化的PCR反应体系及条件:cDNA 2 μL,EAPV-F(10 μmol·L-1)1 μL,EAPV-R(10 μmol·L-1)1 μL,2×PCR mix 12.5 µL,ddH2O 8.5 μL;94℃预变性3 min后94℃ 30 s、55℃ 45 s、72℃ 1 min,共35个循环,最后一轮循环后72℃延伸10 min。取10 μL PCR扩增产物用1.5%琼脂糖凝胶(染料为GelRed)进行电泳检测。PCR产物回收、纯化后,与pMD18-T载体连接,转化大肠杆菌DH5,然后挑选阳性克隆子进行测序。
1.4 EAPV基因组序列扩增及分析
根据小RNA测序后序列拼接结果和GenBank已报道的EAPV基因组序列,设计重叠的特异性引物对(表1),用于扩增EAPV的基因组,5′和3′末端采用cDNA末端快速扩增(RACE)。PCR反应体系和反应条件参照说明书进行。PCR产物回收、纯化后,进行克隆测序,测定的序列采用DNAMAN软件进行拼接,获得EAPV全基因组序列,并提交至GenBank数据库。EAPV基因组的核苷酸和氨基酸序列一致性分析采用NCBI网站上的BLAST程序进行在线比对。

表1 EAPV全基因组扩增引物
1.5 EAPV基因组系统发育分析
应用最大似然法(maximum likelihood,ML)基于多聚蛋白编码区的核苷酸序列重建EAPV系统发育关系。建树前,使用PhyloSuite1.21[8]软件中的MAFFT算法[9]基于密码子(codon)方式进行多重序列比对。应用ModelFinder[10]对数据集的最佳替代模型进行选择,并基于贝叶斯信息标准(Bayesian information criterion,BIC)设置模型参数,应用IQ-TREE 1.6.8[11]重建EAPV系统发育关系。系统发育树各节点的可靠性采用10 000次超快自举检验(Ultrafast bootstrap)和1 000次 SH-aLRT进行评估。以浙贝母病毒Y(fritillary virus Y,FVY)(GenBank 登录号NC_ 010954)为外群(outgroup)。用于系统发育分析的EAPV参考分离物信息如表2所示,寄主均为西番莲。
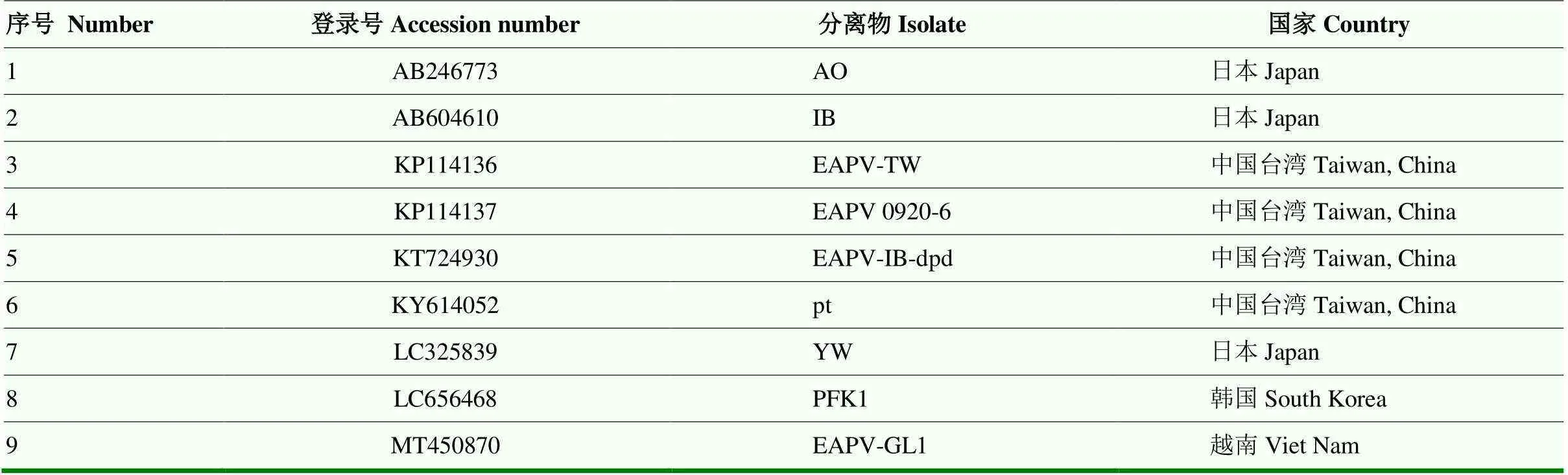
表2 用于系统发育分析的EAPV分离物信息
1.6 EAPV基因组重组分析
先应用SplitsTree 4.14.5[12]重建EAPV系统发育网络并计算成对同质性指数(pairwise homoplasy index,PHI),随后通过RDP 4.101[13]软件包中整合的7种算法进一步进行重组分析,这7种算法包括RDP、Geneconv、Bootscan、Maxchi、Chimaera、Siscan和3Seq。为避免假阳性结果,设置显著性阈值<1.0×10-6,7种方法中超过半数方法都检测到重组事件视为有效。最后,应用Simplot进行序列相似性绘图。
1.7 TC-RT-PCR检测
称取0.1 g西番莲叶片,按1﹕10(W/V)比例加入提取缓冲液(PBST,含0.05% Tween-20),研磨后4℃下10 000 r/min离心5 min,弃去沉淀。取上清100 μL包被PCR管,4℃下包被过夜,PBST洗管3次,每次3 min,ddH2O洗2次后晾干10 min。向PCR管中加入反向引物EAPV-R(10 μmol·L-1)1 μL,ddH2O 10 μL,70℃水浴10 min后立即置于冰上冷却3 min,再加入下列试剂:5×M-MuLV反转录酶缓冲液5 μL,dNTPs(10 mmol·L-1)2 μL,RNasin(40 U·μL-1)1 μL、M-MuLV反转录酶(200 U·μL-1)1 μL。经42℃ 1 h,70℃ 10 min合成cDNA。PCR反应体系和条件同1.3。分别以EPAV、CMV、PLV、HLFPV、TuMV、SMV和TeMV阳性样品为材料,以健康西番莲叶片为阴性对照,进行TC-RT-PCR,测定其特异性;将感染EPAV的西番莲病叶提取液原液进行10倍梯度稀释,然后对原液和各稀释液分别进行TC-RT-PCR,测定其灵敏度。此外,分别取100 μL原液和各稀释液,利用RNA提取试剂盒,参照说明书提取总RNA作为模板,测定普通RT-PCR的灵敏度。
1.8 西番莲样品EAPV的检测
利用建立的TC-RT-PCR检测方法对采集的福建地区西番莲疑似病样进行EAPV检测。样品共60份,各称取0.1 g后按1.7方法进行TC-RT-PCR,检测结果用普通RT-PCR进行验证。
2 结果
2.1 小RNA深度测序结果及病毒筛查
感染EAPV的西番莲病叶样品经小RNA深度测序,共得到8 481 805个原始reads,经质量控制后获得3 276 757个高质量reads,占总reads的38.6%。拼接得到的contig经BLAST比对,与EAPV具有高度一致性的contig有15条。为验证小RNA深度测序结果,利用特异性引物EAPV-F/EAPV-R进行RT-PCR检测,结果显示,从病叶样品中扩增到预期大小(约955 bp)的目的片段(图1)。PCR产物克隆测序结果进一步表明,扩增到的目的片段为EAPV基因的部分片段,其核苷酸序列与已报道的EAPV分离物一致性最高达99%。此外,小RNA深度测序还检测发现了TeMV和CMV两种病毒。
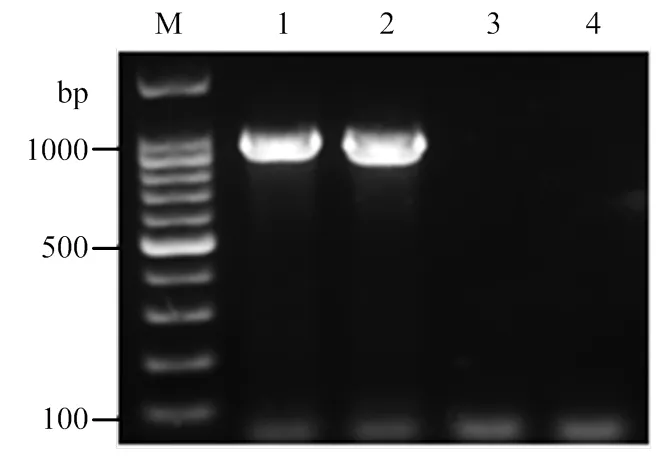
M:DNA分子量标准(100 bp)DNA Marker (100 bp);1、2:EAPV疑似病样EAPV suspected sample;3:阴性对照Negative control;4:空白对照Blank control
2.2 全基因组序列特征
经拼接获得的EAPV-FJ分离物不含poly (A)尾的全基因组长度为10 065 nt(GenBank登录号:ON641738),不同基因的位置、编码分析及与EAPV代表分离物核苷酸和氨基酸序列一致性如表3所示。该分离物的5′端和3′端的UTR长度分别为147和255 nt。148—9 810 nt为多聚蛋白编码区序列,编码一个包含3 220个氨基酸长度的多聚蛋白。EAPV-FJ分离物的9个剪切位点分别为Y/S、G/G、Q/A、Q/S、Q/S、Q/G、E/S、Q/S、Q/T,多聚蛋白被自身编码的蛋白酶切割成10个成熟蛋白,从N′-端到C′-端依次为P1、HC-Pro、P3、6K1、CI、6K2、VPg、NIa、NIb和CP。此外,在P3蛋白内还发现了由移码翻译(相对于P3基因以+2相位编码)产生的PIPO蛋白。在全基因组水平上,EAPV-FJ分离物与4个EAPV代表分离物的核苷酸序列一致性为80%—99%,其中与越南分离物EAPV-GL1的一致性最高,为99%。EAPV-FJ分离物多聚蛋白与4个EAPV代表分离物的核苷酸、氨基酸序列一致性分别为79%—99%、82%—98%。在各基因区段上,该分离物和EAVP AO株系的代表分离物核苷酸序列一致性较高(95%—99%),而与IB株系的代表分离物核苷酸序列一致性较低(70%—87%);在基因编码区,该分离物与EAVP AO株系的代表分离物氨基酸序列一致性较高(94%—100%),而与IB株系的代表分离物氨基酸序列一致性较低(56%—94%)。上述结果表明,EAPV-FJ分离物与EAVP AO株系代表分离物高度同源,亲缘关系较近。
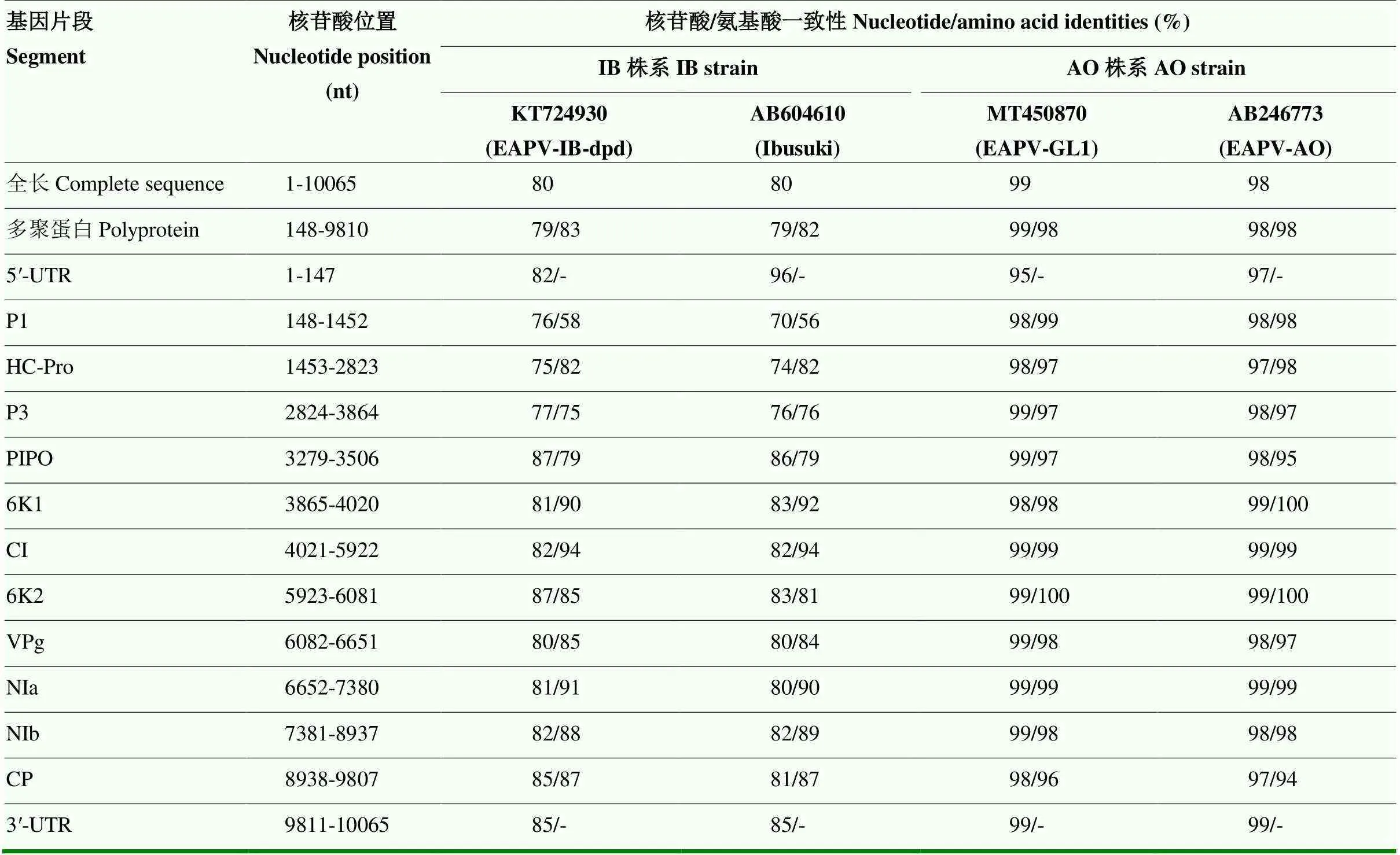
表3 EAPV-FJ与EAPV代表分离物的核苷酸和氨基酸序列一致性
2.3 系统发育分析
基于BIC标准,建树序列最适合的核苷酸替换模型为GTR+G4+F,ML法重建的系统发育树如图2所示。从图2可以看出,EAPV-FJ分离物与AO株系的5个分离物(EAPV-AO、EAPV-YW、EAPV-GL1、EAPV-TW、EAPV-0920-6)形成一个类群(Group I),其中与越南分离物EAPV-GL1单独聚为一簇,IB株系的4个分离物(EAPV-IB、EAPV-PFK1、EAPV-IB-dpd、EAPV-pt)则形成另一个类群(Group Ⅱ)。系统发育分析结果显示,EAPV-FJ分离物在系统发育关系上与越南分离物EAPV-GL1的亲缘关系最近。
2.4 重组分析
SplitsTree计算获得的PHI检验的值小于0.05,表示EAPV基因组中可能存在重组事件。进一步的RDP软件分析发现,EAPV-FJ分离物为非重组分离物,EAPV-IB-dpd分离物(GenBank登录号:KT724930)被鉴定为重组分离物,其可能的亲本为pt分离物和PFK1分离物(7种检测方法值<1.0×10-6)。通过Simplot软件绘图,如图3所示,EAPV-IB-dpd基因组的前半段与pt分离物的序列一致性非常高,而后半段则与PFK1分离物的一致性相近。
2.5 TC-RT-PCR检测的特异性
经反应体系和反应条件优化,建立了用于EAPV检测的TC-RT-PCR技术。TC-RT-PCR具有良好的特异性,仅能从感染EAPV西番莲样品上扩增出预期大小(约955 bp)的目的片段,而从CMV、PLV、HLFPV、TuMV、SMV、TeMV及健康样品上未扩增出目的片段(图4)。PCR扩增产物克隆测序结果显示,目的片段与预期大小完全相符,且与GenBank已报道的EAPV基因序列一致性最高达99%,进一步验证了TC-RT-PCR检测的准确性。
2.6 TC-RT-PCR检测的灵敏度
将感染EPAV的西番莲病叶提取液原液进行10倍梯度稀释后,分别作为模板进行TC-RT-PCR和普通RT-PCR检测。当EPAV病叶提取液原液稀释10倍时目的片段变淡,稀释102倍后无特异性目的片段,说明TC-RT-PCR最低能检测到稀释10倍的EPAV病叶提取液原液(图5-A)。同样,普通RT-PCR的灵敏度也是最低检测到稀释10倍的EPAV病叶提取液原液(图5-B)。上述结果表明,TC-RT-PCR的灵敏度与普通RT-PCR相当。
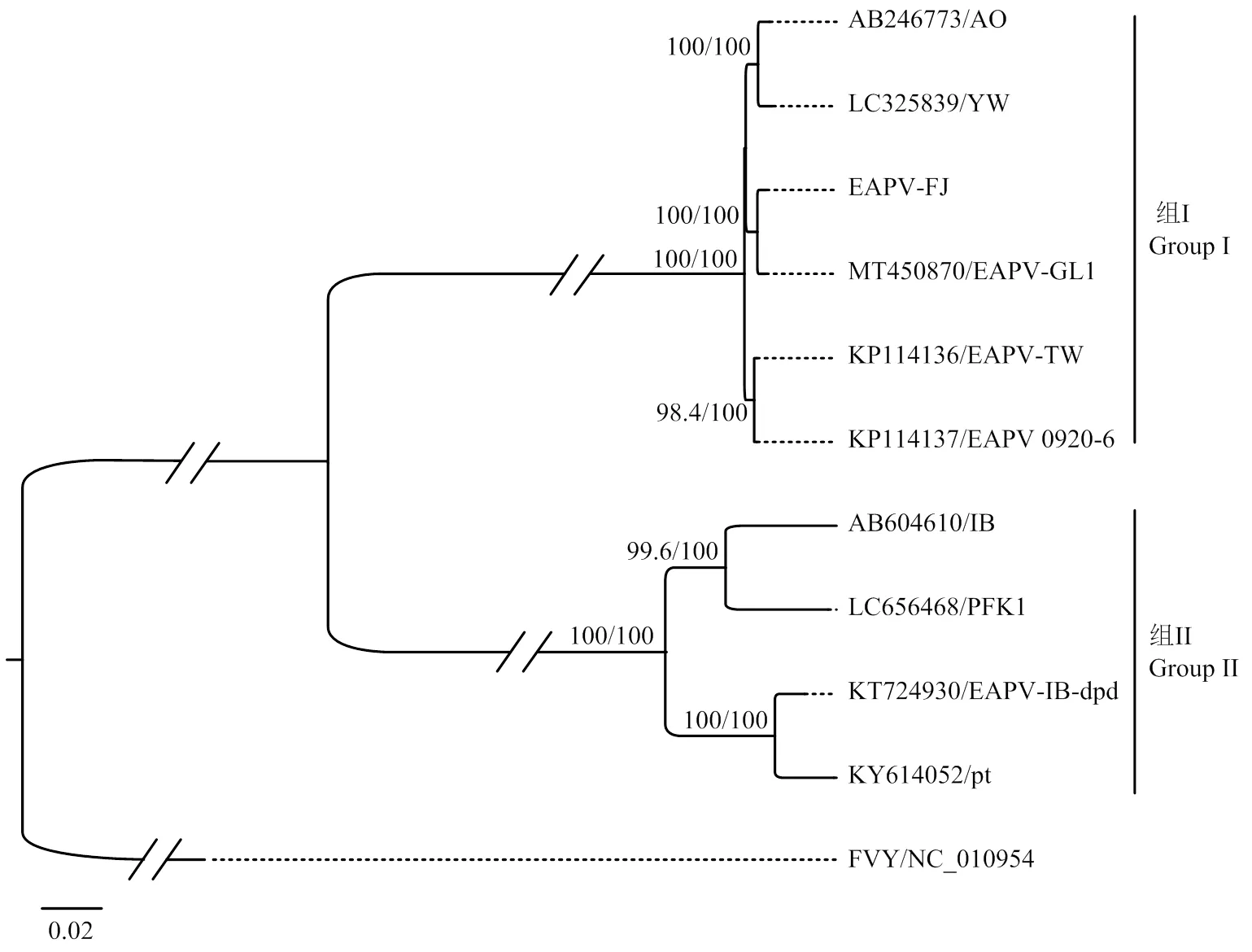
分支上显示数值为SH-aLRT支持率/自举值(>90%),标度尺为替代数/位点

图3 重组分离物EAPV-IB-dpd的Simplot序列一致性分析
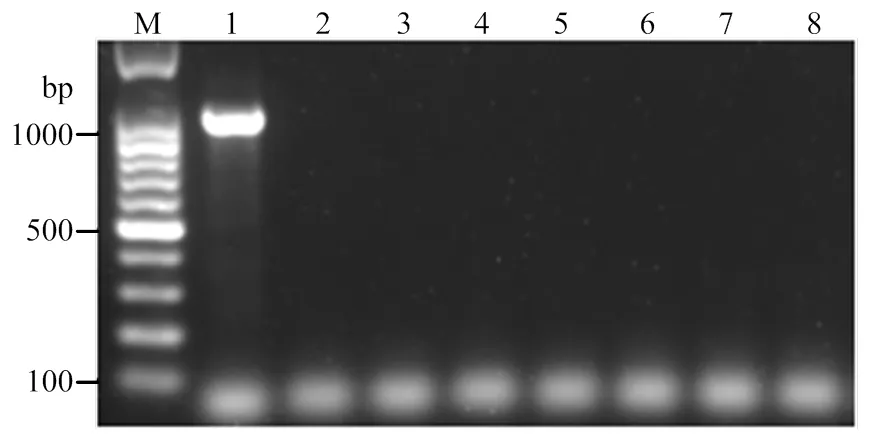
M:DNA分子量标准(100 bp)DNA Marker (100 bp);1:EAPV样品EAPV sample;2:CMV样品CMV sample;3:PLV样品PLV sample;4:HLFPV样品HLFPV sample;5:TuMV样品TuMV sample;6:SMV样品SMV sample;7:TeMV样品TeMV sample;8:阴性对照Negative control
2.7 西番莲样品TC-RT-PCR检测
利用建立的TC-RT-PCR对福建省西番莲果园采集的60份西番莲疑似病样进行检测,结果如图6所示。60份样品中共检出EPAV为阳性的有13份,检出率为21.7%,该结果与普通RT-PCR结果一致,阳性符合率为100%。
3 讨论
EAPV侵染引起的木质化病对西番莲造成极其严重的危害,至今尚无有效的防治药剂。开展EAPV全基因组序列及分子特征研究,对于明确我国EAPV的分类地位、分子变异进化特征及致病机制研究具有重要意义。目前,EAPV仅日本分离物、我国台湾地区分离物全基因组序列有报道,而我国大陆地区全基因组序列尚未报道。近年来,小RNA深度测序技术已越来越多地用于植物病毒的检测鉴定,在未知病毒、新病毒的发现上发挥了重要作用。Li等[14]利用小RNA深度测序技术,从美国和墨西哥番茄病样上检测出马铃薯纺锤形块茎类病毒(potato spindle tuber viroid,PSTVd),获得了其完整基因组序列,同时发现了凤果花叶病毒(pepino mosaic virus,PepMV)和一种新的马铃薯Y病毒属病毒;范旭东等[15]对安尼斯基葡萄病叶进行小RNA深度测序,然后通过RT-PCR扩增,成功地获得了葡萄浆果内坏死病毒(grapevine berry inner necrosis virus,GINV)变种类型l分离物基因组全长序列。本研究以福建地区采集的西番莲疑似病毒症状叶片为材料,首先利用小RNA深度测序对病毒种类进行鉴定,再根据拼接的序列设计引物进行RT-PCR扩增,结合分段克隆、RACE技术,获得了EAPV福建分离物的全基因组序列,分析了其分子特征,并建立了无需RNA提取步骤的操作简便、特异性强、灵敏度高以及成本低的TC-RT-PCR快速检测技术,为预防和控制EAPV危害打下了良好的基础。
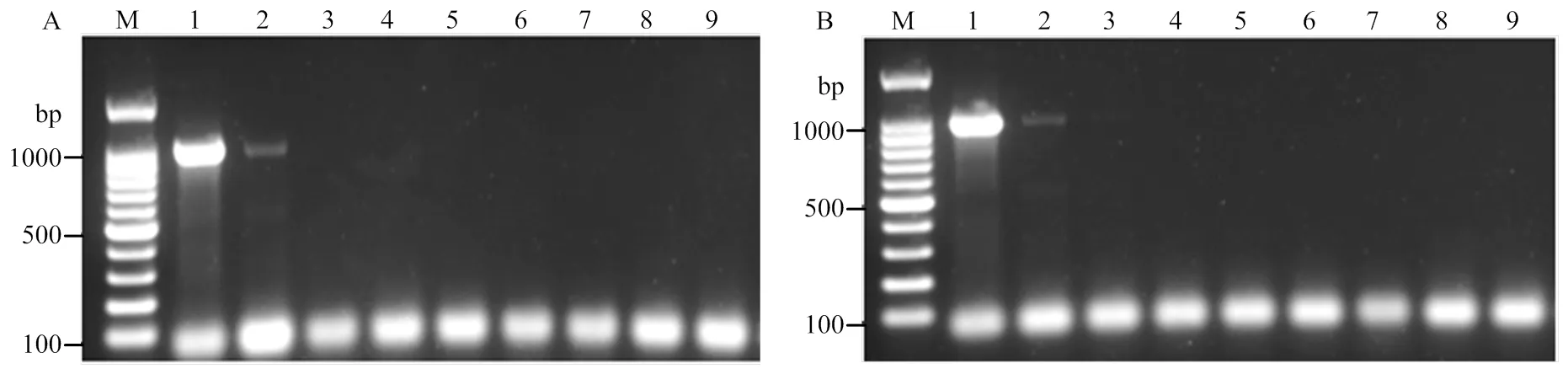
A:TC-RT-PCR;B:普通RT-PCR conventional RT-PCR。M:DNA分子量标准(100 bp)DNA Marker (100 bp);1:EAPV病叶提取液原液(100)Crude extract of EAPV-infected leaf (100);2:101倍稀释101 times dilution;3:102倍稀释102 times dilution;4:103倍稀释103 times dilution;5:104倍稀释104 times dilution;6:105倍稀释105 times dilution;7:106倍稀释106 times dilution;8:107倍稀释107 times dilution;9:阴性对照Negative control
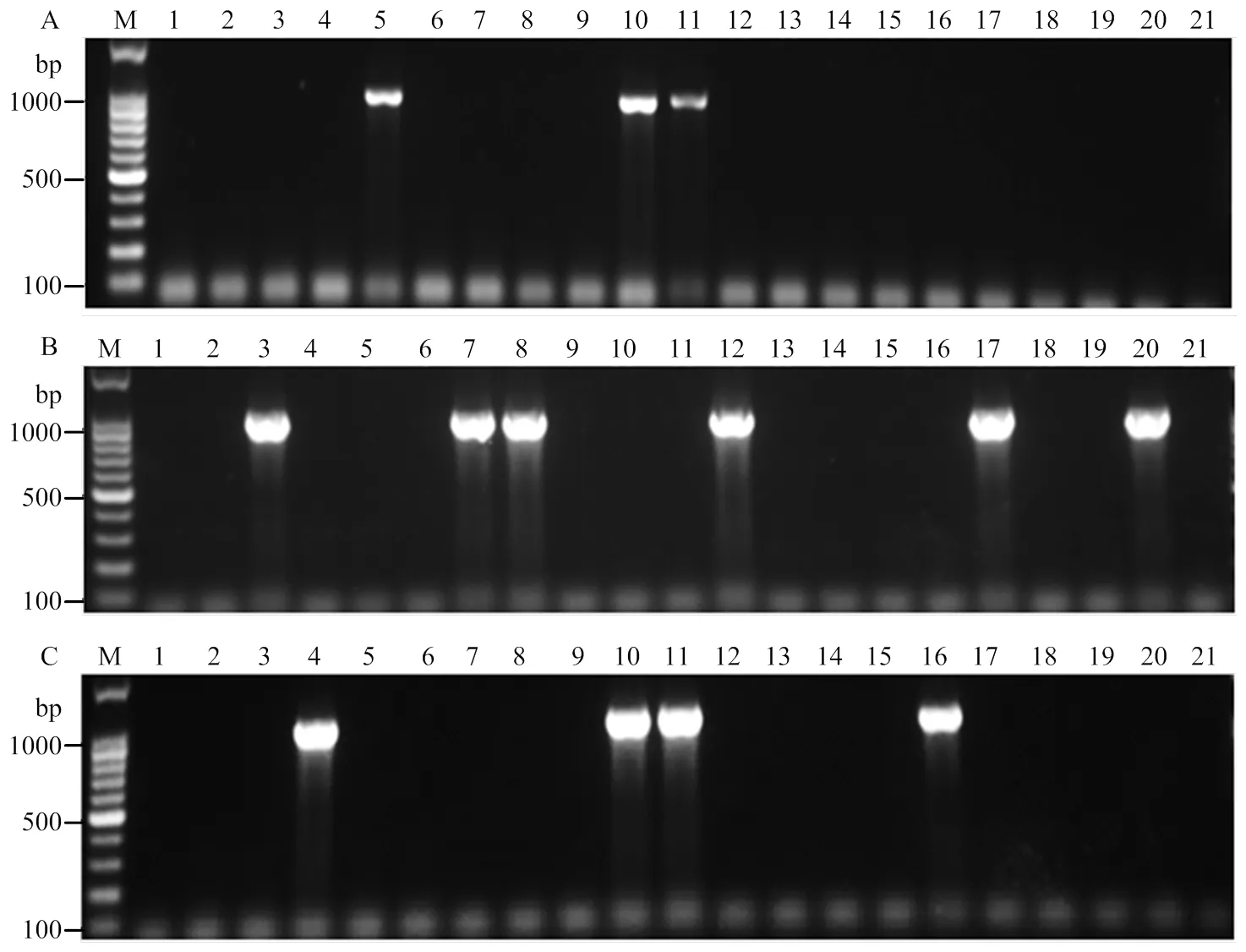
A:1—20号西番莲样品P. edulis sample 1-20;B:21—40号西番莲样品P. edulis sample 21-40;C:41—60号西番莲样品P. edulis sample 41-60。M:DNA分子量标准(100 bp)DNA Marker (100 bp);1—20:西番莲样品P. edulis sample;21:阴性对照Negative control
3.1 EAPV基因组结构
根据国际病毒分类委员会(ICTV)关于马铃薯Y病毒属的分类标准,CP基因的氨基酸序列一致性约低于80%;CP基因或全基因组的核苷酸序列一致性低于76%才可划分为不同的病毒种[16]。基因组结构分析发现,本研究获得的EAPV-FJ全基因组序列与已报道的EAPV代表分离物一致性最高达99%,CP基因的氨基酸、核苷酸序列与已报道的EAPV代表分离物一致性最高分别为98%、96%,均高于马铃薯Y病毒属不同病毒种的界定标准,证实了EAPV-FJ为EAPV的分离物。EAPV-FJ编码的所有蛋白中,P1蛋白相对变异较大,与EAPV IB株系代表分离物氨基酸序列一致性最低(56%—58%),与EAPV AO株系代表分离物氨基酸序列的一致性最高(98%—99%),而CI蛋白则较为保守,无论与EAPV AO株系还是EAVP IB株系代表分离物的氨基酸序列一致性均高于90%,分别为99%和94%。Fukumoto等[5]研究表明,EAPV不同株系间的P1基因氨基酸序列差异最大,而CI基因氨基酸序列差异最小。本研究结果与前人的报道相一致。马铃薯Y病毒属P1基因与寄主的症状表达相关,且其变异性程度与寄主范围密切相关[17-18]。EAPV AO、IB株系的寄主范围和侵染寄主引起的症状有一定的差异,但这是否与P1基因分子变异有关有待进一步研究。此外,本研究也发现,EAPV-FJ编码的P3蛋白内有一个由移码翻译产生的蛋白(PIPO),进一步验证了前人关于马铃薯Y病毒属基因组内存在的报道[19]。
3.2 EAPV系统发育分析
本研究基于全基因组序列的系统发育关系分析显示,EAPV分为AO、IB株系两个类群(Group I和Group Ⅱ),未表现出明显的地理相关性,EAPV-FJ分离物属于AO株系类群,这与笔者前期根据CP基因序列进行的系统发育分析结果一致。重组是促进病毒进化的重要因素,能帮助病毒适应新寄主或扩大寄主范围,在马铃薯Y病毒属病毒中普遍存在[20-23]。本研究针对EAPV全基因组序列进行重组分析,发现EAPV-FJ与其他分离物没有重组现象,但EAPV其他分离物基因组中存在重组信号,EAPV-IB-dpd分离物为潜在的重组分离物,其亲本可能为pt分离物和PFK1分离物。Fukumoto等[1]对日本鹿儿岛地区的EAPV进行重组分析,结果表明不管是CP还是其他蛋白编码区基因序列,均无重组现象;Chiaki等[5]进一步对日本鹿儿岛地区EAPV多聚蛋白重组分析,也未发现重组信号。日本鹿儿岛地区的EAPV分离物以及本研究获得的EAPV-FJ均属于AO株系,由此推测AO株系种群结构相对稳定,无明显遗传差异。尽管重组现象在马铃薯Y病毒属病毒中发生普遍,但本研究仅发现EAPV-IB-dpd分离物为重组分离物,这可能与已报道的EAPV分离物全基因组数量、来源地相对较少有关,因此EAPV是否还有其他重组分离物有待进一步分析确认。
3.3 EAPV的TC-RT-PCR检测
目前,分子生物学检测是包括EAPV在内的西番莲各病毒的主要检测方法,如RT-PCR、RT-LAMP等[7,24-27]。针对EAPV,普通RT-PCR检测方法首先需要提取RNA,然后再进行反转录和PCR。提取的RNA质量直接影响检测结果的准确性。TC-RT-PCR是利用PCR管壁非特异性捕捉病毒粒子,省去RNA提取步骤,将样品提取液包被PCR管后,直接在管内进行反转录和PCR。与普通RT-PCR相比,TC-RT-PCR具有操作简便、检测成本低和不使用有机溶剂的突出优点。沈建国等[28]根据已报道的菜豆荚斑驳病毒(bean pod mottle virus,BPMV)CP基因序列设计特异性引物,建立了适用于大豆种子上BPMV快速检测的TC-RT-PCR技术。本研究建立的TC-RT-PCR检测技术,能够将EAPV与CMV、PLV、HLFPV、TuMV、SMV、TeMV等其他病毒相区分,显示出良好的特异性,同时检测灵敏度与普通RT-PCR检测方法相当,说明TC-RT-PCR检测EAPV具有特异性好、灵敏度高的优点。由于TC-RT-PCR无需提取RNA,因此非常适合大批量样品的分子检测。本研究应用建立的TC-RT-PCR检测技术,对60份从福建省西番莲果园采集的样品进行了检测,验证其实际检测效果,检测结果与普通RT-PCR完全相符,这表明该检测技术可有效用于EAPV的实际检测。
4 结论
测定了EAPV-FJ的全基因组序列,这是我国大陆地区EAPV全基因组序列的首次报道。序列分析结果表明,EAPV-FJ与已报道的EAPV基因组结构一致。系统发育分析结果发现,EAPV-FJ与越南分离物EAPV-GL1亲缘关系最近。基因组重组分析结果显示,EAPV-FJ与其他分离物无重组现象。针对EAPV,建立了简便、高效的TC-RT-PCR快速检测技术。
[1] Fukumoto T, Nakamura M, Ohshima K, Iwai H. Genetic structure and variability of East Asian Passiflora virus population in Amami-O-shima, Japan. Journal of Phytopathology, 2012, 160(7/8): 404-411.
[2] Iwai H, Yamashita Y, Nishi N, Nakamura M. The potyvirus associated with the dappled fruit ofin Kagoshima prefecture, Japan is the third strain of the proposed new species East Asian Passiflora virus (EAPV) phylogenetically distinguished from strains of passion fruit woodiness virus. Archives of Virology, 2006, 151(4): 811-818.
[3] Iwai H, Terahara R, Yamashita Y, Ueda S, Nakamura M. Complete nucleotide sequence of the genomic RNA of an Amami-O-shima strain of East Asian Passiflora potyvirus. Archives of Virology, 2006, 151(7): 1457-1460.
[4] Chiaki Y, Fukumoto T, Nakamura M, Iwai H. Population genetics analysis of East Asian Passiflora virus on Amami Oshima Island. European Journal of Plant Pathology, 2016, 144(1): 109-120.
[5] Fukumoto T, Nakamura M, Rikitake M, Iwai H. Molecular characterization and specific detection of two genetically distinguishable strains of East Asian Passiflora virus (EAPV) and their distribution in southern Japan. Virus Genes, 2012, 44(1): 141-148.
[6] Chong Y H, Cheng Y H, Cheng H W, Huang Y C, Yeh S D. The virus causing passionfruit woodiness disease in Taiwan is reclassified as East Asian passiflora virus. Journal of General Plant Pathology, 2018, 84(3): 208-220.
[7] 谢丽雪, 张立杰, 张小艳, 郑姗, 李韬. 福建西番莲上首次检出东亚西番莲病毒. 园艺学报, 2018, 45(8): 1587-1594.
Xie L X, Zhang L J, Zhang X Y, Zheng S, Li T. First report of East Asian passiflora virus infectingin Fujian, China. Acta Horticulturae Sinica, 2018, 45(8): 1587-1594. (in Chinese)
[8] Zhang D, Gao F L, Jakovlić I, Zou H, Zhang J, Li W X, Wang G T. PhyloSuite: An integrated and scalable desktop platform for streamlined molecular sequence data management and evolutionary phylogenetics studies. Molecular Ecology Resources, 2020, 20(1): 348-355.
[9] Katoh K, Standley D M. MAFFT multiple sequence alignment software version 7: Improvements in performance and usability. Molecular Biology and Evolution, 2013, 30(4): 772-780.
[10] Kalyaanamoorthy S, Minh B Q, Wong T K F, von Haeseler A, Jermiin L S. ModelFinder: fast model selection for accurate phylogenetic estimates. Nature Methods, 2017, 14(6): 587-589.
[11] Nguyen L T, Schmidt H A, von Haeseler A, Minh B Q. IQ-TREE: A fast and effective stochastic algorithm for estimating maximum-likelihood phylogenies. Molecular Biology and Evolution, 2015, 32(1): 268-274.
[12] Huson D H. SplitsTree: analyzing and visualizing evolutionary data. Bioinformatics, 1998, 14(1): 68-73.
[13] Martin D P, Murrell B, Golden M, Khoosal A, Muhire B. RDP4: Detection and analysis of recombination patterns in virus genomes. Virus Evolution, 2015, 1(1): vev003.
[14] Li R G, Gao S, Hernandez A G, Wechter W P, Fei Z J, Ling K S. Deep sequencing of small RNAs in tomato for virus and viroid identification and strain differentiation. PLoS ONE, 2012, 7(5): e37127.
[15] 范旭东, 张尊平, 任芳, 胡国君, 李正男, 张双纳, 董雅凤. 葡萄浆果内坏死病毒变种类型1分离物全长基因组序列分析. 植物病理学报, 2018, 48(3): 423-427.
Fan X D, Zhang Z P, Ren F, Hu G J, Li Z N, Zhang S N, Dong Y F. Complete genome sequence analysis of grapevine berry inner necrosis virus type 1 isolate. Acta Phytopathologica Sinica, 2018, 48(3): 423-427. (in Chinese)
[16] Adams M J, Antoniw J F, Fauquet C M. Molecular criteria for genus and species discrimination within the family. Archives of Virology, 2005, 150(3): 459-479.
[17] Valli A, López-Moya J J, García J A. Recombination and gene duplication in the evolutionary diversification of P1 proteins in the family. Journal of General Virology, 2007, 88(3): 1016-1028.
[18] Shi Y H, Chen J, Hong X Y, Chen J P, Adams M J. A potyvirus P1 protein interacts with the Rieske Fe/S protein of its host. Molecular Plant Pathology, 2007, 8(6): 785-790.
[19] Chung B Y, Miller W A, Atkins J F, Firth A E. An overlapping essential gene in the. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105(15): 5897-5902.
[20] García-Arenal F, Fraile A, Malpica J M. Variation and evolution of plant virus populations. International Microbiology, 2003, 6(4): 225-232.
[21] Roossinck M J. Plant RNA virus evolution. Current Opinion in Microbiology, 2003, 6(4): 406-409.
[22] Moreno I M, Malpica J M, Dı́az-Pendón J A, Moriones E, Fraile A, Garcı́a-Arenal F. Variability and genetic structure of the population of watermelon mosaic virus infecting melon in Spain. Virology, 2004, 318(1): 451-460.
[23] 贺振, 陈春峰, 张志想, 李世访. 马铃薯Y病毒科分子进化研究进展. 植物保护, 2017, 43(3): 13-22.
He Z, Chen C F, Zhang Z X, Li S F. Advances in molecular evolution of viruses in the family. Plant Protection, 2017, 43(3): 13-22. (in Chinese)
[24] 谢丽雪, 张小艳, 郑姗, 张立杰, 李韬. 侵染西番莲的夜来香花叶病毒的分子鉴定及特异性检测. 中国农业科学, 2017, 50(24): 4725-4734.
Xie L X, Zhang X Y, Zheng S, Zhang L J, Li T. Molecular identification and specific detection of telosma mosaic virus infecting passion fruit. Scientia Agricultura Sinica, 2017, 50(24): 4725-4734. (in Chinese)
[25] Xie L X, Gao F L, Shen J G, Zhang X Y, Zheng S, Zhang L J, Li T. Molecular characterization of two recombinant isolates of telosma mosaic virus infectingfrom Fujian Province in China. PeerJ, 2020, 8: e8576.
[26] Xie L X, Gao F L, Zheng S, Zhang X Y, Zhang L J, Li T. Molecular characterization of a new potyvirus infecting passion fruit. Archives of Virology, 2019, 164(7): 1903-1906.
[27] Fu X D, Jian J M, Luo L T, Du Q L, Li X Y, Afandi A, Feng W Z, Xie X. Development of reverse transcription loop-mediated isothermal amplification assay for rapid and visual detection of telosma mosaic virus (TeMV) in passion fruit. Crop Protection, 2021, 150: 105795.
[28] 沈建国, 高芳銮, 廖富荣, 王念武, 郭琼霞, 吴祖建. TC-RT- PCR检测菜豆荚斑驳病毒的研究. 激光生物学报, 2009, 18(1): 108-111.
Shen J G, Gao F L, Liao F R, Wang N W, Guo Q X, Wu Z J. Detection of bean pod mottle virus by TC-RT-PCR. Acta Laser Biology Sinica, 2009, 18(1): 108-111. (in Chinese)
Complete Genome Sequence Characteristics and TC-RT-PCR Detection of East Asian Passiflora Virus Infecting
XIE Lixue, ZHANG Xiaoyan, ZHANG Lijie, ZHENG Shan, LI Tao
Fruit Research Institute, Fujian Academy of Agricultural Sciences, Fuzhou 350013
【Objective】East Asian Passiflora virus (EAPV) is an important virus in. The objective of this study is to determine the complete genome sequence of EAPV Fujian isolate (EAPV-FJ) in mainland China, define the genome sequence characteristics of EAPV-FJ, establish the TC-RT-PCR (tube capture RT-PCR) assay for the specific detection, and to provide theoretical basis and technical support for the detection, monitoring and control of the virus.【Method】The complete genome sequence of EAPV-FJ was determined by using small RNA deep sequencing technology, combined with segmental cloning and RACE technology. The obtained EAPV-FJ sequence was analyzed for sequence characteristics, phylogenetic relationship and recombination. The TC-RT-PCR technology for rapid detection of EAPV was established through optimization of reaction conditions and reaction system. The specificity and sensitivity of the TC-RT-PCR were determined. The established TC-RT-PCR technology was used to detect samples collected fromorchards in Fujian Province, and the conventional RT-PCR was used for verification.【Result】The full length of the obtained EAPV-FJ was 10 065 nt (excluding polyA tail), containing an open reading frame of 9 663 nt in length that encodes a polyprotein of 3 220 aa. The polyprotein was cleavaged into 10 proteins, which are P1, HC-Pro, P3, 6K1, CI, 6K2, VPg, NIa, NIb and CP, respectively. The results of genome sequence identity analysis showed that the nucleotide sequence identity between EAPV-FJ genome and the four EAPV representative isolates registered in GenBank was 80%-99%, among which Vietnamese isolate EAPV-GL1 (GenBank accession number MT450870) of AO strain had the highest identity (99%). The nucleotide and amino acid sequences of the polyprotein were 79%-99% and 82%-98% identical to the four EAPV representative isolates registered in GenBank, respectively. Phylogenetic analysis based on the nucleotide sequence of EAPV-FJ polyprotein showed that EAPV isolates were divided into two groups (Group I was AO strain and Group Ⅱ was IB strain), which did not show obvious geographical correlation. EAPV-FJ belonged to Group I and was most closely related to the reported Vietnamese isolate EAPV-GL1. The results of recombination analysis revealed that EAPV-FJ is not a recombinant of EAPV. The established TC-RT-PCR detection technology showed good specificity and sensitivity, and can only detect EAPV-infected samples of, while other viruses including cucumber mosaic virus (CMV), passiflora latent virus (PLV), hibiscus latent Fort Pierce virus (HLFPV), turnip mosaic virus (TuMV), soybean mosaic virus (SMV), telosma mosaic virus (TeMV) as well as healthy samples cannot be detected. The lowest sensitivity of the TC-RT-PCR could detect the crude leaf extract of EAPV-infected samples ofdiluted by 10times, which was similar to the sensitivity of conventional RT-PCR. A total of 13 EAPV-positive samples were detected from 60 suspected virus disease samples ofcollected fromorchards in Fujian Province by using the established TC-RT-PCR assay, and the results were in good agreement with conventional RT-PCR.【Conclusion】This is the first report of complete genome sequence of EAPV in mainland China. The genome structure of this isolate (EAPV-FJ) is consistent with other reported isolates. EAPV-FJ has the closest relative to the Vietnamese isolate EAPV-GL1 in phylogenetic relationship, and no recombination sites were detected. The established TC-RT-PCR assay has the advantages of convenient operation, strong specificity, high sensitivity and low cost, and can be effectively used for the actual detection of EAPV inorchard samples.
; East Asian Passiflora virus (EAPV); complete genome; TC-RT-PCR detection

10.3864/j.issn.0578-1752.2022.22.007
2022-06-21;
2022-07-25
国家重点研发计划(2016YFF0203200)、福建省公益类科研院所专项(2020R1028005)、福建省农业科学院引导性科技创新项目(YDXM202202)
谢丽雪,E-mail:xielx_faas@126.com。通信作者李韬,Tel:0591-87573907;E-mail:leetao06@163.com
(责任编辑 岳梅)

