乳腺癌化疗耐药微小RNA的筛选
马 旭,范静婧,马斌林
(1新疆维吾尔自治区人民医院急诊科,乌鲁木齐 830001;2新疆医科大学附属肿瘤医院乳腺头颈外科,乌鲁木齐 830011)
乳腺癌作为女性最常见的恶性肿瘤之一[1],严重威胁着女性生命健康与生活质量。化学治疗是乳腺癌临床治疗的主要手段之一,而乳腺癌细胞对化疗药物产生的多重耐药性成为乳腺癌治疗的瓶颈[2]。微小RNA(miRNA)是一种短链非编码RNA,由miRNA介导的基因表达与肿瘤发生、转移及治疗效果相关,并可促使乳腺癌化疗耐药[3]。miRNA可通过调节靶基因表达以调节化疗耐药性等诸多过程。已有研究表明,有多种miRNAs参与了乳腺癌的化疗耐药性[4]及其形成过程[5],故可作为乳腺癌耐药性的预测因子。评估miRNA在化学耐药性中的作用可通过检测靶基因的表达与相关通路的变化,并通过肿瘤细胞系、临床样本及动物实验对差异表达miRNA进行验证,可明确治疗乳腺癌的药物靶标和预后因素[6]。本研究旨在采用生物信息学方法筛选分析与乳腺癌化疗耐药相关的miRNA及其靶基因,以期寻找出与乳腺癌化疗耐药相关的靶基因,为治疗提供潜在靶标。
1 资料与方法
1.1 数据集选择以乳腺癌、化疗耐药及miRNA为关键词在基因表达数据库(gene expressed omnibus,GEO)进行非编码RNA微阵列芯片数据或高通量测序检测的非编码RNA数据的检索,并限定物种为人类,检索出相关数据集。
1.2 筛选差异表达miRNAR语言3.6.3中Limma包对基因芯片数据进行差异表达分析,获得乳腺癌患者化疗耐药组织与化疗敏感组织间差异表达的miRNAs,并进行数据筛选。以P<0.05,且差异倍数(log-FC)≥1为筛选标准。
1.3 靶基因预测通过TargetScan、MiRDB及miRWALK等预测工具预测目标miRNA靶基因,并取三者交集。在基因数据库Genecards中选择化疗耐药相关基因,并提取与上述靶基因交集数据,得出最终靶基因名单。
1.4 靶基因生存分析对TCGA数据库乳腺癌(BRCA)项目中RNAseq数据、临床数据,采用R语言3.6.3中用于可视化的survminer包,用于生存资料的统计分析survival包,对差异倍数绝对值最大的miRNAs靶基因进行生存分析。
1.5 靶基因的GO与KEGG富集分析通过DAVID数据库检索差异表达miRNA的靶基因进行GO与KEGG富集分析。KEGG作为综合型数据库,可分析信号通路及各分子之间的关系,是对关键信号通路进行分析的工具[7]。GO是一种生物信息学工具,用于注释基因并分析其分子功能(molecular function,MF)、生物过程(biological process,BP)和细胞组成(cellular component,CC)等生物学过程[8]。采用R语言3.6.3 ggplot2包进行可视化操作。
2 结果
2.1 数据集检索结果GEO数据库检索得出GSE73736[4]、GSE71142[8]数据集。其中GSE73736数据集包含10例化学耐药组织与10例化学敏感组织。GSE71142数据集包含5例化学耐药组织与5例化学敏感组织,见表1。

表1 GEO数据库检索出的mirRNA数据集
2.2 差异表达miRNAs获取微阵列结果标准化后,共获得27个差异表达miRNAs,其中下调miRNAs 14个,上调miRNAs 13个,见表2。
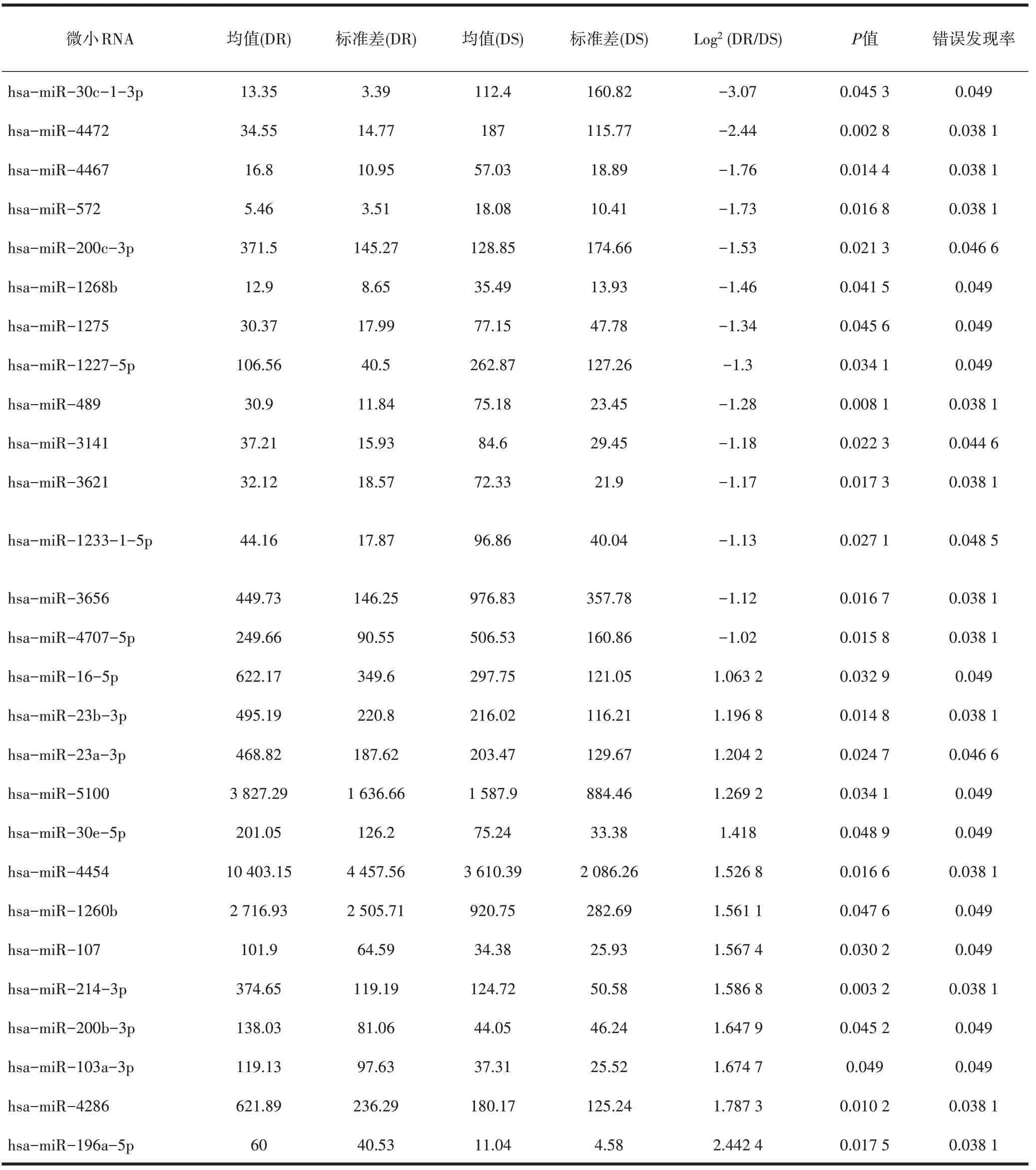
表2 获取差异表达miRNAs结果
2.3 差异表达miRNAs靶基因预测结果通过TargetScan、MiRDB、miRWALK预 测 结 果 的 交 集 与Genecards数据库重合部分结果显示,上调miRNA中差异倍数绝对值最大的是miR-30c-1-3p、miR-4472,下调miRNA中差异倍数绝对值最大的是miR-103a-3p、miR-4286、miR-196a-5p。预 测靶基因结果见表3。

表3 靶基因预测结果
2.4 靶基因生存分析结果TCGA数据库显示,差异表达miRNAs分别对应的前20个靶基因生存分析获得具有统计学意义的有糖原合酶激酶3β(GSK-3β)、肿瘤蛋白53诱导的核蛋白1(TP53INP1)两个靶基因(P<0.05),见图1。
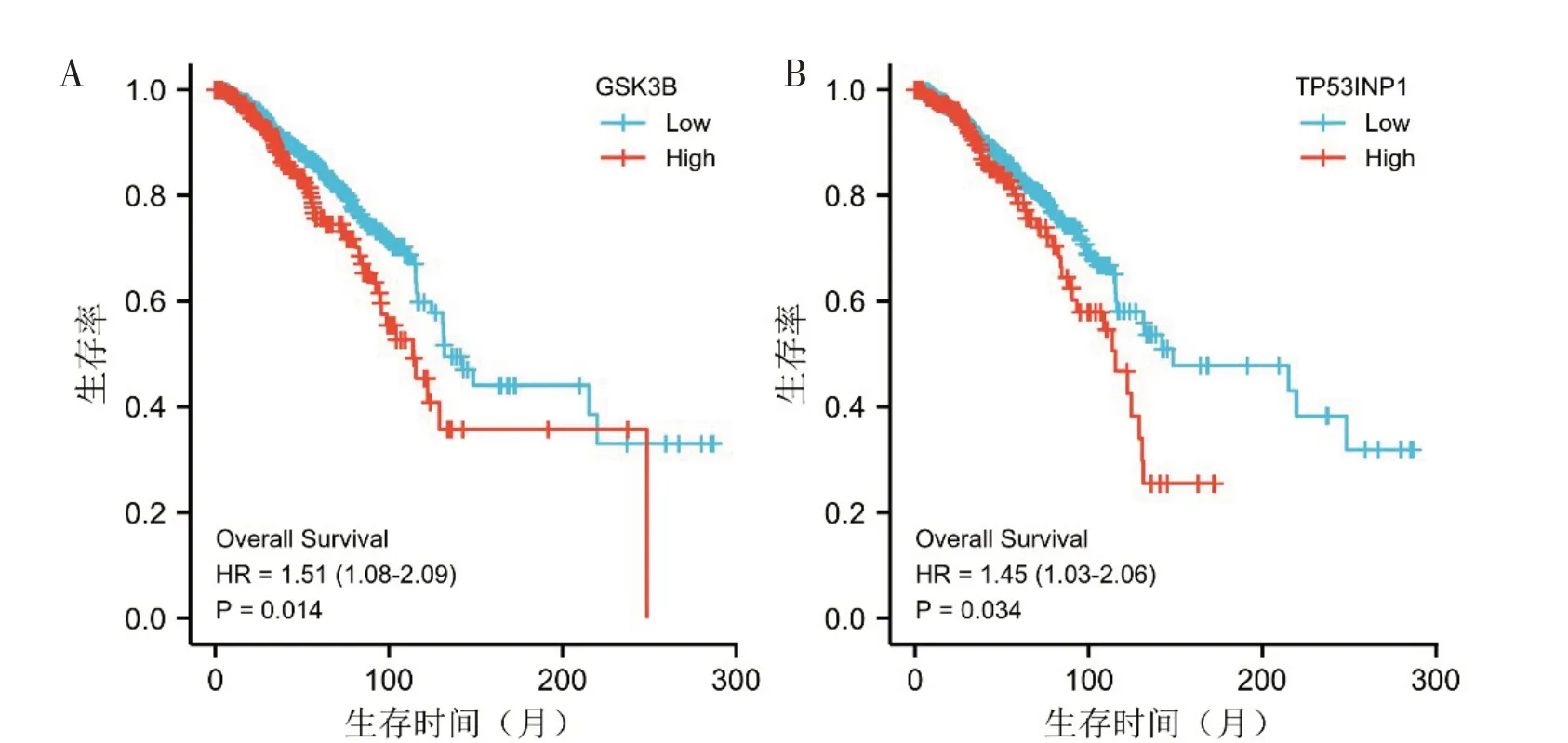
图1 靶基因生存分析结果图
2.5 靶基因GO与KEGG富集分析GO分析结果表明,靶基因生物学过程(BP)主要集中于RNA聚合酶II启动子的转录作用。分子功能(MF)主要集中于细胞核质、细胞膜、胞浆、细胞核及细胞质中。靶基因细胞成分(CC)主要集中于多聚腺苷酸RNA[poly(A)RNA]结合及蛋白质激酶的结合,见图2。KEGG通路分析显示,上调差异表达miRNAs靶基因主要富集于癌症通路,癌症的蛋白聚糖与人类嗜T细胞病毒(HTLV-I)感染。下调差异表达miRNA靶基因主要富集于PI3K-Akt信号通路及病毒导致肿瘤演进,见图3。

图2 差异表达miRNA靶基因GO分析图
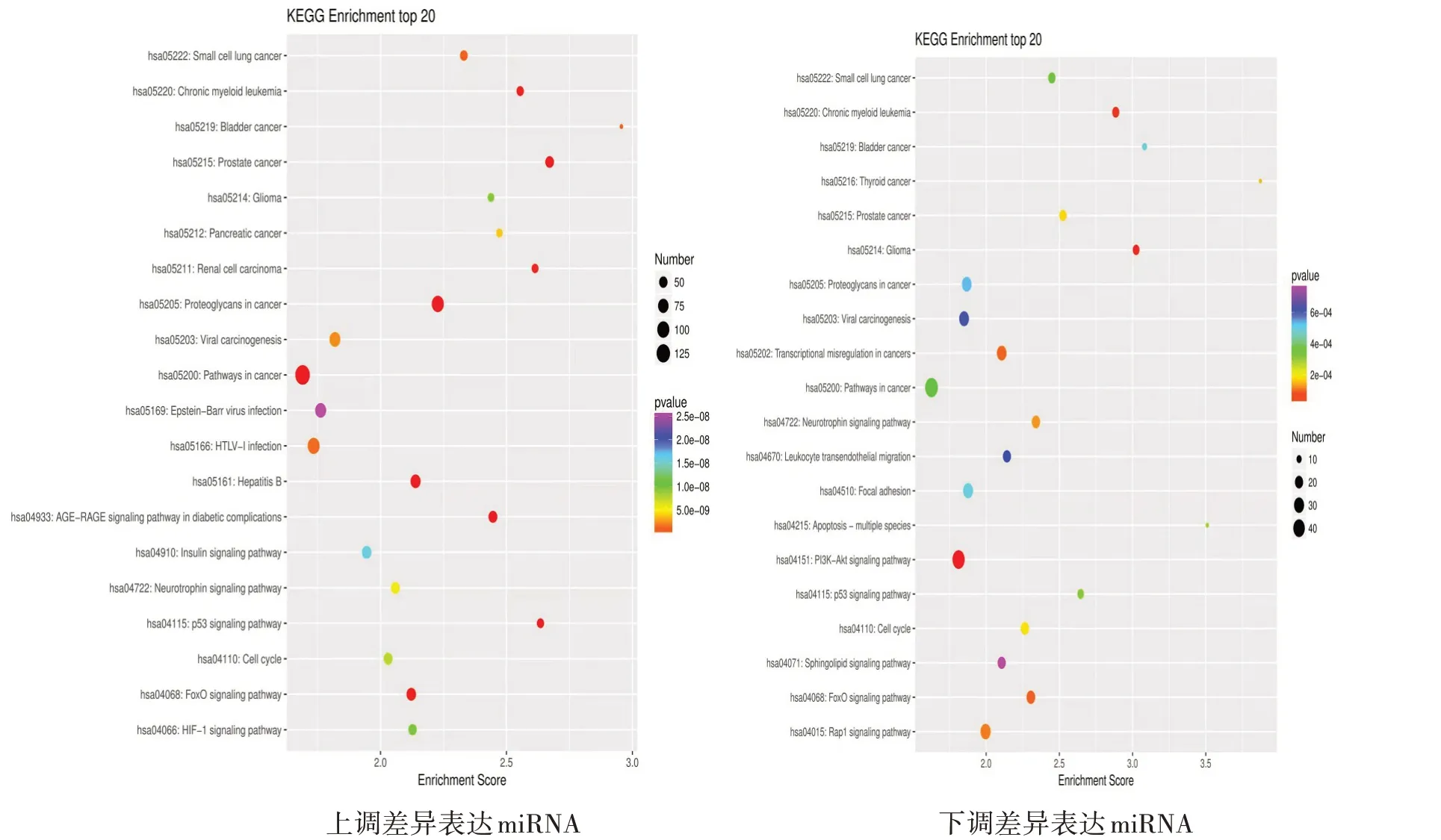
图3 差异表达miRNA靶基因KEGG分析图
3 讨论
目前,乳腺癌的主要治疗手段是以手术治疗、化学治疗、内分泌治疗及靶向治疗联合的综合治疗为主。化学治疗作为乳腺癌常用的治疗方法之一,其治疗效果很大程度上受限于化疗耐药,这种耐药会显著降低乳腺癌的无病生存时间(disease-free survival,DFS)及总生存时间(overall rurvival,OS),为解决这一问题,诸多学者对乳腺癌化疗耐药进程进行研究,发现miRNA在乳腺癌化疗耐药的患者中其表达有差异性,并将其作为化疗耐药的诊断及预后预测靶点[9-10]。有研究显示,乳腺癌中存在的低miR-145与乳腺癌恶性程度密切相关,表明miR-145可能是乳腺癌的潜在诊断及预后标志物[11]。而高水平的外泌体miRNA-21、miRNA-222、miRNA-155均与循环肿瘤细胞存在显著相关,可作为提高乳腺癌诊断和预后的临床工具[12]。MiR-27a使Luminal A型乳腺癌细胞对选择性雌激素受体调节剂的治疗敏感,发现接受内分泌治疗并表现出高miR-27a水平的ER阳性乳腺癌患者的总体存活率增加[13]。上述研究结果说明,乳腺癌患者miRNA的检测不仅可用于早期筛查、预后分析,逆转内分泌耐药、化疗耐药评估,也可作为乳腺癌的治疗靶点。
本研究对获得的27个差异表达miRNAs利用TargetScan,MiRDB及miRWALK数据库预测靶基因,并与Genecards数据库中化疗耐药的基因取交集,进行靶基因的KEGG与GO分析发现,靶基因生物学过程(BP)主要集中于RNA聚合酶II启动子的转录作用。分子功能(MF)主要集中于细胞膜、细胞浆、细胞核及细胞质中。靶基因细胞成分(CC)主要集中于多聚腺苷酸RNA[poly(A)RNA]结合及蛋白质激酶的结合。KEGG通路分析显示,上调差异表达miRNAs靶基因主要富集于癌症通路,癌症的蛋白聚糖与人类嗜T细胞病毒(HTLV-I)感染。下调差异表达miRNA靶基因主要富集于PI3K-Akt信号通路及病毒导致肿瘤演进。生存分析结果发现,GSK-3β、TP53INP1的高表达与不良预后相关,GSK-3β、TP53INP1可作为乳腺癌化疗耐药治疗的潜在靶标。抑制GSK-3β可治疗人类乳腺癌化学耐药性,并确定GSK-3抑制剂9-ING-41是转移性乳腺癌治疗的候选靶向药物[14],可调节三阴性乳腺癌的上皮-间质转化和癌症干细胞特性[15]。这与本研究结果一致。而TP53INP1作为具有肿瘤抑制功能的关键应激蛋白质,其低表达是乳腺癌患者尤其是雌激素受体ERα阳性患者预后不良的独立因素,且认为TP53INP1可能是ERα阳性潜在候选生物标志物与治疗乳腺癌患者的药物靶点[16]。这与本研究结果不一致的原因可能是该研究仅用了43例单一分型的乳腺癌。但miRNA-200a通过拮抗TP53INP1与YAP1增强乳腺癌化学耐药性[17],这说明TP53INP1可作为乳腺癌治疗的潜在靶标。
综上所述,差异表达的miRNA及靶基因可作为潜在的生物标志物,有望成为助于提高乳腺癌化疗耐药患者生存率的药物靶点。

