基于深度学习的胰腺图像自动三维重建
纪建兵,张景林,杨媛媛



摘 要: 胰腺图像的三维重建对于辅助疾病诊断具有重要意义。提出一种全自动的胰腺图像三维重建方法,利用改进的U-Net深度学习网络对图像进行分割,并结合面绘制算法生成三维可视化模型。实验结果表明,该方法重建准确度较高,执行效率快,对辅助诊疗具有积极的作用。
关键词: 胰腺; 深度学习; 面绘制; 三维重建
中图分类号:TP391.9 文献标识码:A 文章编号:1006-8228(2022)01-68-04
Automatic 3D reconstruction of pancreas image based on deep learning
Ji Jianbing1, Zhang Jinglin1, Yang Yuanyuan2
(1. College of Information Engineering, Fujian Business University, Fuzhou, Fujian 350012, China;
2. Department of General Surgery, Fujian Medical University Union Hospital)
Abstract: The 3D reconstruction of the pancreas images is of great significance for assisting disease diagnosis. A fully automatic 3D reconstruction method of the pancreas is proposed, which uses an improved U-Net deep learning network to segment the images, and creates a 3D visual model by surface rendering algorithm. The experiment results show that the proposed method has high reconstruction accuracy and fast execution efficiency. Therefore, it has a positive effect on assisting diagnosis and treatment.
Key words: pancreas; deep learning; surface rendering; 3D reconstruction
0 引言
胰腺癌的治疗主要在于对局部肿瘤做有效的切除手术,但手术存在很大的风险。手术前让病人做计算机断层扫描(Computer Tomography,CT),对扫描图像中的胰腺器官进行三维重建,可以直观地显示出其立体空间结构从而精准判定出内部的实际情况,这对于制定出有效的治疗方案以及减少因判断失误造成的意外具有重要作用[1]。胰腺结构复杂、形状多变,利用Mimics、3D Slicer等软件工具的人工重建方式存在很大的不确定性。因此,研究胰腺图像自动三维重建具有很强的现实意义,也是医学图像处理领域所关注的重要命题。
由于胰腺图像的灰度信息难以概括出某种确定的属性,通过阈值分割、边缘检测等传统的图像处理算法难以达到预期效果。近年来对胰腺图像的自动分割研究主要以深度学习技术展开[2],其原理是通过构建和训练卷积神经网络对图像中的每一个像素进行目标和背景分类,从而完成整个序列图像的分割。图像分割结果仅显示了二维层面的信息,无法全方位直观地观测到目标的细节,因此还需要对分割后的结果进行三维重建。面向医学图像的三维重建方法主要有面绘制[3]和体绘制[4],其中体绘制方法存在计算量大、难以满足实时性要求,而面绘制的方法只需要绘制出三维空间表面,因此成像速度快,更适合于胰腺图像自动三维重建的需求。
本文提出基于深度学习的胰腺图像自动三维重建工作系统。该系统利用改进的U-Net[5]深度学习网络对图像进行分割,并结合面绘制方法的移动立方体(marching cube,MC)[6]算法对分割结果进行三维重建。实验结果表明,所提方法重建速度快,准确度较高,对手术引导具有积极作用。
1 基于深度学习的胰腺图像自动三维重建
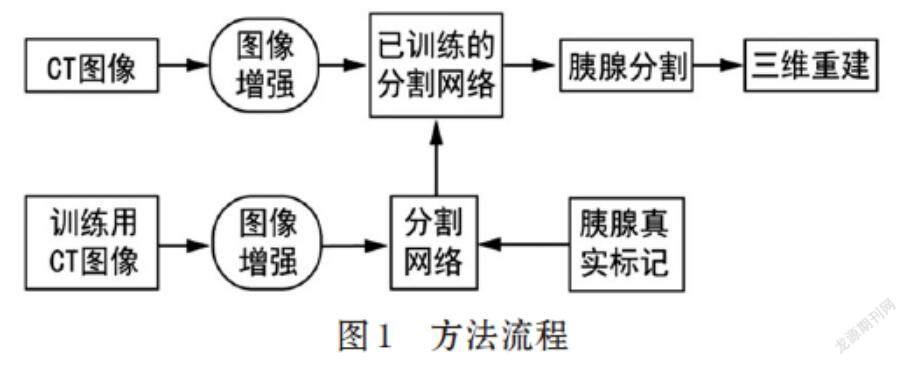
1.1 方法流程
基于深度学习的胰腺图像自动三维重建整体流程如图1所示。流程的关键节点在于图像分割网络的构建及应用。首先使用带有胰腺真实标记的CT图像数据训练深度卷积神经网络,然后通过已训练网络对CT图像中的胰腺进行分割,最后将分割结果重建为三维模型。流程中对所有的CT图像均采用统一的图像增强方法进行预处理。
1.2 图像增强
由于CT图像采集具体流程的差异,如采集设备及技术标准的不同等因素均会对成像产生全局影响,因此在将图像数据导入深度学习网络之前,需要使用图像增强的手段对原始图像做必要的统一处理,从而改善图像视觉效果,便于网络的分析和特征提取。
本文采用的图像增强方式有灰度归一化和直方图均衡化。灰度归一化算法为Max-Min归一化,其本质是将灰度值转化为百分制的形式,可以避免过小值的像素点在深度學习网络的计算中被忽略。直方图均衡化用于调整图像的灰阶分布,通过提高图像对比度来增强图像细节,使网络能更好地提取图像特征。
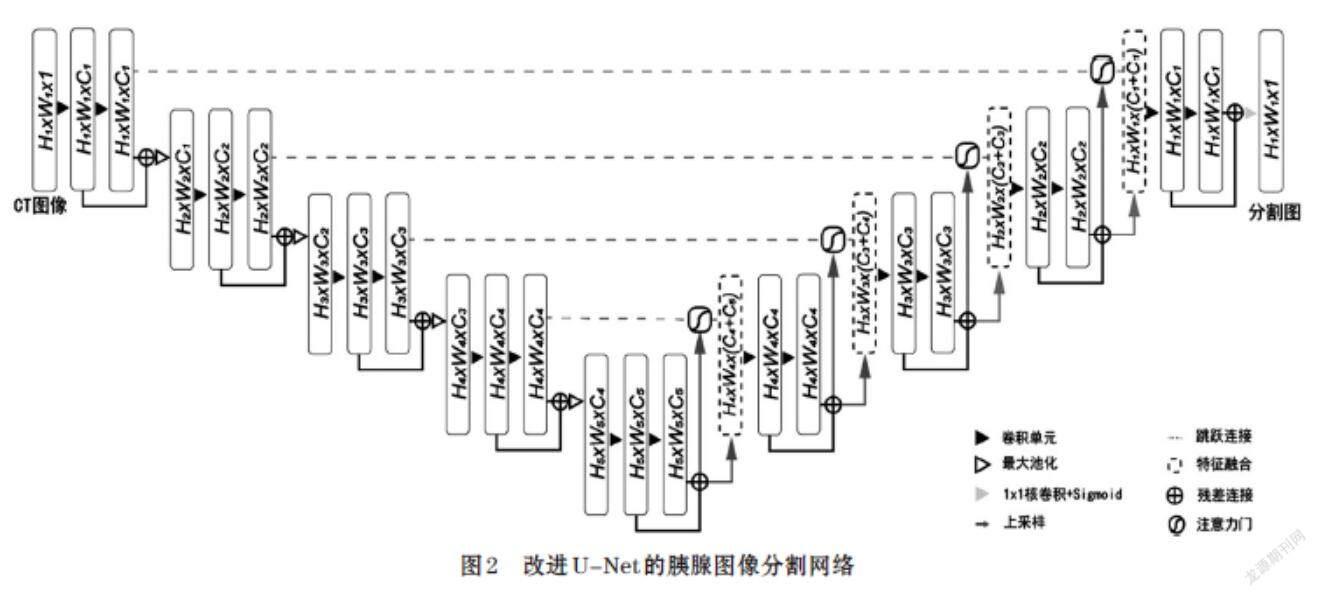
1.3 胰腺图像分割网络
医学图像分割领域使用最为广泛的深度学习网络是由Ronneberger等人提出的U-Net,该网络通过逐级的下采样和上采样,并在相同的层级上加入跳跃连接使得网络在最终输出的时候能够同时考虑浅层信息和深层信息,因此对于大部分的图像分割任务都能产生良好的效果。但由于胰腺在图像中的占比小,以及位置不固定等原因,使用U-Net对胰腺图像分割存在特征提取不充分和存在较多冗余信息,导致分割结果不够理想。
为了获得更好的胰腺图像分割效果,本文在U-Net的基础上进行了分割网络改进。首先,在卷积单元中使用了由HE等人提出的残差连接[7],通过将处理前后的特征信息相加形成残差卷积块,使得网络能够保留更多的原始胰腺特征细节,从而丰富特征提取;然后,在跳跃连接中加入了由Oktay等人提出的注意力门控模块[8]对冗余信息进行过滤,使网络能够专注于胰腺目标区域的学习;模型最后的输出由Sigmoid函数进行归一化,从而输出每一像素的概率图谱。网络整体结构如图2所示。
在网络模型训练过程中,由于胰腺区域和背景区域的像素量差异大,本文使用了Dice损失[9]进行误差计算,定义为:
[Loss=1-2×P×T+εP+T+ε] ⑴
其中P为网络预测输出的分类概率图谱,[ Pi∈0,1],T为真实标记,[T∈0,1],[ϵ]为极小常数的平滑项,用于避免当[P]和[T]都为0时导致分母为0的错误。
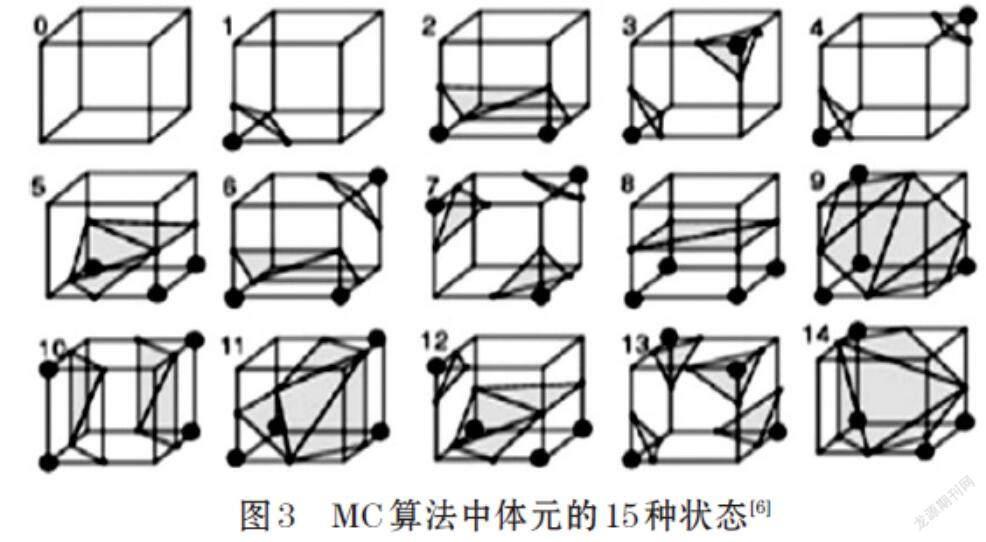
1.4 基于MC算法的胰腺图像三维重建
通过面绘制的三维重建是根据二维图像序列生成可视化三维物体,得到的物体表面是一个闭合的灰度等值面。因此,使用MC算法实现胰腺图像三维重建需要先将分割后的片层图像组成三维数据场,然后提取出等值面,进而绘制成三维可视化模型。算法的主要步骤是:
⑴ 将分割后的胰腺图像序列按深度组成一个三维体数据,并将数据中两组上下相对应的四个顶点组成一个体元;
⑵ 通过体元中8个顶点的灰度值与给定阈值的关系状态进行查表(见图3),得到等值点所在的边编号,然后在边上进行线性插值得到等值顶点的坐标;
⑶ 用三角形将等值点连成三角面并以灰度差分法求取法向量,从而得到一个体元内的等值面;
⑷ 把所有体元内的等值面连接在一起,绘制出整个胰腺的三维可视化模型。
2 實验
2.1 实验数据
实验在NIH胰腺公开数据集(CT-82)上进行,该数据集共有82例有效3D腹部CT数据,扫描的分辨率为512×512像素,片层厚度在1.5mm至2.5mm之间,对影像数据中的胰腺区域进行逐片层的人工标注。该数据集是公开的,可用于方法评估。实验中将数据集解压为二维图像,随机抽取70例用于模型训练,抽取12例用于结果评价。
2.2 评价指标
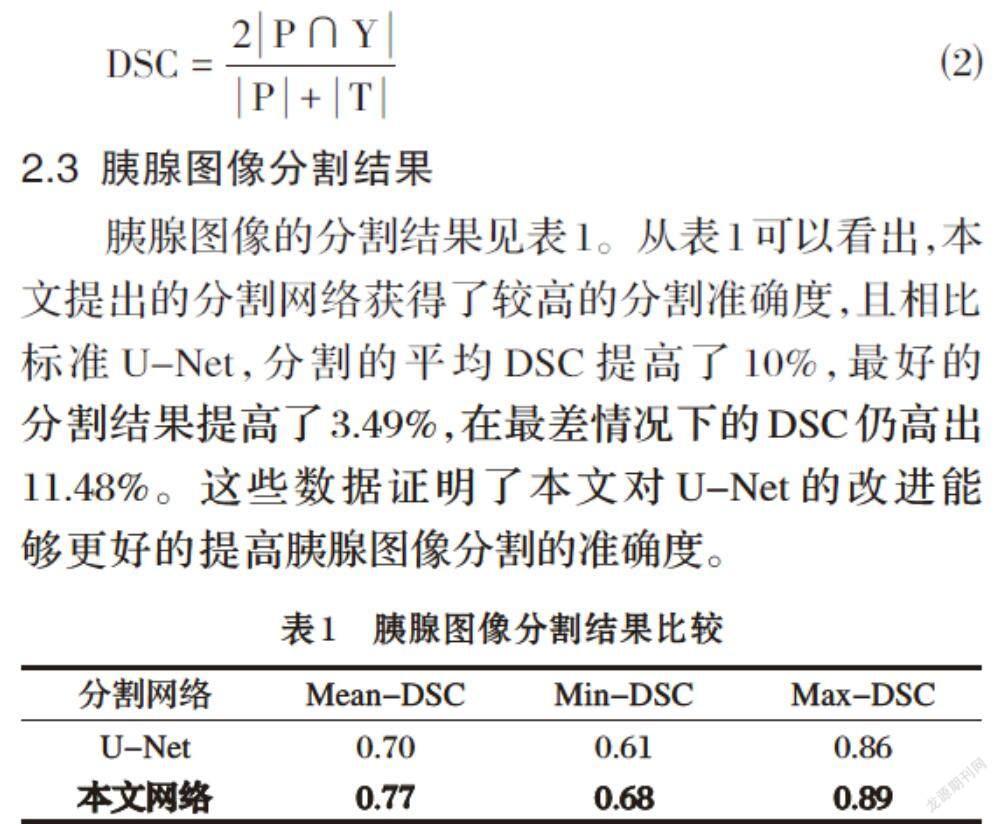
评价指标包括DSC(Dice Similarity Coefficient,集合相似度系数)、系统重建耗时以及与真实标记重建结果的可视化对比。其中,DSC通过网络预测的分割与真实标记相交部分占总体的比值来衡量分割的准确度,以P表示算法预测结果,Y表示真实标记,则DSC定义为:
[DSC=2P∩YP+T] ⑵
2.3 胰腺图像分割结果
胰腺图像的分割结果见表1。从表1可以看出,本文提出的分割网络获得了较高的分割准确度,且相比标准U-Net,分割的平均DSC提高了10%,最好的分割结果提高了3.49%,在最差情况下的DSC仍高出11.48%。这些数据证明了本文对U-Net的改进能够更好的提高胰腺图像分割的准确度。
2.4 三维重建结果
胰腺图像自动三维重建结果与真实标记的比较如图4所示,图中列出了测试集中3组典型样本(CT-82中的第69、75和第77例)的重建结果对比。在结果较好的测试样本中(如第69例),本文方法重建结果与真实标记基本相同,且优于使用标准U-Net的自动重建;重建结果存在的较多误差主要在于多预测了无关的组织(如第75例),其主要原因在于网络存在多分割,但相比U-Net,本文方法的改善效果明显,重建出的三维图像主体明确,误差明显减少;而对于结果较差的样本(如第77例),误差主要表现在漏掉了一部份组织,但本文方法对于局部重建效果仍有明显改善。因此,本文方法对于不同测试样本的重建结果与真实标记基本接近,能够直观反映出胰腺器官的实际情况,且三维重建结果优于使用标准U-Net的方法,证明了本文方法的实用性。
表2中统计了通过两组分割网络进行自动重建的系统耗时。相比使用U-Net网络,本文方法增加了1.2%的网络大小,因此增加了少量的重建耗时,但没有明显影响到整个系统的计算效率。在效率最低的情况下,本文方法用时不到15秒即可完成整个CT样本的自动三维重建,证明了本文方法的高效性。
3 结束语
为更好辅助胰腺疾病诊断,本文提出基于深度学习的胰腺图像自动三维重建系统。利用改进的U-Net网络对CT图像中的胰腺目标进行自动分割,并结合MC算法实现三维重建。本文方法重建速度快,准确度较高,能够全方位、多角度地了解胰腺结构,对辅助医学诊疗具有积极作用。重建的准确度与分割结果关系紧密,下步研究的重点是进一步提升分割的准确度。
参考文献(References):
[1] 周瑞泉,纪洪辰,刘荣.智能医学影像识别研究现状与展望[J].第二军医大学学报,2018.39(8):917-922
[2] 曹正文,乔念祖,卜起荣,等.结合超像素和U型全卷积网络的胰腺分割方法[J].计算机辅助设计与图形学学报,2019,31(10):1777-1785
[3] 贺楠楠.医学图像三维重建面绘制算法研究[J].电脑知识与技术,2017,13(32):210-211,213
[4] 易玮玮,陈子轩,徐泽楷,侯文广.一种基于CPU的三维超声图像体绘制方法[J].生命科学仪器,2019,17(1):32-36,31
[5] Ronneberger O,Fischer P,Brox T.U-net:Convolutional networks for biomedical image segmentation[J].CoRR abs/1505.04597(2015)
[6] rensen W E, Cline H E.Marching cubes:A high resolution 3D surface construction algorithm[J].Acm Siggraph Computer Graphics,1987,21(4):163-169
[7] HE K,ZHANG X,REN S,et al. Deep residual learning for image recognition[C]// Proceedings of the 2016 IEEE Conference on Computer Vision and Pattern Recognition. Piscataway:IEEE,2016:770-778
[8] OKTAY O,SCHLEMPER J,LE FOLGOC L,et al. Attention UNet:Learning where to look for the pancreas[EB/OL].[2019-04-13].https://arxiv.org/pdf/1804.03999.pdf.
[9] Milletari F, Navab N, Ahmadi S A. V-net: fully convolutional neural networks for volumetric medical image segmentation [C]//2016 fourth international conference on 3D vision (3DV). New York: IEEE,2016:565-571

