肿瘤微环境中免疫与基质细胞相关基因在肺腺癌中的预后价值
李世雄,耿华,徐美林
(1.天津医科大学胸科临床学院,天津 300041;2.天津市胸科医院病理科,天津 300300)
肺癌分为小细胞肺癌和非小细胞肺癌。非小细胞肺癌又可分为两种主要病理亚型,即鳞状细胞癌和腺癌。在肺腺癌中,约25%的肿瘤中检测到表皮生长因子受体(EGFR)突变和EML4-ALK重排。此外,还有一些罕见的肺癌驱动基因,如ROS1、RET、BRAF等。基于基因组研究和分子通路分析,靶向治疗和临床试验取得快速进展[1]。近年来,免疫治疗在肺癌患者中的应用越来越广泛。免疫检查点抑制剂,如细胞毒性T淋巴细胞相关抗原-4(CTLA-4)和程序性死亡1(PD-1)证明了治疗效应和安全性[2]。虽然肺腺癌患者已有多种治疗方式,但患者预后仍较差。因此,迫切需要为肺腺癌患者寻找新的、有效的、更准确的预后生物标志物。
肿瘤微环境(TME)由多种成分组成,包括基质细胞、肿瘤成分、先天免疫细胞和获得性免疫细胞[3]。近年来,越来越多的研究表明患者不同的肿瘤微环境特性在介导晚期转移、免疫逃逸和免疫治疗抑制中发挥着重要作用。基质细胞和免疫细胞是TME中的两种非肿瘤成分[4]。TME中的免疫细胞与恶性肿瘤细胞直接作用或通过细胞因子和趋化因子信号转导相互作用,进而影响肿瘤的生物学行为和治疗反应。基质细胞是TME中的另一重要细胞,有研究表明,基质细胞与肿瘤细胞之间存在着动态、双向、复杂的关系[5]。Yoshihara等[6]通过分析TME中免疫细胞和基质细胞特异性基因表达水平,首创了一种名为ESTIMATE的算法来计算肿瘤患者的基质、免疫评分,利用该评分来预测非肿瘤细胞的浸润情况。目前已有相关报道将ESTIMATE应用于胃癌[7],揭示了这种基于大数据的算法的有效性。
在本研究中,笔者对肺腺癌患者临床病理数据以及基质和免疫评分进行相关性分析。根据肺腺癌患者的基质和免疫评分高低筛选出差异表达基因,对差异基因构建相互作用网络并进行富集分析。最后利用生存分析筛选出一组TME相关基因预测肺腺癌患者的预后。
1 资料与方法
1.1 数据收集和预处理 501例肺腺癌患者的基因表达数据来自癌症基因组图谱(TCGA)数据库。数据中女性患者270例(53.9%),男性患者231例(46.1%),年龄38~89岁。利用Illumina平台分析肺腺癌RNA表达谱,所有数值均用log2转换。使用每千个碱基的转录每百万映射读取的片段(FPKM)对在TCGA数据库中注释的19 745个蛋白编码基因进行表达定量。临床资料包括性别、年龄、组织学分级、TNM分期、临床分期、生存时间和随访结局。此外,丢失临床病理信息或生存数据的样本将被删除。
1.2 基质和免疫评分ESTIMATE评分系统包括基质评分和免疫评分。评分基于TCGA数据库中肺腺癌患者的基因表达情况。评分可以通过在线网站(https://bioinformatics.mdanderson.org/estimate/disease)下载。ESTIMATE算法可以通过分析肿瘤样本的转录组数据推断在TME中基质细胞和免疫细胞的浸润情况。
1.3 临床病理特征与基质、免疫评分之间的相关性根据临床病理特征对患者进行分组,分析各组基质和免疫评分高低差异,χ2检验分析基质、免疫评分与临床病理特征之间的相关性。用评分的中位数将这些样本分为高基质评分组、低基质评分组,免疫评分同前分为两组,采用Kaplan-Meier法和log-rank检验分析两组总生存率的差异。
1.4 筛选差异表达基因将肺腺癌患者样本分为两组,即高基质(或免疫)评分组和低基质(或免疫)评分组。以基因表达差异倍数变化>2.0和P<0.01作为筛选差异表达基因(DEGs)的临界值。使用TBtools将DEGs显示在热图上(https://github.com/CJ-Chen/TBtools/releases)[8]。
1.5 蛋白质-蛋白质相互作用(PPI)网络的构建利用基因相互作用检索工具(STRING,https://string-db.org/)[9]检索DEGs的相互作用网络。并通过Cytoscape软件(版本3.7.2)进行基因相互作用的可视化[10]。然后使用Cytoscape的分子复合体检测(Mcode)插件,根据拓扑原理寻找最重要的模块即为连接最密集的区域。选择设置为MCODE分数>5,度截断值=2,节点截断值=0.2,最大深度=100,k-分数=2。
1.6 基因本体论和KEGG通路富集分析可用于基因注释、可视化和集合探索的数据库(DAVID v.6.8)被用来进行基因的富集分析[11]。对基质评分组差异基因和免疫评分组差异基因分别进行细胞成分、分子功能、生物过程等基因本体论分析和KEGG通路的富集分析。FDR校正后的P<0.05被认为在基因本体论分析和KEGG通路富集分析中具有统计学意义。
1.7 关键基因的生存分析采用log-rank方法评价关键基因的预后价值。与肺腺癌患者总生存率显著相关的基因被确定为预后相关基因。
1.8 统计学处理数据均通过SPSS 24.0进行统计学分析。利用χ2检验分析基质、免疫评分与临床病理特征之间的相关性,采用t检验和Cytoscape软件筛选差异表达基因,Kaplan-Meier方法评价肺腺癌TME中关键基因的预后价值。
2 结果
2.1 基质、免疫评分与肺腺癌临床病理特征性分析501例肺腺癌患者的基质评分在-1 959.31~2 098.77,免疫评分分布于-1 355.85~2 905.30。如图1A~D所示,除淋巴结转移分期之外,免疫评分与T分期、M分期、临床分期显著相关(均P<0.05)。而基质评分与M期显著相关(P<0.05)。肺腺癌患者根据评分分为高低两组。如图1E所示,在相同时间高免疫评分组总生存率较低免疫评分组更高(P=0.0093)。
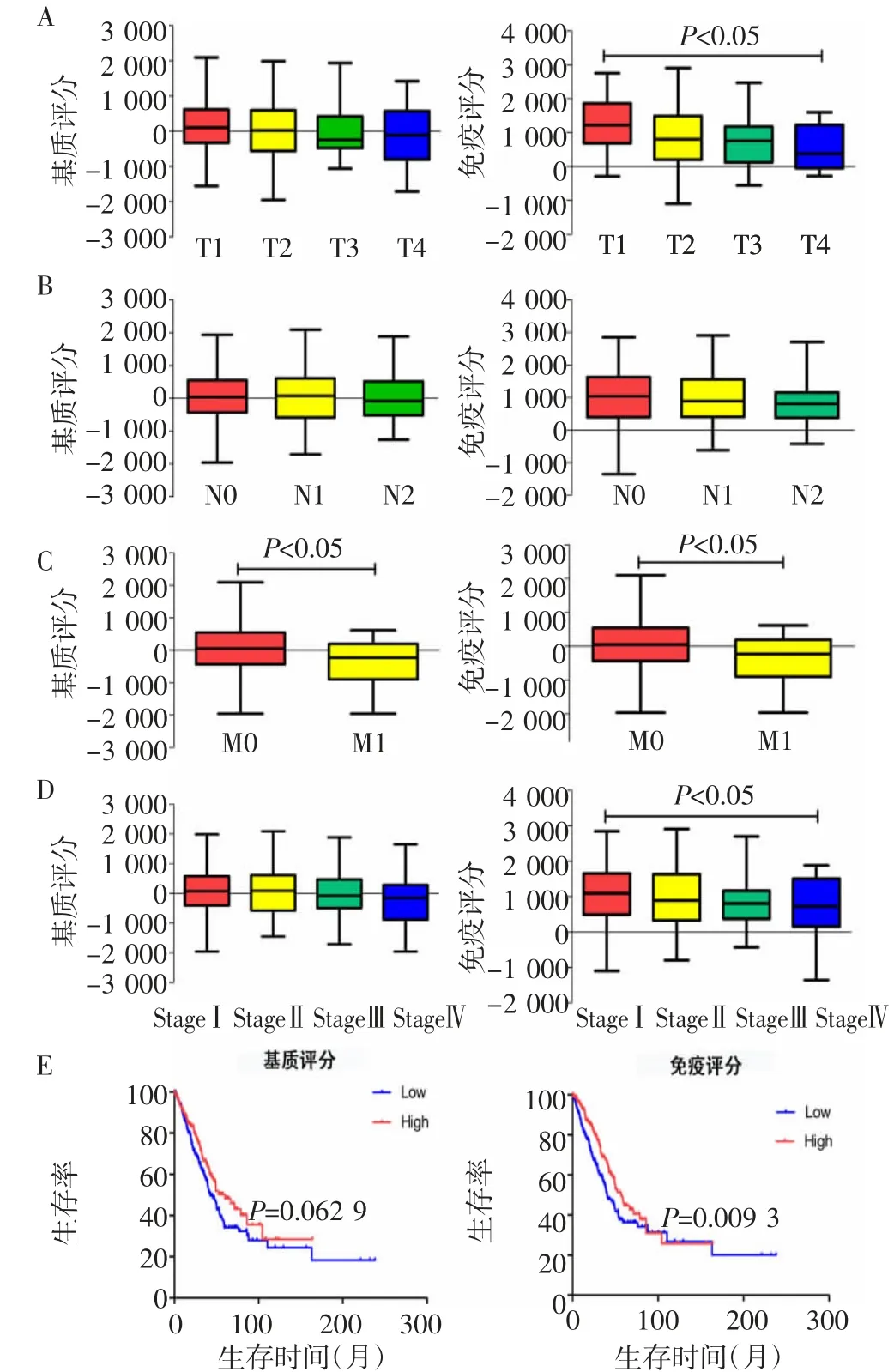
图1 基质、免疫评分与肺腺癌患者临床病理特征和总生存率的相关性Fig 1 Association of stromal/immune scores with clinicopathological features and overall survival rate
2.2 基于免疫、基质评分的DEGs图2显示高免疫评分、基质评分组和低免疫评分、基质评分组之间表达水平有显著差异的基因。与免疫低评分组相比,高评分组共出现257个基因表达上调,5个基因表达下调(表达差异倍数>2.0;P<0.01)。此外,与基质低评分组相比,基质高评分组中有392个基因表达上调,但没有基因表达下调(表达差异倍数>2.0;P<0.01)。利用韦恩图显示免疫评分组和基质评分组共有74个共同的DEGs。

图2 肺腺癌患者基于基质评分和免疫评分的基因表达热图和韦恩图Fig 2 Heat maps and Venn diagrams of gene expression based on stromal score and immune score in lung adenocarcinoma
2.3 DEGs的功能富集分析基因本体论对基质评分差异表达基因的分析显示与免疫反应有很强的相关性(表1)。生物过程(BP)分析主要富集于“补体激活”、“免疫反应调节”、“免疫反应”等。细胞成分(CC)分析主要包括“细胞外区域”、“细胞外泌体”、“细胞膜”等。分子作用(MF)分析包括“抗原结合”、“丝氨酸型内肽酶激活”等。
KEGG信号通路分析显示,许多通路与免疫应答相关(表1),包括“ECM受体相互作用”、“PI3KAkt信号通路”、“细胞因子-细胞因子受体相互作用”等。同样,在免疫评分差异表达基因中,BP分析主要富集在“免疫反应”、“炎症反应”和“多肽或多糖的抗原处理和递呈”等。CC分析主要包括“细胞膜”、“质膜的组成部分”、“MHCⅡ类蛋白质复合物”等。MF分析包括“受体激活”、“受体结合”、“跨膜信号受体激活”等。KEGG通路分析确定了与“金黄色葡萄球菌感染”、“吞噬体”和“同种异体排斥反应”相关的通路(表2)。
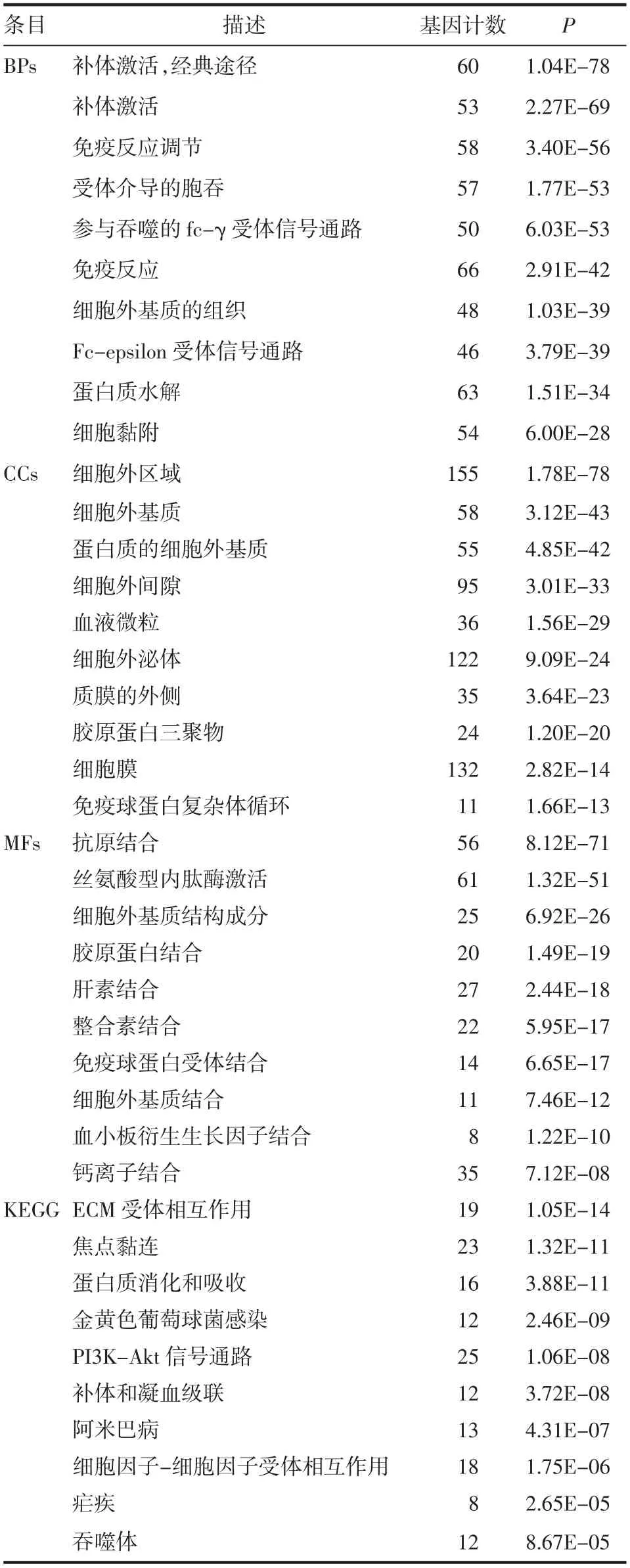
表1 基于基质评分的DEGs功能富集分析Tab1 Functional enrichment analysis of differentially expressed genes based on stromal scores(BPs,CCs and MFs)
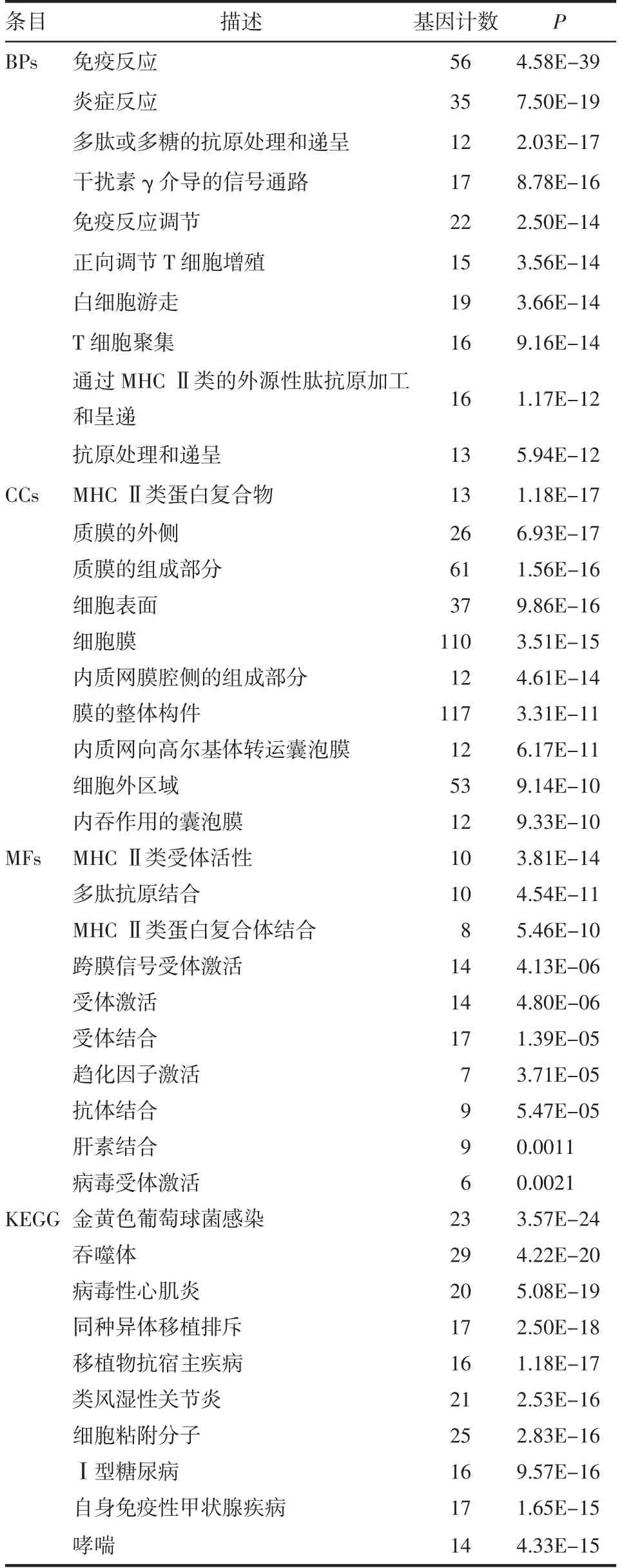
表2 基于免疫评分的DEGs功能富集分析Tab 2 Functional enrichment analysis of differentially expressed genes based on immune scores(BPs,CCs and MFs)
2.4 构建PPI网络和筛选关键基因图3A显示了基质评分的DEGs的PPI网络,并显示了网络中排名前35位的差异基因。筛选出最值得关注的10个节点被认为是关键基因,包括COL1A2、COL4A2、COL3A1、COL1A1、COL4A1、COL5A1、THBS2、BGN、DCN和POSTN。同样,图3B给出了基于免疫评分的DEGs的PPI网络,显示出中排名前29位的基因表达蛋白。排在前10位的关键基因为ITGB2、GSF1R、CD86、SPI1、PLEK、TYROBP、LY86、ITGAM、HCK和CTSS。

图3 基于基质、免疫评分差异表达基因和关键基因的PPI网络Fig 3 PPI networks of differentially expressed genes and key genes based on stromal and immune scores
2.5 生存分析在20个基因中,共有4个DEGs显示与OS时间显著相关(图4),PLEK、LY86、HCK高表达与较好的总生存率显著相关(均P<0.05),POSTN被认为是一个预后不良相关基因(P<0.05)。这4个基因均为上调的DEGs基因。
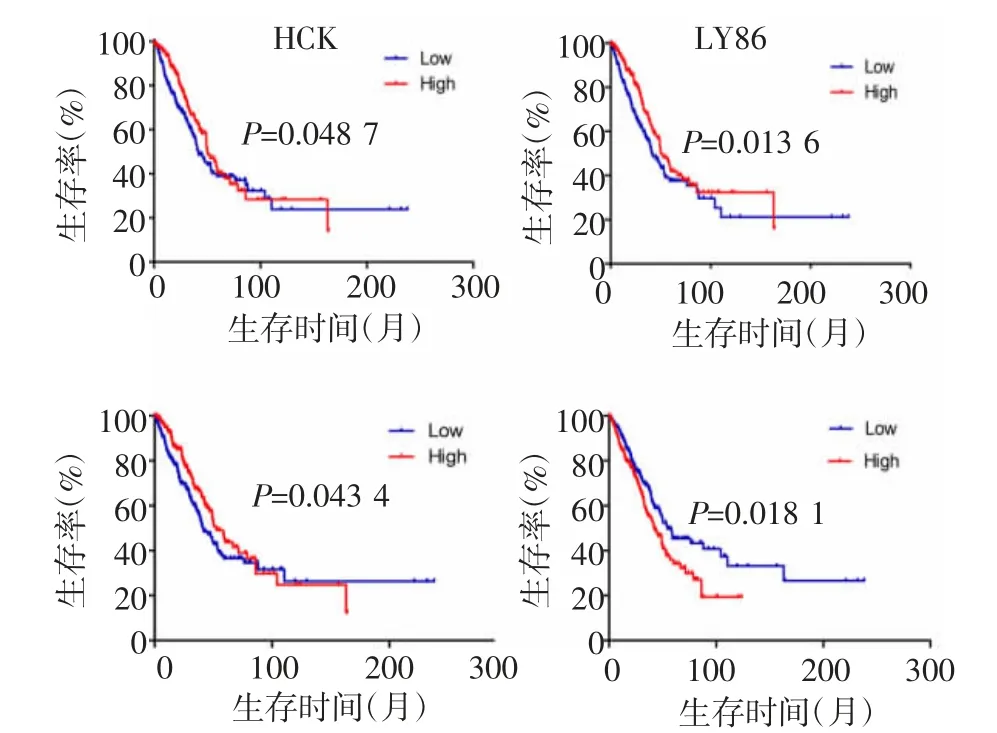
图4 筛选出的4个关键基因在肺腺癌患者中的生存分析Fig 4 Survival analysis of four key genes screened in patients with lung adenocarcinoma
3 讨论
预测肺腺癌患者的预后仍然是研究人员和临床医生面临的挑战。恶性肿瘤细胞与TME的相互作用极大地影响了肿瘤的发展,进而影响了肿瘤的复发、耐药和患者的总体、无进展生存率[12]。免疫细胞和基质细胞在肿瘤内的淋巴细胞浸润已被证实与肺腺癌患者的预后有关[13]。随着知识、技术,甚至临床实践标准的发展,分子表达谱在过去几年中经历了巨大的变化。检测广泛的分子表达水平对所有转移性实体肿瘤患者都是必要的。因此,本研究尝试从TCGA数据库中筛选出影响肺腺癌患者预后的免疫、基质细胞相关基因。本研究创新点在于,这些基因与TME中的基质和免疫成分相关。这将有助于临床工作者分析和预测患者的预后。
笔者通过ESTIMATE数据库获取肺腺癌患者基质细胞和免疫细胞的评分,探讨该评分与肺腺癌患者的临床病理特征和总生存期的相关性。结果显示,随着T分期、M分期和临床分期的进展,患者的免疫评分随之显著降低,但与N分期无相关性。同时,M1期肺腺癌患者的基质评分相较M0期显著下降,但与T分期、N分期和临床分期无关。先前研究表明,晚期肺腺癌患者出现免疫细胞减少,包括B细胞、CD4+T细胞、CD8+T细胞、树突状细胞[14]。另一项研究发现,免疫评分和基质评分与肺腺癌患者的临床特征,包括T期、M期、病理分期和总生存时间显著相关[15]。有较多免疫细胞、基质细胞浸润的肺腺癌患者分期较早,预后较好。此外,基于免疫、基质评分的生存分析也显示出统计学差异。较低的基质、免疫评分与肺腺癌患者较差的总生存率相关。免疫细胞的存在起到了杀死肿瘤细胞和抑制肿瘤生长的作用,因此,从理论上讲,免疫评分高的患者生存期应该会提高。笔者的研究与这一假设是一致的。
按基质细胞和免疫细胞的评分分为高分组和低分组,总共筛选了579个DEGs。随后的GO分析发现它们大多参与免疫和炎症反应及免疫反应调控、T细胞共刺激、抗原与受体结合。这证实了免疫系统在肺腺癌微环境形成中的重要作用。KEGG通路分析也显示大部分的DEGs与免疫应答显著相关,这与之前的报道一致。为了更好地理解DEGs之间的相互作用,笔者构建了PPI网络。在由基质和免疫的差异基因构建的PPI网络中,MCODE被用来选择最重要的网络模块。本研究基于免疫评分确定了10个与免疫细胞相关的关键基因,基于基质评分确定了10个与基质成分相关的关键基因。在肺腺癌患者中筛选出4个TME相关基因,结合生存分析这4个基因的表达与总生存时间显著相关。
普列克底物蛋白(PLEK)位于人类染色体2q14,在心血管疾病、牙周炎、溃疡性结肠炎和类风湿性关节炎中显著过表达,表明其在慢性炎症性疾病中具有重要作用[16]。淋巴细胞抗原(LY86),又被称为MD1、MD-1或MMD-1,敲除LY86导致单核细胞向MCP-1迁移减少,这可能是念珠菌易感性增加的基础。LY86缺乏在结肠炎的进展中至关重要,因此LY86可能是未来炎症性肠病介入治疗的一个潜在靶点[17]。造血细胞激酶(HCK)编码的蛋白是Src家族酪氨酸激酶的成员。它在中性粒细胞迁移和中性粒细胞脱粒中发挥作用。研究发现HCK的高表达与大肠癌患者的低生存率有关,并与活化的巨噬细胞具有相关性[18]。HCK活性的升高也在许多实体恶性肿瘤中被观察到,包括乳腺癌和结肠癌,并与患者存活率降低相关。HCK的过度激活可能是由外周免疫细胞分泌的细胞因子刺激肿瘤细胞,从而增强肿瘤的活化,促进TME的形成。骨膜蛋白(POSTN)的过表达通过促进肿瘤的转移和侵袭与胃癌转移呈正相关[19]。转移相关肺腺癌转录本1(MALAT1)通过负向调控miR-202-3p促进POSTN的表达,该生物传导轴与宫颈癌细胞活力、迁移和侵袭以及上皮细胞向间充质转化的调控密切相关[20]。在本研究中,POSTN被确定为与肺腺癌不良预后相关的基因。而在肺腺癌患者中,PLEK、LY86和HCK的高表达预示着较好的预后。这为阐明肿瘤发展的分子机制提供依据,也对预测患者预后具有重大意义。
在患者的预后预测和个体化治疗时,分子遗传特征与临床病理特征相结合往往发挥重要作用[21]。免疫治疗促进宿主免疫系统对肿瘤的识别,刺激免疫系统对肿瘤细胞生长增殖进行抑制[22]。与以往关注肿瘤细胞本身内在基因激活如何影响TME的报道相比,该研究更多关注于TME中的基因标志物,哪些基因会影响肺腺癌的进展,从而影响患者的预后。该研究为分析肺腺癌和其所在的TME之间复杂的相互作用提供新的思路。
这项研究也有一些局限性。首先,这是一项基于公开数据库的回顾性研究,很难包涵不同地理人种间的差异。其次,考虑到不同肿瘤区域的微环境可能不同,在肿瘤的核心和侵袭边缘进行免疫和基质评分也往往存在不同。最后,笔者的发现还需要进一步实验验证。

