基于Unet的多注意力脑肿瘤图像分割算法
吴 量,付殿臣,程 超
(长春工业大学 计算机科学与工程学院,吉林 长春 130012)
0 引 言
医学图像分割是医学图像分析领域的一个重要内容,通常所说的分割指的是语义分割[1],即通过对图像的每一个像素标记上语义信息,来实现像素级别的预测,属于计算机视觉的范畴。与自然图像分割相比,医学图像分割任务由于边界模糊、数据集小、语义简单等特点,实现的难度会更大。为了提高医学图像分割的效率和准确性,人们开展了越来越多的尝试,其中一个很重要的方向就是脑肿瘤MRI图像的分割[2]。近年来,脑肿瘤在社会上已经成为一种常见的疾病,对人们的生命造成了巨大的威胁。脑肿瘤个体间形状、纹理差异较大,很难提升分割的精度。目前,脑肿瘤医学诊断主要依赖于医生个人知识的积累和实战经验的判断,这种依赖存在较大的潜在问题,比如耗时长,或在高强度的重复工作条件下医生过度疲劳而由此引发的漏诊和误诊。因此,构建一种精确、快速的脑肿瘤分割方法对脑瘤的诊断和早期治疗具有很重要的意义。
1 相关工作
目前脑肿瘤分割算法有很多,主要分为传统图像分割方法和基于深度学习的分割方法。
1.1 传统图像分割方法
在传统的图像分割中,单独使用一种分割方法得到的分割精度较低,研究者经常将传统方法与其他的方法结合使用以提高分割的精度。
阈值法通过设定不同的阈值将分割目标与背景的像素区分,达到分割的目的,特点是计算速度快,但是适用性有限。赖小波等人结合传统机器学习方法随机森林和阈值法提出了一种多模态MRI图像和多特征融合的脑肿瘤分割方法[3],首先进行图像校准和偏置场校正,融合提取的多模态特征构建RF模型进行粗分割,最后根据先验知识设定阈值进行分割。基于区域的方法优点是简单,能够正确分割具有相似属性的区域,缺点是容易造成过度分割。候冬奥等人结合区域生长法提出了一种全自动的脑肿瘤分割方法[4],该方法利用人脑结构对称信息,通过区域生长法实现粗略分割,然后使用改进的测地线活动轮廓(geodesic active contours,GAC)模型对粗略分割区域进行精细分割。童云飞等人采用快速模糊C均值聚类和自动阈值法对肿瘤区域进行粗略轮廓提取[5],而后使用混合水平集算法来提取病灶的边界。张腾达等人针对传统水平集(Level Set)方法对肿瘤MRI图像进行分割时弱边缘分割缺失的现象[6],采用模糊聚类算法与Level Set结合的方式增强了传统方法对脑肿瘤分割的鲁棒性和准确性。
1.2 基于深度学习的图像分割方法
近年来,深度学习技术快速发展,人们结合深度学习,提出了很多优秀的脑肿瘤MRI图像分割方法。研究者首先应用卷积神经网络(CNN)对语义分割进行了探索,并将CNN应用于脑肿瘤MRI图像分割[7],得到了良好的分割结果。但是,由于CNN本身的输出全连接层结构,模型在分割图像时耗费巨大的内存,计算效率很低。另外,CNN只可以对输入图像进行像素的简单分类,且全连接层限制了输入图像必须固定尺寸大小,通常模型的预测图像较输入图像的分辨率会小一些,这大大影响了分割的效率和精度,因此得到的预测分割结果往往不尽如人意。
为解决这一问题,2015年Long等人[8]提出了一种专门适用于图像分割任务的全卷积神经网络(FCN)模型。FCN模型不仅避免了CNN全连接层在计算时重复冗余卷积造成的存储开销过大的问题,也解决了图像尺度的可变换问题,使得网络可以接受任意大小的图像,模型的输出图像可以恢复到与原图大小的分辨率,实现了图像在真正意义上的语义分割。然而,医学图像数据对细节较为敏感,传统FCN的分割精度是远远不够的。邢波涛等人基于FCN模型[9],将多模态MRI图像进行预处理和特征融合,通过结合条件随机场(CRF)来细化FCN的粗病灶分割结果。针对医学图像的分割任务,Ronneberger等人[10]基于FCN提出更加适用于医学图像分割的Unet模型。与FCN模型相比,Unet模型结构对称,其特有的Skip Connection结构充分结合底层空间特征和高层语义特征,对图像的细节特征提取更加敏感,针对医学图像的小目标分割更加精确。Dong等人[11]将Unet模型应用于脑胶质瘤的分割,在BraTS 2015[12]脑肿瘤MRI数据集上取得了优良的分割效果。
因Unet在医学图像分割任务中表现出的优良性能,近几年来提出的医学图像分割模型[13-17]大多基于U型对称结构和应用Unet的特征融合方式,研究者分别在模型深度和特征融合方式等方面对Unet进行了改进。Ibtehaz等人[14]通过结合深度残差网络(ResNet)和Unet模型,加深了模型的深度,并将算法应用于医学图像分割,在5个医学数据集上较Unet提升了分割的精度。最近,Cao等人[15]充分利用密集连接网络[16](DenseNet)特征融合特性,将密集连接(Dense Connection)与Unet模型结合应用电磁图像的分割任务,并通过实验验证了DenseUnet分割性能的提升。医学图像多为3D数据,2D卷积不能充分利用3D数据的空间结构信息,而3D卷积具有较高的计算成本和GPU内存消耗。为了解决这些问题,Li等人[17]提出了一种新的混合密集连接的Unet,并取得了优良的分割指标。针对Unet不同大小的数据集自适应层数问题,Zhou等人[13]提出了Unet++模型,模型可根据不同的数据集自适应调整网络的提取层数和减少训练参数量。随着深度学习理论的完善,人们对不同领域的任务也进行了交叉研究。有研究者尝试把与人类视觉机制相符合的注意力机制应用于图像处理领域。Li等人[18]对注意力门输出层的通道结合方式进行了改进,并用DenseNet结合基础的Unet模型。该文基于Unet重新设计网络提取模型并结合改进后的多注意力机制,提出了一种基于Unet的多注意力脑肿瘤MRI图像自动分割算法。
该文的主要创新点总结如下:
(1)提出了一种快速、准确、端到端的脑肿瘤MRI图像自动分割方法。
(2)在主干特征提取网络部分,重新设计Unet网络的结构,模型与ResBlock结合并且增加并行Dalited Convolution特征提取模块,提升了网络特征提取的性能。
(3)针对小型病灶分割精度低的问题,引入空间注意力和层注意力多注意力机制,使得特征提取网络更加注意某些特征层和空间区域的关键特征,抑制了冗余区域的无效特征。
(4)在BraTS数据集上对比了其他分割网络,验证了该文的模型,得到了较好的分割效果。
2 算法设计
文中算法对四种模态图像数据进行并行处理,最后得到多类肿瘤的分割预测结果,整体的算法流程结构示意图如图1所示。
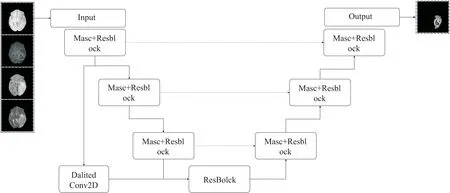
图1 算法流程示意图
2.1 主干特征提取网络的设计
模型基于经典的U型网络,去掉了Unet的池化层并整体采用ResBlock[19]对网络进行加深处理,使得网络能够充分融合浅层和深层语义信息。为保证引入ResBlock后不增加大量的参数,模型去除了两层网络,将各层的通道数调整为原来的1/2。另外,模型增加了一条并行的特征提取模块,提取的特征与最后一层下采样层进行特征叠加。考虑到大幅度的下采样会导致有效特征的丢失,在特征融合前,并行模块采用了膨胀系数为2的Dalited Convolution[20]对第一层提取到的特征进行了二次特征提取,通过增大特征提取卷积的感受野,使得特征下采样过程更加平滑。并行的特征提取网络结构如图2所示。
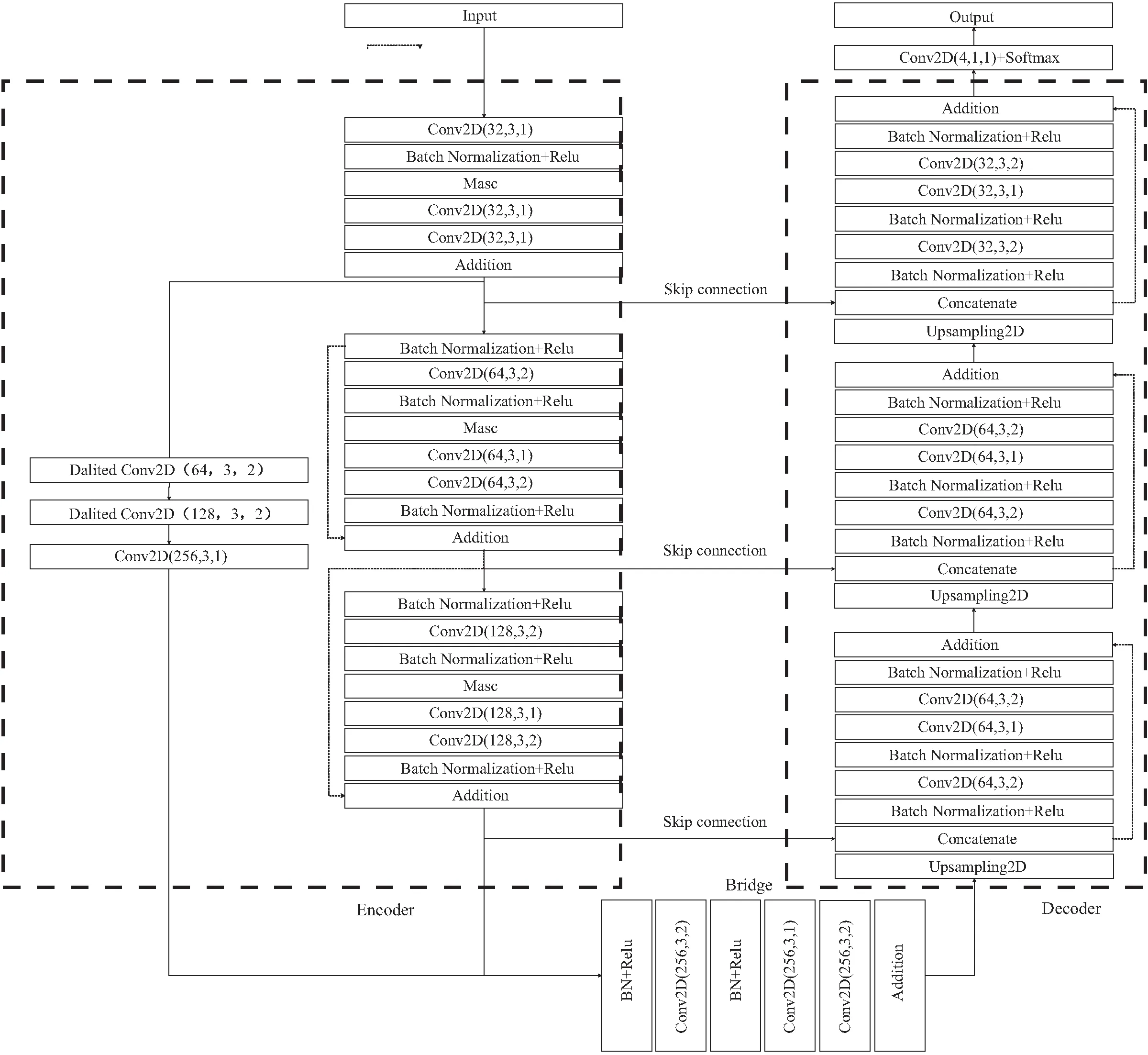
图2 网络模型结构
2.2 多注意力机制Masc模块
该文提出的Masc注意力机制模块,延续了传统注意力机制对输入图像进行通道级别的注意力特征图像级联空间注意力模块的设计思想。在此基础上对模型的输入特征单独应用空间注意力模块进行空间注意特征提取,对两次注意力机制获取的空间注意力特征做了特征融合,使模型更加注重提取图像的二维空间信息。
注意力模块的结构如图3所示,具体的算法流程接下来会详细描述。假设Masc模块的输入是任意一个中间特征F∈RH*W*C,F首先同时输入Masc模块的通道注意力模块和空间注意力模块,分别得到一维通道注意图Fc∈R1*1*C和二维空间注意图Fs2∈RH*W*1,在得到Fc之后,Fc会输入到级联的空间注意力模块得到Fs1∈RH*W*1,最后采取特征相叠加的特征融合方式合并Fs1和Fs2得到最终的注意图Fsc∈RH*W*1。总体的注意图提取过程可以公式化如下:
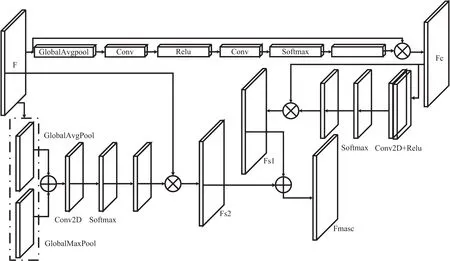
图3 Masc注意力机制模块
Fc=Mc(F∈RH*W*C)
(1)
Fs1=Ms1(F∈RH*W*C)
(2)
Fs2=Ms2(Fsc∈R1*1*C)
(3)
Fsc=Fs1+Fs2
(4)
其中,M*(*)表示特征图进入相应模块进行提取注意特征与原图进行矩阵元素相乘运算。Fsc∈RH*W*1是最终注意力模块的输出结果。
2.2.1 通道注意模块
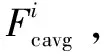

(5)
其中,σ1表示softmax激活函数,σ0表示relu激活函数,w*,b*分别表示卷积层的权重和偏置,⊗ 表示元素乘积运算。下面公式参数同式(5)。
2.2.2 空间注意模块
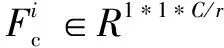
(6)
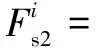
GlobalMaxPool(Fi)))⊗Fi=
(7)
(8)
该文的Masc模块分别对输入的图像进行通道注意力和双空间注意力的提取,更加注重图像2维空间的信息。
3 实 验
3.1 数据集介绍及预处理
数据集采用的是公开数据集BraTS 2019脑胶质瘤MRI数据,其中包含259例高级别胶质瘤(HGG)和76例低级别胶质瘤(LGG)两类患者数据。HGG患者包括水肿(edema),坏死(necrosis),增生(enhancing tumor)肿瘤,非增生(non-enhancing tumor)肿瘤部分,而LGG患者增生(enhancing tumor)可能存在也可能不存在。BraTS 2019数据集[12]包括4类标签,如图4(e)所示,分别是正常组织(标签0)、坏死和非增生肿瘤(标签1)、水肿(标签2)、增生肿瘤(标签4)。分割任务包括三个:(1)完整肿瘤区域(Complete tumor),包括所有三个肿瘤内类,标签是1、2和4。(2)核心肿瘤区域(Core tumor),不包括“edema”区域,标签是1和4。(3)增强肿瘤区域(Enhancing tumor),只包括标签4。实验将BraTS 2019的训练数据分成训练集、验证集和测试集,比例分别是0.6、0.2和0.2。原始数据集是由T1、T2、T1C和FLAIR四个序列构成的3D数据,如图4(a)~图4(d)所示。每例数据由155张厚度为1 mm×1 mm×1 mm,大小为240×240的切片组成。在数据预处理方面,该文取3D数据的2D切片,只对数据做了主要部分的裁剪和空白切片的去除,具体的做法是将(155,240,240)大小的切片处理成(146,192,152)大小,除此之外未做任何的数据增强。另外,该文没有对每种病灶对应相关模态单独处理,四种模态都是单通道图片数据,直接将数据合并成四通道数据送入网络,这种数据处理方式能有效地结合每种模态的特性,更利于模型的训练。
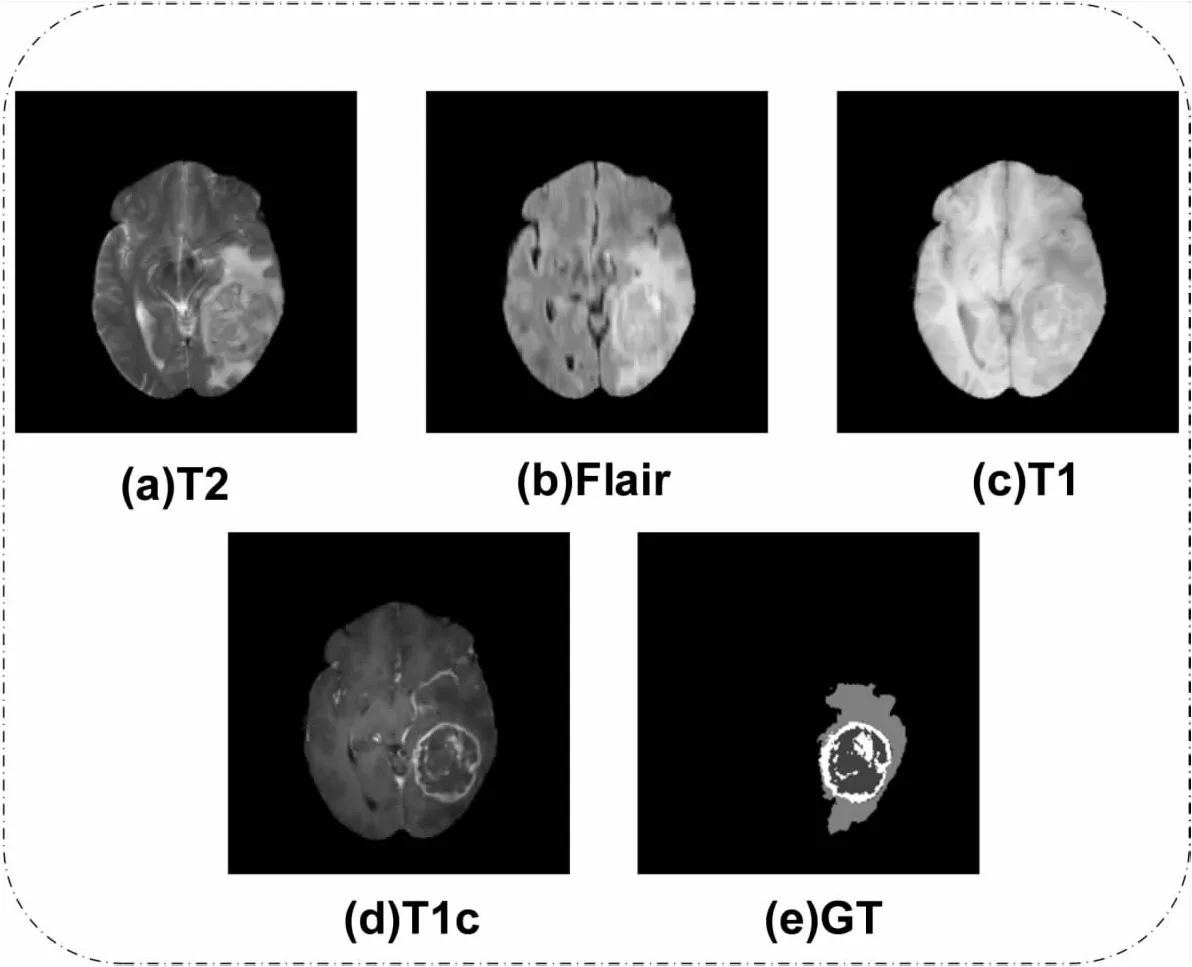
图4 BraTS脑肿瘤数据
3.2 训练环境及模型参数设置
该文的实验环境是Nvidia Tesla V100 GPU,所用框架为基于Tensorflow的Keras框架。模型参数设置:网络优化器采用的是Adam优化器,初始学习率LR设置为1e-3,Batch_Size设置为4,Epoch设置为200。根据不同的实验模型,参数略有调整。
该文的损失函数(Loss Function)采用Sudre[21]提到的Weighted Cross-Entropy (WCE)和Generalized Dice Loss (GDL)相结合的方式。
(9)
(10)
3.3 评价指标
在MRI脑肿瘤分割任务中,最重要的评价指标是Dice相似系数。Dice相关系数有效地衡量了算法分割结果与真实标注标签的重叠程度,具体计算如公式(11):
(11)
其中,TP、FP、FN分别为真阳性、假阳性、假阴性。Dice相关系数越大表示脑肿瘤的分割结果越好。
3.4 实验结果
如表1所示,文中模型MascParallelUnet在参数量并未增加的条件下,分割性能较原始Unet 模型分割Complete Dice提升了1.56%,Core Dice提升了4.06%,Enhancing Dice提升了9.58%。
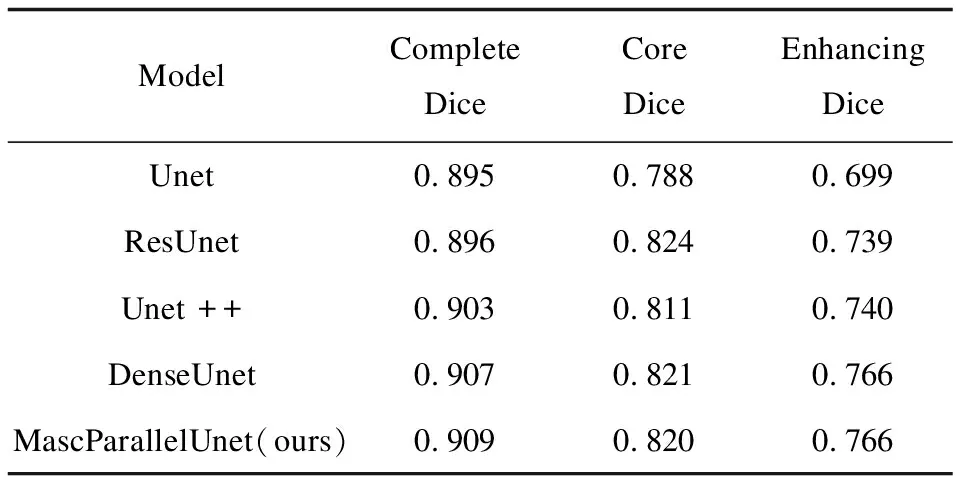
表1 对比实验Dice指标对比
分割效果如图5所示,可以看到文中模型得到的分割结果对于脑肿瘤病灶的细节处理得更好。与现有的模型对比,文中模型比DenseUnet模型的分割精度略低,但是DenseUnet的参数量较该文网络参数量多了2倍左右。
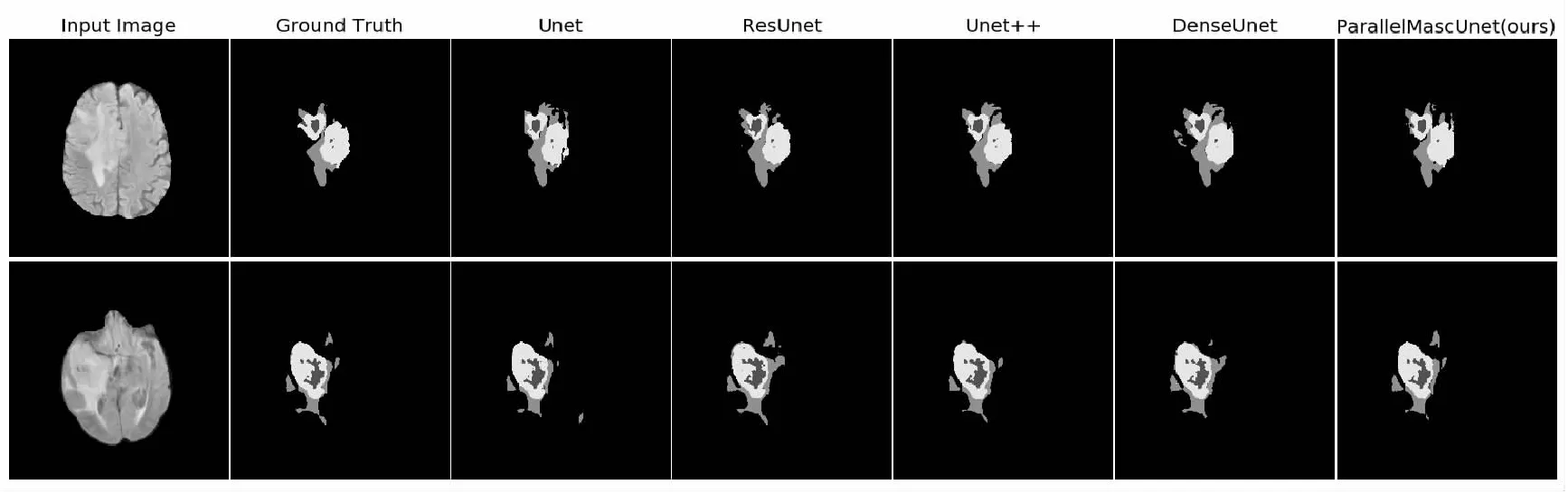
图5 对比实验分割效果图
该文分别测试了结合Masc模块和不结合Masc模块对模型分割精度的影响,并与其他注意力模块进行了对比。如表2所示,可以发现Masc注意力结构加入Unet,Complete Dice没明显提升,而Core Dice和Enhancing Dice有显著提升,分割效果如图6所示。该文在对比实验中加入CBAMUnet和AGUnet,实验结果表明两种注意力模块的Complete Dice和Core Dice都比传统Unet略低,在Enhancing Dice上有所提高。该文提出的新的注意力模块结构较Unet的Core Dice提升了2.03%,在Enhancing Dice上提升了8.73%,可以发现该文提出的Masc模块对小目标病灶的分割更加精确,证明了该文改进的注意力模块的有效性。另外,不加入Masc模块的并行特征提取网络ParallelUnet较Unet模型在Core Dice和Enhancing Dice上也有显著的提升,说明该文提出的并行特征提取网络对总体脑肿瘤分割精度的提升起到了主导作用。
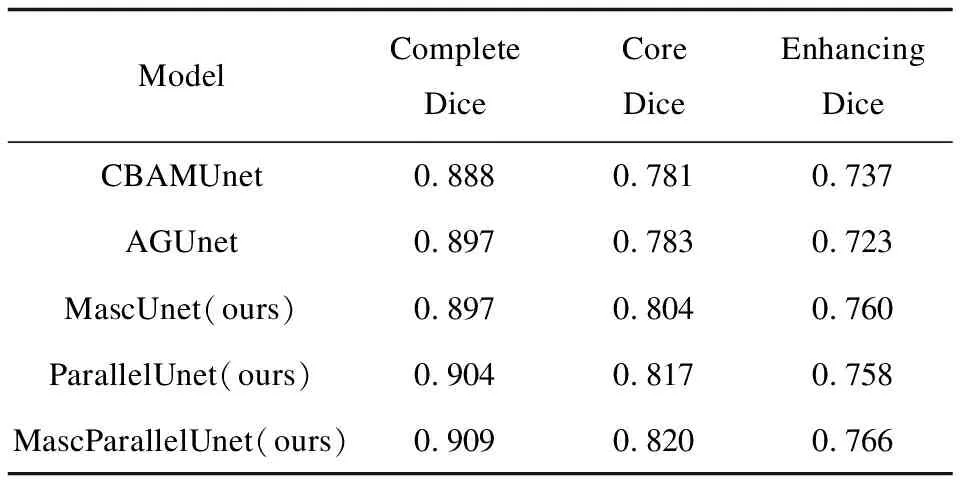
表2 注意力模块Dice指标对比

图6 注意力模块性能分割效果图
4 结束语
为了提升脑肿瘤多类型病灶分割的分割精度,该文提出了一种基于Unet网络的快速、准确的脑肿瘤自动分割模型。首先,重新设计了模型编码网络结构,引入并行Dalited Convolution提取模块,融合浅层空间特征与高层语义特征,使特征提取更加充分。其次,模型结合多注意力机制并对空间注意力模块进行了特征融合加强,使模型更加注意图像的二维空间信息。最后,对比其他基于Unet改进的方法,该文模型的分割精度得到了一定程度的提升。此外,模型是基于2D数据集进行分割的,并没有充分利用数据集3D的空间信息。因此,下一步研究重点是把2D模型拓展到3D模型。

