基于数据库分析CDCA基因家族在胃癌中的表达及预后意义*
罗 倩,詹雪冰,董芳媛,孙恩涛,王书翰,陈 冰
(1.皖南医学院病理教研室,安徽 芜湖 241002; 2.皖南医学院检验学院)
胃癌(Gastric cancer, GC)是全球第五大最常见恶性肿瘤,其致死率在所有肿瘤中排名第三[1]。手术是胃癌患者的主要治疗手段,在过去的几年里诊断和治疗有所进步,但由于大部分患者发病初期无明显症状,就诊时已进入中晚期,并且超过半数的胃癌根治术术后会有复发或伴有远处转移,胃癌患者总体5年生存率低于30%[2]。因此,分子标记物异常表达的检测可能为胃癌患者早期诊断和预后的判断提供有效的参考。
细胞分裂周期相关蛋白(cell division cycle-associated,CDCA)家族有8个成员,即CDCA1(NUF2)、CDCA2、CDCA3、CDCA4、CDCA5、CDCA6(CBX2)、CDCA7、CDCA8。CDCAs基因在生命过程中发挥着重要作用,细胞分裂过程中的失调可能导致恶性肿瘤[3]。据报道,CDCA1的缺失抑制了CD103的抗肿瘤活性,降低了黑色素瘤的检查点反应,促进了黑色素瘤的发生和转移[4]。CDCA2通过调控PI3K/AKT通路,影响结直肠癌细胞体内外的增殖[5]。CDCA3作为miR-145-5的靶基因,参与调控结直肠癌的增殖、转移和上皮细胞-间充质转化[6]。CDCA4是Nrf2信号通路的下游基因,在MCF-7/ADM人乳腺癌细胞系中调控细胞增殖和凋亡[7]。CDCA5通过调节线粒体介导的凋亡、细胞周期调节和PI3k/AKT/mTOR通路激活,在膀胱癌中发挥肿瘤启动子的作用[8]。CDCA6和CDCA7的异常表达在前列腺癌、乳腺癌、肾透明细胞癌和食管癌中被发现[9-10]。CDCA8的表达可被KIF18B激活,进而影响胰腺导管腺癌的增殖[11]。以上说明CDCAs基因在肿瘤的发生发展过程中扮演着重要的角色。
本研究利用Oncomine数据库分析CDCAs基因在胃癌中的表达水平,利用GEPIA数据库验证CDCAs基因在胃癌中的表达水平,结合Kaplan-Meier Plotter数据库的临床信息分析CDCA基因家族成员在胃癌中的预后结果;利用STRING数据库构建CDCAs基因的蛋白质互作网络,并得到与CDCA基因家族共表达的基因;利用metascape数据库将CDCAs基因及其共表达基因的功能信息进行整合,研究其潜在的作用机制。以上生物信息学分析结果有望为寻找胃癌有效的预后标志物提供新思路。
1 对象与方法
1.1Oncomine数据库分析 本研究利用Oncomine数据库(https://www.oncomine.org/)分析正常胃组织和胃癌组织。设定筛选条件:(1)gene:CDCA1、CDCA2、CDCA3、CDCA4、CDCA5、CDCA6、CDCA7、CDCA8;(2)fold change >2,P<0.05,gene rank = top 10%。
1.2GEPIA数据库分析 GEPIA数据库(http://gepia.cancer-pku.cn/)可利用TCGA和GTEx直接进行CDCAs基因的表达分析。设置限定条件:|Log2FC|Cutoff: 1,P值 Cutoff: 0.05。
1.3Kaplan-Meier Plotter数据库分析 Kaplan-Meier Plotter(https://kmplot.com/analysis)数据库采用Kaplan-Meier法对多种肿瘤患者的生存资料进行预后分析。筛选条件如下:(1)cancer:gastric cancer;(2)gene symbol:CDCA1、CDCA2、CDCA3、CDCA4、CDCA5、CDCA6、CDCA7、CDCA8;(3)Split patients by: best cutoff;(4)survival:OS。在进行总生存期(overall survival,OS)分析的同时,计算危险比(hazard radio,HR)、95%置信区间(confidence interval,CI)及LongrankP<0.05检验差异,LogrankP<0.05差异有统计学意义。
1.4STRING数据库分析 STRING数据库(https://string-db.org/)是一个在线搜索已知的蛋白质互作关系的数据库,本研究于2020年12月28日访问STRING数据库11.0版。选择Multiple Proteins板块,条件设置为:(1)List Of Names: CDCA1、CDCA2、CDCA3、CDCA4、CDCA5、CDCA6、CDCA7、CDCA8;(2)Organism: Homo sapiens;(3)minimum required interaction score: medium confidence(0.400)。
1.5metascape数据库分析 metascape数据库(http://metascape.org)整合了GO和KEGG等多个数据资源,可完成通路富集和相关基因的蛋白质网络分析等。条件设置为:(1)Multiple Gene List: CDCA1、CDCA2、CDCA3、CDCA4、CDCA5、CDCA6、CDCA7、CDCA8;(2)Input as species and Analysis as species: H.sapiens。最后进行通路富集分析。
2 结果
2.1Oncomine数据库中胃癌患者CDCA基因的表达情况 Oncomine数据库结果显示:CDCA1、CDCA2、CDCA3、CDCA4、CDCA5、CDCA6、CDCA7、CDCA8在胃癌组织中显著升高(图1)。分析三个数据集“Cui Gastric”,“Derrico Gastric”和“Cho Gastric”,得到CDCA基因家族在胃癌组织和正常组织的差异表达分析结果(表1)。
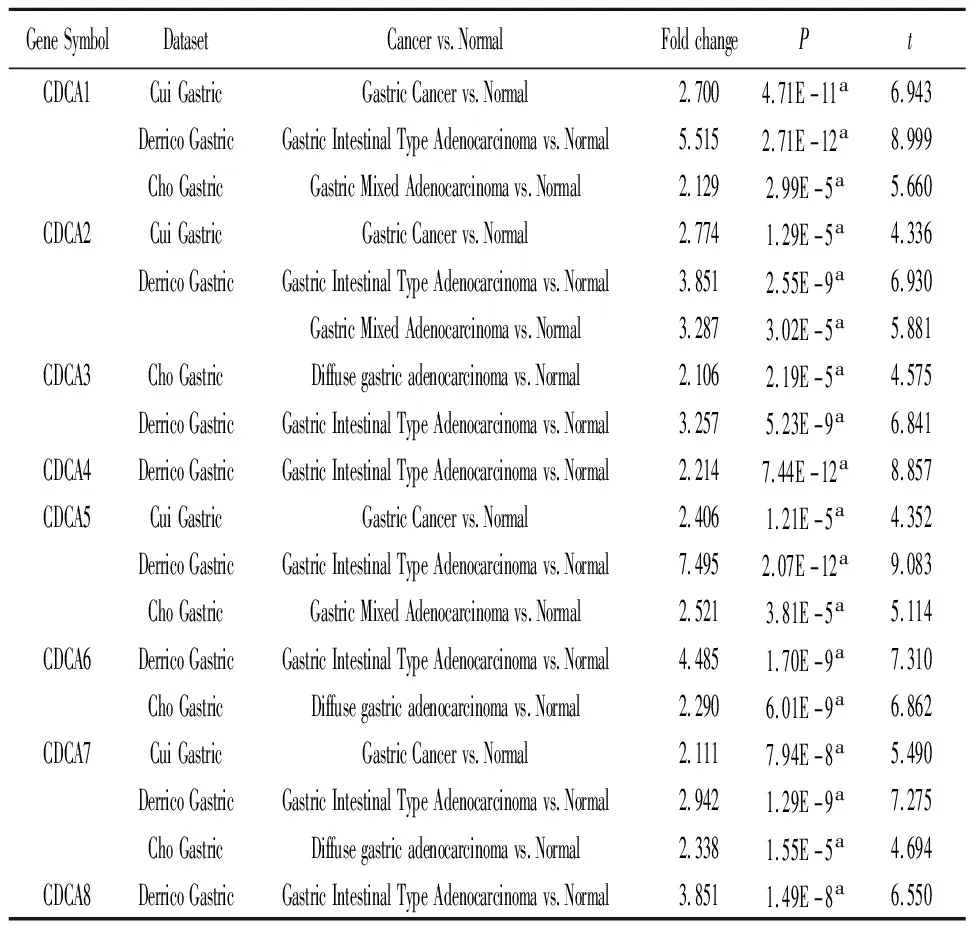
表1 不同类型胃癌与正常肺组织之间CDCA转录水平的表达差异显著
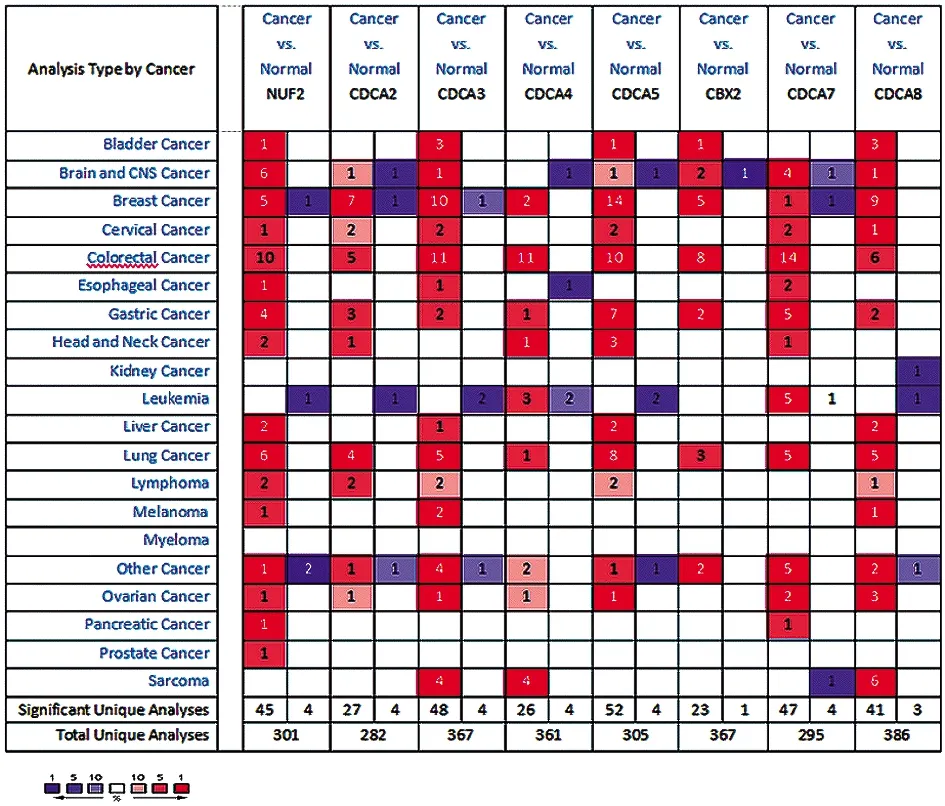
图1 CDCA在不同肿瘤中的转录水平表达
2.2GEPIA数据库中胃癌患者CDCA基因的表达情况 结果显示,CDCA1、CDCA2、CDCA3、CDCA4、CDCA5、CDCA6、CDCA7、CDCA8的表达水平在胃癌组织中相对于在正常组织中上调(肿瘤样本:n=408,正常样本:n=211,P<0.05)(图2)。
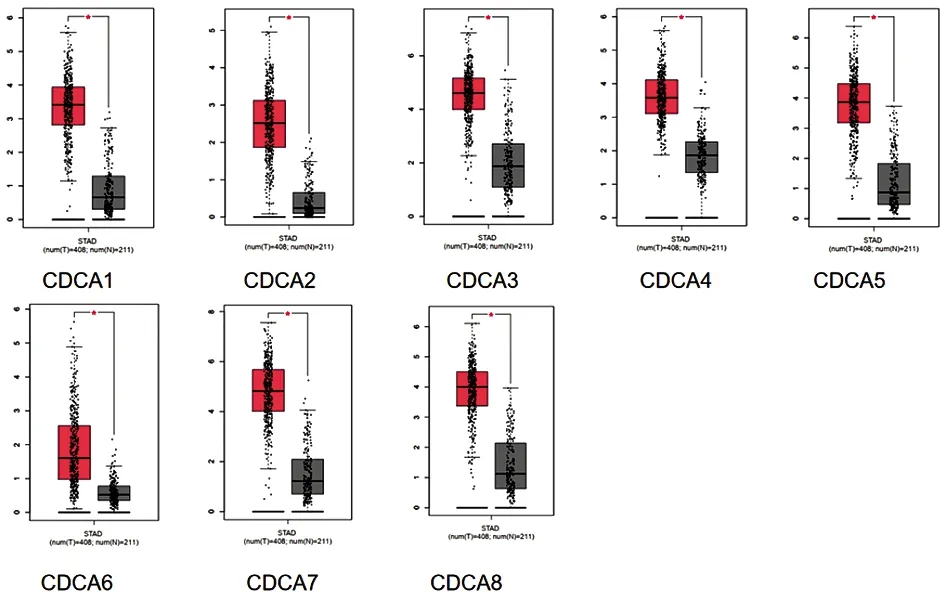
图2 CDCA基因家族在胃癌组织和正常组织中的表达
2.3Kaplan-Meier Plotter数据库中CDCA基因家族在胃癌组织中的表达与患者临床病理资料的生存分析 结果显示,CDCA1、CDCA2、CDCA3、CDA4、CDCA5、CDCA7、CDCA8的表达高低与预后相关。以HER2(+)/(-)和不同临床分期为前提条件进行生存分析,CDCA1、CDCA2、CDCA3、CDCA5、CDCA7、CDCA8基因高表达胃癌患者的总生存期较低表达胃癌患者的总生存期显著延长,而CDCA4基因低表达胃癌患者预后更好(图3)。见表2、3。
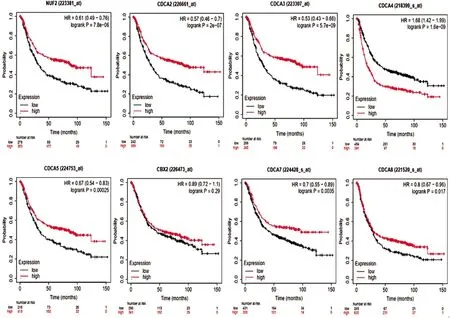
图3 胃癌患者CDCA基因家族的OS分析
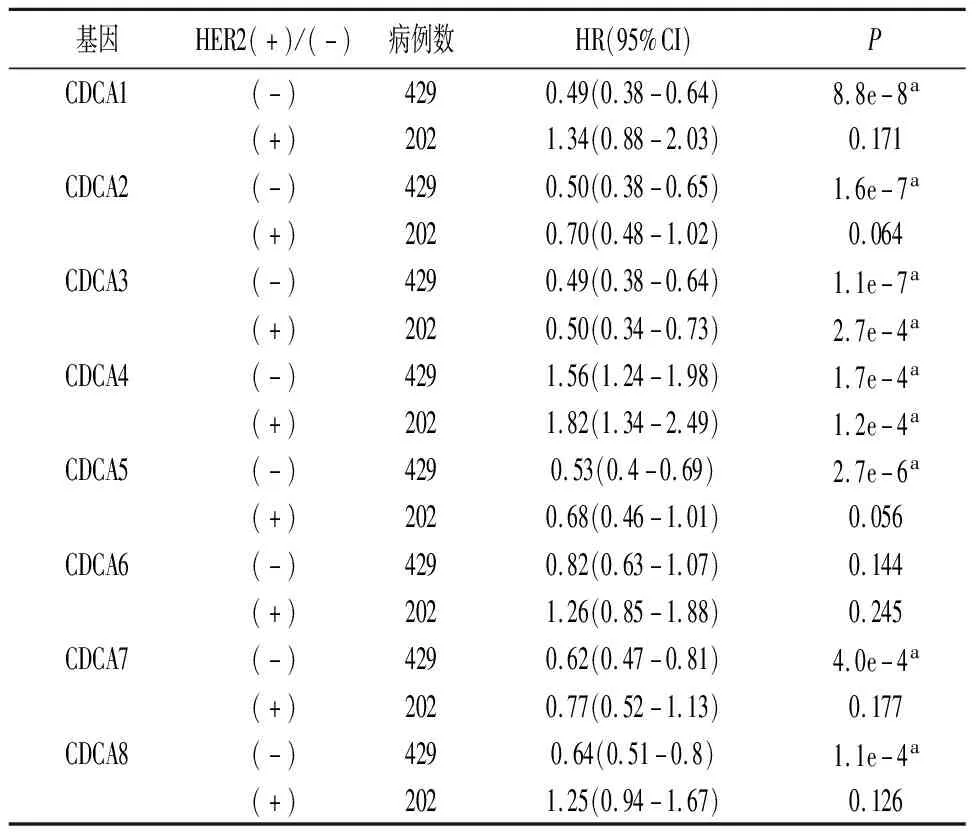
表2 CDCA基因家族表达与胃癌患者HER2(-)/(+)的相关性
2.4STRING数据库中胃癌患者CDCA基因的通路富集分析 结果显示,CDCAs基因在胃癌中呈正相关的共表达基因为:CDCA2、CDCA7、CDCA4、CDCA1(NUF2)、CDCA3、CDCA6(CBX2)、CDCA8、CDCA5、NDC80、SPC25、BIRC5、BUB1、AURKB、CDC20、INCENP、SPC24、CCNB1和CENPE,作用评分均大于0.99(图4a、4b)。

表3 CDCA基因家族表达与胃癌患者分期的相关性
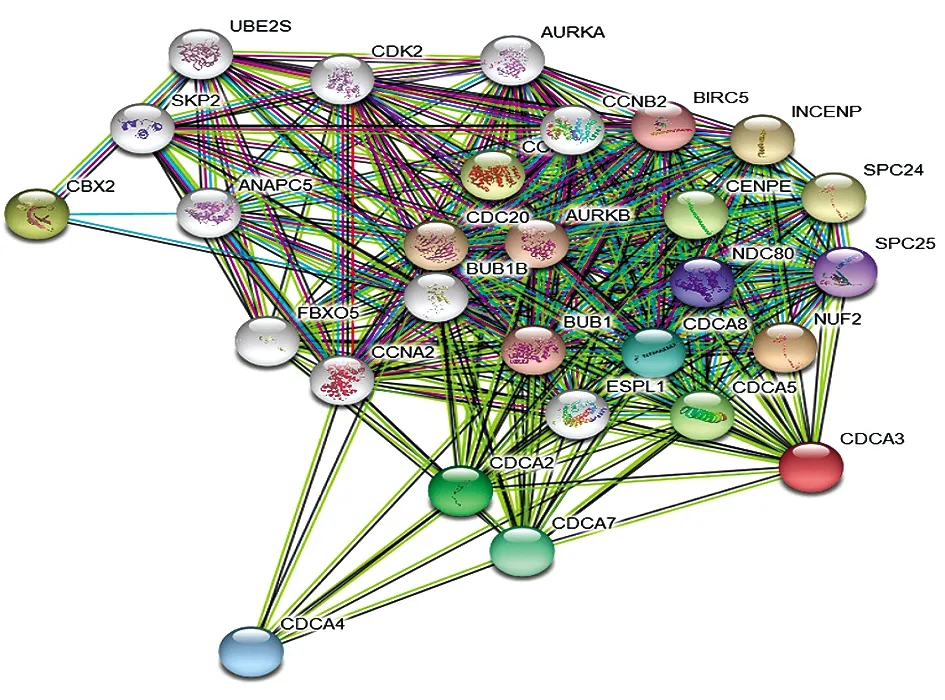
图4a CDCAs基因基于STRING数据库的蛋白质互作分析
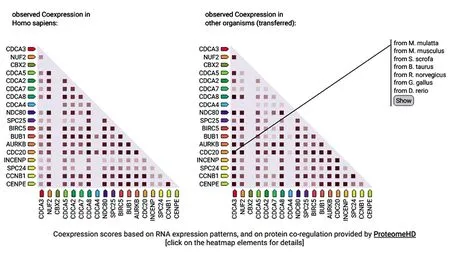
图4b CDCAs基因基于STRING数据库的基因共表达分析
2.5metascape数据库中胃癌患者CDCA基因的功能预测 结果显示,CDCA基因的改变影响了以下几个过程:(1)R-HAS-2500257: 分辨姐妹染色单体粘连;(2)M129: PID PLK1通路;(3)M14: PID AURORA B通路;(4)CORUM:127: NDC80着丝点复合体;(5)GO:0030261: 染色体凝缩;(6)GO:0140013: 减数分裂(图5a、5b和5c)。
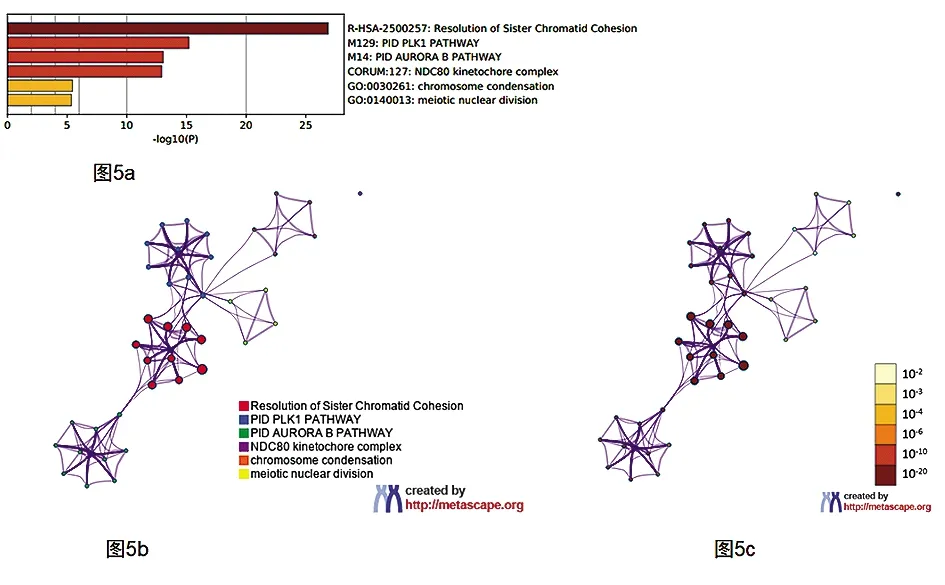
图5 CDCAs基因的功能以及与CDCAs基因改变显著相关的功能
3 讨论
近年来有研究表明CDCA基因家族可能有助于肿瘤的发展[12],但目前较少有胃癌中CDCA基因家族的表达情况和预后分析的相关报道。细胞周期异常调节所介导的细胞转化和不受控制的细胞生长为肿瘤的增殖提供了有利条件,因此肿瘤的发生发展离不开细胞分裂[13]。值得注意的是CDCA基因家族部分家族成员在肿瘤中高表达,具有调节肿瘤细胞周期、促进肿瘤细胞增殖、减少肿瘤细胞凋亡的作用,导致预后不良[14]。本研究利用Oncomine数据库和GEPIA数据库进行分析,均提示CDCA基因家族8个成员的mRNA在胃癌组织中呈高表达,说明CDCA基因家族成员可能在胃癌的发生发展中起着一定的作用。
有报道CDCA基因家族的一些成员可能在乳腺癌、肺腺癌和胰腺癌中上调,这些基因的高表达水平被认为是这些肿瘤生存的不良预测因素[12]。本研究利用Kaplan-Meier Plotter数据库进行生存分析,结果表明除CDCA6基因差异无统计学意义,CDCA基因家族另外7个成员中,CDCA1、CDCA2、CDCA3、CDCA5、CDCA7、CDCA8的高表达与胃癌患者的总生存期延长有关,CDCA4在胃癌中的高表达,与胃癌患者的总生存期减少有关。本研究以HER2(+)/(-)和不同临床分期为前提条件进行分析,CDCA4基因表达下调,CDCA1、CDCA2、CDCA3、CDCA5、CDCA7、CDCA8基因表达部分上调。已有报道胃癌HER2状态和胃癌的预后有一定的相关性[15]。值得注意的是,本研究中以HER2(+)/(-)和不同临床分期为前提条件的结果与胃癌患者总生存期结果相比结果有差异,说明CDCA基因家族基因在胃癌组织中的表达情况可能与HER2通路有关,还需进一步研究证实。另外,本研究CDCA6基因的mRNA在胃癌组织中的表达高于正常组织,但其表达水平对胃癌患者生存期无明显影响,推测可能CDCA6的功能主要作用在于肿瘤的发生,对胃癌患者后期影响较小。
此外,为探讨CDCA基因家族成员影响胃癌预后的机制,本研究利用STRING数据库和metascape数据库进行了KEGG和GO的分析,找出CDCAs基因表达和突变频率最高的连锁基因与胃癌预后的关系,结果是CDCAs基因改变会影响以下途径:(1)R-HAS-2500257: 分辨姐妹染色单体粘连;(2)M129: PID PLK1通路;(3)M14: PID AURORA B通路;(4)CORUM:127: NDC80着丝点复合体;(5)GO:0030261: 染色体凝缩;(6)GO:0140013: 减数分裂。凝缩后的染色体包含两个姊妹染色单体,两单体由着丝粒连接。研究发现,PLK1是有丝分裂激酶信号的主要调节者和协调者,而AURORA B就是有丝分裂激酶成员之一[16]。PLK1通过调节AURORA B的活性确定着丝粒的身份,AURORA B激酶通过磷酸化着丝粒上的NDC80蛋白,调控NDC80寡聚化作用来纠正错误的微管-动粒连接,动粒与微管正确连接的前提是姐妹染色单体的正常粘连[16-18]。本研究推测CDCAs基因及其突变频率最高的连锁基因可能通过调控PID PLK1和PID AURORA B信号通路影响NDC80着丝点复合体,进而干扰动粒与微管的连接影响姐妹染色单体粘连,最终影响染色体的稳定性,使肿瘤细胞的生长失控。因此,对CDCAs基因表达的调控,有望通过调节上述信号通路影响染色体的稳定性,将胃癌细胞阻滞在有丝分裂期,使胃癌细胞发生凋亡,进而影响胃癌的发生发展。
综上所述,本研究利用生物信息学,探讨CDCAs基因的表达水平与胃癌发生发展的关系,提示CDCAs基因表达水平可能与胃癌的预后相关。筛选CDCAs基因在胃癌中的共表达基因然后进行GO和KEGG富集分析,探讨了其可能在胃癌发生发展中的机制。目前,检测CDCAs基因的异常表达可能为胃癌患者早期诊断和预后提供新思路,并可能为胃癌的预后诊疗提供新的方法和新的理论依据。同时,深入研究CDCA基因家族成员在肿瘤的发生发展也为研究其他胃肠道肿瘤提供新的思路和方向。

