CRISPR/Cas系统的核酸诊断技术应用进展
刘晓玫 魏双施★ 于农
作者单位:1.中国科学院苏州生物医学工程技术研究所,中国科学院生物医学检验技术重点实验室,江苏,苏州215163
2.苏州高新区人民医院检验科,江苏,苏州215129
自1987年在大肠杆菌(Escherichia coli,E.coli)中发现规则成簇间隔短回文重复序列(Clustered Regularly Interspaced Short Palindromic Repeats,CRISPR)以来,对生物学,尤其是基因组编辑和基因工程领域的基础研究和临床应用带来了巨大的改变[1]。细菌和古细菌的原核细胞中存在的CRISPR/Cas 系统,通过依赖于指导RNA 的DNA 或RNA 核酸酶活性提供针对外来遗传元件的适应性免疫,保护生物体免受外源核酸的引入[2-4]。CRISPR 基因座由重复序列组成,长度约为20~40 bp,被独特的间隔20~58 bp 的间隔子隔开[5]。CRISPR 基因座的下游为编码Cas 蛋白的序列,而且其中一些蛋白质以前被认为与DNA 修复有关[3]。病毒DNA 或质粒首次进入细胞后,它将被Cas 蛋白降解并插入重复序列之间的特定位点。如果细菌宿主确实具有CRISPR 系统,则这些序列会在细胞中转录生成CRISPR RNA(crRNA),并与Cas 蛋白形成干扰复合物。在crRNA的指导下,该复合物可指导crRNA 与病毒DNA 之间氢键的形成,从而催化外源DNA的分裂[5]。
随着研究的逐步深入,人们对CRISPR 和CRISPR 相关(Cas)蛋白的结构以及功能有了更深的了解,发现具有核酸酶、解旋酶和聚合酶等多种活性的不同类型的Cas 蛋白。根据最新的分类,CRISPR/Cas 系统被分为两个主要的大类、六种型以及48 个亚型。其中第一类的Cas 蛋白为多亚基效应复合物,包括Ⅰ型的Cas3、Ⅲ型的Cas10、Ⅳ型的Csf1;第二类的Cas 蛋白为单亚基效应蛋白,包括Ⅱ型的Cas9、Ⅴ型的Cas12 和Ⅵ型Cas13 等[6-7]。
CRISPR 系统因具有有效的核酸识别和编辑能力的固有优势,已被证明是核酸检测的有力工具。此篇综述简要总结CRISPR/Cas 系统在检测中的几种常用酶,及其在传染病病原及肿瘤核酸诊断中的应用进展,旨在为该领域研究人员提供参考。
1 核酸检测中的Cas 蛋白
1.1 Cas9
CRISPR/Cas9 的工作原理是,Cas9 与引导RNA(guide RNA,gRNA)协同工作。gRNA 包含一个CRISPR RNA(crRNA),其3′端与tracrRNA 配对,crRNA 的5′游离端是DNA 的靶序列,Cas9 抓住tracrRNA,形成二元复合体。这使Cas9 可以将crRNA 靶序列引导至DNA 分子上的互补区(前间隔序列)。crRNA 与前间隔序列的配对将使匹配的第二条DNA 链分开,并形成Cas9-gRNA-前间隔序列三元复合物。取决于前间隔序列和前间隔序列相邻基序(protospacer adjacent motif,PAM)的存在,crRNA 目标序列可指导Cas9 切割前间隔序列中两条DNA 链(图1A)。为了方便进行基因组编辑,crRNA 和tracrRNA 被设计为单个嵌合体,称为单向导RNA(single guide RNA,sgRNA)[8-9]。2016年,Keith Pardee 等[10]首次报道了一种基于CRISPR/Cas9的序列特异性核酸酶活性的新技术,应用于寨卡病毒检测。作者利用依赖核酸序列的扩增技术(Nuclear Acid Sequence Based Amplification,NASBA),扩增过程的中间产物双链DNA 作为Cas9 核酸内切酶的底物,如果Cas9 切割了该dsDNA 中间体,则体外转录产生截短的RNA;而非目标分子中间体不被切割产生完整的RNA,从而激活Toehold 反应。靶标RNA 的检测通过纸盘上的颜色变化来指示(图2A)。该技术可实现区分具有单碱基差异的寨卡病毒毒株,例如非洲和美国Zika 变体,而区分不同的Zika变体可用于基因分型和确定感染的起源。
随后,研究人员相继开发了CRISPR/Cas9 与指数扩增反应(Exponential amplification reaction,EXPAR)和滚环扩增(Rolling circle amplification,RCA)相结合的高度敏感和特异性的核酸检测器。Huang及其同事将CRISPR/Cas9 切割与EXPAR 结合在一起,提出了一种称为CAS-EXPAR 的单链核酸生物传感方法[11]。在PAM 呈递寡核苷酸(PAM-presenting oligonucleotide,PAMmer)的协助下,将ssDNA 或RNA 在特定位点切割,以释放短片段,这些片段起引物作用以启动EXPAR 过程,从而产生丰富的ds-和ssDNA 扩增子。EXPAR强大的指数扩增能力使CAS-EXPAR 的分析灵敏度达到了0.82 aM。就特异性而言,CAS-EXPAR可以区分恰好位于切割位点的单碱基错配,因为该错配会阻碍EXPAR 的引物延伸,从而消除读数。CAS-EXPAR 技术已成功应用于单核细胞增生李斯特菌mRNA 的检测。
1.2 Cas13
Cas13 是一种RNA 引导的核糖核酸酶(RNase)。Cas13a 在相应的crRNA 的引导下,特异性切割带有互补间隔序列的单链RNA(ssRNA)。而且,结合到ssRNA 靶标后,Cas13a 蛋白被激活并转化为非特异性内切酶,对附近的非靶标ssRNA具有附带的裂解活性(图1C)[12-13]。2016年,East-Seletsky及其同事首次尝试使用LbuCas13a 检测噬菌体λRNA 和内源性β-肌动蛋白mRNA。Cas13a被ssRNA 靶标激活,进而降解了荧光淬灭剂标记的ssRNA(ssRNA-FQ)报告基因,从而增加了荧光强度,并实现了检出限(Limit of detection,LOD)低至1-10 pM 的快速检测[12]。
2017年,张锋等[14]报道了基于Cas13a 的“SHERLOCK”核酸诊断工具。首先,将重组酶聚合酶扩增(Recombinase Polymerase Amplification,RPA)或RT-RPA 用作预扩增,然后进行T7 体外转录,这会产生大量RNA 扩增子,这些扩增子随后触发ssRNA-FQ 报告基因的Cas13 侧链切割,从而产生荧光读数(图2B)。整个过程在37℃条件下的1~3 h 内实现了阿摩尔(attomolar,aM 或10-18M)浓度的RNA 和DNA 检测。值得关注的是,将该技术试剂制成冻干滤纸可用于即时检测(point-ofcare testing,POCT),同时保持了阿摩尔的灵敏性,而且成本低廉,只需0.61 美元/反应。随后,该团队推出了名为SHERLOCKv2 的二代技术,它增加了四点优势:①可实现单管四重检测;②通过调节RPA 引物来控制指数预扩增的进程,从而在低至阿摩尔水平的各种浓度范围内进行定量;③由Cas13a 侧链切割触发的Csm6 核酸酶活性的级联激活提高敏感性;④90 分钟内通过肉眼读数灵敏度可达2 aM。总的来说,SHERLOCK 和先进的SHERLOCKv2 是具有aM 级的核酸检测灵敏度和单碱基特异性的多功能、快速、便携式且经济高效的核酸检测技术,这对于POCT、流行病监测和病原体检测非常需要,尤其适用于基础设施比较不完善的欠发达地区。
1.3 Cas12
2 类V-A 型的Cas12a 系统(以前称为Cpf1),是由有序的cas12a-cas4-cas1-cas2-CRISPR 阵列组成。Cas12a蛋白含有RuvC 内切核酸酶结构域,可识别富含T 的PAM 序列以进行靶标切割[15]。此外,Cas12,特别是LbCas12a,与其目标DNA 的结合释放了非特异性的ssDNA 切割活性(图1B)。DETECTR技术(DNA Endonuclease Targeted CRISPR Trans Reporter)和HOLMES 技术(one-HOur Low-cost Multipurpose highly Efficient System)正是基于Cas12的这种靶向激活的附带ssDNA 切割活性而开发的[15]。其工作原理是通过RPA 或RT-RPA 等温扩增靶核酸,然后结合Cas12a-sgRNA 复合物并触发ssDNA荧光猝灭剂报告ssDNA 的裂解,从而产生荧光信号。见图2C。

图2 基于CRISPR/Cas 的核酸检测工具[23]Figure 2 Nucleic acid detection tool based on CRISPR/Cas
1.4 Cas14
Cas14 是一种很小的Cas 蛋白,大小仅为其他Cas 蛋白的一半。Cas14 蛋白能够进行靶向单链DNA(ssDNA)切割,而无需PAM 进行激活(图1D)。此外,Cas14 识别靶标会触发非特异性ssDNA分子裂解活性,类似于含RuvC 的酶Cas12a[18]。Cas14a 也可应用到DETECTR 平台中,以生成一个新的ssDNA 检测系统Cas14a-DETECTR。它可以在没有PAM 限制的情况下进行高保真DNA 单核苷酸多态性(SNP)以及病毒DNA 检测[19]。
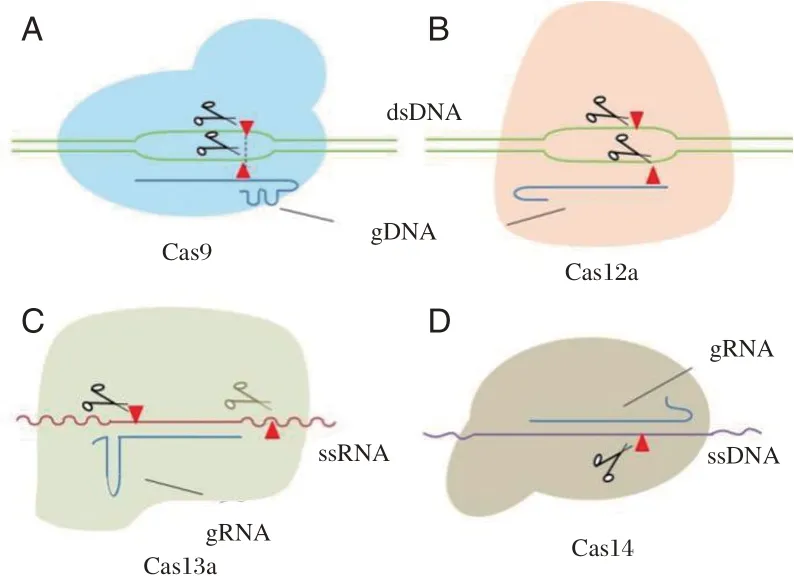
图1 CRISPR/Cas 核酸识别示意图[16-17]Figure 1 Schematic diagram of CRISPR/Cas nucleic acid recognition
2 CRISPR/Cas 核酸检测系统的应用
2.1 病原检测
2.1.1 人乳头瘤病毒检测
人乳头瘤病毒(Human papillomavirus,HPV)是一种Papillomaviridae 家族的DNA 病毒,可引起某些常见的人类癌症,尤其是宫颈癌,而宫颈癌是全世界女性第二大最常见的癌症死亡原因。准确地鉴定HPV 基因型对于确定那些处于HPV 相关癌症风险中的人至关重要。最近报道了三种CRISPR-分型PCR(CRISPR-typing PCR,ctPCR)的方法。其原理是通过一对Cas9-sgRNA 复合物切割靶DNA,然后将切割的DNA 连接形成线性或环状分子,再通过PCR 或qPCR 扩增检测[20-22]。除Cas9 系统外,前文也提到了基于“附带”裂解活性的Cas12a 的DETECTR 检测平台也可用于HPV 检测[15]。
2.1.2 SARS-CoV-2(severe acute respiratory syndrome coronavirus-2)检测
当前由SARS-CoV-2 引起了(新冠肺炎)(COVID-19)全球大流行。建立快速、准确、经济的SARS-CoV-2 诊断对于疾病的早期治疗和控制至关重要。研究者们基于CRISPR 的诊断原理,利用Cas12、Cas13 识别切割特性开发出一系列快速、精准的方法来检测SARS-CoV-2。此外,Moon 等[24]报道了一种基于CRISPR/dCas9 系统的病毒诊断方法,并成功检测出SARS-CoV-2、pH1N1 和pH1N1/H275Y 突变病毒。
2.1.3 结核分枝杆菌(Mtb)检测
结核病主要由结核分枝杆菌(Mycobacterium tuberculosis,M.tb)感染引起的传染病,是一项严重的全球健康挑战,预防和控制结核病的关键是开发低成本、快速且可靠的诊断方法,尤其在资源匮乏的地区及早诊断结核病[25]。Zhang 等[26]将dCas9分别连接到一对萤火虫荧光素酶(NFluc 和CFluc)的N 端和C 端,在sgRNA 的指导下,分别与靶DNA序列的上游和下游片段互补。当两个相邻片段的底物DNA 被一对dCas9 结合时,荧光素酶的整体催化活性就会产生荧光。国内的张文宏团队建立了一种基于Cas12a 的Mtb 快速精准检测系统[27]。
2.2 癌症诊断
监测肿瘤来源的细胞外囊泡(Tumor-derived EVs,TEVs)并检测其内含的生物标志物,可能是在“液体活检”中筛查癌症的一种有前途的方法。Li 等通过将特异性结合TEV 上蛋白质靶标的适配体,与基于PCR 的指数扩增和CRISPR/Cas12a 实时DNA 检测相结合,开发了一种称为aptamer-CRISPR/Cas12a 检测的新技术,可快速、方便地检测血清中超低浓度的CD109+和EGFR+TEV,可用于鼻咽癌的诊断和预后评价[28]。
MicroRNA(miRNA)作为癌症早期临床诊断的潜在生物标记已被广泛地研究[29]。Zhou 等[30]构建了一个由CRISPR/Cas13a 驱动的便携式电化学发光(Electrochemiluminescence,ECL)芯片(PECL-CRISPR)。其中靶标miRNA 激活Cas13a 以裂解系统中设计的预引物,并触发随后的指数扩增和ECL 检测,miR-17 的检测限达到1×10-15M。
3 小结与展望
基于CRISPR 的核酸检测系统经过几年的发展,已经显示出其巨大的优势,比如可以提供快速、灵敏、特异性、低成本以及现场即时定量分析,并且可以实现多重化检测。该技术已被用于医学诊断的多个领域,例如,它们可以用于确定病原体的基因型,从而实现量身定制的治疗。它们还可以用于检测人、动物、植物或环境病原体。此外,还可用于植物病原体的鉴定和基因型检测以指导农药施用的数量和类型;关键病原体的检测来快速评估水质或评估生物恐怖威胁;食品中的病原体检测以预防食源性疾病等。随着当前技术的应用发展以及新型功能强大的Cas 直系同源物的不断开发,相信CRISPR/Cas 系统将成为核酸检测不可替代的重要工具。

