基于9种RNA的肾透明细胞癌预后模型建立与验证
管波 单卫民
肾细胞癌是起源于肾实质泌尿小管上皮系统的恶性肿瘤,又称肾腺癌,简称为肾癌。在过去的20年里,肾癌的发病率增加了约2%[1],占肾脏恶性肿瘤的80%~90%[2]。男性为女性的1.5倍,发病高峰在60~70岁[3]。2016年WHO根据肾癌起源以及基因改变等特点将其分为肾透明细胞癌(clear cell renal cell carcinoma, ccRCC)(60%~85%)、乳头状肾细胞癌(7%~14%)、嫌色细胞癌(4%~10%)、集合管癌(1%~2%)和其他罕见类型等[4]。ccRCC是肾癌最常见的病理类型,界限清楚,通常无包膜,切面呈黄色,常伴有出血和坏死。染色体3p缺失和von Hippel-Lindau(VHL)基因在3p25号染色体上的突变是最常见基因改变,其他的肿瘤抑制基因包括SETD2、BAP1和PBRM1等[5]。与乳头状肾细胞癌和肾嫌色细胞癌相比,ccRCC的预后更差[6]。癌症基因组图谱(The Cancer Genome Atlas, TCGA)是美国政府发起的癌症和肿瘤基因图谱计划,采用大规模的基因组测序,旨在找到所有致癌和抑癌基因的微小变异,了解癌细胞发生、发展的机制。分子标签是指将一定数量的分子标志物进行组合,形成一个新的变量,并用此变量来判断或定义某些生物学特性。与单个标志物的分子模式不同,分子标签不单纯以单基因功能为研究基础,而是更加注重基因之间的共同协调作用,从整体和系统水平上对某种特定的生物学特性进行描述。
本研究利用TCGA高通量数据库数据,通过对数据再分析构建新的ccRCC预后分子标签,希望能用于临床判断ccRCC预后,并为ccRCC的病因及发病机制研究提供新的思路。
对象与方法
一、研究对象
从TCGA(https://www.cancer.gov)下载ccRCC 611例RNA-Seq测序数据(其中正常样本数据72例,肿瘤样本539例)和530例患者的临床数据,如患者性别、年龄、种族、病理分期、吸烟状况、放疗及化疗情况、存活状况等。
二、研究方法
1.ccRCC组织与正常肾组织差异分析:利用R语言(版本4.0.0)分析ccRCC组织与正常肾组织差异分析RNA,本研究确定差异表达的标准为校正后伪发现率(false discovery rate, FDR)<0.05且倍数变化绝对值≥2。对筛选出的差异表达的RNA进行聚类分析,并绘制热图和火山图。
2.ccRCC预后标志物的筛选:采用Lasso回归筛选变量,并利用Cox回归构建预后分子标签。评分风险(Risk score)=βgene1×EXPgene1+βgene2×EXPgene2+……+βgene11×EXPgene11(EXP:基因的表达水平;β:回归相关系数)。代入相关数据后,计算分子标签得分。通过RNA分子标签得分的中位数将ccRCC患者划分为高表达组和低表达组。
3.联合ccRCC患者临床数据构建列线图模型:将分子标签联合TCGA数据库中ccRCC患者的临床数据(包括性别、年龄、种族、病理分期、吸烟状况、放疗及化疗情况)进行单因素和多因素Cox回归分析,根据Cox回归模型中各个参数对总生存期的贡献程度(回归系数的大小),给每个参数的每个取值水平进行赋分,将每个变量和各自的协变量的分值以一簇平行线段的形式绘制在平面坐标系中,构建ccRCC预后列线图模型。
结 果
一、纳入对象的基本情况
病例总数为530例,其中男344例(64.91%),女186例(35.09%);死亡177例(33.40%),存活353例(66.60%)。患者基本特征见表1。

表1 TCGA数据库中ccRCC患者基本特征[例(%)]
二、ccRCC组织和正常肾组织差异表达基因
从TCGA下载ccRCC 611例RNA-Seq测序数据,其中正常样本数据72例,肿瘤样本539例(部分患者有多个肿瘤样本数据,同一患者的数据取平均值处理),差异分析得到ccRCC和正常肾组织中差异表达的RNA 5 759种,其中在肿瘤组织中上调表达的基因有4 492种,下调表达的基因有1 267种。对筛选出的差异表达的RNA进行聚类分析,并绘制热图和火山图(图1)。
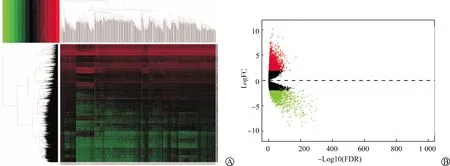
图1 ccRCC组织和正常肾组织中5 759种差异表达基因的热图(A)及火山图(B)
三、ccRCC预后标志物的筛选及分子标签构建
将筛选出的5 759种在ccRCC和正常肾组织中差异表达的RNA结合生存数据进行单因素Cox分析,得到1 610种与ccRCC预后相关的基因(P<0.001)。
将530例肿瘤组织样本ccRCC转录组数据按30%和70%随机分为两组,70%组为训练组,用于筛选预后分子标签;30%组为测试组,用于测试筛选出的分子标签预测预后效果。
将单因素回归分析筛选出的1 610种与ccRCC预后相关的基因进一步做Lasso回归分析(图2),筛选出20种RNA,包括AC011700.1、AC064847.1、AC091153.2、AC091812.1、AC104958.1、AC245100.6、ADAMTS14、AL133255.1、AL355796.1、AL592494.1、DYNLL1P4、FIRRE、KCNMB2-AS1、LINC00896、LINC01956、PLG、RDH16、SNORA70B、Z99289.2、ZIC2。再进行多因素Cox分析,最终从中筛选出9种与ccRCC预后相关的基因(图3,表2),分别为 DYNLL1P4、LINC01956、PLG、SNORA70B、ZIC2、AC011700.1、AC091812.1、AC104958.1、AL133255.1。利用9种基因联合回归系数构建分子标签,Risk score=βgene1×EXPgene1+βgene2×EXPgene2+……+βgene11×EXPgene11(EXP:基因的表达水平;β:回归相关系数)。代入相关数据后,计算分子标签值。
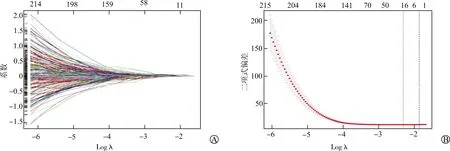
A:不同RNA系数变化情况;B:二项式偏差的曲线图图2 Lasso回归筛选ccRCC预后相关分子
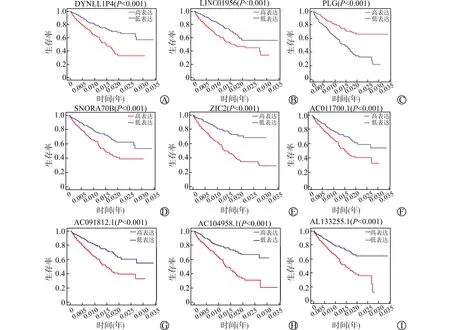
A:DYNLL1P4生存曲线;B:LINC01956生存曲线;C:PLG生存曲线;D:SNORA70B生存曲线;E:ZIC2生存曲线;F:AC011700.1生存曲线;G:AC091812.1生存曲线;H:AC104958.1生存曲线;I:AL133255.1生存曲线图3 分子标签中9种RNA生存曲线
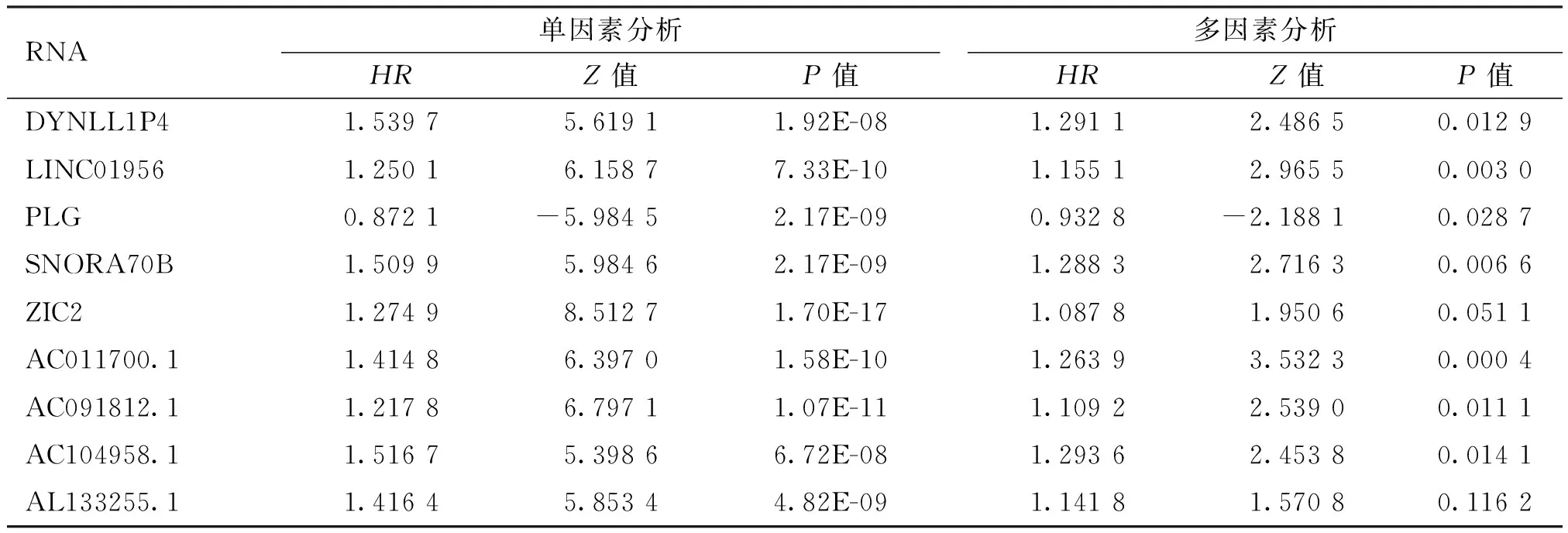
表2 筛选出的9种ccRCC预后基因单变量及多变量Cox分析结果
分子标签表达式为:Risk score=0.255 5×EXPDYNLL1P4+0.144 2×EXPLINC01956-0.069 5×EXPPLG+0.253 4×EXPSNORA70B+0.084 2×EXPZIC2+0.234 2×EXPAC011700.1+0.103 6×EXPAC091812.1+0.257 5×EXPAC104958.1+0.132 6×EXPAL133255.1。
代入相关数据后,计算分子标签分值。通过分子标签得分的中位数将患者划分为高分组和低分组,结合TCGA中患者生存时间及生存状态在训练组和测试组分别绘制生存曲线,并比较高分组和低分组生存曲线差异有无统计学意义(图4),如图所示分子标签值在训练组、测试组、全部数据组与ccRCC患者生存期显著相关,分子标签值越高患者预后越差;并以1年、3年、5年生存期绘制受试者工作特征(receiver operating characteristic, ROC)曲线(图4),其中全部数据组1年、3年、5年ROC曲线下面积分别为0.802、0.758、0.805。

A:训练组生存曲线;B:测试组生存曲线;C:全部数据生存曲线;D:训练组1年、3年、5年ROC曲线;E:测试组1年、3年、5年ROC曲线;F:全部数据1年、3年、5年ROC曲线图4 分子标签的生存曲线及ROC曲线
四、结合临床参数构建ccRCC预后列线图模型
纳入参数包括分子标签、性别、年龄、种族、病理分期、肿瘤分级、吸烟状况和放疗、化疗情况,单因素Cox回归分析发现分子标签、年龄、化疗情况、病理分期、肿瘤分级、吸烟为ccRCC的有统计学意义的预后因素,将单因素分析P<0.05的临床参数进行多因素Cox分析,最终模型保留的参数有分子标签、年龄、病理分期、肿瘤分级、是否化疗为预后因素,整体模型的P<2.2e-16,各参数P值见表3,根据多因素Cox回归模型中分子标签、年龄、病理分期、肿瘤分级、是否化疗对总生存期的贡献程度(回归系数的大小),给每个参数的每个取值水平进行赋分,将每个变量和各自的协变量的分值以一簇平行线段的形式绘制在平面坐标系中,构建ccRCC预后列线图模型,并绘制校准图评价模型(图5)。
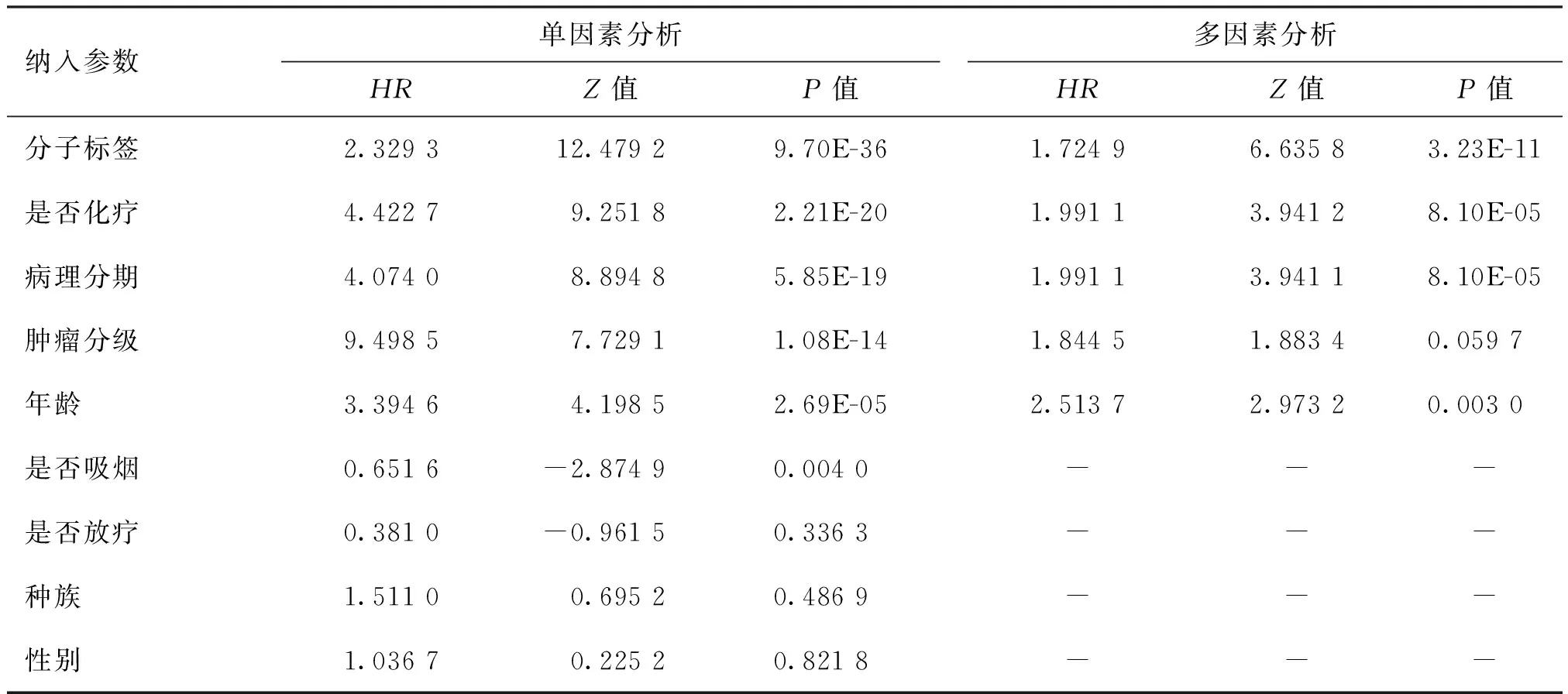
表3 分子标签及临床参数单变量及多变量Cox分析结果
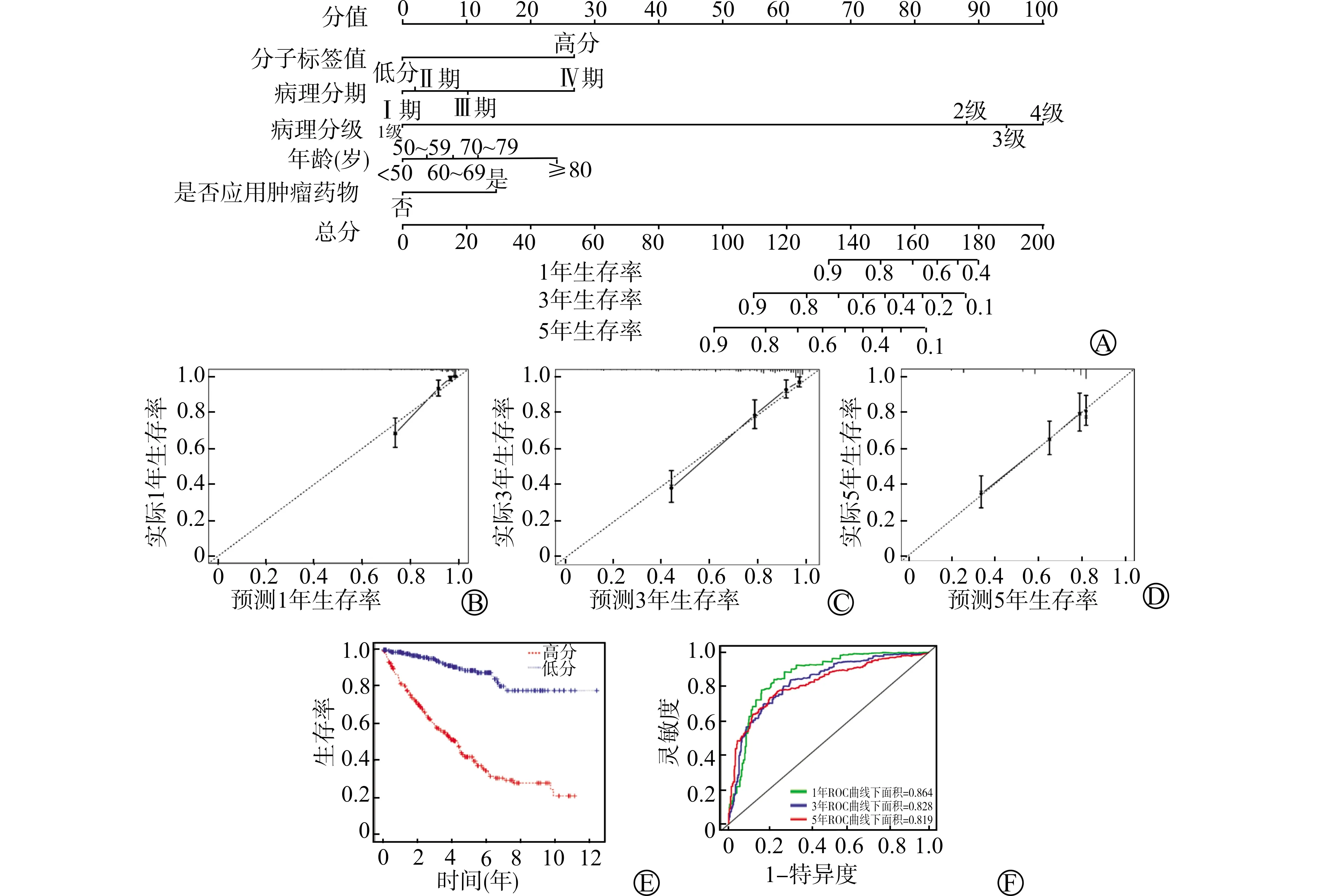
A:ccRCC预后列线图;B、C、D分别为1年、3年、5年生存的校准图;E、F分别为列线图生存曲线和ROC曲线图5 ccRCC预后列线图
讨 论
肾癌为泌尿系统三大恶性肿瘤之一,ccRCC是肾癌最常见的亚型,在发生转移的肾癌中占80%~85%,其对传统的放疗和化疗均不敏感[7]。晚期ccRCC的治疗主要依赖生物治疗,但效果有限。临床上需要ccRCC预后标志物,用以判断其预后,给予患者更早期的有效干预。
二代测序技术作为21世纪的重大科学技术进步之一,为肿瘤基因组学研究提供了极大的帮助,随着肿瘤基因组数据库和患者样本信息的不断丰富,科研人员对肿瘤基因的分析日趋深入,新的快速检测方法也不断更新,使二代测序等技术应用于临床成为可能[8]。
本研究通过提取TCGA数据库中ccRCC患者临床数据以及ccRCC转录组数据,采用Lasso-Cox回归分析结合生存数据筛选其预后相关的转录组数据,并构建ccRCC预后分子标签。首先筛选出530例ccRCC和正常肾组织中差异表达的RNA 5 759种,差异基因结合ccRCC生存数据经过单因素Cox分析得到1 610个与ccRCC预后相关RNA(P<0.001);再将530例肿瘤组织样本数据按30%和70%随机分为两组,70%组为训练组,用于筛选预后分子标签;30%组为测试组,用于测试筛选出的分子标签预测预后效果。采用Lasso-Cox回归分析从中筛选出9种与ccRCC预后相关的RNA,分别为DYNLL1P4、LINC01956、PLG、SNORA70B、ZIC2、AC011700.1、AC091812.1、AC104958.1、AL133255.1,并联合这9种RNA构建了ccRCC预后分子标签,通过该分子标签判断ccRCC预后效果显著。
本研究尚存在一定局限性。第一,未对筛选出的RNA进行相关机制的研究,这些RNA是如何影响ccRCC的生长和凋亡,进而影响其预后的机制仍有待研究;第二,未验证构建的RNA标签能否预测中国ccRCC患者的预后,这些问题均需要后续研究进一步探讨。
综上所述,本研究利用TCGA数据库的挖掘,筛选并构建了新的ccRCC预后分子标签,整合成组合标志物后有望用于临床ccRCC预后预测,为其病因、发病机制、治疗、预后判断提供新的研究思路和方向。

