基于生物信息学途径食管鳞癌lncRNA预后风险模型的构建
蔡河源,谢春莹,邹健勇,罗红鹤
(中山大学附属第一医院胸外科,广东广州510080)
食管癌是全球最常见肿瘤之一,最新全球癌症统计,其发病率占第11位,而死亡率高居第6位[1]。食管癌按照病理类型分为食管鳞癌(esophageal squamous cell carcinoma,ESCC)和食管腺癌(esoph⁃ageal adenocacinoma,EAC)。在我国,ESCC占食管癌的90%左右,且90%病理分期为中晚期[2],预后非常差,癌症死亡率居第4位[3],5年整体生存率不到30%[4-6],亟需寻找新的早期诊断及治疗方法。长链非编码RNA(long non-coding RNA,lncRNA)是指长度大于200 bp,且转录后不编码翻译产生蛋白质的一类RNA,参与并调节人体70%的基因的复制,转录,翻译等细胞生物学过程,从而调节机体各 项 生 命 活 动[7-10]。Linc02471,UCA1、CASC9、DLEU1等,lncRNA通过调控转录翻译促进ESCC的发生发展,并可以作为ESCC的诊断治疗潜在靶点[11-14];但lncRNA与ESCC预后关系尚不明确。本研究基于癌症基因组图谱(the cancer genome at⁃las,TCGA)数据库食管癌转录组基因表达谱,分析ESCC组织与正常食管上皮组织lncRNA表达差异,并构建ESCC预后风险模型,旨在预测ESCC患者预后,寻找ESCC潜在生物标记物,指导ESCC临床诊治。
1 材料与方法
1.1 数据下载整理
从TCGA数 据 库(https://cancergenome.nih.gov/)下载食管癌基因表达数据及临床资料。剔除病理类型为腺癌的病例,剔除生存时间小于30 d的病例,数据集包含基因转录表达数据样本90例,80例为ESCC组织样本,10例为癌旁正常组织样本,进一步提取数据得到食管鳞癌lncRNA表达矩阵。数据采集时间为2021年1月16日。
1.2 差异基因筛选
采用R 4.0.3软件limma包对lncRNA表达矩阵进行差异基因筛选,并设定错误发现率(false dis⁃covery rate,FDR)<0.05和log2|fold change|>1作为临界值,得到ESCC与正常食管组织表达差异的lncRNA。并使用pheatmap包和ggplot2包绘制差异基因热图及火山图。
1.3 预测模型的构建
合并ESCC样本临床生存资料及差异lncRNA表达数据,使用R 4.0.3软件survival包进行单因素COX及多因素COX回归分析,α=0.05。选取单因素COX及多因素COX分析均有统计学差异的ln⁃cRNA构建lncRNA风险预测模型。
模型构建公式为:

其中N,expi,coefi分别代表模型lncRNA,ln⁃cRNA表达量和表达系数。
计算80例ESCC样本的Riskscore,并按中位数排列由低到高将80例ESCC样本分为低风险组及高风险组,进一步使用Kaplan-Meier生存曲线分析高低风险组生存预后差异。进一步使用pheatmap包绘制高低风险组生存状态分布图及模型lncRNA表达热图,使用survival ROC包绘制ESCC样本1年、2年和3年总体生存率ROC曲线图。
1.4 临床特征与Riskscore模型预测性能比较
使用R 4.0.3软件survival包对ESCC临床特征及Riskscore分别进行单因素COX及多因素COX回归分析,探索ESCC独立预后影响因素,并绘制ROC曲线比较不同临床特征和风险模型的预测性能。
1.5 临床特征与Riskscore模型评分相关性分析
分别提取ESCC样本临床特征如性别、年龄、肿瘤TMN分期与对应模型Riskscore评分合并,使用ggpubr包wilcoxon秩和检验进行相关性分析,α=0.05,并绘制相关性箱式图。
1.6 高低风险组基因表达矩阵主成分分析
采用主成分分析(Principal component analy⁃sis,PCA)分别对ESCC样本lncRNA表达矩阵及模型lncRNA矩阵进行降维分析,并使用scatterplot3d包绘制高低风险组3D散点图。
1.7 GO,KEGG功能富集分析
通过基因富集分析(Geneset enrichment analy⁃sis,GSEA)对高低风险组进一步行基因本体(gene ontology,GO),京都基因与基因组百科全书(kyoto encyclopedia of genes and genomes,KEGG)功能富集分析,探索高低风险组在不同细胞通路基因富集的差异性。从(http://www.gsea-msigdb.org/)下载GSEA软件,GO基因集,KEGG基因集,分析并绘制基因富集图。
2 结果
2.1 食管鳞癌样本临床特征
90例食管上皮测序样本纳入分析,80例为ES⁃CC组织样本,10例为癌旁正常组织样本。80例ESCC组织样本中,男性68人,女性12人,平均年龄58.43(30~90)岁,肿瘤分期以Ⅱ,Ⅲ期多见,T分期中T2-3占85%,N分期中N0期最为高发,N1次之,且87.5%ESCC患者未见远处转移,其他临床特征见表1。

表1 80例食管鳞癌组织样本临床特征Table 1 Clinical characteristics of 80 cases of esoph⁃ageal squamous cell carcinoma [n(%)]
2.2 差异表达的lnc RNA筛选结果
80例ESCC组织样本,10例癌旁正常组织样本经筛选得到差异lncRNA 174个,其中ESCC样本表达上调lncRNA 126个,表达下调lncRNA 48个(图1)。

图1 差异表达lncRNA热图及火山图Fig.1 Heat map and volcano plot of differentially expressed lncRNA
2.3 差异lnc RNA单因素及多因素COX回归分析
结果显示单因素COX回归有3个lncRNA有统计学意义(P<0.05),多因素COX回归分析有2个lncRNA有统计学意义(P<0.05),AC108449.2和AL033384.1可作为ESCC的独立预后因素,AC108449.2的风险比为0.218,是ESCC预后的保护因素,AL033384.1的风险比为3.681,是ESCC预后的危险因素(表2、3)。

表2 差异表达lncRNA单因素COX回归分析Table 2 Univariate Cox regression analysis of differentially expressed lncRNA
2.4 Lnc RNA风险模型构建及高低风险组生存分析
经单因素及多因素COX回归分析得到模型算式Riskscore=1.303×AL033384.1-1.525×AC108449.2,Riskscore中位值为1.025,根据中位值由低到高排列将ESCC样本分为低风险组和高风险组。生存状态图显示相较于高风险组,低风险组患者存活数更多,死亡患者存活时间更长,且低风险组AL033384.1低表达,AC108449.2高表达,而高风险组相反,结果见图2。进一步Kaplan-Meier生存分析显示低风险组1年,3年,5年生存率均高于高风险组(P<0.001),预后较好(图3)。Riskscore模型时间依赖性ROC曲线图显示该模型预测ESCC患者1年、2年和3年总体生存率效能分别为0.750、0.768和0.796,具有良好的预测性能(图4A)。
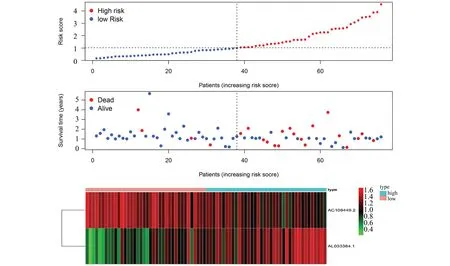
图2 高低风险组生存状态分布及lncRNA表达Fig.2 Survival-distribution plot and lncRNA-expression in high and low risk groups

图3 高风险组和低风险组生存率的Kaplan-Meier生存分析曲线Fig.3 Kaplan-Meier survival analysis curve in high and low risk groups
2.5 临床特征单因素及多因素COX回归分析及ROC曲线
对年龄、stage肿瘤分期、T分期、M分期、N分期和Riskscore进行单因素及多因素COX回归分析。单因素COX回归分析显示stage分期、N分期和Riskscore有统计学意义(P<0.05),进一步对stage分期、N分期和Riskscore做多因素COX回归分析,显示只有Riskscore有统计学意义(P<0.05),提示风险模型Riskscore可作为ESCC生存独立预后因素(表4、5)。进一步比较风险模型Riskscore和各临床特征的预测效能,预测1年生存率的ROC曲线下面积显示Riskscore模型为0.750,高于临床特征N分期0.695,stage肿瘤分期0.667,T分期0.550,M分期0.549,年龄0.520,提示该模型相比于传统TNM分期能更准确判断ESCC患者预后(图4B)。

图4 Riskscore模型及临床特征的ROC曲线图Fig.4 ROC curves of Riskscore model and clinical characteristics

表3 差异表达lncRNA多因素COX回归分析Table 3 Multivariate Cox regression analysis of differentially expressed lncRNA

表4 临床特征单因素COX回归分析Table 4 Univariate Cox regression analysis of clinical characteristics
2.6 临床特征与Riskscore相关性分析
分析各ESCC样本Riskscore评分与性别、年龄、生存状态、肿瘤分期、T分期、N分期和M分期等临床特征的相关性,结果显示男性及死亡患者的Riskscore评分更高(P<0.05),女性及生存患者的Riskscore评分相对较低(P<0.05),在年龄和stage分期中Riskscore评分差异没有统计学意义(P>0.05),在T分期,N分期中差异性不显著,M分期(M0/M1:70/4)未行相关性检验(图5)。

图5 临床特征与Riskscore相关性分析Fig.5 Correlation analysis between clinical characteristics and Riskscore
2.7 高低风险组主成分分析
对ESCC样本lncRNA表达矩阵和模型lncRNA(AC108449.2,AL033384.1)表达矩阵按照高低风险组进行主成分分析,3D散点图显示原有ESCC ln⁃cRNA表达矩阵经降维后仍不能有效区分高低风险组患者,而模型lncRNA表达矩阵经降维后高低风险患者区分较为明显,提示模型lncRNA(AC108449.2,AL033384.1)是ESCC高低风险组分布的主要影响因素(图6)。

图6 高低风险组主成分分析图Fig.6 Principal component analysis plot in high and low risk group
2.8 高低风险组GO及KEGG基因功能富集分析
使用GSEA软件对高低风险组行GO、KEGG分析,富集标准为|NES|>1,NOMp-value<0.05,FDRq-value<0.25,结果显示高风险组主要富集在表皮细胞的角质化和细胞内的高代谢等过程,低风险组主要富集在细胞内氨基酸的甲基化过程,提示高风险组患者食管上皮磷化过程及细胞高代谢促进了ESCC的发生发展,是导致高风险组不良预后的内在影响因素(图7)。

图7 高低风险组GSEA富集分析图Fig.7 GSEA enrichment analysis plot in high and low risk group
3 讨论
食管癌是全世界最常见的肿瘤之一,早期难以发现,一经诊断90%的患者即为中晚期,5年生存率不到30%。然而,对于食管鳞癌的发病机制研究尚有争议,近年来,随着分子研究的不断发展,ln⁃cRNA作为明星分子被发现在基因复制、转录、表达等各个环节影响着各种癌症的发生发展。大量研究证明lncRNA与食管鳞癌的发生发展密不可分。Li等[15]人研究发现lncRNANLIPMT可通过调节miR-320/survivin轴抑制食管鳞癌细胞增殖和迁移并促进细胞周期停滞、凋亡;Xu等[16]人指出ln⁃cRNAZEB2-AS1可通过调节miR-574-3p/HMGA2轴促进食管鳞癌的增殖,迁移和侵袭;Li等[17]研究证实lncRNAMIR205HG通过调节miR-214/SOX4轴加速食管鳞癌的进展。可见食管鳞癌发生发展与lncRNA的调控息息相关。

表5 临床特征多因素COX回归分析Table 5 Multivariate Cox regression analysis of clinical characteristics
然而,关于lncRNA与食管鳞癌预后的关系国内尚无相关报导,已有研究提示基于4个lncRNA构建的风险模型可以有效预测胶质母细胞瘤的预后[18],胃癌患者中表达的AC097478.1、AC097478.3、AL354719.2也可有效预测胃癌患者的预后[19]。本研究旨在通过分析TCGA食管鳞癌表达样本基因分子特征差异,寻找食管鳞癌预后的生物标志物,以指导临床诊疗。我们发现食管鳞癌上皮与正常食管上皮基因表达存在差异,单因素及多因素COX回归分析提示AC108449.2,AL033384.1这两个ln⁃cRNA与食管鳞癌的预后相关,且高风险组AC108449.2表达下调,AL033384.1表达上调,总体生存率较差,而低风险组相反,已有研究显示AC108449.2是肾脏透明细胞癌预后因素,在高风险组中低表达,在低风险组中高表达[20],另有研究指出AL033384.1与食管鳞癌预后相关,AL033384.1高表达的食管鳞癌患者预后较差[21],这与我们的研究发现类似。我们进一步根据预后相关基因构建了食管鳞癌预后风险模型,并将食管鳞癌样本按照模型中位值划分为高低风险组,预后风险模型的预测效能均高于TNM分期等临床特征,具有良好的预测效能,可作为食管鳞癌患者临床预后分析的补充。进一步主成分分析(PCA)提示模型lncRNA(AC108449.2,AL033384.1)能较好区分高低风险组的患者,GSEA富集分析提示高风险组的患者基因富集在表皮细胞的角质化及细胞的高代谢活动,提示高风险组患者通过促进表皮鳞化和加速肿瘤增殖导致了高风险组食管鳞癌的发生发展,这为进一步深入研究其内在机制奠定了基础。
然而,本研究仍存在一定的局限性。首先,纳入模型构建的食管鳞癌样本均来自TCGA数据库,可能与我国食管鳞癌的情况有所差异。其次,我们的研究发现Stage肿瘤分期与模型Riskscore评分不相关,T分期和N分期与模型Riskscore具有较弱的相关性,我们认为样本量不足与其他临床因素如术后有无辅助治疗等导致了这种现象,后续扩大样本量以及进一步亚组分析术后治疗对预后的影响可能有助于探讨TNM分期与风险Riskscore评分的关系。最后,作为一项初步的探索性研究,只对ln⁃cRNA与食管鳞癌患者预后风险鉴别有定性作用,尚未能精确定量lncRNA与食管鳞癌患者预后的关系,应用价值有限,仍需要多中心的大样本的研究进一步验证。
综上所述,通过差异表达lncRNA与食管鳞癌患者预后关系揭示食管鳞癌2个lncRNA潜在生物标志物,本研究成功构建了预后风险模型,是鉴别和预测食管鳞癌患者预后的重要尝试,是现有基于TNM分期预测食管鳞癌患者预后的有益补充手段。

