基于WGCNA分析和风险评分建模的骨肉瘤转移标志物筛选和预后研究*
仲 涛 朱英会 仲伟俍 王志坚 于英楠△ 田 康
【提 要】 目的 应用WGCNA分析筛选骨肉瘤转移标志物,通过风险评分建立骨内瘤预后的预测模型,为评估骨内瘤转移和预后提供方法指导。方法 基于GSE14359数据集(n=20),WGCNA法确定骨内瘤转移模块中的中心基因;获取TARGET公共数据库骨内瘤样本(n=86),在单因素Kaplan-Meier预后分析基础上,Cox多因素计算风险评分构建评估患者生存的预测模型。结果 中心基因HLA-DRA和FLI1与骨内瘤转移密切相关(P<0.05);风险评分=0.149×G0S2-0.572×ARHGDIB+0.048×CD74+0.242×HLA-DMA-0.473×MGAT1-0.813×PLD3+0.230×EPAS1,有较好的预后评估能力(P<0.001;HR=2.72)。结论 WGCNA分析能够有效筛选OS转移标志物,风险评分能识别更灵敏的预后模型。
骨肉瘤(Osteosarcoma,OS)是骨骼系统中一种侵袭性恶性肿瘤,其病情发展迅速且预后不良,已经成为儿童和青少年的主要致死性疾病[1]。虽然近年来的新辅助化疗不断发展,手术切除技术不断进步[2],但是由于缺少早期筛查标记物,约20%OS患者在诊断时就已经出现转移,特别是肺转移[3],因此其5年生存率仍然没有得到显著提升[4]。因此,寻找潜在的生物标志物来评估OS的转移和预后具有重要的临床应用价值。
WGCNA分析[5-6],即加权基因共表达网络分析,首先通过计算基因之间的表达相关性,将具有相似表达模式的基因聚类到一个模块中并筛选出中心基因,然后再分析该模块与样本特征(包括临床病理参数和治疗方法等)之间的相关性。目前,由于骨肉瘤的异质性,基于小样本的差异表达分析的骨肉瘤转移生物标志物往往敏感度和特异度较低,因而缺乏临床应用价值[7]。WGCNA则从复杂的多样本转录组数据中快速地提取出与转移相关的模块及基因,在比较模块内连接性和基因重要性基础上,获得适用性更广泛的生物标志物[8-9]。风险评分建模基于Cox回归赋予多个基因的风险系数后计算个体预后风险高低[10],并分析其与患者生存的相关性,多项研究证实多基因模型预后评估的准确性往往高于单一基因[11-13]。
本研究将WGCNA分析用于骨肉瘤转移标志物的筛选;采用风险评分建模进行预后模型的构建,同时绘制ROC曲线评估多基因模型的准确性。
材料与方法
1.数据来源
本研究收集的数据分别下载自GEO和TARGET数据库。其中,GEO来源的GSE14359数据集包括2例正常样本和18例OS组织,后者中有8例是转移瘤;GSE32981包括5例未转移OS及11例转移瘤样本。从TARGET数据库获取的86例OS患者中,有57例生存,29例死亡,中位生存时间为1323(0~5840)天。
2.统计分析方法
(1)基因差异表达分析:使用R语言limma包分析差异表达基因,并绘制火山图进行可视化。
(2)加权基因共表达网络分析(WGCNA):通过计算尺度独立性(R2)和平均连通性以确定表征基因符合无尺度分布的软阈值,Pearson法选定与“转移”相关的模块及其中心基因。
(3)预后分析:风险评分是通过多因素Cox回归计算风险系数得到的[14],绘制受试者工作特征曲线(ROC曲线)来表征预后模型的灵敏度;log-rank单因素Kaplan-Meier法用于分析单个指标或者风险评分与OS预后的相关性[15]。
结 果
1.识别OS样本构成的模块
分析GSE14359数据差异基因发现,相比于正常样本,OS中有上调基因1108个,下调基因1419个(图1A)。进一步WGCNA分析显示,通过尺度独立性和平均连通性比较发现基因间联系软阈值为5后(图1B),绘制聚类树状图得到了29个关键模块(图1C)。

图1 WGCNA法基于差异基因确定的软阈值并识别共表达网络模块
2.确定OS转移相关的中心基因
深入分析发现绿色、蓝色和棕色模块与骨转移密切相关(图2A);为了进一步验证模块中参与调控OS转移的关键基因(n=48),将其与OS转移和非转移的差异基因取交集,结果发现只有HLA-DRA和FLI1在OS转移过程中表达失调(图2B)。

图2 筛选与OS转移相关的中心基因
3.分析中心基因与OS患者预后的相关性
通过单因素Kaplan-Meier法分析48个中心基因与OS患者预后的相关性发现,PLD3、ARHGDIB、G0S2、MGAT1、CD74、HLA-DMA高表达时,患者生存时间延长;而EPAS1高表达时,患者预后不良(图3),ROC曲线分别评估它们,预测预后1、3、5年的灵敏度和特异度(图4)。

图3 中心基因和OS预后的Kaplan-Meier曲线
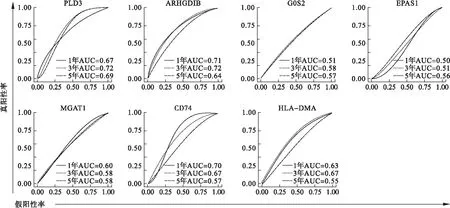
图4 中心基因预测预后的ROC曲线
4.构建OS患者预后评估最佳模型
将上述7个基因纳入多因素Cox回归分析后得到预后模型:风险评分=0.149×G0S2-0.572×ARHGDIB+0.048×CD74+0.242×HLA-DMA-0.473×MGAT1-0.813×PLD3+0.230×EPAS1(图5A)。ROC曲线显示该模型具有较好的准确性(AUC>0.7)(图5B)。此外,相比于单因素,七基因组合与预后的相关性更显著,提示更强的评估能力(P<0.001;HR=2.72),见图5C。
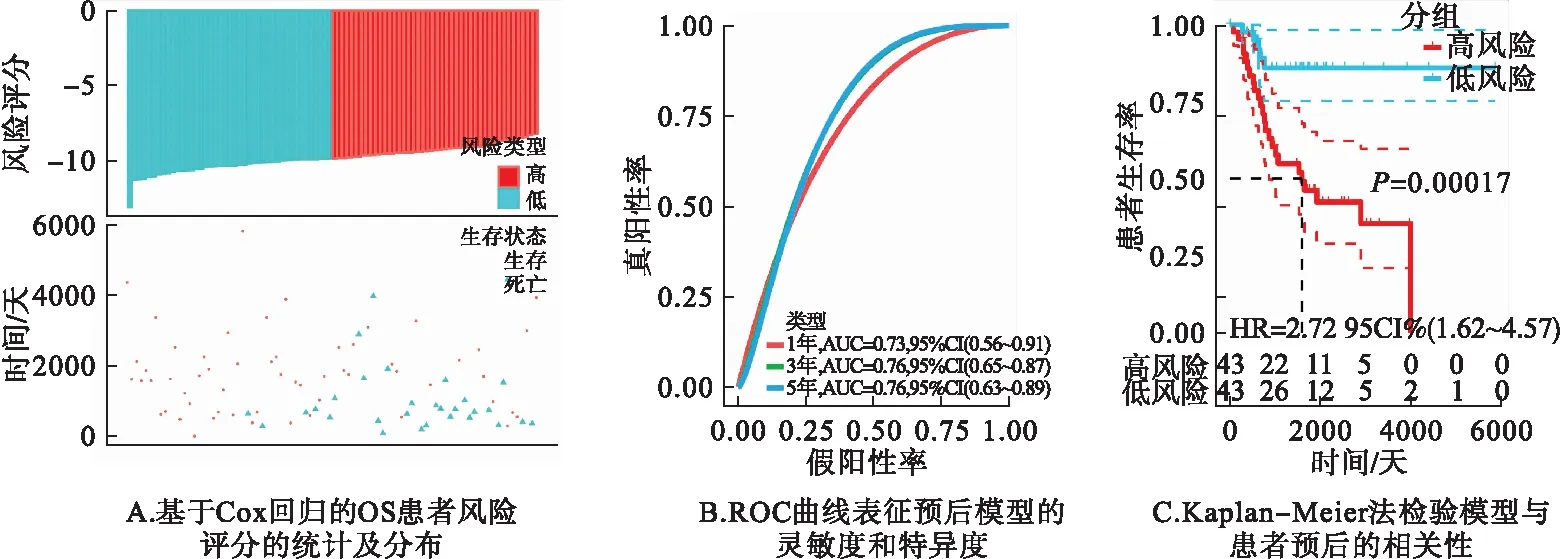
图5 通过计算风险评分构建预测OS患者生存的高效模型
讨 论
OS是最常见的原发性骨恶性肿瘤,在年轻患者中表现出高度侵袭性和早期转移性[3]。为了实现OS的及时诊断和治疗,寻找潜在的生物标志物已经成为目前亟待完成的任务。本研究基于GSE14359和GSE32981数据集,采用WGCNA法筛选出2个与OS转移显著相关的中心基因(HLA-DRA和FLI1);进一步分析TATGET数据库发现,通过Cox回归风险评分得到的“七基因预后模型”有更好的评估能力。
在本研究中,通过WGCNA法筛选得到三个与OS转移特性密切相关的模块中的48个中心基因(P<0.05)。新近研究发现,Tian等同样基于OS样本的差异表达基因通过WGCNA法筛选出能分型转移的SVM过滤器[16];此外,Zhang等使用WGCNA法分析GSE21257数据来确定区分OS转移的预测标记[17];Wang等则通过分析收集的52例OS样本的RNA-seq结果[18],采用WGCNA法模块性状分析得到其与转移正相关在内的关键模块及中心基因,为骨肉瘤的分子机制提供见解。值得注意地是,为了精确筛选预测因子,本研究进一步通过交集GSE32981中转移与否样本的差异基因,最终确认HLA-DRA和FLI1参与OS转移调控。由此,本研究在WGCNA法的基础上进行深入过滤,对筛选OS转移潜在生物标志物方法进行了优化。
本研究基于单因素Kaplan-Meier法探讨OS转移相关的中心基因对患者预后的影响,结果发现,PLD3、ARHGDIB、G0S2、MGAT1、EPAS1、CD74及HLA-DMA参与了患者生存时间的调节(P<0.05)。为了构建出更好的预后标记,本研究进一步通过计算七个基因的风险系数赋予每个患者相应的预后评分。结果发现,七基因模型的预后评估效能明显提升(P<0.001;HR=2.72)。有研究报道,Niu等发现EGR1、CXCL10、MYC和CXCR4均可作为OS预后的潜在生物标志物[19];Nakka等的结果也表明miR-21 和miR-221在OS中的表达差异对患者预后具有重要意义[20]。然而,以上证据只提供了单个基因的生存评估作用,本研究则与Liu等[11]基于Cox回归模型发现两基因(PML-EPB41)模型比单独基因具有更好的预后预测价值相似。本研究通过计算风险评分将患者分为高或低风险组,并证实高或低风险组患者生存时间差异更显著,从而进一步证实多基因模型能更有效地评估OS预后。

