柱花草磷饥饿响应基因SgPHR1和SgPHR2的克隆与表达分析
黄杰 宋剑灵 安娜 邹晓燕 李季肤 刘国道 陈志坚

摘 要:低磷胁迫是限制作物生长和产量的重要因素之一。磷饥饿响应因子PHR(phosphate starvation response)是植物磷信号调控网络中的关键因子,具有调控植物磷平衡的生物学功能。本研究在柱花草(Stylosanthes guianensis)中克隆到转录因子SgPHR1和SgPHR2基因。SgPHR1和SgPHR2基因cDNA全长分别为1413 bp和849 bp,编码470和282个氨基酸残基,蛋白分子量分别为51.4 kD和30.9 kD。SgPHR1和SgPHR2均为SANT家族成员,包含MYB蛋白结构域和CC蛋白结构域,并具有多个潜在的磷酸化位点。亚细胞定位预测表明,SgPHR1和SgPHR2均定位于细胞核中。实时定量PCR结果表明,SgPHR1基因在柱花草根和叶中的表达量高于茎中的表达量,而SgPHR2基因在叶中表达量显著高于根和茎。缺磷(-P)和缺氮(-N)处理均显著增强了SgPHR1和SgPHR2在柱花草根中的表达。不同缺磷时间处理结果进一步表明了SgPHR1和SgPHR2在转录水平上响应低磷胁迫,暗示SgPHR1和SgPHR2可能参与了柱花草对低磷胁迫的应答。本研究结果为解析柱花草响应低磷胁迫的分子机制提供了候选基因。
关键词:低磷胁迫;柱花草;PHR;基因表达;转录因子
Abstract: Low phosphorus (P) stress is one of the constraints limiting crop growth and yield. PHR (phosphate starvation response) is the key factor in P signaling network, regulating plant phosphate (Pi) homeostasis. In this study, two transcription factors, SgPHR1 and SgPHR2, were cloned in stylo (Stylosanthes guianensis). The full length of SgPHR1 and SgPHR2 was 1413 bp and 849 bp, encoding 470 and 282 amino acid residues, respectively. The protein molecular weight of SgPHR1 and SgPHR2 was 51.4 kD and 30.9 kD, respectively. Both SgPHR1 and SgPHR2 belonged to the SANT family and contained MYB protein domain and CC protein domain. Both SgPHR1 and SgPHR2 included a set of potential phosphorylation sites. Subcellular localization prediction indicated that both SgPHR1 and SgPHR2 localized to the nucleus. Real-time quantitative PCR results showed that the transcript of SgPHR1 in leaves and roots was higher than that in stems, while SgPHR2 exhibited the highest expression in leaves. Furthermore, the expression of SgPHR1 and SgPHR2 was up-regulated by both -P and nitrogen (-N) deficient treatments in stylo roots. The transcription level of SgPHR1 and SgPHR2 was found to be increased during -P treatments, suggesting that SgPHR1 and SgPHR2 involved in stylo response to Pi starvation. This study would provide candidate genes for investigating the mechanism of stylo response to P deficiency.
Keywords: low phosphorus stress; Stylosanthes; phosphate starvation response; gene expression; transcription factor
磷(phosphorus,P)是植物生长发育所必需的大量营养元素之一,参与植物多种生理生化过程,如光合作用、能量和脂类代谢等[1-3]。然而,土壤磷的有效性低,特别是在酸性土壤中,磷容易被铝(Al)和铁(Fe)等金属元素结合形成不能被植物直接吸收和利用的难溶性磷[4-5]。因此,低磷胁迫是酸性土壤上限制作物生长和产量的重要因素之一[6]。在低磷环境下,植物表现出植株矮小,生长缓慢,叶色暗绿和根系发育受阻等胁迫症状[7]。為应对低磷胁迫,植物形成了一系列的适应性机制,如植物可以通过改变根系形态和构型增加对土壤磷的吸收[8-9];通过分泌有机酸和酸性磷酸酶来活化利用土壤难溶性磷和有机磷[10-12];通过诱导磷转运基因表达增强对磷的吸收和转运[13]。另外,磷信号网络在维持植物细胞磷平衡中具有重要调控作用[14-15]。
磷饥饿响应因子(phosphate starvation response,PHR)属于MYB-CC型转录因子,在磷信号网络中发挥关键作用[16]。PHR具有Myb DNA结合结构域,C端具有卷曲螺旋(CC)结构域。PHR一般定位于细胞核,具有转录调控的作用,能调控下游磷饥饿响应基因的表达,如PT、PHT和MIR399等[17]。在低磷条件下,PHR会形成二聚体,通过CC结构域结合在磷饥饿响应基因启动子P1BS(GNATATNC)顺式作用元件上,从而调控靶基因表达[16]。绿色衣藻(Algachamycary)CrPSR1是最早发现的MYB-CC型转录因子,其在调节绿色衣藻响应低磷胁迫中起作用[17]。AtPHR1是拟南芥(Arabidopsis thaliana)磷信号网络中的重要调节因子,其可通过调控PT等下游基因表达响应低磷环境[18-19]。近年来,在水稻(Oryza sativa)[20]、玉米(Zea mays)[21]、大豆(Glycine max)[22]和菜豆(Phaseolus vulgaris)[23]等植物中相继克隆并鉴定了PHR同源基因,暗示PHR在植物适应低磷胁迫中起重要作用。
柱花草(Stylosanthes spp.)原产于美洲和非洲热带和亚热带地区,是重要的热带豆科牧草[24]。柱花草广泛用于牧草饲料和绿肥覆盖等方面。柱花草具有良好的适应低磷酸性土壤的能力,其被认为是研究植物适应酸性土壤低磷胁迫的重要热带豆科植物。迄今为止,已从柱花草中克隆了一系列低磷响应基因,如SgPT1、SgPAP7/10/23/26和SgSPX1[25-27],但是,对柱花草磷信号网络调控因子基因的克隆和研究较少。因此,本研究克隆了柱花草2个磷饥饿响应转录因子基因SgPHR1和SgPHR2,并对这两个基因进行了生物信息学和基因表达分析,可为进一步解析柱花草适应低磷胁迫的分子调控网络提供基因资源。
1 材料与方法
1.1 材料
本研究所用的实验材料为‘热研2号圭亚那柱花草(Stylosanthes guianensis)。柱花草种子由中国热带农业科学院热带作物品种资源研究所草业研究中心提供。
1.2 方法
1.2.1 材料与处理 柱花草种子去种皮后于80 ℃水浴3 min,在湿润的滤纸上萌发2 d。将萌发后的柱花草幼苗移到1/2 Hoagland营养液中进行正常培养。柱花草培养14 d后,进行不同磷浓度处理,分别为0.5 μmol/L KH2PO4(低磷,LP)和250 μmol/L KH2PO4(正常磷,CK)。处理14 d后收获样品,测定生物量和磷含量。每个处理设置3个生物学重复。
1.2.2 磷含量测定 柱花草样品置于105 ℃杀青30 min,75 ℃烘干至恒重。将样品粉碎后,称取0.07 g样品,在马弗炉中600 ℃灰化10 h,然后采用钼锑抗显色法测定植株磷含量。
1.2.3 RNA提取和cDNA合成 总RNA提取参考TRIzol Universal提取试剂(Tiangen,中国)说明书。称取0.1 g根系样品,加入1 mL TRIzol提取液充分研磨并在室温放置5 min。4 ℃,12 000 r/min离心3 min,吸取上清液至1.5 mL离心管中,加入预冷的5 mol/L氯化钠和氯仿,充分混匀。4 ℃,12 000 r/min离心10 min,取上清液,加入等体积的异丙醇,充分混匀,室温放置10 min。4 ℃,12 000 r/min离心10 min,弃上清液,加入1 mL 75%乙醇。4 ℃,7500 r/min离心5 min,弃上清液,风干沉淀,加入无RNase ddH2O充分溶解RNA。
参考RevertAid First Strand cDNA Synthesis试剂盒(Thermo Fisher,美国)方法合成cDNA第一链。在PCR管中加入2 μg RNA、1 μL Oligo (dT)18,并加入无RNase ddH2O至12 μL。于65 ℃反应5 min后,加入4 μL 5×Reaction Buffer、2 μL dNTP Mix、1 μL RNase Inhibitor和1 μL RevertAid M-MuL V Reverse transcriptase。反应混合物于42 ℃反应60 min,70 ℃反应5 min。反应结束后,cDNA样品于–20 ℃保存备用。
1.2.4 SgPHR1和SgPHR2全长克隆 基于本课题组柱花草根系低磷响应转录组结果,筛选获得SgPHR1和SgPHR2基因全长序列,根据序列设计SgPHR1-ORF-F/R和SgPHR2-ORF-F/R引物(表1)。以根系cDNA为模板,通过PCR扩增SgPHR1和SgPHR2基因。回收PCR产物并连接到克隆载体pMD18-T(TaKaRa,日本),进一步转化大肠杆菌DH5α并进行测序分析,获得SgPHR1和SgPHR2全长序列。
1.2.5 生物信息学分析 利用KinasePhos(http://kinasephos.mbc.nctu.edu.tw/)进行磷酸化位點预测;利用WoLF PSORT(https://www.genscript. com/ wolf-psort.html)进行亚细胞定位预测分析;运用ClustalX1.8进行多序列比对分析;采用MAGA-X构建系统进化树;利用NCBI(https://www.ncbi.nlm.nih.gov/)网站进行保守结构域分析。
1.2.6 实时定量PCR分析 使用QuantStudioTM Real-Time PCR(Thermo Fisher,美国)系统和SYBR Green(Vazyme,中国)定量试剂盒,进行实时定量PCR分析。定量PCR反应体系为10 μL 2×SYBR Green PCR master mix、1 μL引物(10 μmol/L)、2 μL稀释30倍的cDNA模板,并加入ddH2O至20 μL。反应程序为:95 ℃ 1 min,95 ℃ 15 s,58 ℃ 15 s,72 ℃ 30 s,40个循环。基因相对表达量=目的基因(SgPHRs)表达量/内参基因(SgEF1a)表达量。定量PCR引物见表1。
1.3 数据处理
数据通过Microsoft Excel 2010软件进行平均数、标准误计算和作图,采用SPSS数据分析软件(V23.0,SPSS Institute,美国)进行方差分析。
2 结果与分析
2.1 不同磷浓度处理对柱花草干重和磷含量的影响
从图1A可以看出,相对正常磷(250 μmol/L KH2PO4,CK)处理,低磷(0.5 μmol/L KH2PO4,LP)处理显著抑制了柱花草地上部干重,但对根部干重没有显著影响。低磷处理下的地上部干重仅为正常磷条件下的46%(图1A)。相对正常磷(CK)处理,低磷(LP)处理显著降低了柱花草地上部和根部磷含量,低磷处理下的地上部和根部磷含量分别仅为正常磷条件下的17%和15%(图1B)。
2.2 柱花草SgPHR1和SgPHR2的克隆
研究基于前期柱花草轉录组测序结果获得的SgPHR1和SgPHR2基因序列,设计PCR引物,以低磷处理下柱花草根系cDNA为模板,扩增出SgPHR1和SgPHR2基因cDNA全长。如图2所示,SgPHR1和SgPHR2基因全长分别为1413 bp和849 bp。利用ExPASyProt软件分析发现SgPHR1基因编码470个氨基酸残基,蛋白分子量为51.4 kD,理论等电点为5.4,为亲水性蛋白。SgPHR2基因编码282个氨基酸残基,蛋白分子量为30.9 kD,理论等电点为7.7,也为亲水性蛋白。
2.3 柱花草SgPHR1和SgPHR2蛋白理化性质分析
利用WoLF PSORT进行亚细胞定位预测,结果表明SgPHR1和SgPHR2蛋白均定位于细胞核上,暗示其为核蛋白。利用KinasePhos网站对SgPHR1和SgPHR2蛋白的磷酸化位点进行预测,发现SgPHR1蛋白具有14个丝氨酸、4个苏氨酸和4个酪氨酸等潜在的磷酸化位点,而SgPHR2蛋白具有11个丝氨酸、6个苏氨酸和1个酪氨酸等潜在的磷酸化位点(表2)。此外,SgPHR1蛋白的磷酸化位点主要分布在N端,而SgPHR2蛋白的磷酸化位点主要分布在C端(图3)。
2.4 柱花草SgPHR1和SgPHR2蛋白保守结构域和多序列比对分析
CDD Tools预测表明,SgPHR1和SgPHR2都属于SANT家族成员,均含有PHR蛋白的保守序列,即myb_SHAQKYF和Myb_CC_LHEQLE特征序列(图4)。SgPHR1蛋白在248~300和331~378氨基酸范围内,分别包含myb_SHAQKYF结构域和Myb_CC_LHEQLE结构域;SgPHR2蛋白在20~76和106~152氨基酸范围内,分别包含myb_ SHAQKYF结构域和Myb_CC_LHEQLE结构域。
多序列比对分析发现,SgPHR1和SgPHR2蛋白与已报道的不同物种PHR蛋白均包含myb_SHAQKYF和Myb_CC_LHEQLE保守结构域。此外,不同物种PHRs蛋白N端和C端同源性较低(图5)。
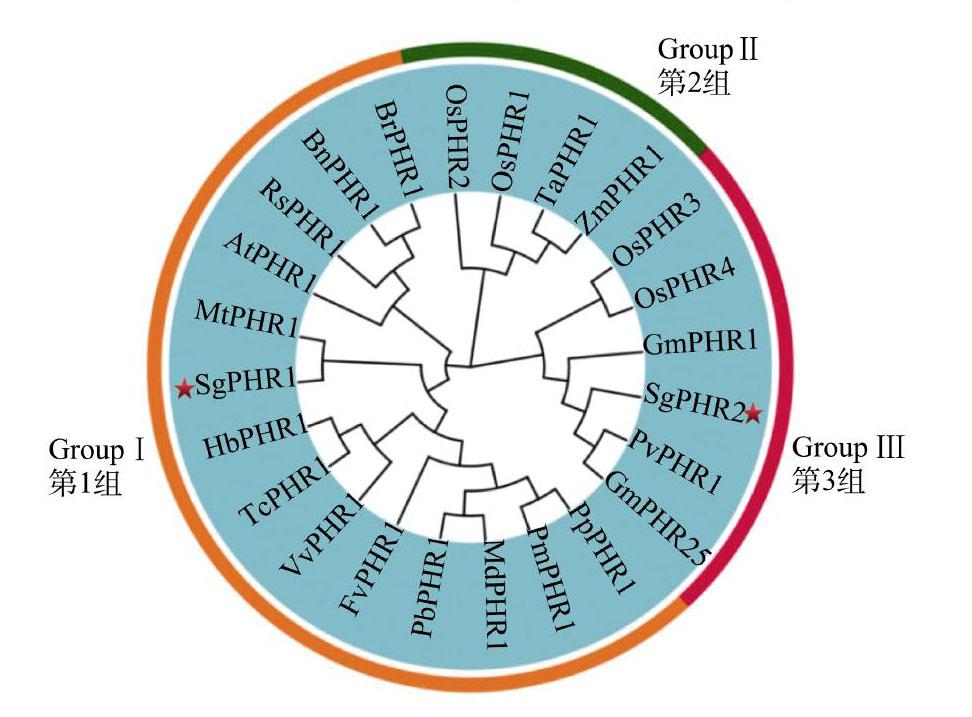
2.5 系统进化树分析
系统进化树分析表明,PHRs蛋白可以分为3大组(图6)。第Ⅰ组包括拟南芥、截形苜蓿(Medicago truncatula)、草莓(Fragaria vesca)和甘蓝型油菜(Brassica naus)等植物PHR蛋白,如拟南芥AtPHR1和甘蓝型油菜BnPHR1。柱花草SgPHR1被分在第Ⅰ组中,与截形苜蓿MtPHR1属于同一分枝,同源性最高。第II组包括水稻、玉米和小麦(Triticum aestivum)等植物PHR蛋白,如水稻OsPHR1、OsPHR2、小麦TaPHR1和玉米ZmPHR1。第III组包括水稻、大豆、菜豆和柱花草等植物PHR蛋白。柱花草SgPHR2与菜豆PvPHR1、大豆GmPHR1和GmPHR25同源性最高(图6)。
蛋白序列编号包括:柱花草SgPHR1、拟南芥(AtPHR1,NP_194590)、玉米(ZmPHR1,JF831533.1)、水稻(OsPHR1,AK063486.1)、草莓(FvPHR1,XP_004289982.1)、甘蓝型(BnPHR1,JN806156.1)和大豆(GmPHR1,HQ007311);红色方框内为MYB结构域,蓝色方框内为CC结构域。
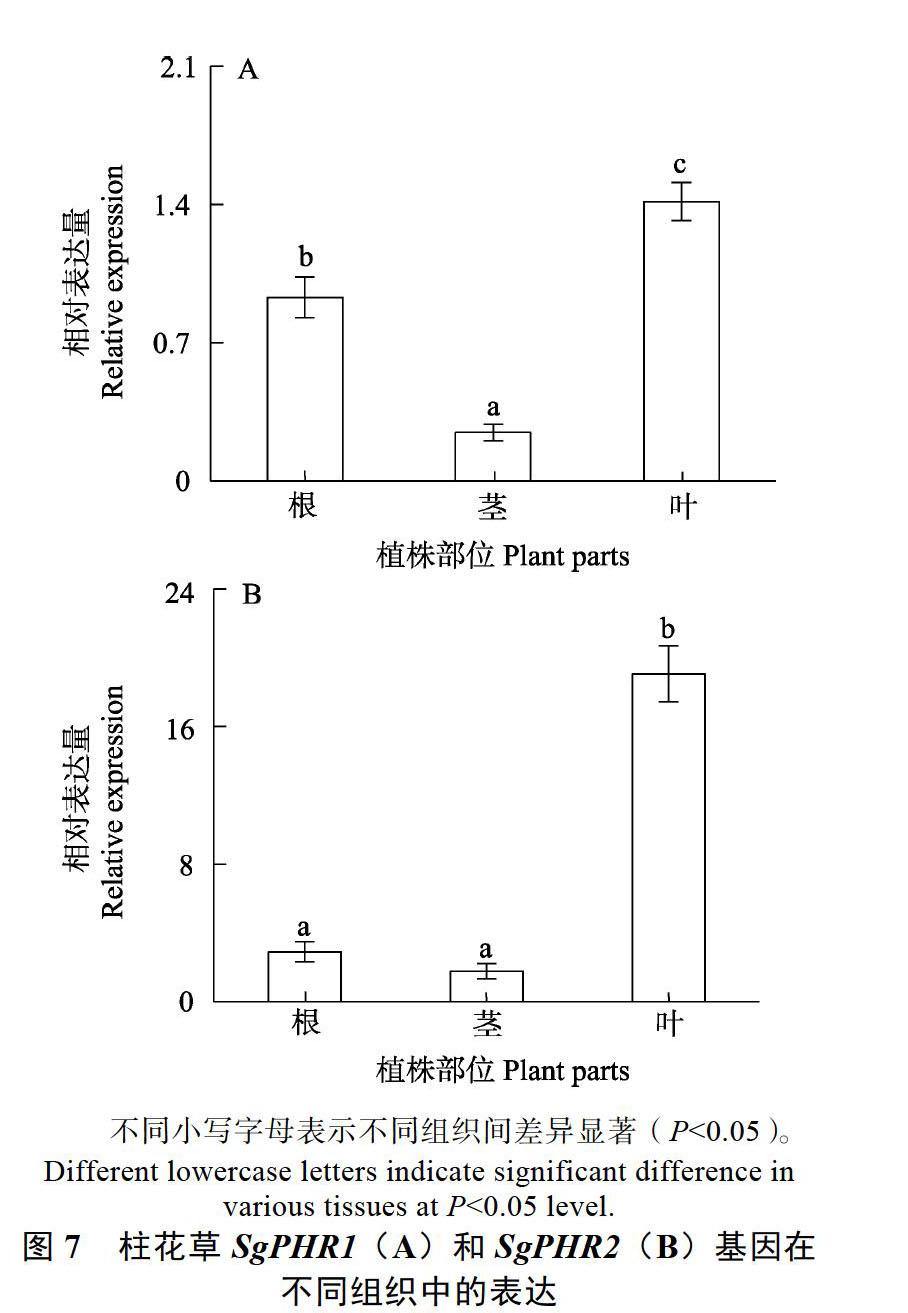
2.6 柱花草SgPHR1和SgPHR2基因表达分析
本研究对SgPHR1和SgPHR2基因表达进行分析。结果表明,SgPHR1和SgPHR2基因在根、茎和叶中均有表达,其中,SgPHR1在叶中表达量最高,根次之,茎中表达量最低,SgPHR2基因在叶中表达量显著高于茎和根(图7)。由于PHR基因被报道参与了植物对养分胁迫的应答,本研究进一步分析了不同缺素处理对柱花草SgPHR1和SgPHR2基因表达的影响。结果发现,与对照(CK)相比,缺氮(–N)、缺磷(–P)和缺钾(–K)处理均显著增强了SgPHR1基因在柱花草根系中的表达(图8A);与对照相比,缺氮和缺磷处理显著增强了SgPHR2基因在柱花草根系中的表达,而缺钾处理对SgPHR2基因的表达无显著影响(图8A)。其中,缺磷处理下SgPHR1和SgPHR2基因表达量分别是对照条件下的1.3和2.6倍(图8A)。
从图8B可见,随着低磷处理时间的延长,SgPHR1和SgPHR2基因表达量逐渐增加,并在低磷处理第7天基因表达量最高,SgPHR1和SgPHR2Genebank序列号包括:拟南芥(AtPHR1,NP_194590)、玉米(ZmPHR1,JF831533.1)、大豆(sGmPHR25,NP_001350613.1)、菜豆(PvPHR1,ACD13206.1)、水稻(OsPHR1,AK063486.1;OsPHR2,AK100065.1;OsPHR3,A2X0Q0.1;OsPHR4,XP_015644151.1)、小麦(TaPHR1,AGH13375.1)、草莓(FvPHR1,XP_004289982.1)、甘蓝型油菜(BnPHR1,JN806156.1)、梅花(PmPHR1,XP_016650539.1)、桃李(PpPHR1,XP_020425868.1)、甜樱桃(PaPHR1,XP_021831311.1)、海棠(MdPHR1,XP_008372382.1)、梨(PbPHR1,XP_018506029.1)、截形苜蓿(MtPHR1,XP_003625354.1)、可可树(HcPHR1,XP_007050189.2)、巴西橡胶树(HbPHR1,XP_021670552.1)、葡萄(VvPHR1,XP_002270511.1)、萝卜(RsPHR1,XP_018469653.1)和大豆(GmPHR1,HQ007311)等。代表柱花草PHRs蛋白。
3 讨论
磷是维持植物正常生长发育所必需的大量营养元素之一,低磷胁迫抑制作物的生长和产量。在水稻、大豆和玉米等植物中的研究发现,低磷胁迫显著抑制了这些植物的植株生物量和磷含量[20-22]。类似的,本研究结果发现,低磷处理显著抑制了柱花草地上部干重,并降低了柱花草地上部和根部磷含量(圖1)。在应对低磷胁迫过程中,植物形成了如改变根系形态构建型等适应性机制,进而增强对土壤磷的吸收和利用[8]。低磷胁迫下植物的适应性变化受到磷信号网络的精细调控,其中,转录因子PHR基因在植物磷信号网络中发挥关键作用[15]。因此,为探索柱花草响应低磷胁迫的磷信号网络,本研究克隆了柱花草MYB-CC型转录因子SgPHR1和SgPHR2(图2)。
近年来,对MYB型转录因子PHR功能研究较为广泛,PHR在磷信号网路中起重要作用[14]。
据报道,拟南芥包括14个AtPHRs、大豆包括35个GmPHRs、水稻包括4个OsPHRs[19,22,28]。PHR转录因子具有高度保守的与DNA结合的MYB结构域(myb_SHAQKYF)和与二聚体形成相关的螺旋CC结构域(Myb_CC_LHEQLE)。PHR通过与下游磷饥饿诱导基因的启动子结合以调控细胞内的磷平衡。研究表明,MYB结构域和卷曲螺旋CC结构域都是PHR结合下游靶基因所必需的结构域[29]。柱花草SgPHR1和SgPHR2均属于SANT蛋白家族成员,包含myb_SHAQKYF和Myb_ CC_LHEQLE特征序列(图4),表明柱花草SgPHR1和SgPHR2具备典型的PHR蛋白特性和结构特征。
系统进化树分析表明,柱花草SgPHR1被分在第Ⅰ组中(图6),这一组包括拟南芥AtPHR1、油菜BnPHR1和草莓FvPHR1等蛋白,这些蛋白被认为是植物响应低磷胁迫的重要因子[28,30]。如,AtPHR1定位在细胞核内,是拟南芥关键的磷信号调控因子[18]。BnPHR1是甘蓝型油菜响应低磷的重要调控因子,具有调控下游磷饥饿响应基因表达,从而促进植物对磷的吸收和维持磷平衡的生物学功能[31]。草莓FvPHR1定位于细胞核内,FvPHR1被报道可以直接激活下游磷饥饿响应基因的表达[30]。柱花草SgPHR2与大豆GmPHR1和GmPHR25以及菜豆PvPHR1等被分在第III组中。大豆GmPHR25定位在细胞核中,具有调控大豆磷稳态的生物学功能[22]。PvPHR1被报道是调控菜豆磷转运和稳态相关基因的正调节因子[23]。因此,SgPHR1和SgPHR2可能具有植物PHR蛋白保守的生物学功能。
氮、磷和钾是植物正常生长和发育所必需的大量营养元素。研究表明,MYB转录因子家族基因的表达受到氮、磷和钾等元素缺乏的影响[14,32,33]。如,在水稻中,缺磷处理能够增强水稻根中MYB家族基因OsPHR4的表达[31]。在大豆中,缺氮处理增强了7个GmPHR家族基因在根中的表达[22]。另外,转录组分析结果表明,缺钾处理也能调控水稻和番茄(Lycopersicon esculentum)多个MYB家族基因的表达[34-35]。本研究结果发现,缺磷和缺氮处理均显著增强了SgPHR1和SgPHR2在柱花草根系中的表达,表明SgPHR1和SgPHR2可能受到氮磷协同调控,进而参与了柱花草对氮和磷胁迫的应答。另外,SgPHR1和SgPHR2表达量随着磷饥饿处理时间的延长而增加(图8)。类似的,缺磷处理增加了OsPHR3和OsPHR4在水稻根部中的表达量[31],而玉米ZmPHR1在根和叶中的表达也会随着磷饥饿程度增加而增加[21]。然而,研究发现,拟南芥AtPHR1、水稻OsPHR1和OsPHR2等PHR基因在转录水平上不受外源磷的影响,这些PHRs基因可能通过翻译后水平调控植物响应低磷胁迫[19,32]。虽然低磷处理对拟南芥AtPHR1基因表达的影响不明显,但是,与野生型拟南芥相比,phr1突变体缺失对磷饥饿响应基因的调控,表明AtPHR1在拟南芥磷信号网路中起重要的转录调控作用[18-19]。因此,SgPHR1和SgPHR2可能作为重要的转录调控因子参与了柱花草对低磷胁迫的响应。
参考文献
Chen S S, Luo Y, Ding G D, et al. Comparative analysis of Brassica napus plasma membrane proteins under phosphorus deficiency using label--free and MaxQuant-based proteomics approaches[J]. Journal of Proteomics, 2016, 133: 144-152.
Liu Y, Xie Y R, Wang H, et al. Light and ethylene coordinately regulate the phosphate starvation response through transcriptional regulation of phosphate starvation response1[J]. The Plant Cell, 2017, 29(9): 2269-2284.
Murakawa M, Ohta H, Shimojima M, et al. Lipid remodeling under acidic conditions and its interplay with low Pi stress in Arabidopsis[J]. Plant Molecular Biology Reporter, 2019, 101(1/2): 81-93.
Yang Z B, Rao I M, Horst W J, et al. Interaction of aluminium and drought stress on root growth and crop yield on acid soils[J]. Plant and Soil, 2013, 372(1-2): 3-25.
Qu X J, Zhou J Q, Masabni J, et al. Phosphorus relieves aluminum toxicity in oil tea seedlings by regulating the metabolic profiling in the roots[J]. Plant Physiology and Biochemistry, 2020, 152: 12-22.
Koopmans G F, Chardon W J, Willigen P, et al. Phosphorus desorption dynamics in soil and the link to a dynamic concept of bioavailability[J]. Journal of Environmental Quality, 2004, 33(4): 1393-1402.
Tania Galindo‐Castaeda, Kathleen M Brown, Jonathan P Lynch, et al. Reduced root cortical burden improves growth and grain yield under low phosphorus availability in maize[J]. Plant Cell and Environment, 2018, 41(7): 1579-1592.
Güsewell Sabine. Regulation of dauciform root formation and root phosphatase activities of sedges (Carex) by nitrogen and phosphorus[J]. Plant & Soil, 2017, 415(1-2): 57-72.
馬若囡, 刘 庆, 李 欢, 等. 缺磷胁迫对甘薯前期根系发育及养分吸收的影响[J]. 华北农学报, 2017, 32(5): 171-176.
Li C C, Li C F, Zhang H Y, et al. The purple acid phosphatase GmPAP21 enhances internal phosphorus utilization and possibly plays a role in symbiosis with rhizobia in soybean[J]. Physiologia Plantarum, 2017, 159(2): 215-227.
Mehdi Y H, Ali I D, Ali M M, et al. Agrobacterium rhizogenes transformed soybeans with AtPAP18 gene show enhanced phosphorus uptake and biomass production[J]. Biotechnology and Biotechnological Equipment, 2018, 32(4): 865-873.
Chen Z C, Liao H. Organic acid anions: An effective defensive weapon for plants against aluminum toxicity and phosphorus deficiency in acidic soils[J]. Journal of Genetics and Genomics, 2016, 43(11): 631-638.
Chen G H, Yan W, Yang S P, et al. Overexpression of rice phosphate transporter gene OsPT2 enhances tolerance to low phosphorus stress in soybean[J]. Journal of Agricultural Science and Technology, 2015, 17(2): 469-482.
Amit Sharma, Alice Mühlroth, Juliette Jouhet, et al. The Myb-like transcription factor Phosphorus Starvation Response (PtPSR) controls conditional P acquisition and remodeling in marine microalgae[J]. New Phytologist, 2020, 225(6): 2380-2395.
Xuan L T H, Du N H N, Nguyen B A T, et al. Transcription factors and their roles in signal transduction in plants under abiotic stresses[J]. Current Genomics, 2017, 18(6): 483-497.
Stanislas Thiriet-Rupert, Gregory Carrier, Beno?t Chénais, et al. Transcription factors in microalgae: genome-wide prediction and comparative analysis[J]. BMC Genomics, 2016, 17(1): 1-16.
Wykoff D D, Grossman A R, Weeks D P, et al. Psr1, a nuclear localized protein that regulates phosphorus metabolism in Chlamydomonas[J]. Proceedings of the National Academy of Sciences of the United States of America, 1999, 96(26): 15336-15341.
Rubio V, Linhares F, Solano R, et al. A conserved MYB transcription factor involved in phosphate starvation signaling both in vascular plants and in unicellular algae[J]. Genes and Development, 2001, 15(16): 2122-2133.
Nilsson L, Müller R, Nielsen T H, et al. Increased expression of the MYB-related transcription factor, PHR1, leads to enhanced phosphate uptake in Arabidopsis thaliana[J]. Plant Cell and Environment, 2007, 30(12): 1499-1512.
Guo M, Ruan W Y, Li C Y, et al. Integrative comparison of the role of the Phosphate Response1 subfamily in phosphate signaling and homeostasis in rice[J]. Plant Physiology, 2015, 168(4): 1762-1776.
Wang X H, Bai J R, Liu H M, et al. Overexpression of a maize transcription factor ZmPHR1 improves shoot inorganic phosphate content and growth of Arabidopsis under low-phosphate conditions[J]. Plant Molecular Biology Reporter, 2013, 31(3): 665-677.
Xue Y B, Xiao B X, Zhu S N, et al. GmPHR25, a GmPHR member up-regulated by phosphate starvation, controls phosphate homeostasis in soybean[J]. Journal of Experimental Botany, 2017, 68(17): 4951-4967.
Valdés L O, Arenas H C, Ramírez M, et al. Essential role of MYB transcription factor: PvPHR1 and microRNA: PvmiR399 in phosphorus-deficiency signalling in common bean roots[J]. Plant Cell and Environment, 2008, 31(12): 1834-1843.
严琳玲, 张 瑜, 白昌军. 20份柱花草营养成分分析与评价[J]. 湖北农业科学, 2016(1): 128-133.
孙丽莉, 陈志坚, 刘攀道, 等. 柱花草磷转运蛋白SgPT1的克隆和表达分析[J]. 草业学报2013, 22(4): 187-196.
劉攀道, 董荣书, 丁西朋, 等. 不同磷效率柱花草基因型对外源DNA活化利用能力的比较分析[J]. 分子植物育种, 2018, 16(4): 1085-1091.
孙丽莉, 田 江, 陈志坚, 等. 柱花草SgSPX1基因的克隆与表达分析[J]. 热带作物学报, 2012, 33(10): 1794-1799.
Ren F, Guo Q Q, Chang L L, et al. Brassica napus PHR1 gene encoding a MYB-like protein functions in response to phosphate starvation[J]. PloS One, 2012, 7(8): e44005.
Jiang M Q, Sun L F, Isupov M N, et al. Structural basis for the target DNA recognition and binding by the MYB domain of phosphate starvation response 1[J]. The Febs Journal, 2019, 286(14): 2809-2821.
Wang Y, Zhang F, Cui W X, et al. The FvPHR1 transcription factor control phosphate homeostasis by transcriptionally regulating miR399a in woodland strawberry[J]. Plant Science, 2019, 280: 258-268.
Ruan W Y, Guo M N, Wu P, et al. Phosphate starvation induced OsPHR4 mediates Pi-signaling and homeostasis in rice[J]. Plant Molecular Biology Reporter, 2017, 93(3): 327-340.
Tiwari J K, Buckseth T, Zinta R, et al. Transcriptome analysis of potato shoots, roots and stolons under nitrogen stress[J]. Scientific Reports, 2020, 10(1): 921-941.
Du X Q, Wang F L, Li H, et al. The transcription factor MYB59 regulates K/NO translocation in the Arabidopsis response to low K stress[J]. The Plant Cell, 2019, 31(3): 699-714.
Zhang X Q, Jiang H, Wang H, et al. Transcriptome analysis of rice seedling roots in response to potassium deficiency[J]. Scientific Reports, 2017, 7(1): 5523.
Zhao X M, Liu Y, Liu X, et al. Comparative transcriptome profiling of two tomato genotypes in response to potassium-deficiency stress[J]. International Journal of Molecular Sciences, 2018, 19(8): 2402-2426.
責任编辑:沈德发

