苹果叶片不定芽再生过程的差异表达基因鉴定与分析
刘锴,何闪闪,张彩霞,张利义,卞书迅,袁高鹏,李武兴,康立群,丛佩华,韩晓蕾
苹果叶片不定芽再生过程的差异表达基因鉴定与分析
刘锴,何闪闪,张彩霞,张利义,卞书迅,袁高鹏,李武兴,康立群,丛佩华,韩晓蕾
中国农业科学院果树研究所/农业部园艺作物种质资源利用重点实验室/国家苹果育种中心,辽宁兴城 125100
【】筛选分析‘GL-3’苹果叶片不定芽再生过程中的差异表达基因(differentially expressed gene,DEG),进一步解析苹果叶片不定芽再生的潜在分子机制,为提高苹果的遗传转化效率提供理论参考。‘GL-3’苹果继代组培苗叶片外植体接种在再生培养基上,分别于0、3、7、14和21 d后取样并提取RNA,构建mRNA文库后采用Illumina Nova seq平台进行测序。筛选出各时间点的DEGs,根据GO(Gene ontology)和KEGG(Kyoto encyclopedia of genes and genomes)注释结果以及官方分类,使用R软件中的phyper函数对筛选到的DEGs进行GO和KEGG富集分析;利用BLAST软件进行基因比对注释;重点分析植物再生相关的激素、酶、转录因子、多胺等DEGs;采用qRT-PCR对DEGs进行定量验证。再生培养基上培养3、7、14和21 d的苹果叶片外植体与对照组相比,分别筛选到5 250、4 937、6 852、6 493个DEGs,4个时间点共有的DEGs有3 027个。DEGs的GO功能富集显示,4个时间点筛选到的共有DEGs中上调表达的DEGs主要与氧化还原过程、细胞外围、蛋白激酶活性和有机环化合物结合等功能有关;下调表达的DEGs主要与单细胞代谢过程、钙离子结合、光合膜和类囊体部分等功能有关。DEGs的KEGG通路富集分析显示,4个时间点筛选到的共有DEGs中上调表达的DEGs主要富集在磷酸戊糖途径、植物激素信号转导、植物-病原菌相互作用和内质网蛋白质加工等途径中;下调表达的DEGs主要富集在-亚麻酸代谢、苯丙烷生物合成、碳代谢和光合作用等途径中。对与植物离体叶片再生相关的激素、酶、转录因子和多胺等相关DEGs的表达模式进行分析发现,这些DEGs大部分呈上调表达趋势。经qRT-PCR验证后,所检测基因的表达趋势与转录组测序结果一致。通过对苹果叶片不定芽再生过程中不同时间点的基因表达谱进行检测和对比分析,获得了大量与苹果叶片不定芽再生相关的基因,研究结果为深入探讨苹果离体叶片再生机理提供了理论依据。
苹果;不定芽再生;RNA-Seq;差异表达基因;影响因子
0 引言
【研究意义】苹果是广受欢迎的大众水果,在全球温带地区广泛种植,我国苹果产业发展迅速,据2016年统计数据显示,我国苹果栽培面积和产量均居世界首位,已成为世界上最大的苹果生产国[1]。苹果分子生物学[1]在栽培苹果()测序完成后发展迅速[2],分子辅助选择技术也逐步应用到苹果育种中,提高了育种效率,缩短了育种周期[3]。然而苹果的遗传转化效率低,严重制约着苹果分子生物学的发展[4-5]。根癌农杆菌介导的遗传转化体系是目前苹果遗传转化的最优体系,主要以离体叶片作为基因转化的受体材料,离体叶片不定芽再生能力是影响苹果遗传转化效率的关键因素之一[6],然而苹果离体叶片不定芽再生的分子机制目前尚不明确。叶外植体的芽再生与基因型密切相关[7-8],研究发现,‘嘎拉’‘乔纳金’和‘自由’等品种的再生能力较强,而‘富士’‘平邑甜茶’等品种的离体叶片很难再生不定芽[9]。‘GL-3’是由沈阳农业大学选育的‘嘎拉’实生后代,具有再生能力强和对农杆菌敏感的特性[6]。近年来,RNA-Seq在植物再生关键基因挖掘和植物再生机制研究中发挥着越来越重要的作用[10]。利用RNA-Seq鉴定和分析‘GL-3’苹果离体叶片再生不定芽过程中的差异表达基因,进一步解析苹果再生调控过程的分子机制,为提高苹果遗传转化效率奠定理论基础。【前人研究进展】离体叶片不定芽再生是一个多级发育过程,其中包括体细胞对植物激素信号的感知与传递,启动细胞分裂增殖,获得具有器官再生能力的去分化以及形成器官的再分化等,这是一个复杂的基因调控过程[11]。近年来,已鉴定出许多植物不定芽离体发生过程中的关键基因和转录因子。Che等[12]利用RNA-Seq分析了拟南芥不定芽离体发生过程中的基因表达谱。在愈伤组织形成过程中,IAA-氨基合成酶基因、GCN5-relatedN-乙酰基转移酶基因等显著上调表达;在不定芽分化过程中,与细胞分裂素信号转导有关的基因如等差异表达最为显著。研究发现,拟南芥S突变体完全丧失芽再生能力,表明是调控芽再生的关键基因[13]。过量表达植物年龄相关miR156亚家族基因可以显著提高离体叶片的茎尖再生数[14]。最新研究发现,介导的组蛋白H3K4me2去乙酰化是离体器官不定芽再生的重要条件,敲除突变体的芽再生能力显著下降[15]。【本研究切入点】当前对苹果叶片不定芽再生能力的研究多集中在植株基因型和不定芽再生体系创建上,相关的分子机制研究较少,对苹果再生调控过程的分子机制尚不清晰。【拟解决的关键问题】本研究以遗传转化效率高、再生能力强的‘GL-3’苹果叶片为研究试材,对叶片外植体不定芽再生过程中不同时间点的试材进行转录组测序,通过差异基因筛选、GO富集分析、KEGG富集分析和关键基因表达模式分析,解析苹果叶片不定芽再生的分子机制。
1 材料与方法
试验于2019年8月至2020年4月在中国农业科学院果树研究所/农业部园艺作物种质资源利用重点实验室/苹果育种中心实验室进行。
1.1 叶片接种培养
本试验所用植物材料为沈阳农业大学果树分子生物学实验室惠赠的苹果‘GL-3’组培苗。外植体选择继代培养30 d左右的‘GL-3’组培苗顶部的幼嫩叶片,且叶片生理状态基本一致。将叶片垂直主脉横切3—5刀(不切断叶缘)后,叶背朝下接种于再生培养基上培养(25℃、避光)。再生培养基配方为:MS+TDZ 2.0 mg∙L-1+NAA 0.1 mg∙L-1+蔗糖35 g∙L-1+琼脂6.2 g∙L-1,pH 5.8。分别于0、3、7、14和21 d后取样。0 d取样的叶片作为对照组,3、7、14和21 d取样的叶片作为处理组,对照组及处理组均设置3次生物学重复。
1.2 转录组测序
提取上述样品的总RNA,检测合格后委托北京贝瑞和康生物技术有限公司采用Illumina Nova seq测序平台进行二代双向测序。测序得到的数据经碱基识别分析后得到原始数据raw reads,过滤掉低质量、含N比例大于10%、接头污染的reads后得到适合分析的数据clean reads。使用比对软件BWA[16]将数据比对‘金冠’苹果基因组。
1.3 差异表达基因的筛选
使用基于泊松分布的DEGseq算法[13]进行DEGs的检测,将差异倍数为两倍以上(fold change,FC≥2)且Q-value≤0.001(adjusted P-value≤0.001)的基因定义为显著差异表达基因,将每个时间点差异倍数最大且FPKM≥10的30个DEGs定义为极显著差异表达基因。
1.4 差异表达基因的功能分析
根据GO和KEGG注释结果以及官方分类,使用R软件中的phyper函数对筛选到的DEGs进行GO和KEGG富集分析。
1.5 差异基因的qRT-PCR荧光定量分析
反转录采用PrimeScriptTMRT reagent Kit with gDNA Eraser试剂盒(TaKaRa公司),qRT-PCR荧光定量分析采用TB Green染料。用苹果肌动蛋白基因为内标参照[17],荧光定量引物由金唯智生物科技有限公司合成(引物序列见表1),样品3次重复,基因表达倍数通过2-△△Ct法计算[18]。

表1 差异表达基因的定量引物验证
2 结果
2.1 离体叶片不定芽再生过程的形态变化
对再生培养基上培养的离体叶片状态进行定期观察,结果如图1所示,叶片刚接种到培养基时叶面平整,紧贴培养基;3 d时叶片切口处微微隆起,叶片膨胀变大;7 d时叶片切口处隆起更加明显,且切口处已有少许愈伤组织形成,叶片继续膨胀变大;14 d时叶片切口处的愈伤组织变密变多,且有少量芽点出现,叶片不再膨胀变大;21 d时不定芽数量明显增多,并开始变绿和进行伸长生长。
2.2 差异表达基因筛选
对‘GL-3’苹果叶片不定芽再生过程中的处理组与对照组的差异表达基因(差异表达倍数FC≥2及Q-value≤0.001)进行筛选。再生培养基上培养3 d的叶片外植体与对照组相比共筛选到5 250个DEGs,其中1 681个上调表达,3 569个下调表达;培养7 d的叶片外植体与对照组相比,共筛选到4 937个DEGs,其中1 882个上调表达,3 055个下调表达;培养14 d的叶片外植体与对照组相比共筛选到6 852个DEGs,其中2 705个上调表达,4 147个下调表达;培养21 d的苹果叶片外植体与对照组相比共筛选到6 493个DEGs,其中2 602个上调表达,3 891个下调表达(图2-A)。基因表达水平通过RPKM方法进行计算(图2-B)。对这些DEGs作韦恩图进行分析,叶片外植体接种于再生培养基3、7、14和21 d时共有的DEGs有3 027个(图2-C)。这些数据表明‘GL-3’苹果离体叶片不定芽再生过程在转录水平上发生明显变化。

图1 离体叶片不定芽再生过程的形态变化

A:各时间点差异表达基因上下调数量统计;B:各时间点差异表达的散点图;C:各时间点差异表达基因韦恩图
2.3 4个时间点共有差异表达基因的GO功能富集分析
叶片外植体接种于再生培养基3、7、14和21 d时共有的DEGs有3 027个,其中1 046个上调表达,1 981个下调表达。GO富集结果显示,上调表达的DEGs主要与氧化还原过程(oxidation reduction process)、细胞外围(cell periphery)、蛋白激酶活性(protein kinase activity)和有机环化合物结合(organic cyclic compound binding)等功能有关(图3-A);下调表达的DEGs主要与单细胞代谢过程(single organism metabolic process)、钙离子结合(calcium ion binding)、光合膜(photosynthetic membrane)和类囊体部分(thylakoid part)等功能有关(图3-B)。
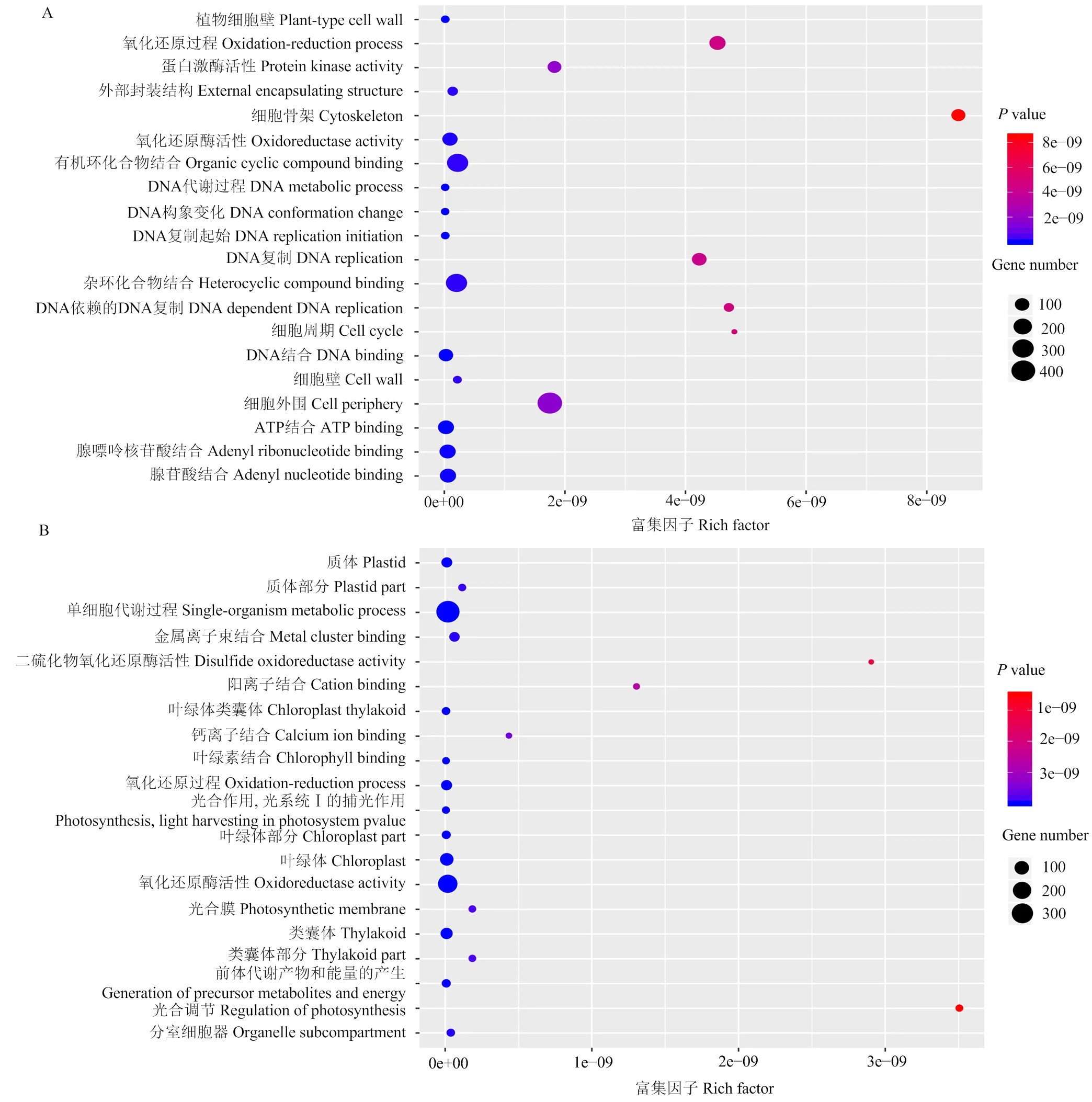
A、B分别为4个时间点共有的差异表达基因中上调表达和下调表达差异基因的GO富集结果
2.4 4个时间点共有差异表达基因的KEGG功能富集分析
对4个时间点共有差异表达基因进行KEGG富集分析发现,上调表达的DEGs主要富集在磷酸戊糖途径(pentose phosphate pathway)、植物激素信号转导(plant hormone signal transduction)、植物-病原菌相互作用(plant pathogen interaction)和内质网蛋白质加工(protein processing in endoplasmic reticulum)等途径中(图4-A);下调表达的DEGs主要富集在-亚麻酸代谢(alpha linolenic acid metabolism)、苯丙烷生物合成(phenylpropanoid biosynthesis)、碳代谢(carbon metabolism)和光合作用(photosynthesis)等途径中(图4-B)。
2.5 极显著差异表达基因的功能分析
4个时间点共有的极显著差异表达基因主要与细胞分裂素代谢过程、氧化还原过程和磷酸化信号转导系统等功能有关,显著富集在玉米素生物合成、植物激素信号转导、生物碱的生物合成及戊糖及葡萄糖醛酸转换等途径中(表2)。
2.6 植物再生相关差异表达基因分析
2.6.1 植物激素相关差异表达基因分析 外源生长物质对离体器官分化的诱导作用依赖于对内源激素平衡的调节。生长素(IAA)和细胞分裂素(CTK)与植物不定芽再生关系最为密切,对IAA和CTK相关的DEGs的表达模式进行分析,发现IAA和CTK相关的DEGs上调表达和下调表达的数目大致相等,且IAA相关的DEGs明显多于CTK相关的DEGs(图5)。
2.6.2 转录因子相关差异表达基因分析 转录因子在植物离体叶片不定芽再生过程中发挥至关重要的作用。在苹果叶片外植体再生过程的各时间点均检测到大量差异表达的转录因子,其中以AP2-EREBP、ARR、HD-ZIP、bZIP、LBD为主,并且这些转录因子主要呈上调表达趋势(图6)。
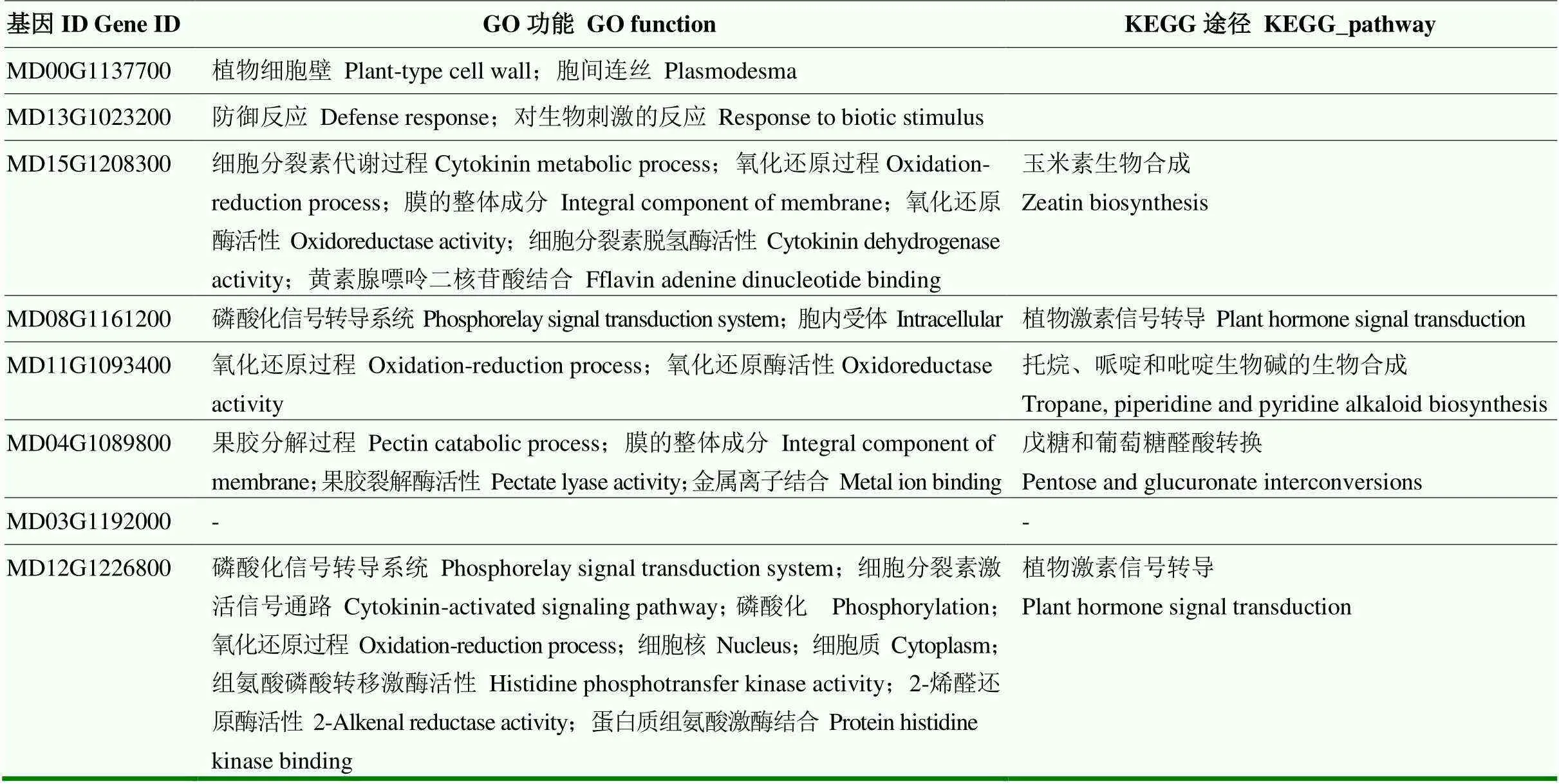
表2 极显著差异表达基因的功能分析
2.6.3 酶相关差异表达基因分析 植物离体器官再生相关的酶类主要有超氧化物歧化酶(SOD)、过氧化物酶(POD)、过氧化氢酶(CAT)。4个时间点筛选到的编码POD的DEGs最多,其次是编码SOD的DEGs,筛选到编码CAT的DEGs最少。对编码这些酶的DEGs的表达模式进行分析,编码SOD的DEGs主要呈上调表达趋势,编码POD、CAT的DEGs主要呈下调表达趋势(图7)。
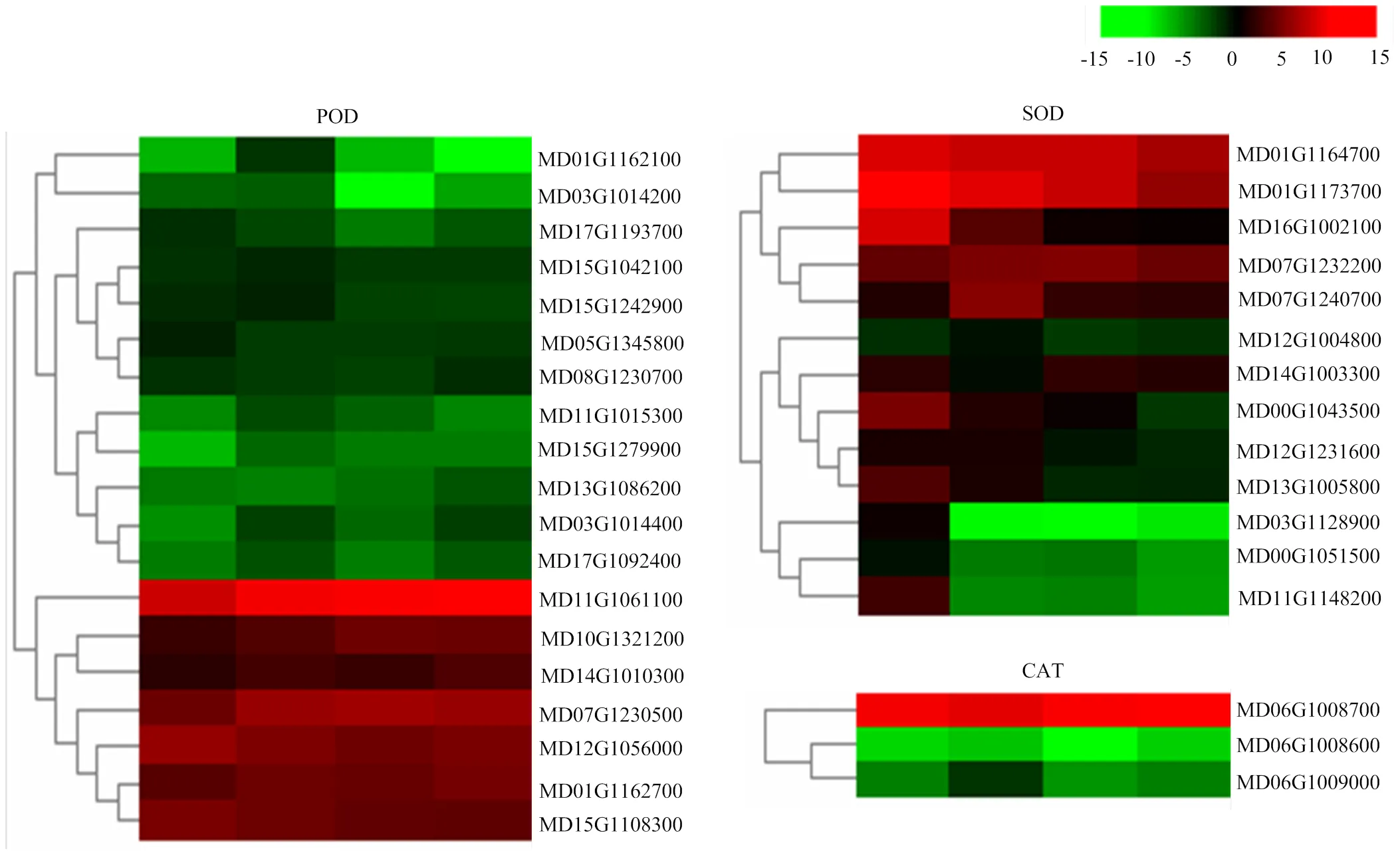
图7 编码POD、SOD、CAT的DEGs的热图
2.6.4 多胺分析 植物离体器官再生相关的多胺主要有腐胺(Put)、精胺(Spm)和亚精胺(Spd)。对编码这些多胺的DEGs表达模式进行分析,编码这3类多胺的DEGs主要呈上调表达趋势且上调表达的倍数较小,21 d时,一半的DEGs下调表达(图8)。
2.7 差异表达基因的qRT-PCR检测
选择IAA相关基因、和,CTK相关基因,SOD相关基因,POD相关基因、,编码植物再生相关转录因子的基因、、、、进行qRT-PCR验证。选定的12个(8个上调表达,4个下调表达)DEGs都在对照组和处理组之间差异表达,差异倍数较大且与分析结果密切相关,qRT-PCR验证结果与转录组测序结果基本一致(图9)。
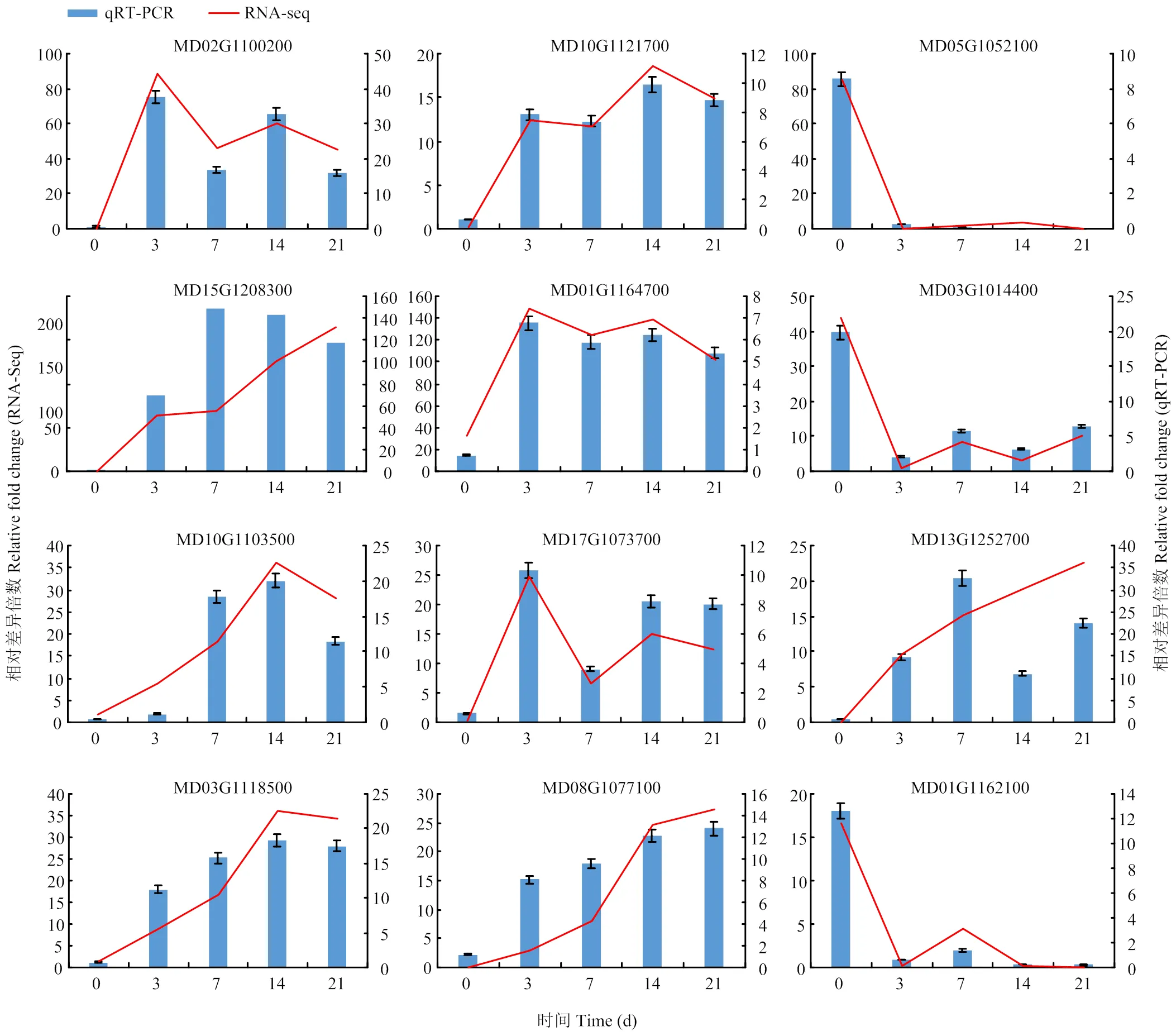
图9 差异表达基因的qRT-PCR验证
3 讨论
苹果遗传转化效率低下,严重限制了苹果功能基因组学研究的进程,进而阻碍了苹果分子生物学的发展。叶片外植体再生困难是导致苹果遗传转化效率低的主要原因之一,然而离体叶片再生不定芽的分子机制目前尚不明确。离体叶片不定芽再生是一个多级发育过程,涉及大量的转录和重编程[19-20]。利用RNA-Seq技术从全局角度分析苹果叶片不定芽再生的分子机制,为建立一个高效、稳定的离体叶片不定芽再生体系提供理论参考,对于苹果遗传转化效率的提高、基因功能验证和性状改良等方面具有重要意义。
植物离体叶片不定芽发生分为两种途径:一种是通过愈伤组织分化产生不定芽,一种是由叶片直接再生不定芽[21]。苹果的遗传转化体系主要通过愈伤组织上再生不定芽的方式再生植株[6]。愈伤组织形成和不定芽再生是不定芽形成的2个关键阶段,大量基因在这2个阶段显著差异表达并对细胞去分化和再分化的过程进行调控[22],本研究对这2个阶段基因的差异表达情况进行了分析。0—14 d是愈伤组织形成阶段,在3 d和7 d分别筛选到5 250和4 937个DEGs,推测这些DEGs与诱导愈伤组织形成相关。14—21 d愈伤组织分化产生不定芽,在14 d和2 1d分别筛选到6 852和6 493个DEGs,推测这些DEGs与愈伤组织分化产生不定芽相关。在14 d和21 d筛选到的DEGs明显多于3 d和7 d,推测14 d时离体叶片细胞基因表达程序发生剧烈变化,这些14 d才开始差异表达的基因在愈伤组织向不定芽分化的过程中起到重要的调控作用。
植物已分化的细胞经过脱分化变成愈伤组织细胞,细胞内部会发生一系列反应,造成异染色质发生大规模解聚和重新排布,外端粒的长度、端粒酶的活性也会发生改变[23]。细胞通过磷酸戊糖途径可以产生大量的NADPH,为各种反应提供还原剂[24]。推测磷酸戊糖途径在苹果离体叶片愈伤组织形成过程中发挥重要作用。植物细胞向愈伤组织细胞转化过程中,细胞壁会发生大规模的解离[25]。木质素主要是由苯丙烷类组成的复杂化合物[26],是构成植物细胞壁的成分之一[27]。由此推测苯丙烷生物合成相关的DEGs下调表达促进了植物细胞壁的解离过程,加速已分化细胞完成脱分化。研究发现,脂肪酸会对中柱鞘类细胞分生能力关键基因的表达进行抑制,从而对愈伤组织形成过程进行调控[28]。推测-亚麻酸是植物体内限制愈伤组织形成的关键信号物质。由于愈伤组织再生不定芽的过程快速且不易观察,此过程控制细胞谱系发展的机制并不十分清晰[29]。本研究对14 d和21 d筛选到的DEGs进行功能分析发现,DEGs主要与单细胞代谢过程、质体、ATP结合和有机环结合等功能相关。推测这些功能在离体叶片细胞从脱分化向再分化转变,完成不定芽的再生过程中发挥重要作用。
细胞分裂素、生长素是外植体再生不定芽所必需的,细胞分裂素与生长素比值高有助于外植体分化不定芽,而外源生长物质必须通过对内源激素平衡的调节才发挥作用,从而影响器官分化[30]。本研究发现生长素和细胞分裂素相关的DEGs上调表达和下调表达的数目大致相等,筛选到的与生长素相关的DEGs数目明显多于细胞分裂素。研究发现,生长素通过自身极性运输调节自身浓度梯度分布并决定干细胞的位置[31],生长素主要分布于将要产生茎顶端分生组织的愈伤组织周围,在离体器官再生过程中起到了决定性的作用[32]。推测植物主要通过调节内源生长素的浓度变化来促进离体叶片不定芽的再生。筛选到的生长素相关的DEGs中,MD02G1100200差异表达最为显著,在21 d和0 d的表达量相差200多倍,该基因序列与拟南芥中的序列高度相似。是生长素早期响应基因,其蛋白产物能够特异性结合生长素响应因子ARF,进而调控生长素响应基因的表达,在整个植物生长素信号转导过程中具有重要作用[33]。筛选到的细胞分裂素相关的DEGs中,MD15G1208300差异表达最为显著,在21 d和0 d的表达量相差160多倍,该基因序列与拟南芥中的序列高度相似,编码细胞分裂素氧化酶(CKX),在植物细胞内的细胞分裂素浓度调节中发挥重要作用[34]。推测MD02G1100200和MD15G1208300分别通过对生长素和细胞分裂素浓度的调节在苹果叶片再生不定芽的过程中发挥重要作用。植物中存在多个转录因子家族,相关报道表明AP2、LBD、bZIP、HD-ZIP、ARR家族的转录因子在不定芽再生过程起主要的转录调控作用[35]。LBD类转录因子中的LBD16、LBD17、LBD18受到生长素信号因子ARF7和ARF19的调控,超表达其中任何一个LBD转录因子都能促进植物外植体愈伤组织的形成[36]。拟南芥LBD转录因子还可以与bZIP家族转录因子bZIP59形成转录复合体来调控愈伤组织形成[37]。是植物不定芽再生的关键基因[38],细胞分裂素信号途径关键转录因子B型ARR可以直接结合到启动子区域,激活的表达[39]。此外,B型ARRs还可以与HD-ZIPIII特异性结合,形成转录复合体激活表达[22]。AP2家族转录因子有144个成员,参与植物多个生长发育过程[40],多个AP2家族成员参与植物离体器官不定芽再生过程,如生长素可以诱导、和的表达,而3个转录因子又可激活根分生组织的特征基因和以及芽再生特征基因和的表达,从而赋予愈伤组织再生芽的能力[41]。本研究筛选到了大量差异表达的AP2、LBD、bZIP、ARR家族转录因子,并且这些转录因子主要呈上调表达趋势,推测这些转录因子在一定程度上参与了叶片不定芽再生的调控过程。植物离体培养过程中,酶的活性和种类的变化会对离体器官的发生产生重要影响,与离体器官发生相关的酶类主要有SOD[42]、POD[43]、CAT[44]等。SOD在植物脱分化及愈伤组织、根、芽分化期间的活性一般增强,可以将IAA氧化分解,从而影响植物体内激素的平衡,是离体器官发生过程中重要的酶[45]。本研究筛选到的SOD相关的DEGs在不定芽再生过程中显著上调表达。推测SOD通过对内源生长素浓度的调节影响苹果叶片离体再生不定芽的过程。多胺是生物体内的一类活性物质,在植物细胞中主要有腐胺、精胺和亚精胺[46]。研究发现,内源激素对外植体形态发生的影响可能通过多胺来实现或协同作用,多胺在调控形态发生时主要起第二信使的作用[47-48]。本研究筛选到的腐胺、精胺和亚精胺相关的DEGs均显著上调表达,推测在苹果叶片离体再生过程中,植物激素变化引起细胞内多胺变化,随后引起生理生化变化,最终影响到形态发生。
4 结论
再生培养基上培养3、7、14和21 d的苹果叶片外植体与对照组相比,分别筛选到5 250、4 937、6 852和6 493个DEGs,4个时间点共有的DEGs有3 027个。4个时间点共有的DEGs主要与分子功能、细胞成分和生物过程相关,显著富集在植物激素信号转导、磷酸戊糖途径、苯丙烷生物合成和-亚麻酸合成等途径中。苹果叶片外植体在外源激素的诱导下,通过对大量植物再生相关基因的转录激活,对植物体内多种功能和代谢途径进行调节,从而使离体叶片通过愈伤组织分化产生不定芽,内源IAA和CTK的相互作用在这个过程中发挥至关重要的调控作用。
[1] 丛佩华, 张彩霞, 韩晓蕾, 田义, 张利义, 李武兴. 我国苹果育种研究现状及展望. 中国果树, 2018(6): 1-5.
CONG P H, ZHANG C X, HAN X L, TIAN Y, ZHANG L Y, LI W X. Current research situation and prospect of apple breeding in China. China Fruits, 2018(6): 1-5. (in Chinese)
[2] VELASCO R, ZHARKIKH A, AFFOURTIT J, DHINGRA A, CESTARO A, KALYANARAMAN A, FONTANA P, BHATNAGAR S K, TROGGIO M, PRUSS D, SALVI S, PINDO M, BALDI P, CASTELLETTI S, CAVAIUOLO M, COPPOLA G, COSTA F, COVA V, Dal RI A, GOREMYKIN V,. The genome of the domesticated apple (×Borkh.). Nature Genetics, 2010, 42(10): 833-839.
[3] 常源升, 程来亮, 王海波, 何平, 李慧峰, 李林光. 苹果分子标记及辅助育种研究进展. 园艺学报, 2017, 44(9): 1658-1680.
CHANG Y S, CHENG L L, WANG H B, HE P, LI H F, LI L G. Review of molecular marker and marker assisted breeding of apple. Acta Horticulturae Sinica, 2017, 44(9): 1658-1680. (in Chinese)
[4] JAMES D J, PASSEY A J, BARBARA D J, BEVAN M. Genetic transformation of apple (Mill.) using a disarmed Ti-binary vector. Plant Cell Reports, 1989, 7(8): 658-661.
[5] EUN S S, KWAN J S, SUNG J, CHANG Y Y, ILL M C. Silver nitrate and aminoethoxyvinylglycine affect Agrobacterium -mediated apple transformation. Plant Growth Regulation, 2005, 45(1): 75-82.
[6] DAI H Y, LI W R, HAN G F, YANG Y, MA Y E, LI H, ZHANG Z H. Development of a seedling clone with high regeneration capacity and susceptibility toin apple. Scientia Horticulturae, 2013, 164: 202-208.
[7] VIDAL N, MALLÓN R, VALLADARES S, MEIJOMÍN A M, VIEITEZ A M. Regeneration of transgenic plants by- mediated transformation of somatic embryos of juvenile and mature. Plant Cell Reports, 2010, 29(12): 1411-1422.
[8] RICHARD L B, RALPH S, DELORES L. Adventitious shoot regeneration of pear (spp.) genotypes. Plant Cell, Tissue and Organ Culture (PCTOC), 2012, 108(2): 229-236.
[9] 赵政阳, 付润民, 税守岐, 张秀琴, 黄英. 苹果试管苗叶片再生植株研究. 陕西农业科学, 1992(6): 18-19.
ZHAO Z Y, FU R M, SHUI S Q, ZHANG X Q, HUANG Y. Study on the regeneration of apple plantlets from the leaves. Shaanxi Journal of Agricultural Sciences, 1992(6): 18-19. (in Chinese)
[10] IIZASA S, IIZASA E, WATANABE K, NAGANO Y. Transcriptome analysis reveals key roles of AtLBR-2 in LPS-induced defense responses in plants. BMC Genomics, 2017, 18(1): 995.
[11] 向亚男, 黄蕊蕊, 顾婷婷, 甘立军. 基于RNA-Seq的拟南芥不定芽再生过程的基因表达谱分析. 南京农业大学学报, 2018, 41(2): 308-320.
XIANG Y N, HUANG R R, GU T T, GAN L J. Analysis of RNA- Seq-based expression profiles during adventitious shoot regeneration in. Journal of Nanjing Agricultural University, 2018, 41(2): 308-320. (in Chinese)
[12] CHE P, LALL S, NETTLETON D, HOWELL S H. Gene expression programs during shoot, root, and callus development intissue culture. Plant Physiology, 2006, 141(2): 620-637.
[13] MAYER K F X, SCHOOF H, HAECKER A, LENHARD M, JÜRGENS G, LAUX T. Role of WUSCHEL in regulating stem cell fate in theshoot meristem. Cell, 1998, 95(6): 805-815.
[14] ZHANG T Q, LIAN H, TANG H B, DOLEZAL K, ZHOU C M, YU S, CHEN J H, CHEN Q, LIU H T, LJUNG K, WANG J W. An intrinsic MicroRNA timer regulates progressive decline in shoot regenerative capacity in plants. The Plant Cell, 2015, 27(2): 349-360.
[15] HIROYA I, KAORU S, PAUL T T, HARUKA T, SATOSHI K, YAYOI I, TAKUYA S, TAKU S, MITSUHIRO A, TAKAMASA S, SOICHI I, KENGO M, MOTOAKI S, TETSUJI K, ELLIOT M M, SACHIHIRO M. Primed histone demethylation regulates shoot regenerative competency. Nature Communications, 2019, 10(1): 1786.
[16] LI H, DURBIN R. Fast and accurate short read alignment with Burrows-Wheeler transform. Bioinformatics, 2009, 25(14): 1754-1760.
[17] OH Y, DONOFRIO N, PAN H Q, COUGHLAN S, BROWN D E, MENG S W, MITCHELL T, DEAN R A. Transcriptome analysis reveals new insight into appressorium formation and function in the rice blast fungus. Genome Biology, 2008, 9(5): R85.
[18] LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) Method. Methods (San Diego, Calif.), 2001, 25(4): 402-408.
[19] ATTA R, LAURENS L, BOUCHERON-DUBUISSON E, GUIVARC'H A, CARNERO E, GIRAUDAT-PAUTOT V, RECH P, CHRIQUI D. Pluripotency ofxylem pericycle underlies shoot regeneration from root and hypocotyl explants grown. The Plant Journal, 2009, 57(4): 626-644.
[20] SUGIMOTO K, JIAO Y L, MEYEROWITZ E M.regeneration from multiple tissues occurs via a root development pathway. Developmental Cell, 2010, 18(3): 463-471.
[21] DUCLERCQ J, SANGWAN-NORREEL B, CATTEROU M, SANGWAN R S. De novo shoot organogenesis: from art to science. Trends in Plant Science, 2011, 16(11): 597-606.
[22] ZHANG T Q, LIAN H, ZHOU C M, XU L, JIAO Y L, WANG J W. A two-step model for de novo activation of WUSCHEL during plant shoot regeneration. The Plant Cell, 2017, 29(5): 1073-1087.
[23] GRAFI G, BEN-MEIR H, AVIVI Y, MOSHE M Y, DAHAN Y, ZEMACH A. Histone methylation controls telomerase-independent telomere lengthening in cells undergoing dedifferentiation. Developmental Biology, 2007, 306(2): 838-846.
[24] WILLIAMS A C, FORD W C L. Functional significance of the pentose phosphate pathway and glutathione reductase in the antioxidant defenses of human sperm. Biology of Reproduction, 2004, 71(4): 1309-1316.
[25] FAJKUS J, FULNEČKOVÁ J, HULÁNOVÁ M, BERKOVÁ K, ŘÍHA K, MATYÁŠEK R. Plant cells express telomerase activity upon transfer to callus culture, without extensively changing telomere lengths. Molecular and General Genetics MGG, 1998, 260(5): 470-474.
[26] BHATIA R, DALTON S, ROBERTS L A, MORON-GARCIA O M, IACONO R, KOSIK O, GALLAGHER J A, BOSCH M. Modified expression of ZmMYB167 inandleads to increased cell wall lignin and phenolic content. Scientific Reports, 2019, 9(1): 8800.
[27] TAKEDA Y, KOSHIBA T, TOBIMATSU Y, SUZUKI S, MURAKAMI S, YAMAMURA M, RAHMAN M M, TAKANO T, HATTORI T, SAKAMOTO M, UMEZAWA T. Regulation of CONIFERALDEHYDE 5-HYDROXYLASE expression to modulate cell wall lignin structure in rice. Planta, 2017, 246(2): 337-349.
[28] SHANG B, XU C, ZHANG X, CAO H, XIN W, HU Y. Very-long- chain fatty acids restrict regeneration capacity by confining pericycle competence for callus formation in. Proceedings of the National Academy of Sciences of the United States of America, 2016, 113(18): 5101-5106.
[29] 孙贝贝, 刘杰, 葛亚超, 盛李宏, 陈吕琴, 胡小梅, 杨仲南, 黄海, 徐麟. 植物再生的研究进展. 科学通报, 2016, 61(36): 3887-3902.
SUN B B, LIU J, GE Y C, SHENG L H, CHEN L Q, HU X M, YANG Z N, HUANG H, XU L. Recent progress on plant regeneration. Chinese Science Bulletin, 2016, 61(36): 3887-3902. (in Chinese)
[30] GAJ M D. Factors influencing somatic embryogenesis induction and plant regeneration with particular reference to(L.) heynh. Plant Growth Regulation, 2004, 43(1): 27-47.
[31] GRIENEISEN V A, XU J, MARÉE A F M, HOGEWEG P, SCHERES B. Auxin transport is sufficient to generate a maximum and gradient guiding root growth. Nature, 2007, 449(7165): 1008-1013.
[32] GORDON S P, HEISLER M G, REDDY G V, OHNO C, DAS P, MEYEROWITZ E M. Pattern formation during de novo assembly of theshoot meristem. Development (Cambridge, England), 2007, 134(19): 3539-3548.
[33] ANGELA K S, SANG H L, JONATHAN P W, NATHALIE G, HIRONORI I, DIRK I, WENDY A P, ANGUS S M, PAUL J O, WILLIAM M G. The SAUR19 subfamily of SMALL AUXIN UP RNA genes promote cell expansion. The Plant Journal, 2012, 70(6): 978-990.
[34] BARTRINA I, OTTO E, STRNAD M, WERNER T, SCHMÜLLING T. Cytokinin regulates the activity of reproductive meristems, flower organ size, ovule formation, and thus seed yield in. The Plant Cell, 2011, 23(1): 69-80.
[35] 许智宏, 张宪省, 苏英华, 胡玉欣, 徐麟, 王佳伟. 植物细胞全能性和再生. 中国科学(生命科学), 2019, 49(10): 1282-1300.
XU Z H, ZHANG X S, SU Y H, HU Y X, XU L, WANG J W. Plant cell totipotency and regeneration. Science in China (Series C), 2019, 49(10): 1282-1300. (in Chinese)
[36] FAN M Z, XU C Y, XU K, HU Y X. LATERAL ORGAN BOUNDARIES DOMAIN transcription factors direct callus formation inregeneration. Cell Research, 2012, 22(7): 1169-1180.
[37] XU C Y, CAO H F, ZHANG Q Q, WANG H Z, XIN W, XU E J, ZHANG S Q, YU R X, YU D X, HU Y X. Control of auxin-induced callus formation by bZIP59–LBD complex in Arabidopsis regeneration. Nature Plants, 2018, 4(2): 108-115.
[38] LAUX T, MAYER K F, BERGER J, JÜRGENS G. The WUSCHEL gene is required for shoot and floral meristem integrity in. Development (Cambridge, England), 1996, 122(1): 87-96.
[39] MENG W J, CHENG Z J, SANG Y L, ZHANG M M, RONG X F, WANG Z W, TANG Y Y, ZHANG X S. Type-BRESPONSE REGULATORs specify the shoot stem cell niche by dual regulation of WUSCHEL. The Plant Cell, 2017, 29(6): 1357-1372.
[40] HORSTMAN A, WILLEMSEN V, BOUTILIER K, HEIDSTRA R. AINTEGUMENTA-LIKE proteins: hubs in a plethora of networks. Trends in Plant Science, 2014, 19(3): 146-157.
[41] KAREEM A, DURGAPRASAD K, SUGIMOTO K, DU Y J, PULIANMACKAL A J, TRIVEDI Z B, ABHAYADEV P V, PINON V, MEYEROWITZ E M, SCHERES B, PRASAD K. PLETHORA genes control regeneration by a two-step mechanism. Current Biology, 2015, 25(8): 1017-1030.
[42] SHAFI A, GILL T, SREENIVASULU Y, KUMAR S, AHUJA P S, SINGH A K. Improved callus induction, shoot regeneration, and salt stress tolerance inoverexpressing superoxide dismutase from. Protoplasma, 2015, 252(1): 41-51.
[43] TANG W, HARRIS L C, OUTHAVONG V, NEWTON R J. Antioxidants enhanceplant regeneration by inhibiting the accumulation of peroxidase inpine (Mill.). Plant Cell Reports, 2004, 22(12): 871-877.
[44] SRIVASTAVA S, DWIVEDI U N. Plant regeneration from callus of Cuscuta reflexa-an angiospermic parasite- and modulation of catalase and peroxidase activity by salicylic acid and naphthalene acetic acid. Plant Physiology & Biochemistry, 2001, 39(6): 529-538.
[45] CHAI M L, JIA Y F, CHEN S, GAO Z S, WANG H F, LIU L L, WANG P J, HOU D Q. Callus induction, plant regeneration, and long-term maintenance of embryogenic cultures in[L.] Merr. Plant Cell, Tissue and Organ Culture (PCTOC), 2011, 104(2): 187-192.
[46] FLORES H E, GALSTON A W. Osmotic stress-induced polyamine accumulation in cereal leaves I. physiological parameters of the response. Plant Physiology, 1984, 75(1): 102-109.
[47] SHOEB F, YADAV J S, BAJAJ S, RAJAM M V. Polyamines as biomarkers for plant regeneration capacity: Improvement of regeneration by modulation of polyamine metabolism in different genotypes of indica rice. Plant Science, 2001, 160(6): 1229-1235.
[48] MUKHOPADHYAY A, CHOUDHURI M M, SEN K, GHOSH B. Changes in polyamines and related enzymes with loss of viability in rice seeds. Phytochemistry, 1983, 22(7): 1547-1551.
Identification and Analysis of Differentially Expressed Genes in Adventitious Shoot Regeneration in Leaves of Apple
LIU Kai, HE ShanShan, ZHANG CaiXia, Zhang LiYi, BIAN ShuXun, YUAN GaoPeng, LI WuXing, KANG LiQun, CONG PeiHua, HAN XiaoLei
Research Institute of Pomology, Chinese Academy of Agricultural Sciences/Key Laboratory of Horticultural Crop Germplasm Resources Utilization, Ministry of Agriculture and Rural Areas/National Apple Breeding Center, Xingcheng 125100, Liaoning
【】In this study, the differentially expressed genes (DEGs) in adventitious shoot regeneration of ‘GL-3’ apple leaves were screened. The potential mechanism of adventitious shoot regeneration of apple leaves was analyzed, which will contribute to develop an efficient genetic transformation system for apple. 【】The explants of ‘Gl-3’ apple were cultured on regeneration medium. Samples were taken for RNA extraction and construction of mRNA library at 3, 7, 14 and 21 d post culture, respectively, further sequenced on the Illumina Nova seq platform. On the basis of the Kyoto Encyclopedia of Gene and Genome (KEGG) and Gene ontology (GO), the terms and pathway enrichment were then analyzed using the Phyper function with R software. Gene annotation was performed by using BLAST software. The DEGs related to plant regeneration, such as hormones, enzymes, transcription factors (TFs) and polyamines were analyzed, the expression levels of DEGs were verified by qRT-PCR. 【】Compared with the control group, 5 250, 4 937, 6 852 and 6 493 DEGs were identified at 3, 7, 14 and 21 d post culture, respectively, and 3 027 DEGs were shared in all four points. GO functional enrichment analysis showed that the up-regulated DEGs shared in all four points were mainly related to oxidation reduction process, cell periphery, protein kinase activity and organic cyclic compound binding, while the down-regulated DEGs were mainly related to single organism metabolic process, calcium ion binding, photosynthetic membrane and thylakoid part. KEGG pathway enrichment analysis indicated that the up-regulated DEGs shared in all four points were significantly enriched in pentose phosphate pathway, plant hormone signal transduction, plant pathogen interaction and protein processing in endoplasmic reticulum, while the down-regulated DEGs were significantly enriched in alpha linolenic acid metabolism, phenylpropanoid biosynthesis, carbon metabolism and photosynthesis. In addition, the DEGs encoding transcription factors, enzymes, and components of hormone biosynthesis and signaling pathways were analyzed. The results of qRT-PCR showed that most of these DEGs were up-regulated, which was consistent with data of RNA-Seq. 【】Through the detection and comparative analysis of large-scale gene expression profiles in adventitious shoot of ‘GL-3’ apple leaves at different time points, a number of genes related to adventitious shoot regeneration of apple leaves were obtained, which could provide a basis for further study on the mechanism of apple leaves in vitro regeneration.
apple; leaves regeneration; RNA-Seq; differentially expressed genes; impact factors

10.3864/j.issn.0578-1752.2021.16.011
2020-09-19;
2021-01-07
中央级公益性科研院所基本科研业务费专项(Y2019XK09)、国家现代农业产业技术体系建设专项(CARS-27)、中国农业科学院科技创新工程(CAAS-ASTIP-2016-RIP-02)
刘锴,E-mail:liukai2429@163.com。通信作者韩晓蕾,E-mail:hanxiaolei@caas.cn
(责任编辑 赵伶俐)

