假禾谷镰孢转录因子FpAPSES的鉴定与功能分析
赵静雅,夏荟清,彭梦雅,凡卓,殷悦,徐赛博,张楠,陈文波,陈琳琳
假禾谷镰孢转录因子FpAPSES的鉴定与功能分析
赵静雅,夏荟清,彭梦雅,凡卓,殷悦,徐赛博,张楠,陈文波,陈琳琳
河南农业大学植物保护学院/小麦玉米作物学国家重点实验室,郑州 450002
【】假禾谷镰孢()侵染小麦引起的小麦茎基腐病严重影响中国小麦的安全生产。查找假禾谷镰孢中的APSES转录因子,分析其在病原菌致病过程中的作用,为解析假禾谷镰孢的致病机制及小麦茎基腐病的防治提供理论依据。从GenBank获得物种中已知的APSES氨基酸序列,利用BLASTP方法在假禾谷镰孢中查找APSES同源蛋白,利用pfam软件预测蛋白结构域,采用MEGA5.05构建APSES蛋白的系统进化树。利用实时荧光定量PCR(qRT-PCR)方法分析A组APSES转录因子基因和在假禾谷镰孢侵染过程中的表达。通过PEG介导的原生质体转化和PCR筛选和基因缺失的突变体菌株。在PDA培养基上测定假禾谷镰孢野生型菌株Wz2-8A、Δ和Δ突变体菌株的菌丝形态和生长速率;测定在CMC培养液中培养后的分生孢子产生情况、形态以及在无菌水中的萌发率;利用菌丝块和分生孢子接种小麦胚芽鞘和大麦叶片以及盆栽试验测定其致病性;采用ELISA方法测定小米培养基中脱氧雪腐镰刀菌烯醇(DON)的含量。假禾谷镰孢中有4个APSES同源蛋白,均含有保守的DNA结合结构域HTH。与其他物种的APSES蛋白构建系统进化树,发现FpAPSES1、FpAPSES2和FpAPSES4属于A组APSES,FpAPSES3分布在C组。和在侵染阶段诱导表达,推测可能参与假禾谷镰孢的致病。分别获得2个和基因缺失的突变体菌株Δ-T10、Δ-T27和Δ-T1、Δ-T2。表型测定结果显示,与野生型相比,和基因缺失突变体在PDA平板上的生长速率明显减慢、色素积累明显增多;基因缺失突变体菌丝形态无差异,而基因缺失突变体菌丝弯曲、分支明显增多;和基因缺失突变体在CMC液体中的分生孢子产生明显减少,分别比野生型下降了99.5%和97.4%,产生的分生孢子变短、隔膜减少、萌发率有所降低;与野生型相比,和基因缺失突变体菌丝体和分生孢子对小麦胚芽鞘的致病力明显降低,菌丝在小麦胚芽鞘表皮细胞中的扩展明显受阻;和基因缺失突变体对大麦叶片和小麦根部的致病力也明显降低;和基因缺失突变体在小米培养基中产生的DON毒素分别比野生型下降了约78%和44%。编码A组APSES同源蛋白的和对假禾谷镰孢的菌丝生长、分生孢子产生和致病力均具有重要作用。
小麦茎基腐病;假禾谷镰孢;APSES转录因子;致病力
0 引言
【研究意义】假禾谷镰孢()是一种土传病原真菌,其引起的小麦茎基腐病已成为威胁中国麦区小麦生产的主要病害[1-2]。假禾谷镰孢也可以侵染小麦穗部造成赤霉病,并产生脱氧雪腐镰刀菌烯醇(deoxynivalenol,DON)等毒素危害人和家畜的健康[3-4]。但是,目前对该病菌了解不深,尤其缺乏抗病小麦品种及有效防治策略。研究假禾谷镰孢致病的分子机制,对制定合理有效的小麦茎基腐病和赤霉病防治策略具有重要意义。【前人研究进展】转录因子与特定的DNA位点结合,通过DNA-蛋白互作、蛋白-蛋白互作或者染色质结构的改变来激活或抑制基因的转录。APSES(Asm1p、Phd1p、Sok2p、Efg1p和StuAp)蛋白是在真菌中非常保守的一类转录因子,广泛作用于真菌的生长、发育和次级代谢等过程[5]。APSES类转录因子包含一段约100 bp的保守的DNA结合结构域,其核心结构域可以形成一个典型的基础螺旋-环-螺旋(bHLH/HTH)结构[6]。在不同真菌中,APSES类转录因子可以分成A、B、C和D 4个组,A组包括Mbp1、Swi4和Swi6蛋白,主要通过调控细胞周期作用于真菌的生长发育。在稻瘟病菌()中,MoSwi6与丝裂原活化激酶(MAPK)MoMps1互作,Δ突变体在菌丝生长、分生孢子形态、附着胞形成和致病力等方面表现异常[7]。B组由Xbp1蛋白组成[8]。C组APSES蛋白在不同真菌中研究报道比较多,又分成C-Ⅰ(Efg1、Efh1、Phd1和Sok2)和C-Ⅱ(StuA/Asm1 的同源蛋白)。在酿酒酵母()中,Phd1在营养缺陷条件中能够激活酵母假菌丝的生长[9];白色念珠菌()Efg1及其同源蛋白Efh1p主要作用于真菌的形态建成、代谢、生物膜形成和致病力等[10-11];StuA/Asm1是最先鉴定到的APSES转录因子,其同源蛋白的功能已在多种植物病原真菌中报道是真菌生长、产孢和侵染等过程的关键因子,包括稻瘟病菌的Mstu1、禾谷镰孢()的FgStuAp和拟轮枝镰孢()的FvStuA等[12-15]。D组APSES蛋白目前主要通过生物信息学预测,其功能还不明确。【本研究切入点】目前,APSES类转录因子在假禾谷镰孢中的分布和功能还未见报道。【拟解决的关键问题】通过生物信息学系统分析假禾谷镰孢中的APSES转录因子,并通过实时荧光定量PCR(qRT-PCR)、遗传转化和生物学表型分析,探讨A组APSES转录因子FpAPSES1和FpAPSES4的功能,为进一步揭示APSES类转录因子在植物病原真菌中的作用以及解析假禾谷镰孢的致病机制提供理论依据。
1 材料与方法
试验于2019年5月至2020年11月在河南农业大学农作物病害检测与防控实验室完成。
1.1 材料
假禾谷镰孢菌株Wz2-8A由河南农业大学农作物病害检测与防控实验室分离并保存。
假禾谷镰孢Wz2-8A高感小麦品种矮抗58和周麦27由国家小麦工程技术研究中心提供,啤酒大麦种子购于河北兴农富民种子销售有限公司。
1.2 方法
1.2.1 FpAPSES蛋白的筛选与分析 根据酿酒酵母、粗糙脉孢菌()、构巢曲霉()和稻瘟病菌中已知的APSES类蛋白,利用BLASTP在假禾谷镰孢基因组数据库中查找同源蛋白[16]。获得的候选蛋白利用Pfam软件分析蛋白结构域[17],具有保守HTH结构域的蛋白被认定为APSES同源蛋白。采用MEGA 5.05软件构建APSES蛋白的系统发育进化树[18]。
1.2.2 实时荧光定量PCR 假禾谷镰孢野生型菌株Wz2-8A在马铃薯琼脂培养基(potato dextrose agar,PDA:马铃薯200 g、葡萄糖20 g、琼脂粉20 g,水1 L)上活化后转接到新的PDA培养平板上,25℃、黑暗培养3 d,用打孔器在菌落边缘取直径5 mm的菌饼置在分生孢子诱导培养基(carboxymethyl cellulose,CMC:carboxymethyl cellulose 10 g、NH4NO31 g、酵母提取物1 g、MgSO4·7H2O 0.5 g,水1 L)中,25℃、150 r/min摇培3 d收集分生孢子。将分生孢子在YEPD培养基中,25℃、150 r/min摇培20 h收集菌丝体。对假禾谷镰孢感病的小麦品种矮抗58种子在25℃、保湿萌发生长4 d,取2 µL分生孢子悬浮液(1×107/mL)接种于小麦幼苗胚芽鞘,25℃、黑暗、保湿培养,分别在侵染18、30、48 h、3、5和7 d取样,置于液氮中保存备用。
利用RNAprep Pure多糖多酚植物总RNA提取试剂盒(天根,北京)对上述收集的样品提取总RNA。利用PrimeScriptTMRT reagent Kit with gDNA Eraser试剂盒(宝生物,大连)去除基因组DNA,并合成cDNA。采用实时荧光定量PCR(Applied Biosystems 7300,美国)方法检测在不同阶段的表达水平,作为内参基因,所用引物见表1。荧光定量PCR反应体系:TB Green Premix Ex TaqⅡ6 µL,Primer F(10 µmol·L-1)0.5 µL,Primer R(10 µmol·L-1)0.5 µL,cDNA 1 µL,ddH2O 4 µL。反应程序:95℃ 2min预变性,40个循环(95℃15 s,60℃15 s,68℃20 s),95℃15 s,60℃ 15 s和95℃ 15 s,获得熔解曲线。将假禾谷镰孢菌丝体待分析基因表达量定为1,分析比较相应基因在分生孢子和侵染过程中的相对表达量。
1.2.3 基因敲除及检测 为了获得基因缺失的突变体菌株,利用split-marker策略获得目标基因同源重组序列[19-20],所用引物见表1。第1轮PCR扩增,以假禾谷镰孢菌丝体基因组DNA为模板,利用引物F1+R1和F2+R2分别进行PCR扩增获得目标基因的上游(A1)和下游(A2)约1 000 bp的DNA序列;以pKOV21(含潮霉素基因)载体为模板,利用引物HYG/F+HYG/R进行PCR扩增获得潮霉素片段(H)。PCR反应体系包含Premix Taq 25 µL,正反向引物各1 µL,假禾谷镰孢基因组DNA 100 ng,用ddH2O补足50 µL。PCR反应条件为94℃5 min预变性,35个循环(94℃30 s,58℃30 s,72℃1 min),72℃10 min再延伸。第2轮PCR扩增将以第1轮PCR纯化后产物以A1﹕A2﹕H=1﹕1﹕3浓度比混合作为模板。反应体系为10×LA Taq buffer 5 µL,dNTPs 2 µL,LA Taq 0.2 μL,ddH2O补足50 µL。PCR反应条件为94℃ 5 min预变性,15个循环(94℃30 s,58℃30 s),72℃3.5 min再延伸。第3轮PCR扩增将直接以第2轮PCR扩增产物为模板,分别利用引物F1+HY/R和YG/F+R1进行PCR扩增。PCR反应体系为Premix Taq 25 µL,引物2 µL,模板2 µL,ddH2O补足50 µL。PCR反应程序为95℃5 min预变性,35个循环(95℃30 s,58℃30 s,72℃2 min),72℃10 min再延伸。第3轮PCR扩增获得的产物纯化后通过PEG介导的原生质体转化方法导入假禾谷镰孢野生型菌株原生质体中[21]。
利用潮霉素抗性筛选阳性转化子。利用引物H2F/H2R、F3/H1R、H1F/R3和G1+G2依次进行PCR扩增转化子中的潮霉素基因、目标基因上游和潮霉素融合片段、潮霉素和目标基因下游融合片段以及目标基因,确定目标基因被潮霉素替换的转化子。

表1 本研究中所用的引物
1.2.4 菌丝生长测定 假禾谷镰孢野生型和突变体菌株活化后,转接到新的PDA平板上,25℃、黑暗培养3 d,利用直径5 mm的打孔器取菌落边缘菌丝块,转接到新的PDA平板上,25℃、黑暗培养3 d,观察菌落形态、色素积累,拍照并测量菌落直径。将灭菌的载玻片蘸取融化好的PDA培养基,放置在培养皿中,将菌丝块接种在载玻片上。待长出菌落后,盖上盖玻片在显微镜下观察并拍照。所有试验重复3次,每次试验每个样品设置3个重复。利用T测验统计分析样品间的差异。
1.2.5 分生孢子产生及其萌发测定 假禾谷镰孢野生型和突变体菌株活化后,转接到新的PDA平板上,25℃、黑暗培养3 d,利用直径5 mm的打孔器取两块边缘菌丝块,转接60 mL CMC培养液中诱导分生孢子的产生,野生型和突变体菌株在25℃、150 r/min摇培3—7 d。用一层miracloth滤布过滤收集分生孢子,用血球计数板计数,并用显微镜观察分生孢子的形态。收集的分生孢子用无菌水清洗两遍,制备浓度为4×105/mL的孢子悬浮液。取100 μL置于灭菌载玻片上,25℃、黑暗保湿培养3—6 h,取10 μL经萌发处理孢子悬浮液滴于新的载玻片上,用显微镜观察分生孢子萌发芽管长度并统计萌发数量。所有试验重复3次,每次试验每个样品设置3个生物学重复。利用T测验统计分析样品间的差异。
1.2.6 致病力测定 假禾谷镰孢野生型和突变体菌株活化后,转接到新的PDA平板上,25℃、黑暗培养3 d,待用。选择在温室内对假禾谷镰孢感病的小麦品种周麦27于培养砵中,25℃、16 h光照﹕8 h黑暗条件下培养4 d,至小麦胚芽鞘长约5 cm时备用。利用直径5 mm的打孔器取活化的假禾谷镰孢菌落边缘菌丝块置于胚芽鞘上,25℃、黑暗、保湿培养24 h,移去菌丝块并将小麦移到25℃、16 h光照﹕8 h黑暗条件下继续培养3 d,拍照并记录病斑长度。撕取接种的小麦胚芽鞘内表皮,利用尼康Ti-s显微镜观察假禾谷镰孢菌丝在小麦细胞中的侵染和扩展(因突变体菌株生长减慢,侵染延长至5 d)。收集分生孢子配制成1×106/mL浓度的孢子悬浮液。将0.5 cm×5 cm的灭菌吸水纸缠绕在生长3 d的小麦胚芽鞘上,并滴加50 μL分生孢子悬浮液,25℃、黑暗保湿培养4 d,拍照并记录病斑长度。
啤酒大麦种子种于培养砵中,25℃、16 h光照﹕8 h黑暗培养7 d。利用直径5 mm的打孔器取活化的假禾谷镰孢菌落边缘菌丝块置于离体叶片上,每片叶接种两个菌丝块。25℃、黑暗、保湿培养24 h,移去菌丝块继续培养3 d,拍照并记录病斑直径。
根据实验室已发表的盆栽试验方法[22],分别将活化的假禾谷镰孢野生型和突变体菌丝块接种到小米培养基上扩繁,25℃培养7 d。按质量比0.5%的比例将菌混入灭菌土中,每个种植钵放置200 g菌土,对照为等比例的小米和无菌土。将催芽后的周麦27小麦种子播于种植钵内。25℃、16 h光照﹕8 h黑暗培养7 d,拍照并通过观察小麦的株高、根系长度以及叶鞘和茎秆变褐程度确定小麦茎基腐病发病程度。
每种致病力测定试验至少重复3次,每次试验每个样品设置5个重复。利用T测验统计分析样品间的差异。
1.2.7 DON毒素测定 将假禾谷镰孢野生型和突变体菌株在PDA培养基上活化,用直径5 mm的打孔器取菌落边缘菌丝块转接到小米培养基中,25℃培养10 d至小米表面完全长满菌。取5 g小米样品粉碎后,利用北京华安麦科生物技术有限公司的呕吐毒素ELISA检测试剂盒(HEM0896),参照说明书操作,检测接种不同假禾谷镰孢菌株的小米培养基中的DON即呕吐毒素的含量。因Δ-T10菌株接种小米培养基后有细菌污染,所以未测定。试验重复3次,每次试验每个处理重复3次,数据利用T测验进行统计分析。
2 结果
2.1 FpAPSES蛋白的筛选与分析
根据物种中已知的APSES类转录因子,通过BLASTP的方法,在假禾谷镰孢中查找到5个APSES候选蛋白。利用Pfam预测候选蛋白的功能域(图 1-A),发现FPSE_10424、FPSE_07209、FPSE_02622和FPSE_07396包含APSES转录因子保守的DNA结合结构域HTH,初步确定为APSES同源蛋白,并根据其与禾谷镰孢APSES蛋白的同源性,分别命名为FpAPSES1、FpAPSES2、FpAPSES3和FpAPSES4。候选蛋白FPSE_05854无明显的HTH结构域。从表2可以看出,FpAPSES蛋白分子量较大,除FpAPSES2外,其余3个FpAPSES蛋白分子量均在75 kD以上;FpAPSES蛋白均呈弱酸性。

A:FpAPSES蛋白结构域分析 Domains analysis of FpAPSES proteins;B:FpAPSES蛋白进化分析 Phylogenetic analysis of FpAPSES proteins
将FpAPSES蛋白与物种中已知的APSES蛋白构建系统进化树(图1-B),结果显示FpAPSES1、FpAPSES2和FpAPSES4属于A组APSES蛋白,FpAPSES3分布在C组。分析不同物种APSES蛋白的相似度可以发现,FpAPSES与禾谷镰孢的APSES蛋白同源性最高,其次分别为曲霉、稻瘟病菌、粗糙脉孢菌和酿酒酵母,符合物种进化的方向。

表2 FpAPSES转录因子的基本特性
2.2 FpAPSES1和FpAPSES4在侵染阶段诱导表达
C组APSES蛋白在植物病原真菌中的功能已有较多报道,而A组APSES蛋白在镰孢菌中的功能还不清楚,因此在假禾谷镰孢中选择了两个A组APSES同源蛋白FpAPSES1和FpAPSES4做进一步的功能分析。为了预测FpAPSES1和FpAPSES4在病原菌致病中的作用,利用qRT-PCR方法分析其在假禾谷镰孢侵染过程中的表达(图2)。结果显示和在侵染阶段均明显被诱导表达,其中在侵染48 h表达量最高,与菌丝阶段相比,上调表达约8倍。而在侵染18 h和5 d时表达量最高,是菌丝阶段表达量的7倍以上。以上结果说明,FpAPSES1和FpAPSES4可能参与假禾谷镰孢的致病过程。

图2 FpAPSES1和FpAPSES4在假禾谷镰孢侵染阶段的表达分析
2.3 FpAPSES1和FpAPSES4基因缺失突变体的构建
为了分析和在假禾谷镰孢中的生物学功能,利用split-marker PCR的方法分别扩增获得和基因上游-潮霉素以及潮霉素-目标基因下游的融合片段(图3-A)。通过PEG介导的遗传转化方法将重组片段转入假禾谷镰孢Wz2-8A菌株的原生质体中,利用潮霉素筛选阳性菌株,通过PCR检测和基因缺失的菌株。如图3-B所示,在转化子Δ-T10和Δ-T27中能够检测到潮霉素(750 bp)、上游-潮霉素(1 598 bp)以及潮霉素-1下游(1 508 bp)的融合片段,但无法检测到(811 bp),因此获得2个基因缺失的突变体菌株。如图3-C所示,在转化子Δ-T1和Δ-T2中能够检测到潮霉素(750 bp)、上游-潮霉素(1 540 bp)以及潮霉素-下游(1 520 bp)融合片段,但是无法检测到(975 bp),同样获得2个基因缺失的突变体菌株。
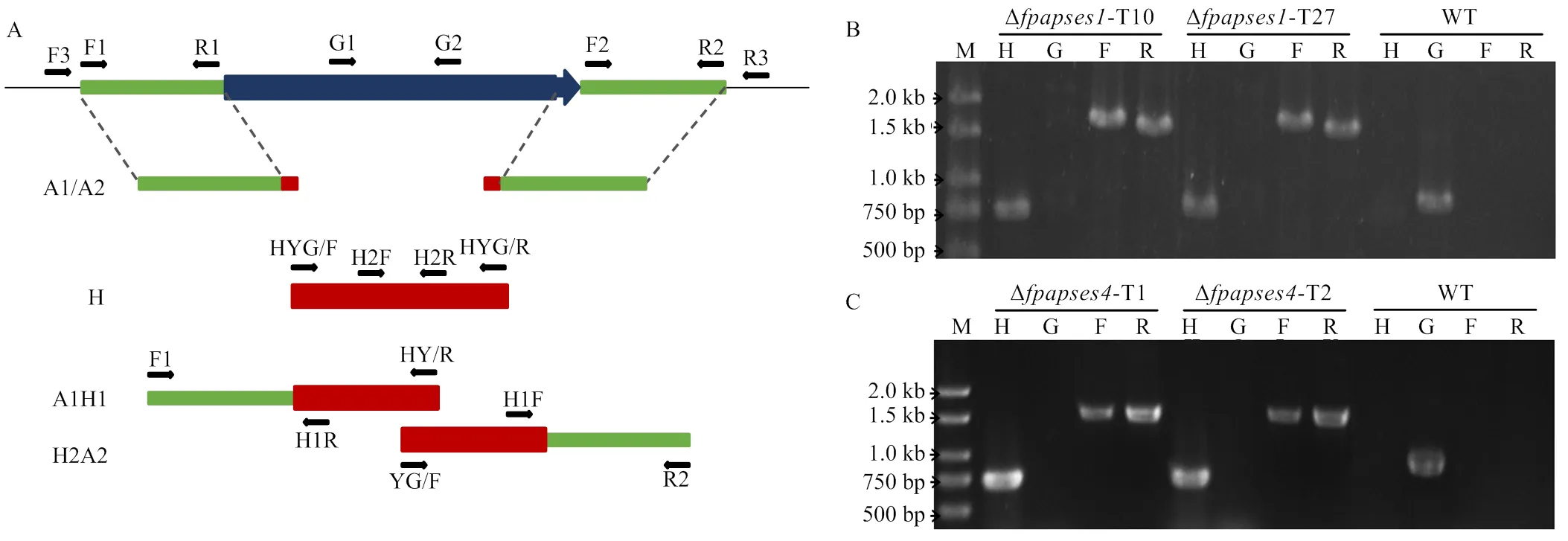
A:假禾谷镰孢基因敲除策略及引物示意图Schematic representation of the target gene deletion strategy and primers;B:FpAPSES1基因缺失突变体PCR检测PCR products for the FpAPSES1 gene replacement construct;C:FpAPSES4基因缺失突变体PCR检测PCR products for the FpAPSES4 gene replacement construct
2.4 FpAPSES1和FpAPSES4参与假禾谷镰孢的菌丝生长
在PDA培养基上测定野生型菌株Wz2-8A、Δ和Δ突变体菌株的菌丝生长速率及菌落形态。结果显示,与野生型菌株相比,Δ和Δ突变体菌丝生长速度均明显减慢,色素积累明显增多(图4-A、4-B)。显微观察发现,与野生型相比,Δ的菌丝形态无明显差异,而Δ的菌丝弯曲、分支明显增多(图4-A)。说明FpAPSES1和FpAPSES4对假禾谷镰孢的生长非常关键。
2.5 FpAPSES1和FpAPSES4调控假禾谷镰孢分生孢子的产生和萌发
在CMC培养液中检测野生型菌株Wz2-8A、Δ和Δ突变体菌株分生孢子的产生及形态。假禾谷镰孢野生型菌株摇培3 d,即可获得大量分生孢子,此时Δ和Δ突变体分生孢子非常少。显微观察发现,与野生型分生孢子相比,Δ和Δ突变体的分生孢子变短,分隔减少(图5-A)。为了明确Δ和Δ突变体分生孢子减少是否是其生长速率减慢造成的,继续摇培至7 d。结果显示,假禾谷镰孢野生型菌株分生孢子量持续增多,而Δ和Δ突变体分生孢子并没有明显增多(图5-B)。将收集的野生型、Δ和Δ突变体分生孢子在无菌水中调整浓度至4×105/mL,置于载玻片上25℃、黑暗保湿培养3—6 h。显微观察结果显示,与野生型相比,Δ和Δ突变体分生孢子萌发6 h的芽管长度较短(图5-A),萌发率有所降低(图5-C)。以上结果说明FpAPSES1和FpAPSES4作用于假禾谷镰孢分生孢子的产生、形态建成和萌发。

A:假禾谷镰孢野生型菌株Wz2-8A、Δfpapses1和Δfpapses4突变体菌株在PDA培养基上的菌落和菌丝形态,标尺=20 µm Colony and mycelial morphology of F. pseudograminearum WT, Δfpapses1 and Δfpapses4 strains grown on PDA medium, bar=20 µm;B:假禾谷镰孢野生型菌株Wz2-8A、Δfpapses1和Δfpapses4突变体菌株在PDA平板上的菌落直径Colony diameters of F. pseudograminearum WT, Δfpapses1 and Δfpapses4 strains grown on PDA plates
2.6 FpAPSES1和FpAPSES4基因缺失影响假禾谷镰孢的致病力
为了明确FpAPSES1和FpAPSES4是否参与假禾谷镰孢的致病过程,通过小麦胚芽鞘接种、大麦叶片接种和盆栽试验分析了野生型菌株Wz2-8A、Δ和Δ突变体菌株的致病力。结果显示,与野生型相比,Δ和Δ突变体菌株侵染的小麦病斑均明显减小,但仍形成了明显的侵染病斑(图6-A、6-B)。收集野生型菌株Wz2-8A、Δ和Δ突变体菌株的分生孢子,制备成1×106/mL浓度的孢子悬浮液,接种到生长4 d的周麦27小麦胚芽鞘上,25℃、保湿培养4 d。结果显示,野生型菌株侵染的小麦胚芽鞘有明显扩展的坏死病斑,而Δ和Δ突变体分生孢子接种的小麦胚芽鞘只在接种点的位置形成坏死病斑,无明显扩展(图6-A、6-C)。
为了明确Δ和Δ突变体菌丝侵染病斑减小是否由其生长减慢造成,显微观察了菌丝在小麦表皮细胞中的扩展。结果显示,假禾谷镰孢野生型菌丝可以在小麦表皮细胞中正常扩展,而Δ和Δ突变体的菌丝在小麦表皮细胞间扩展过程中受到明显阻碍(图6-A)。
对大麦叶片侵染试验结果显示,与野生型相比,Δ和Δ突变体菌株侵染的大麦叶片病斑明显减小(图7-A、7-B)。通过盆栽试验模拟土壤中假禾谷镰孢对小麦的侵染。结果显示,与接种假禾谷镰孢野生型的小麦相比,接种Δ和Δ菌株的小麦生长较好,根和茎基部发病明显减轻(图7-C)。以上结果说明,FpAPSES1和FpAPSES4调控假禾谷镰孢的致病力,且主要影响病原菌在寄主细胞中的扩展。
2.7 FpAPSES1和FpAPSES4参与假禾谷镰孢DON毒素的合成
DON毒素即呕吐毒素,在假禾谷镰孢侵染过程中非常重要[3,23]。利用ELISA检测野生型菌株Wz2-8A、Δ和Δ突变体菌株DON毒素的合成。结果显示,假禾谷镰孢野生型菌株可以产生大量的DON毒素,含量可达到12.8 mg·kg-1。与其相比,Δ和Δ突变体接种的小米培养基中DON毒素含量明显减少,分别下降了78%和44%(图 8)。说明FpAPSES1和FpAPSES4均调控假禾谷镰孢DON毒素的合成。

A:假禾谷镰孢野生型菌株Wz2-8A、Δfpapses1和Δfpapses4突变体菌株菌丝块接种大麦叶片 Barley leaves inoculated with mycelial blocks of WT, Δfpapses1 and Δfpapses4 strains;B:接种4 d的病斑直径Lesion diameters measured at 4 dpi;C:小麦盆栽试验 Wheat seedlings from the pot-culture experiment
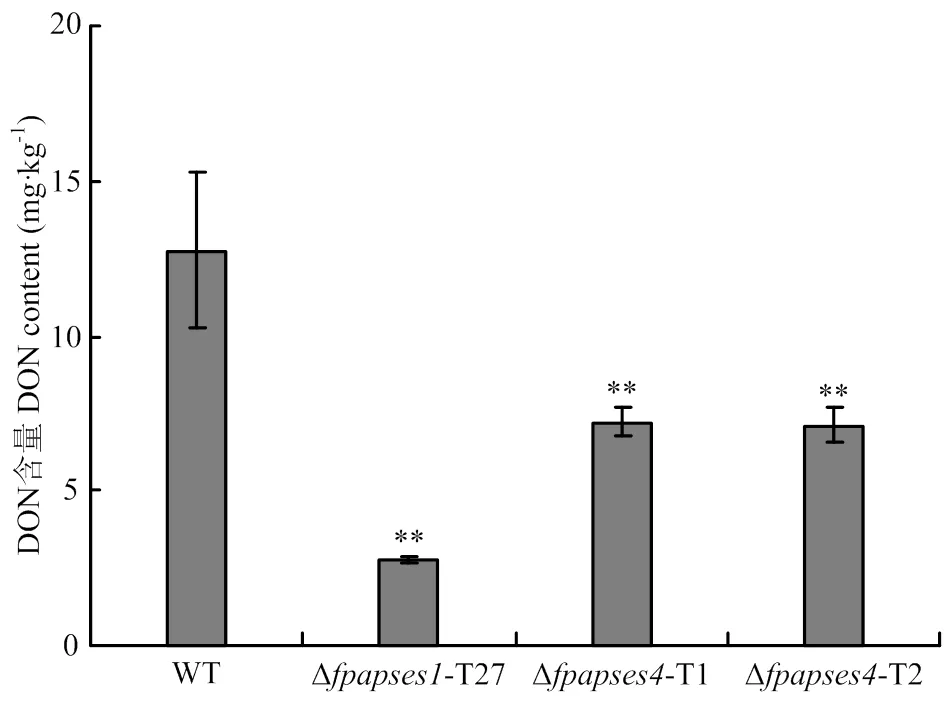
图 8 假禾谷镰孢野生型、Δfpapses1和Δfpapses4菌株产DON分析
3 讨论
APSES类转录因子广泛作用于真菌的细胞分化、营养生长、孢子产生,且与人和植物等多种真菌病害相关[5]。在曲霉和粗糙脉孢菌等丝状真菌中通常包含5个APSES蛋白,根据蛋白序列比对,本研究在假禾谷镰孢中也筛选到5个APSES同源蛋白,其中4个为包含APSES保守DNA结合结构域的蛋白FpAPSES1—4。进化分析发现,FpAPSES1、FpAPSES2和FpAPSES4属于A组APSES蛋白,FpAPSES3属于C组APSES蛋白。StuA作为APSES家族中最早被鉴定的成员,在人类和植物病原真菌中已有广泛研究报道,其对真菌的生长、产孢和致病等均有重要的调控作用[12,14,24-25],但对A组APSES蛋白在镰孢菌致病过程中的作用了解较少。
目前已报道的真菌A组APSES蛋白主要包括Mbp1、Swi4和Swi6。在酿酒酵母中,Swi6是转录因子复合体激活转录过程中非常常见的亚单位,但其没有DNA结合活性,所以Swi6常结合Mbp1形成MBF复合体,或者结合Swi4形成SBF复合体起作用[26-27]。MBF复合体调控下游多个途径相关基因的表达,尤其是丝裂原蛋白激酶信号途径,与Mpk1结合作用于细胞的完整性[28-30]。在稻瘟病菌中,Mbp1(PCG2)具有一种新的有翼螺旋-转-螺旋(wHTH)结构域的DNA结合模式,缺失突变体表现为菌丝生长缓慢、产孢量下降、附着胞形成和侵染、扩展能力显著降低[6,31];MoSwi6与稻瘟病菌丝裂原活化激酶MoMps1互作,且缺失突变体菌株在多个层面表现缺陷,包括菌丝生长、分生孢子萌发、附着胞形成、细胞壁完整性、过氧化氢耐受性以及致病性等,缺失也导致过氧化物酶和漆酶等多种胞外酶的转录和活性降低[7]。在本研究中,选择Mbp1的同源蛋白FpAPSES1和MoSwi6的同源蛋白FpAPSES4,通过qRT-PCR方法分析发现,和在假禾谷镰孢侵染阶段诱导表达,推测其可能参与病原菌的致病过程。
利用遗传学的方法分别获得和的缺失突变体。表型分析发现,和缺失突变体的菌丝生长明显减慢、分生孢子产生减少、致病力减弱、DON毒素合成减少。对比和在禾谷镰孢中的同源基因和的功能,和缺失突变体菌丝生长也明显减慢,但是分生孢子的数量并没有明显差异,而且突变体菌株完全不致病[32]。推测APSES蛋白在物种间的功能已有分化,但是和缺失突变体的回补和功能有待验证。
4 结论
APSES转录因子基因和在假禾谷镰孢的侵染阶段高表达,突变菌株分析推测这些转录因子影响了菌丝生长、分生孢子产生和致病过程。研究结果将为进一步解析假禾谷镰孢的致病机制提供参考。
[1] LI H L, YUAN H X, FU B, XING X P, SUN B J, TANG W H. First report ofcausing crown rot of wheat in Henan, China. Plant Disease, 2012, 96(7): 1065.
[2] ZHOU H F, HE X L, WANG S, MA Q Z, SUN B J, DING S L, CHEN L L, ZHANG M, LI H L. Diversity of the Fusarium pathogens associated with crown rot in the Huanghuai wheat-growing region of China. Environmental Microbiology, 2019, 21(8): 2740-2754.
[3] KAZAN K, GARDINER D M. Fusarium crown rot caused byin cereal crops: recent progress and future prospects. Molecular Plant Pathology, 2018, 19(7): 1547-1562.
[4] OBANOR F, NEATE S, SIMPFENDORFER S, SABBURG R, WILSON P, CHAKRABORTY S.andcaused the 2010 head blight epidemics in Australia. Plant Pathology, 2013, 62(1): 79-91.
[5] ZHAO Y, SU H, ZHOU J, FENG H H, ZHANG K Q, YANG J K. The APSES family proteins in fungi: Characterizations, evolution and functions.Fungal Genetics and Biology, 2015, 81: 271-280.
[6] LIU J F, HUANG J G, ZHAO Y X, LIU H A, WANG D W, YANG J, ZHAO W S, TAYLOR I A, PENG Y L. Structural basis of DNA recognition by PCG2 reveals a novel DNA binding mode for winged helix-turn-helix domains. Nucleic Acids Research, 2015, 43(2): 1231-1240.
[7] QI Z Q, WANG Q, DOU X Y, WANG W, ZHAO Q, LV R L, ZHANG H F, ZHENG X B, WANG P, ZHANG Z G. MoSwi6, an APSES family transcription factor, interacts with MoMps1 and is required for hyphal and conidial morphogenesis, appressorial function and pathogenicity of. Molecular Plant Pathology, 2012, 13(7): 677-689.
[8] MILES S, LI L, DAVISON J, BREEDEN L L. Xbp1 directs global repression of budding yeast transcription during the transition to quiescence and is important for the longevity and reversibility of the quiescent state. PLoS Genetics, 2013, 9(10): e1003854.
[9] GIMENO C J, FINK G R. Induction of pseudohyphal growth by overexpression of, agene related to transcriptional regulators of fungal development. Molecular and Cellular Biology, 1994, 14(3): 2100-2112.
[10] PARK H, MYERS C L, SHEPPARD D C, PHAN Q T, SANCHEZ A A, EDWARDS J E, FILLER S G. Role of the fungal Ras-protein kinase A pathway in governing epithelial cell interactions during oropharyngeal candidiasis. Cellular Microbiology, 2005, 7(4): 499-510.
[11] STOLDT V R, SONNEBORN A, LEUKER C E, ERNST J F. Efg1p, an essential regulator of morphogenesis of the human pathogen, is a member of a conserved class of bHLH proteins regulating morphogenetic processes in fungi. The EMBO Journal, 1997, 16(8): 1982-1991.
[12] LYSOE E, Pasquali M, Breakspear A, Kistler H C. The transcription factor FgStuAp influences spore development, pathogenicity, and secondary metabolism in. Molecular Plant-Microbe Interactions, 2011, 24(1): 54-67.
[13] Nishimura M, Fukada J, Moriwaki A, Fujikawa T, Ohashi M, Hibi T, Hayashi N. Mstu1, an APSES transcription factor, is required for appressorium-mediated infection in. Bioscience, Biotechnology and Biochemistry, 2009, 73(8): 1779-1786.
[14] Rath M, Crenshaw N J, Lofton L W, Glenn A E, Gold S E. FvSTUA is a key regulator of sporulation, toxin synthesis, and virulence in. Molecular Plant-Microbe Interactions, 2020, 33(7): 958-971.
[15] 周欣, 云英子, 郭谱胜, 吴凯莉, 陈伟钟, 李江江, 周晓莉. 尖孢镰刀菌番茄专化型中APSES转录因子FolStuA的功能分析. 分子植物育种, 2018, 16(3): 772-780.
ZHOU X, YUN Y Z, GUO P S, WU K L, CHEN W Z, LI J J, ZHOU X L. Functional analysis of APSES transcription factor FolStuA inf. sp.. Molecular Plant Breeding, 2018, 16(3): 772-780. (in Chinese)
[16] Gardiner D M, Benfield A H, Stiller J, Stephen S, Aitken K, Liu C, Kazan K. A high-resolution genetic map of the cereal crown rot pathogenprovides a near-complete genome assembly. Molecular Plant Pathology, 2018, 19(1): 217-226.
[17] Finn R D, BATEMAN A, CLEMENTS J, COGGILL P, EBERHARDT R Y, EDDY S R, HEGER A, HETHERINGTON K, HOLM L, MISTRY J, SONNHAMMER E L L, TATE J, PUNTA M. Pfam: The protein families database. Nucleic Acids Research, 2014, 42: D222-D230.
[18] Hall B G. Building phylogenetic trees from molecular data with MEGA. Molecular Biology and Evolution, 2013, 30(5): 1229-1235.
[19] Catlett N L, Lee B, Yoder O C, Turgeon B G. Split-marker recombination for efficient targeted deletion of fungal genes. Fungal Genetics Reports, 2003, 50: 9-11.
[20] 张承启, 廖露露, 齐永霞, 丁克坚, 陈莉. 禾谷镰孢核孔蛋白基因的功能分析. 中国农业科学, 2021, 54(9): 1894-1903.
ZHANG C Q, LIAO L L, QI Y X, DING K J, CHEN L. Functional analysis of the nucleoporin gene. Scientia Agriculture sinica, 2021, 54(9): 1894-1903. (in Chinese)
[21] LIU Z H, Friesen T L. Polyethylene glycol (PEG)-mediated transformation in filamentous fungal pathogens. Methods in Molecular Biology, 2012, 835: 365-375.
[22] Chen L L, Geng X J, Ma Y M, Zhao J Y, Li T L, Ding S L, Shi Y, Li H L. Identification of basic helix-loop-helix transcription factors reveals candidate genes involved in pathogenicity of. Canadian Journal of Plant Pathology, 2019, 41(2): 200-208.
[23] Tunali B, Obanor F, Erginbas G, Westecott R A, Nicol J, Chakraborty S. Fitness of three Fusarium pathogens of wheat.FEMS Microbiology Ecology, 2012, 81(3): 596-609.
[24] Chung D, Upadhyay S, Bomer B, Wilkinson H H, Ebbole D J, Shaw B D.ASM-1 complements the conidiation defect in amutant of. Mycologia, 2015, 107(2): 298-306.
[25] Miller K Y, Toennis T M, Adams T H, Miller B L. Isolation and transcriptional characterization of a morphological modifier: thestunted () gene. Molecular and General Genetics, 1991, 227(2): 285-292.
[26] Foord R, Taylor I A, Sedgwick S G, Smerdon S J. X-ray structural analysis of the yeast cell cycle regulator Swi6 reveals variations of the ankyrin fold and has implications for Swi6 function. Nature Structural Biology, 1999, 6(2): 157-165.
[27] Koch C, Moll T, Neuberg M, Ahorn H, Nasmyth K. A role for the transcription factors Mbp1 and Swi4 in progression from G1 to S phase.Science, 1993, 261(5128): 1551-1557.
[28] Amigoni L, Colombo S, Belotti F, Alberghina L, Martegani E. The transcription factor Swi4 is target for PKA regulation of cell size at the G1to S transition in. Cell Cycle, 2015, 14(15): 2429-2438.
[29] Jiang L, Wang J, Asghar F, Snyder N, Cunningham K W. CaGdt1 plays a compensatory role for the calcium pump CaPmr1 in the regulation of calcium signaling and cell wall integrity signaling in.Cell Communication and Signaling, 2018, 16(1): 33.
[30] Medina E M, Walsh E, Buchler N E. Evolutionary innovation, fungal cell biology, and the lateral gene transfer of a viral KilA-N domain. Current Opinion in Genetics and Development, 2019, 58/59: 103-110.
[31] 王大伟. 稻瘟病菌APSES转录因子Pcg2的作用及其机理的研究[D]. 北京: 中国农业大学, 2014.
WANG D W. Functional characterization of the APSES transcription factor Pcg2 in the rice blast fungus[D]. Beijing: China Agricultural University, 2014. (in Chinese)
[32] Son H, Seo Y S, Min K, Park A R, Lee J, Jin J M, Lin Y, Cao P, Hong S Y, Kim E K,. A phenome-based functional analysis of transcription factors in the cereal head blight fungus,. PLoS Pathogens, 2011, 7(10): e1002310.
Identification and Functional Analysis of Transcription Factors FpAPSES in
ZHAO JingYa, XIA HuiQing, PENG MengYa, FAN Zhuo, YIN Yue, XU SaiBo, ZHANG Nan, CHEN WenBo, CHEN LinLin
College of Plant Protection, Henan Agricultural University/National Key Laboratory of Wheat and Maize Crop Science, Zhengzhou 450002
【】Fusarium crown rot (FCR) caused byseriously affects wheat production in China. The objective of this study is to find and analyze the functions of APSES transcription factors in, and to provide theoretical basis for revealing the pathogenic mechanism ofand prevention and treatment of FCR.【】The known APSES proteins were obtained from GenBank, and APSES candidates inwere found by BLASTP. Domains of FpAPSES proteins were determined using Pfam. The phylogenetic tree of APSES proteins was constructed using MEGA 5.05. The relative expression levels ofandduring infection were examined by quantitative RT-PCR (qRT-PCR).anddeletion mutants were generated by polyethylene glycol (PEG)-mediated protoplast fungal transformation, and screened by PCR. The wild type, Δand Δstrains were activated on PDA medium, and cultured on PDA for mycelial growth and morphology assays; mycelial blocks were introduced into liquid CMC medium to assess conidiation and conidia morphology; conidia were cultured in sterile distilled water to explore conidia germination; mycelial blocks or conidia suspension were prepared and inoculated wheat coleoptiles and barely leaves to explore pathogenicity. The pot-culture experiment was also used for virulence assay. ELISA was used to detect deoxynivalenol (DON) in inoculation millet. 【】Four APSES candidates were found in, which all contained the conserved DNA binding domain HTH. The phylogenetic tree analysis showed that FpAPSES1, FpAPSES2 and FpAPSES4 were divided into A group, while FpAPSES3 belonged to the C group. The qRT-PCR analysis revealed thatandwere up-regulated at infection stages, which suggested that FpAPSES1 and FpAPSES4 might play important roles in infection. Twodeletion mutants (Δ-T10 and Δ-T27) and twodeletion mutants (Δ-T1 and Δ-T2) were obtained. Compared with the wild type strain, both- and-deleted mutants exhibited significantly reduced colony growth rates.-deleted mutants had normalhyphal branches, while-deleted mutants exhibited curved and more branched hypha. both- and-deleted mutants exhibited significantly reduced conidiation in CMC, and the conidia numbers were respectively reduced by 99.5% and 97.4% comparing with that of the wild type. Furthermore, conidia of- and-deleted mutants were shorter, less septa and lower germination rates. The wheat coleoptiles were point-inoculated with mycelial blocks or conidia, and the virulence of- and-deleted mutants was significantly reduced comparing to the wild type. Reduced pathogenicity was further observed by barely leaves inoculation and pot culture experiment. the DON levels in millet upon infection with- and-deleted mutants were reduced by 78% and 44% comparing to the wild type, respectively.【】Both A group APSES homologsandplay important roles in growth, conidiation and pathogenicity of.
Fusarium crown rot;; APSES transcription factor; pathogenicity

10.3864/j.issn.0578-1752.2021.16.006
2020-11-26;
2021-01-08
国家自然科学基金(U2004140)、河南省青年人才托举工程项目(2020HYTP043)、大学生创新创业训练计划(201910466015)
赵静雅,E-mail:zhaojy0108@163.com。通信作者陈琳琳,E-mail:llchensky@163.com
(责任编辑 岳梅)

