藜麦NLP转录因子家族的鉴定及表达分析
朱满喜,张玉荣,杨雅舒,杨小兰,王创云,邓 妍,赵 丽,张丽光,秦丽霞,杨利艳
(1.山西师范大学 生命科学学院,山西 临汾 041000;2.山西农业大学 农学院,山西 太原 030031)
氮素是植物生长的必需营养物质,同时也是蛋白质、叶绿素、核苷酸和植物激素的主要成分,对植物的生长发育和产量形成具有重要的作用[1-2]。近年来,为挖掘作物最大的生产潜力,氮肥被大量施用[3],但氮肥的利用效率并不高,大约50%的氮肥被流失到环境中,导致土壤、水体和空气受到污染,并产生如一氧化二氮这样氮含量很高的温室气体[4]。
藜麦(ChenopodiumquinoaWilld.)是原产于南美洲安第斯地区的一年生作物,传统上种植于贫瘠的土地上,对瘠薄土壤有较强的适应性;同时藜麦还具有均衡的油脂、必需氨基酸、碳水化合物、矿物质、维生素和膳食纤维[5],由于藜麦突出的全营养价值和对不利环境表现出的极强适应性,2013年被宣布为国际藜麦年[6-7],被认为是一种对全球特别是发展中国家粮食安全有重要价值的作物[8-9]。因此,了解藜麦对氮素的利用机制将有助于提高作物对氮素的利用效率,降低环境污染,促进农业的可持续发展。
NIN-Like Protein(NLP)是一个植物特异性转录因子家族,在响应氮胁迫和植物氮代谢调节过程中发挥着重要作用[10]。NIN(Nodule inception)蛋白首次发现于豆科植物莲花中,是共生根瘤发育的关键调节因子[11-12],后来发现其广泛存在于拟南芥、水稻[13]、甘蓝型油菜[14]、小麦[15]和玉米[16]等植物中。NLP转录因子包含RWP-RK和PB1等2个保守结构域,其中,RWP-RK结构域与DNA结合,目前已发现,含有RWP-RK结构域的硝酸酯酶(NIT2)是衣藻硝酸盐信号转导途径的关键调控因子[17];PB1结构域位于羧基端,参与蛋白互作过程[18]。此外,GAF结构域是一个普遍存在于NLP家族中的重要结构域,在信号感知和传导中发挥作用[19]。对拟南芥NLP转录因子研究表明,NLP蛋白(AtNLP)通过与硝酸盐反应顺式元件结合,在协调初级氮反应中发挥关键作用[10,20-21]。硝酸盐-CPK-NLP调控网络的发现进一步阐明了NLP家族在硝酸盐信号转导中的作用,以及NLP磷酸化在生长协调中的重要性[22]。AtNLP7与氮反应途径关键的基因ANR1、LBD37/38、NRT1.1、NRT2和NIA1结合,通过激活或抑制下游基因的转录调节氮的同化和代谢[23-24]。AtNLP8被证明是硝酸盐促进种子萌发的主要调节因子,可以直接与脱落酸(ABA)分解酶基因(CYP707A2)的启动子结合,并以硝酸盐依赖性的方式降低ABA水平[25]。目前,已经对许多豆科植物和非豆科植物中NLP转录因子家族基因进行了鉴定,但藜麦中 NLP相关信息尚未见报道。
本研究利用已公布的藜麦全基因组信息对NLP转录因子家族进行了鉴定,并对其基因结构、染色体定位、启动子元件、蛋白理化性质、保守结构域、保守基序以及系统进化关系和氮胁迫下表达模式进行了分析,旨在为进一步理解藜麦NLP家族的分子进化和生物学功能提供一定理论基础。
1 材料和方法
1.1 试验材料及氮素处理
供试藜麦品种为ZK7(Chenopodiumquinoacv. ZK7),由山西省农业科学院作物科学研究所提供。
用10%的次氯酸钠溶液消毒5 min,冲洗干净后将种子置于湿润的滤纸上,发芽后置于人工气候培养箱,用Hoagland全营养液(N含量为7.1 mmol/L)进行培养,培养条件为光照 16 h,黑暗 8 h,温度为光照23 ℃,黑暗18 ℃;待幼苗长到6~8片叶后,选取生长一致的幼苗进行处理:低氮处理(LN). 20%N Hoagland营养液培养,N浓度为1.42 mmol/L;缺氮处理(DN). 0%N Hoagland营养液处理(用0.5 mol/L KCl 和 0.5 mol/L CaCl2代替KNO3和CaNO3),N浓度为0;对照(CK). Hoagland全营养液处理(N浓度为7.1 mmol/L)。处理时间分别为5,30 d。每个处理设 3个生物学重复。采集的幼苗叶片立即置于液氮中速冻,超低温保存,用于转录组及筛选基因的qRT-PCR分析。
1.2 藜麦NLP转录因子家族成员的生物信息学分析
1.2.1 基因序列和转录组数据来源 藜麦基因组数据下载于藜麦全基因组数据库ChenopodiumDB(https://www.cbrc.kaust.edu.sa/chenopodiumdb/),转录组数据由山西师范大学细胞生物学实验室提供,拟南芥9个NLP转录因子和水稻12个NLP转录因子的氨基酸序列分别从拟南芥全基因组数据库TAIR(https://www.arabidopsis.org/)和水稻全基因组数据库RAP-DB(http://rapdb.dna.affrc.go.jp/)获得。
1.2.2 藜麦NLP 转录因子家族成员的识别 从拟南芥数据库获得9个AtNLP基因的蛋白序列作为查询序列,运用TBtools软件的Blast Compare Two Seq功能在藜麦基因组中进行BlastP比对,设E 值为10-5;将得到的藜麦蛋白序列在NCBI中再次进行Blastp比对,去除近缘家族的成员;在CDD数据库查询鉴定的藜麦NLP家族成员保守结构域,去除无NLP典型保守结构域RWP-RK和PB1的蛋白序列。
1.2.3 藜麦NLP基因分析 利用TBtools软件的GFF3/GTF Gene Position(info.)Parse功能,从藜麦基因组注释文件中(Cq_PI614886_gene_V1_pseudomolecule.gff)获得NLP基因的位置信息,提交到在线软件MG2C(http://mg2c.iask.in/mg2c_v2.0/),用于基因染色体定位分析。运用在线服务器GSDS 2.0(http://gsds.cbi.pku.edu.cn/),输入藜麦NLP基因的全长序列和CDS序列,分析其基因结构特征。通过TBtools的Fasta Extract功能从藜麦基因组中提取NLP基因转录起始位点前2 000 bp核苷酸序列作为启动子序列,将所有的CqNLP启动子序列提交至PlantCARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/),进行启动子顺式作用元件的预测分析。
1.2.4 藜麦NLP蛋白分析 利用在线分析软件ExPasy(http://au.expasy.org/tool.html)的Compute pI/MW功能分析蛋白序列的长度、分子量、等电点等理化性质。通过BioEdit软件的ClustalW Multiple alignment功能对保守结构域的氨基酸进行多序列比对,在桌面版Jalview(Jalview 2.10.5)程序中对比对结果进行编辑与可视化,在IBS软件中绘制保守结构域位置图。通过MEME网站(http://memesuite.org/tools/meme) 对藜麦NLP 蛋白保守基序进行分析。参数如下:基序数目(Motif number)设置为10,基序宽度(Motif width)设置为6~100。运用TBtools软件的Gene Structure View和Batch MEME Motif Viz功能对结果进行可视化。
1.2.5 藜麦NLP转录因子家族的系统进化分析 利用BioEdit软件的ClustalW Multiple alignment功能对藜麦、水稻和拟南芥的NLP家族蛋白序列进行多重比对,比对结果在MEGA 7.0软件中采用Neighbor-Joining法构建无根进化树(Bootstrap=1 000)。氨基酸替代模型选择Poisson model。
1.3 藜麦NLP基因在氮胁迫条件下的表达模式分析
1.3.1 转录组分析 藜麦在不同氮素水平下叶片的转录组分析由北京百迈克生物科技有限公司完成。从转录组数据库中提取NLP基因家族在不同氮胁迫条件下的表达量数据,将藜麦NLP基因家族各成员的表达量FPKM值进行log2(FPKM)换算后,利用TBtools软件绘制NLP基因家族各成员的表达热图。
1.3.2 RNA提取及qRT-PCR分析 利用TRIzol法提取总RNA(TIANGEN 试剂盒),用超微量核酸蛋白测定仪(scandrop100)检测RNA OD值(A260/280的值)。采用Aidlab公司反转录试剂盒(TUREscript 1st Stand cDNA SYNTHESIS Kit)进行cDNA第1条链的合成。利用实时荧光定量PCR分析其在不同氮素水平下的表达特性,以各样品的cDNA为模板进行实时荧光定量PCR分析,以EF1a作为藜麦的内参基因。扩增程序为:95 ℃预变性3 min;95 ℃变性10 s,58 ℃退火30 s,39个循环。熔解程序为:60~95 ℃,每个循环增加1 ℃,持续时间为4 s;反应体系为10.0 μL,其中,2×SYBR Green Supermix 5.0 μL,上下游引物(10 μmol/L)各0.5 μL。采用2-ΔΔCt方法计算基因的相对表达量。基因特异性引物序列如表1所示。

表1 qRT-PCR引物序列Tab.1 Primer sequences for qRT-PCR
2 结果与分析
2.1 藜麦NLP基因家族成员鉴定及基本信息分析
以拟南芥中已鉴定好的9个NLP蛋白序列为参考,运用双向BlastP方法获得蛋白序列,在CDD数据库中去除无NLP典型保守结构域RWP-RK和PB1的蛋白序列,最终鉴定到9个藜麦NLP家族成员;根据其在染色体上的位置依次命名为CqNLP1~CqNLP9(表2),其中,CqNLP6(9 192 bp)最长,CqNLP4(3 854 bp)最短;CqNLP4的CDS序列长度最短(2 157 bp),其他NLP基因的CDS序列长度均在2 800 bp左右。通过ExPasy网站分析NLP蛋白理化性质,统计发现,编码的氨基酸数目为718~952个;分子质量为80.48~104.86 ku,氨基酸数目和分子质量均以CqNLP4最小、CqNLP8最大;等电点为5.31~6.50,均小于7,偏酸性。
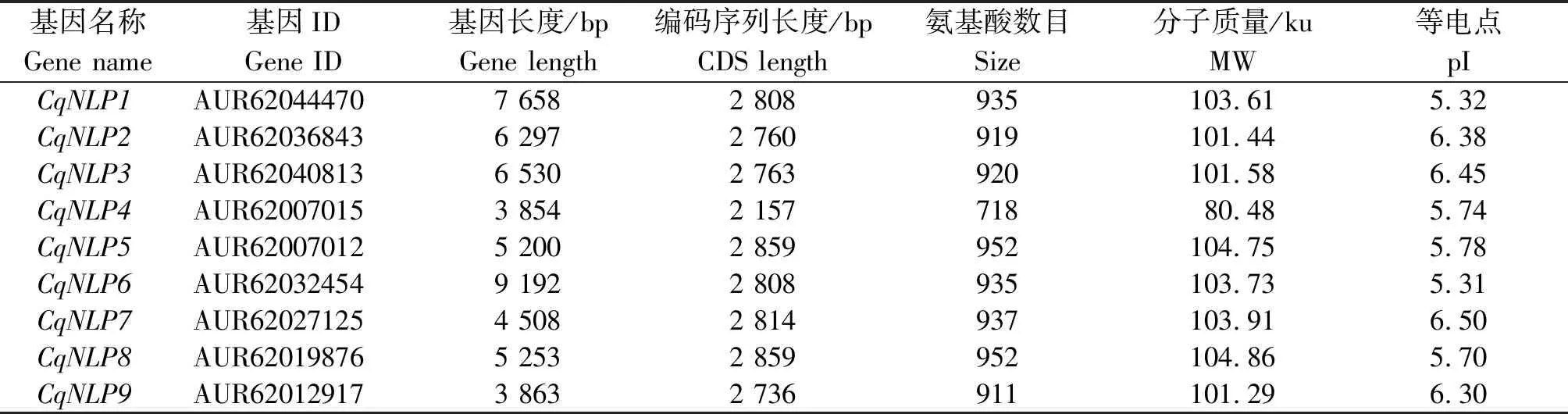
表2 藜麦NLP基因家族成员基本信息Tab.2 Basic information of NLP gene family members in quinoa
2.2 藜麦NLP基因染色体定位
基于藜麦基因组注释文件(Cq_PI614886_gene_V1_pseudomolecule.gff)进行染色体定位,分析结果发现(图1),9 个藜麦NLP基因不均匀地分布在不同的染色体上,其中,CqNLP3、CqNLP6、CqNLP7、CqNLP8、CqNLP9分别分布于Chr10、Chr14、Chr15、Chr16、Chr17上,CqNLP1和CqNLP2分布于Chr00上,CqNLP4和CqNLP5分布于Chr13上,并且大部分藜麦NLP基因定位于染色体两端的区域。基因定位结果显示,CqNLP4和CqNLP5位于13号染色体末端,且距离为41 bp。有研究认为,在染色体上距离小于50 bp的重复序列是串联重复序列,且多分布于染色体的末端[26]。因此,预测在藜麦的NLP家族中存在串联重复事件,即CqNLP4和CqNLP5为一对串联重复基因。

图1 藜麦NLP基因在染色体上的分布情况Fig.1 Distribution of CqNLPs on chromosome
2.3 藜麦NLP基因结构分析
藜麦NLP 基因结构分析结果表明(图2),该基因家族成员含有4~5个外显子、3~4个内含子;CqNLP4和CqNLP9没有3′UTR区,其他基因同时均具有3′UTR区和5′UTR区;另外,9个藜麦NLP基因形成了4对旁系同源基因,旁系同源基因在长度和结构上具有很高的相似性,如CqNLP5和CqNLP8为一对旁系同源基因,其基因长度仅相差53 bp,CDS长度完全相等,且同时包含5个外显子和4个内含子,推测其具有相似的功能。

图2 藜麦NLP家族基因结构分析Fig.2 Gene structure analysis of NLPs in quinoa
2.4 藜麦NLP启动子分析
截取藜麦NLP基因上游2 000 bp碱基序列作为启动子区域,提交至PlantCARE网站进行顺式作用元件预测,结果发现,9个CqNLP启动子除了含有生长所必需的TATA-box和CAAT-box外,还存在多种应答元件,如激素应答元件ABRE(脱落酸)、ERE(乙烯)、P-box(赤霉素)、TCA(水杨酸)、TGA(生长素);非生物胁迫应答元件MBS(干旱)、ARE(低氧)、LTR(低温)和生物胁迫应答元件W-box(病原菌);此外,在CqNLP3和CqNLP4的启动子区域分别预测到1个氮素响应元件GCN4(表3),推测其在氮胁迫应答中发挥作用。
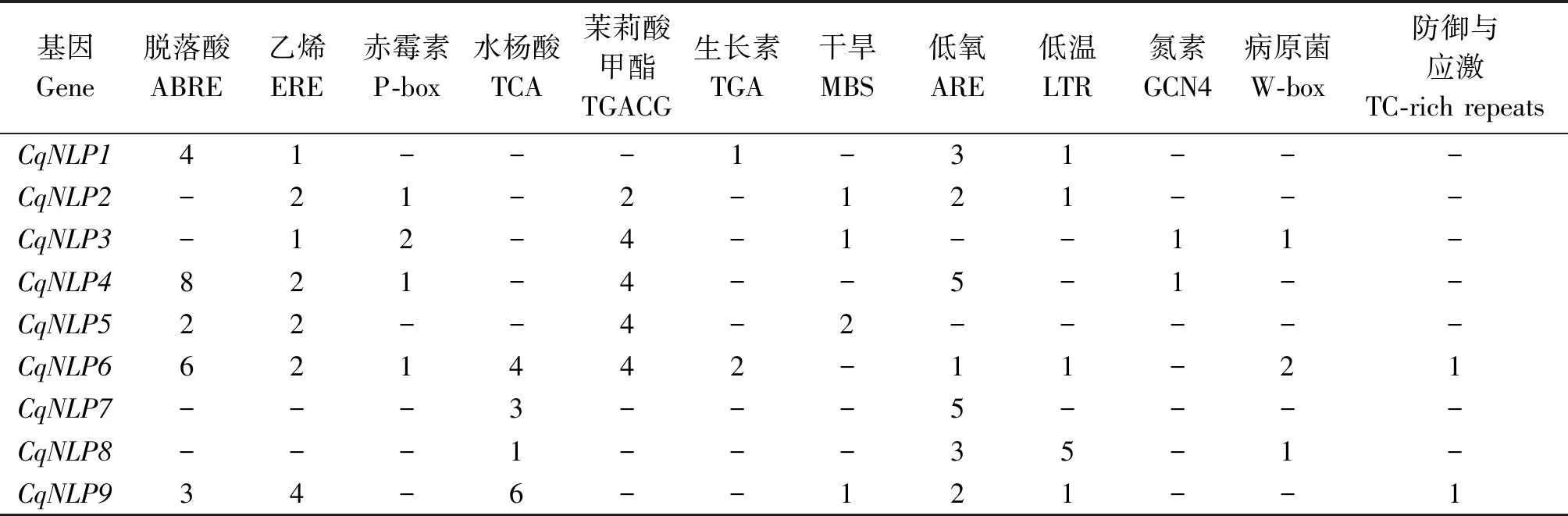
表3 藜麦NLP启动子顺式作用元件分析Tab.3 Analysis of cis-acting elements of NLP promoters in quinoa
2.5 藜麦NLP蛋白保守结构域分析
通过NCBI的CDD数据库验证藜麦NLP蛋白保守结构域,结果发现,9个藜麦NLP蛋白均含有保守结构域RWP-RK和PB1,且2个结构域都位于蛋白序列的N端(图3);同时,每一对旁系同源基因的蛋白长度和保守结构域位置都具有很高的相似性,且CqNLP4蛋白长度明显比其他藜麦NLP蛋白短,结合其保守结构域位置和基因结构,推测其可能在进化过程中发生5′端缺失突变。
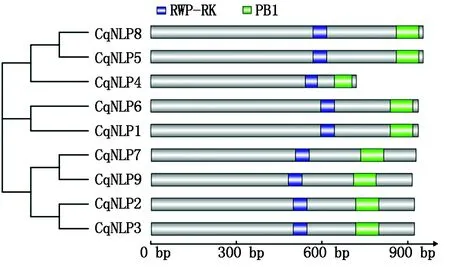
图3 藜麦NLP蛋白保守结构域位置Fig.3 Conserved domain location of CqNLP proteins
将藜麦NLP蛋白RWP-RK和PB1结构域的氨基酸序列进行多重比对,进一步分析2个结构域的保守性发现(图4),RWP-RK结构域由43~49个氨基酸组成,PB1结构域由61~81个氨基酸组成,且2个结构域中均存在多个氨基酸保守位点,尤其在RWP-RK结构域中只有极个别位点保守性较差,推测这些保守位点在NLP蛋白行使功能过程中发挥重要作用。CqNLP4蛋白的2个保守结构域较其他NLP蛋白保守性差,均出现3′缺失现象,结合对CqNLP4蛋白的分析,推测其在进化的过程中可能出现了新的功能。
2.6 藜麦NLP蛋白保守基序分析
对9个藜麦NLP蛋白保守基序的位置和序列进行分析,结果如图5-A所示,共发现10个Motif,在藜麦NLP蛋白家族每个成员中均存在,依次命名为Motif1~Motif10;通过CDD数据库验证,Motif1对应RWP-RK结构域,Motif5和Motif9对应PB1结构域。
由图5-B可知,Motif1、Motif5和Motif9的氨基酸序列比其他基序的保守性高,与结构域的保守性一致;此外,Motif1中存在RWP-RK结构域的特征氨基酸序列组成“RWPSRK”,但只有在CqNLP4中缺失2个氨基酸位点,一致性较差,该结果进一步印证了对保守结构域的分析结果。
2.7 藜麦、拟南芥和水稻NLP家族蛋白系统进化分析
本研究用藜麦和已经鉴定的拟南芥、水稻全部NLP 蛋白序列构建系统发育树,对藜麦NLP家族蛋白的进化关系进行分析,结果如图6所示,OsNLP6由于RWP-RK保守结构域缺失被分到外类群,其余29个NLP蛋白被明显分为3组,分别命名为Class Ⅰ、ClassⅡ和ClassⅢ;在藜麦NLP蛋白中,CqNLP2和CqNLP3属于Class Ⅰ组,CqNLP7和CqNLP9归到ClassⅡ组,CqNLP1、CqNLP4、CqNLP5、CqNLP6和CqNLP8聚到Class Ⅲ组。通过对3个物种NLP蛋白的同源性进行分析,结果只发现了10对物种内的旁系同源基因,没有发现物种间的直系同源基因,表明这3个物种的NLP家族在进化过程中按照各自物种的特性进行了分化,以更好地适应环境,这种现象普遍存在于各种植物中。CqNLP4、CqNLP5、CqNLP8与AtNLP7的亲缘关系最近。

图4 藜麦NLP蛋白保守结构域多序列比对Fig.4 Multiple sequences alignment of conserved domain of NLP proteins

图5 藜麦NLP蛋白Motif分析Fig.5 Motif analysis of NLP proteins in quinoa
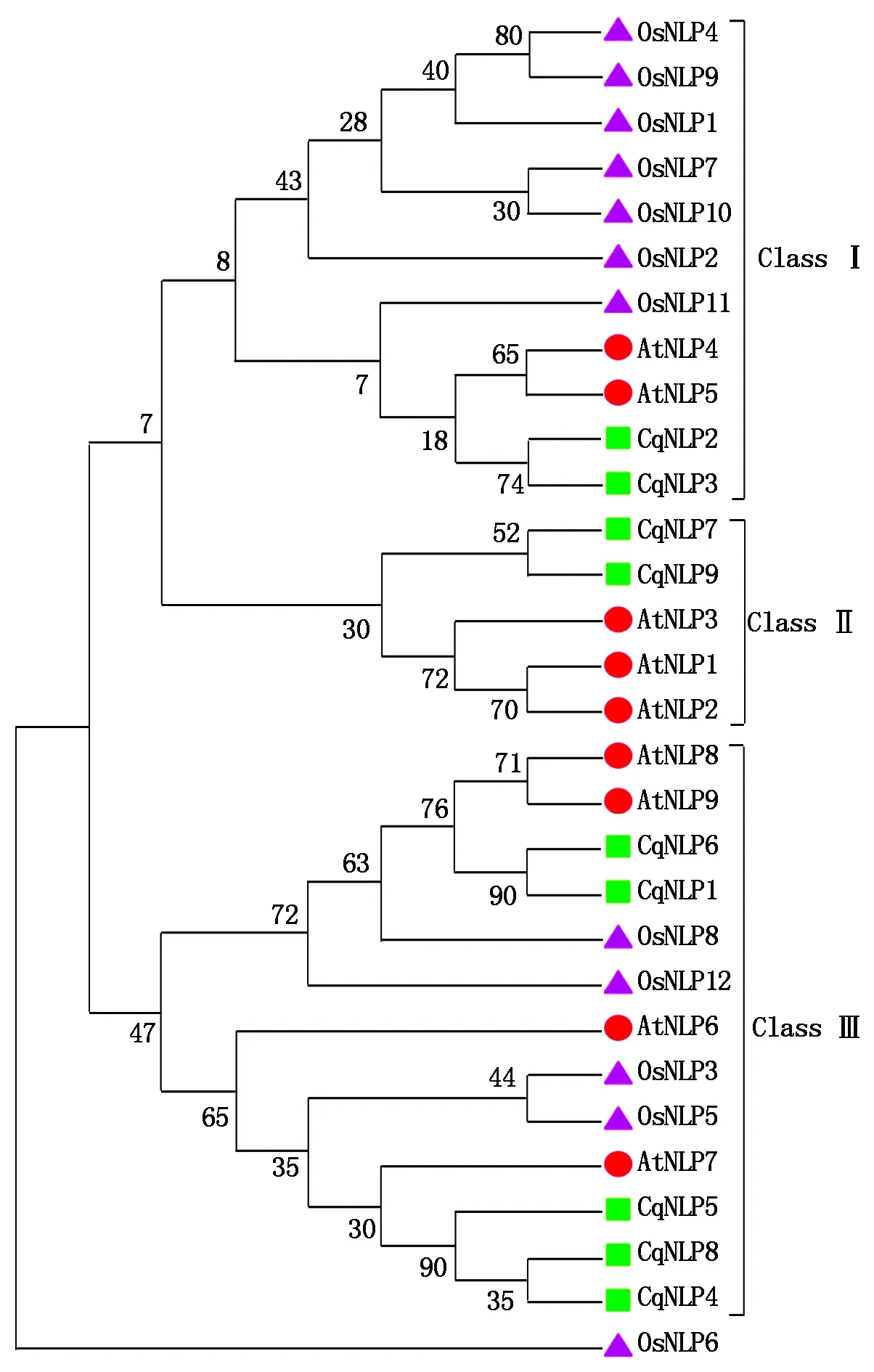
圆表示拟南芥NLP蛋白;三角形表示水稻NLP蛋白;正方形表示藜麦NLP蛋白。The circle represents the Arabidopsis NLP protein; The triangle represents riceNLP protein;The square represents the Chenopodium quinoa NLP protein.
2.8 藜麦NLP基因家族在氮素胁迫下的表达分析
以山西师范大学细胞生物学实验室已有的藜麦品种ZK7不同氮素水平处理下的叶片转录组数据为基础,提取NLP基因家族表达数据,将不同样品的FPKM值进行log2(FPKM)标准化处理后绘制藜麦NLP基因家族成员的表达热图,结果显示(图7),9个藜麦NLP基因在不同氮素水平处理时间(5,30 d)、不同处理浓度(CK、LN和DN)均有表达;整体来看,处理组(LN和DN)的表达量高于对照组(CK),且LN组的表达量高于DN组,处理30 d的表达量明显高于处理5 d的表达量。
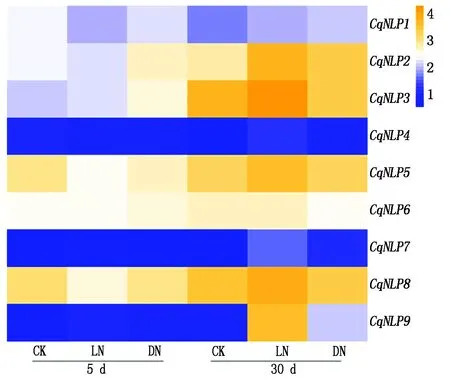
图7 藜麦NLP基因在不同氮素水平处理下的表达热图Fig.7 Expression heat map of quinoa NLPs geneunder different nitrogen levels
为进一步明确氮胁迫下藜麦NLP基因的表达模式,从表达图谱中选取表达量明显高于其他家族成员的CqNLP2、CqNLP3、CqNLP5、CqNLP8、CqNLP9进行实时荧光定量PCR分析,结果表明(图8),藜麦NLP基因表达量受不同氮素水平的诱导发生变化;总体上,处理后期(30 d)藜麦NLP基因的表达量高于前期(5 d),其中,CqNLP2、CqNLP3、CqNLP5、和CqNLP8表达趋势基本一致,在不同氮素水平处理前期(5 d),CqNLP2在低氮处理(LN)时表达量显著增加,CqNLP3、CqNLP5、CqNLP8相对表达量的变化不显著;后期(30 d) 达量发生显著变化;低氮处理(LN)显著上调,缺氮处理(DN)显著下调,以CqNLP5变化最为显著,与对照相比,其在低氮处理组表达上调74.1%,缺氮处理组表达下调59.5%;CqNLP9的表达模式较为特殊,在处理后期(30 d),不同氮胁迫程度下表达量均有增加,与CqNLP2、CqNLP3、CqNLP5和CqNLP8的表达趋势明显不同(低氮上调、缺氮下调),尤其低氮处理后期CqNLP9的表达量急剧增加,为对照组的23.6倍。实时荧光定量PCR 分析结果与转录组表达热图分析结果基本一致。
3 结论与讨论
转录因子作为基因表达上游的重要调控因子,在植物生长发育[27]、信号转导[28]和逆境响应[20,29]方面发挥着关键作用,其中,研究较多的有WRKY[30-31]、NAC[32]、MYB[33]、bZIP[34]等,有关NLP转录因子功能在拟南芥和豆科植物中有少量报道,对藜麦NLP的功能研究尚未见报道。
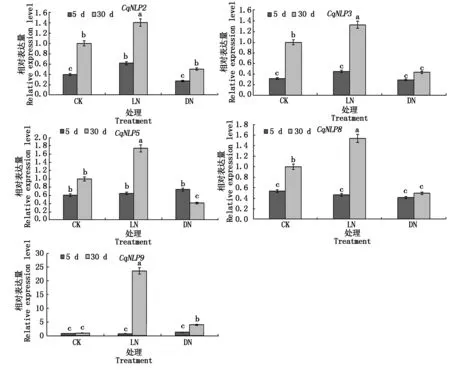
字母 a、 b、 c表示不同样本之间存在显著差异(P<0.05)。a,b,and c showed significant difference between different samples(P<0.05).
本研究对藜麦NLP转录因子家族进行了鉴定,在藜麦全基因组中分析发现了9个NLP家族成员,与拟南芥NLP家族成员数目相同[13],相比水稻中有6个NLP家族成员[35]、高粱中有5个NLP家族成员,藜麦NLP家族成员的功能可能更多样化,但藜麦基因组(1.39 Gb)具有与小基因组拟南芥(135 Mb)相同数量的NLP家族成员,表明NLP家族在进化上的保守性及其对维持植物正常生长发育的重要性。为研究CqNLP基因的功能,本研究对其基因结构进行了分析,结果发现,CqNLP含有不同数目的外显子和内含子,大多数CqNLP含有UTR区,只有CqNLP4和CqNLP9没有3′UTR区,主要基因结构是5个外显子和4个内含子,表明基因结构具有多样性且与其他物种的NLP基因结构相似[16,36]。CqNLP基因结构的差异是由内含子的变化引起的,基因结构的不同状态可能与CqNLP基因的进化有关,有助于进化基因选择新的功能,提高植物对环境的适应性。植物启动子主要包括一些特异性的顺式作用元件,通过与多种调控因子结合决定基因的表达模式和强度。通过预测发现,藜麦NLP家族基因的启动子区除了包含生长必需的元件外,还存在多种胁迫应答元件,其中,在CqNLP3和CqNLP4的启动子中各发现了1个氮素响应元件GCN4,该响应元件在苹果[37]、小麦[15]中也有发现,推测其可能在藜麦氮胁迫响应过程中发挥作用,并具有一定的保守性。蛋白序列分析及保守结构域比对发现,藜麦NLP家族成员均具有RWP-RK和PB1保守结构域,其中,RWP-RK包括一个“螺旋-环-螺旋-转角-螺旋”亮氨酸拉链结构[13],这一结构是NLP转录因子与DNA相互作用的关键结合位点,能够激活或抑制下游基因的转录;PB1结构域是一个蛋白质-蛋白质相互作用结构域,能够在含有PB1结构域的蛋白质之间进行异源二聚反应[38],NIN和NLP通过其羧基末端的PB1结构域进行物理相互作用,触发NLP从胞浆到细胞核的重新定位,抑制NIN激活CRE1的表达,在硝酸盐抑制共生结瘤过程中发挥核心作用[39]。系统发育分析表明,藜麦NLP家族被分为3组,这与前人的研究结果一致[13],藜麦和拟南芥的NLP家族成员存在于各个分支,水稻的NLP家族成员只存在Class Ⅰ和Class Ⅲ组中,并且藜麦与拟南芥NLP转录因子的亲缘关系比水稻更近,说明NLP基因家族的分化在单子叶植物水稻分化之前。CqNLP4、CqNLP5、CqNLP8与AtNLP7亲缘关系最近。前人研究表明,AtNLP7通过与氮反应途径的关键基因结合来调节氮的同化和代谢[24-25],推测这3个转录因子在藜麦氮代谢途径中可能起重要调控作用。
NLP转录因子是植物非生物胁迫响应过程的重要调控因子。前人研究表明,拟南芥AtNLP7突变体表现出叶片失水减少和对干旱胁迫的耐受性[20],但具体机制尚不明确。苹果MdNLP5启动子中存在一个响应氮的GCN4作用元件,说明该类转录因子与氮素有密不可分的关系[37]。本研究分析了氮胁迫下藜麦NLP家族基因的表达情况,氮胁迫处理前期(5 d)藜麦NLP基因表达无明显变化,这可能与取材部位有关,本试验取材于藜麦叶片,氮素经吸收和转运,最后作用于叶片存在一定的延时;处理后期(30 d)随着氮胁迫程度的加深藜麦NLP基因表达量先升高后降低,此时藜麦叶片对氮素的变化做出响应,NLP基因的表达量发生显著变化,其中,低氮处理后期CqNLP9的表达量急剧增加,后期可作为响应氮胁迫候选基因对其功能进一步验证。
本研究结果表明,在藜麦全基因组中共鉴定出9个NLP基因家族成员,不均匀地分布在7条染色体上;系统发育分析将其分为Class Ⅰ、Class Ⅱ和Class Ⅲ共3组,成员均具有典型保守结构域RWP-RK和PB1;启动子区包含多种激素和胁迫应答顺式作用元件;氮素胁迫处理前期除CqNLP2外各基因表达量无显著变化,胁迫处理后期CqNLP2、CqNLP3、CqNLP5和CqNLP8的表达量显著变化(低氮上调、缺氮下调),CqNLP9受低氮显著诱导表达。本研究结果可为后续CqNLPs基因克隆和氮素胁迫分析提供一定参考依据。

