HLA-DQB1和HLA-DRB1基因多态性与龋病相关性的系统评价与Meta分析
陈越,吴泽钰,貊燕丽,敬英豪,刘奕杉,3
1.新疆医科大学第一附属医院(附属口腔医院)儿童口腔科-口腔预防科,新疆维吾尔自治区 乌鲁木齐(830054);2.新疆医科大学第一附属医院(附属口腔医院)牙体牙髓病科,新疆维吾尔自治区 乌鲁木齐(830054);3.新疆维吾尔自治区口腔医学研究所,新疆维吾尔自治区 乌鲁木齐(830054)
龋病是以细菌为主的多因素作用下,牙体硬组织发生的慢性进行性破坏[1-2]。随着龋病病因学的深入研究,遗传因素在龋病病因学中的作用被逐渐重视,基因多态性与龋病相关性的研究也越来越多被报道。人类白细胞抗原(human leukocyte antigen,HLA)主要包括高度多态性的HLAⅠ类和HLAⅡ类基因[3]。HLA超级基因座是染色体位置6p21的一个基因组区域,它编码6个经典的HLA基因和至少132个蛋白质编码基因,这些基因在免疫系统等调控中具有重要作用[4]。HLA-DR单倍型由多个编码和非编码HLA-DR基因的拷贝组成。高度多态性的DRB1等位基因存在于所有单倍型中[5]。人类基因组的这一小部分已与100多种不同疾病相关,如糖尿病、系统性红斑狼疮和各种其他自身免疫性疾病[6-7]。近年关于HLA与龋病相 关 性 的 研 究 被 广 泛 报 道[8-11],Valarini等[12]对15~19岁学生进行了对比分析,按照DMFT>1和DMFT=0进行分组:结果证实HLA基因与龋病的发生密切相关。但HLA-DQB1和HLA-DRB1基因多态性与龋病的相关性未能达成统一结论,笔者通过Meta分析探讨HLA-D基因多态性与龋病的相关性,为龋病的早期预防提供理论依据及数据支持。
1 资料和方法
1.1 注册
使用系统综述和荟萃分析首选报告条目(preferred reporting items for systematic reviews and metaanalyses,PRISMA)检查表作为指南。本Meta分析在国际前瞻性系统综述登记研究PROSPERO数据库中登记。登记编号:CRD42021224938,https://www.crd.york.ac.uk/prospero/display_record.php?ID=CRD42021224938。
1.2 检索策略
检索截至2020年12月之前发表在英文电子数据库(Cochrane Library、Embase、PubMed、Scopus和Web of science)及中文电子数据库(知网、万方、维普和中国生物医学文献数据库)中的文献。使用“Dental Caries”和“HLA-D Antigens”为检索词,在PubMed中检索Mesh主题词,以Mesh主题词+自由词检索方式进行检索。对性别、年龄和研究人群的语言、种族和地区没有限制。同时对检索到的文章的参考文献进行交叉检索,以避免遗漏任何研究。
1.3 纳入标准
1.3.1 研究对象 病例组人群为DMFT≥1,临床诊断为龋病的患者。对照组为DMFT=0的健康人群。
1.3.2 暴露因素 HLA-DQB1和HLA-DRB1等位基因多态性。
1.3.3 结局指标 龋病的发病风险。
1.3.4 研究类型 HLA-DQB1和HLA-DRB1基因多态性与龋病相关性的病例-对照研究。
1.4 排除标准
①未使用DMFT=0的健康人群作为对照组的文献;②病例组患者除有龋病外,还有糖尿病等其他慢性疾病史;③数据不充分的研究;④重复发表的文章;⑤基础科学或动物研究;⑥综述、病例报告和病例系列、会议摘要、给编辑的信函以及定性研究。
1.5 文献筛选、资料提取和偏倚风险评估
根据文献纳入和排除标准,在选择文献时,隐去对筛选者可能产生影响的文章信息(期刊名、作者、作者单位、发表年份、资金资助情况),并且由2名研究者采用盲法独立进行。若存在分歧,进行复核,并请专家评议。从纳入的每篇文献中提取以下信息:第一作者的姓名、发表文章的日期、国家、检测样本类型、种族、年龄、HLA-DQB1和HLADRB1基因分型和等位基因的频率、研究的等位基因数量、基因检测方法及病例组和对照组的样本量。根据Newcastle–Ottowa量表评估偏倚风险,由2名研究者使用相同的标准对纳入的每篇文献进行打分。
1.6 统计学分析
使用R 4.0.2软件对提取的数据进行合并。通过Z检验测定了ORs值和95%CIs,以评价HLADQB1和HLA-DRB1等位基因与龋病风险的相关性。通过Cochran Q检验和I2检验检查研究间的异质性。I2取值越大,异质性越大。当I2<50%时,选用固定效应模型计算ORs值和95%CIs;当I2>50%时,选用随机效应模型合并效应量以减小部分异质性对结果的影响。按不同国家进行亚组分析以探讨异质性来源。测量漏斗图不对称的大小,以评估发表偏倚。采用敏感性分析评价结果的稳定性。
2 结果
2.1 文献筛选流程及结果
初检得到52篇文献,经过筛选,最终有10篇纳入本篇Meta分析。筛选流程见图1。
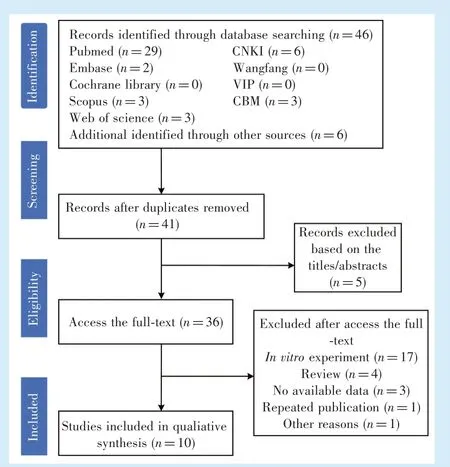
Figure 1 Literature screening procedure and results图1 文献筛选流程及结果
2.2 纳入研究的基本特征及偏倚风险评价
各研究的基本信息如表1所示。10项研究中,7项采集唾液进行检测,仅3项采集血液进行检测。除此之外,所有研究均采用WHO标准,即dmft/DMFT记录口腔情况,并进行分组。
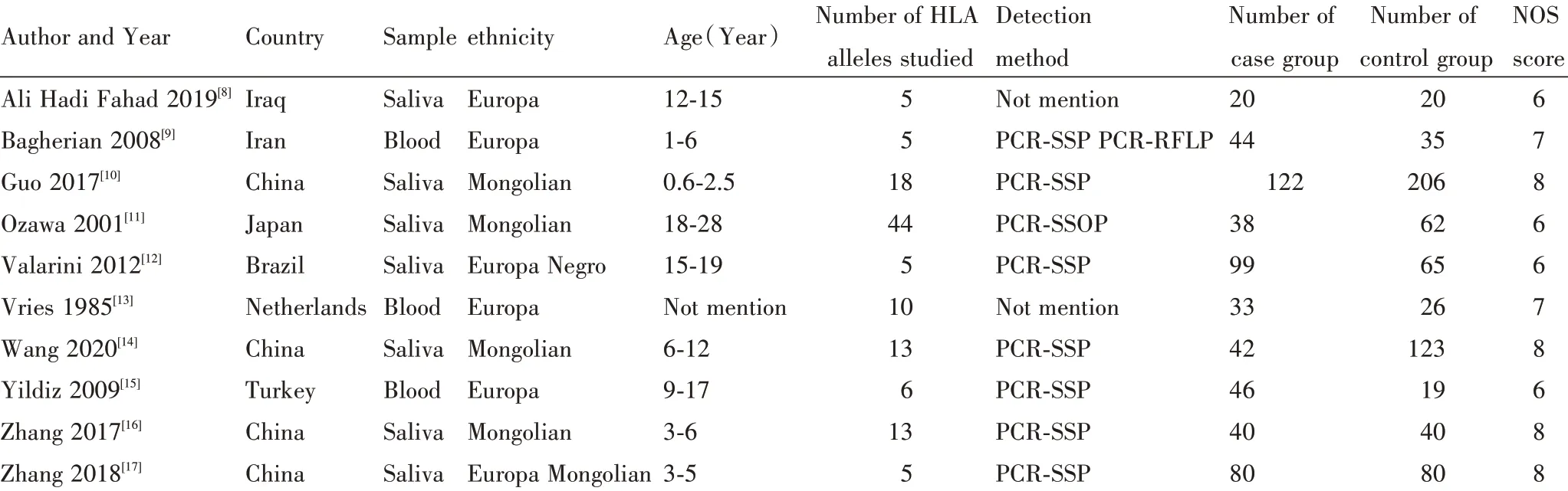
表1 纳入研究的基本特征Table 1 Characteristics of the studies in the meta-analysis
2.3 Meta分析的结果
2.3.1 HLA-DQB1等位基因家族多态性与龋病关系的Meta分析结果 纳入的研究中,确定了5个HLA-DQB1等位基因(HLA-DQB1*02、HLA-DQB1*03、HLA-DQB1*04、HLA-DQB1*05和HLA-DQB1*06)与龋病的相关性(表2)。结果显示仅HLADQB1*02(OR=0.52,95%CI=0.29~0.93,P=0.03)差异有统计学意义,是龋保护因素(图2)。
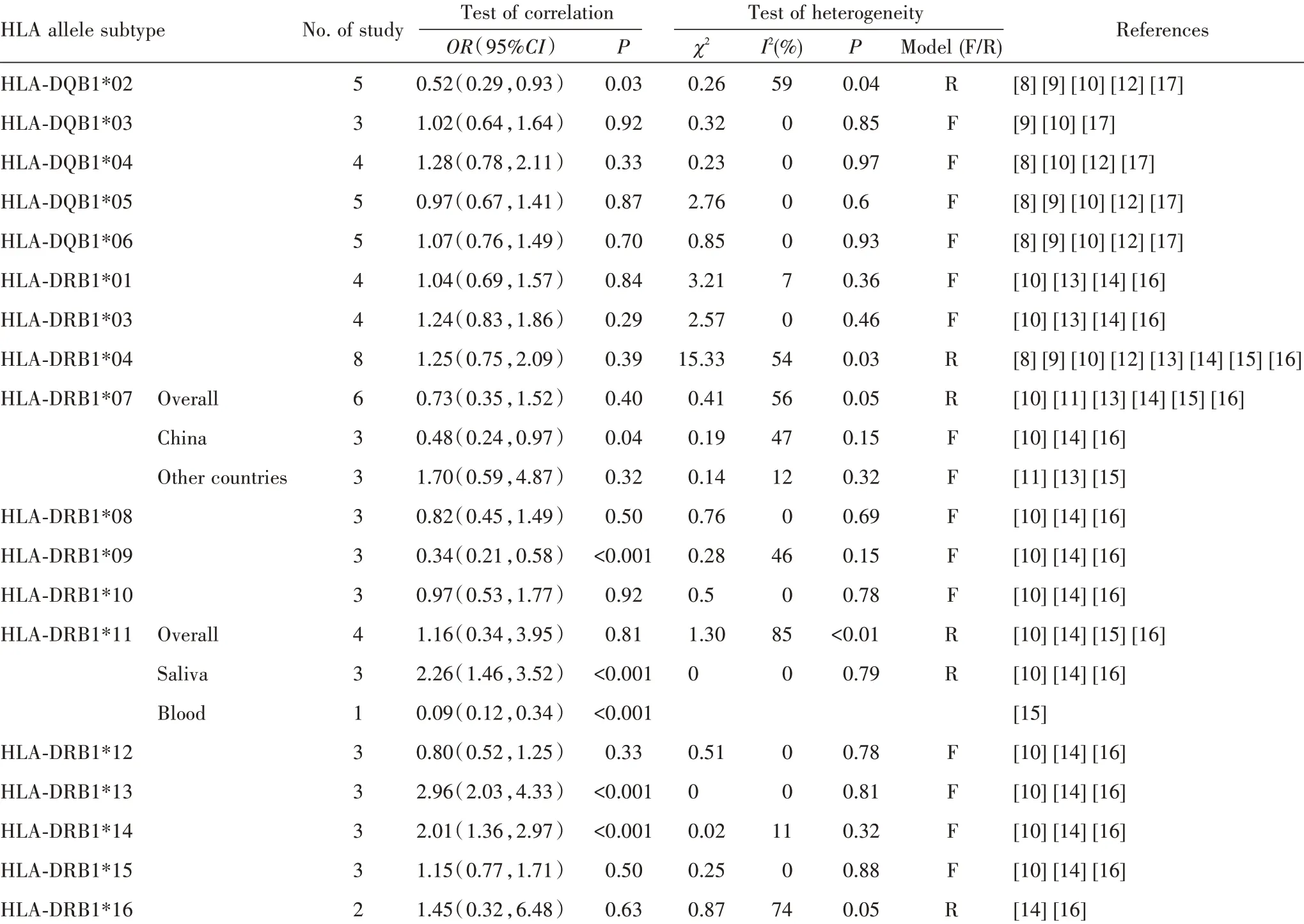
表2 HLA基因多态性与龋病相关性的Meta分析结果Table 2 Meta-analysis of the correlation between HLA gene polymorphisms and dental caries
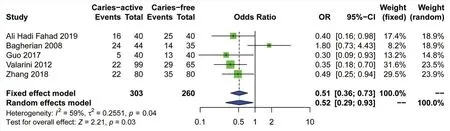
Figure 2 Forest plot of the correlation between HLA-DQB1*02 and caries图2 HLA-DQB1*02与龋病相关性的森林图
2.3.2 HLA-DRB1等位基因家族多态性与龋病关系的Meta分析结果 共对13个HLA-DRB1等位基因进行了分析,探讨多态性与龋病之间的相关性(表2)。以下7个等位基因与龋病的相关性无统计学意义:HLA-DRB1*01、HLA-DRB1*03、HLADRB1*04、HLA-DRB1*08、HLA-DRB1*10、HLADRB1*12、HLA-DRB1*15和HLA-DRB1*16。仅观察 到HLA-DRB1*09(OR=0.34,95%CI=0.21~0.58,P<0.001)是龋保护因素(图3)。相反,HLADRB1*13(OR=2.96,95%CI=2.03~4.33,P<0.001)和HLA-DRB1*14(OR=2.01,95%CI=1.36~2.97,P<0.001)增加龋病发病风险(图4、图5),是龋易感因素。HLA-DRB1*07亚组分析结果显示:在中国人群中是龋保护因素;HLA-DRB1*11亚组分析结果显示:在唾液组中为龋易感因素。
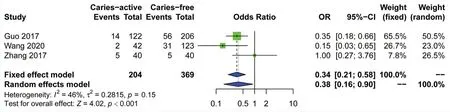
Figure 3 Forest plot of the correlation between HLA-DRB1*09 and caries图3 HLA-DRB1*09与龋病相关性的森林图
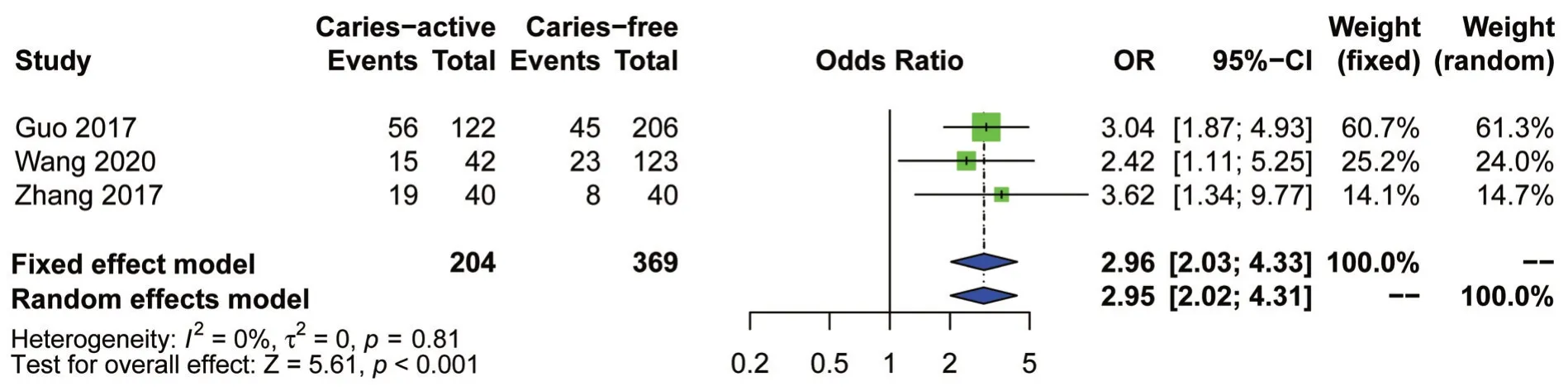
Figure 4 Forest plot of the correlation between HLA-DRB1*13 and caries图4 HLA-DRB1*13与龋病相关性的森林图

Figure 5 Forest plot of the correlation between HLA-DRB1*14 and caries图5 HLA-DRB1*14与龋病相关性的森林图
2.3.3 亚组分析 对HLA-DRB1*07和HLA-DRB1*11进行亚组分析,分析结果显示HLA-DRB1*07在中国人群中是龋保护因素(图6);HLA-DRB1*11在唾液组中为龋易感因素(图7)。
2.4 敏感性分析与发表偏移
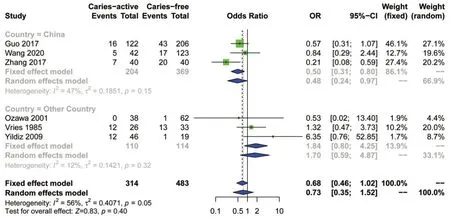
Figure 6 Forest plot of the correlation between HLA-DRB1*07 and caries in populations from different countries图6不同国家人群中HLA-DRB1*07与龋病相关性的森林图
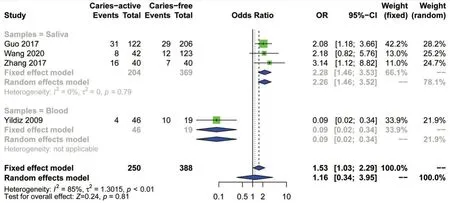
Figure 7 Forest plot of the correlation between HLA-DRB1*11 and caries in different samples图7 不同样本中HLA-DRB1*11与龋病相关性的森林图
在分析HLA-DRB1*04多态性与龋病的相关性时,在排除张瑞涵等[16]的研究后,异质性从54%下降到20%(图8),合并ORs未发生实质性变化,表明结果具有统计学稳健性。在分析HLA-DQB1*02多态性与龋病的相关性时,在排除Bagherian等[9]的研究后,异质性从59%下降到0%(图9)。漏斗图的形状在所有基因中基本对称(以HLA-DRB1*04为例,图10),表明本次Meta分析结果的发表偏倚较低。
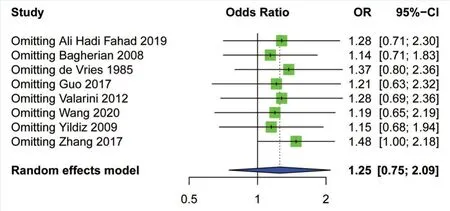
Figure 8 Sensitivity analysis of the correlation between HLA-DRB1*04 and dental caries图8 HLA-DRB1*04与龋病相关性的敏感性分析
3 讨论
随着经济的发展,龋病发生率逐年升高[18],在拉美、中东和东南亚国家最常见[19]。龋病的发生除了四联因素以外,遗传和环境因素也起到共同作用[20]。多种基因与龋病相关性的研究已被报道,Sharifi等[21]通过Meta分析证实釉蛋白(ENAM)rs3796704的G等位基因和GG基因型与龋病风险增加相关。龋病相关的HLA区域有HLA-DQB1和HLA-DRB1位点,目前已发表的HLA-DQB1、HLADRB1等位基因家族多态性与龋病之间相关性的研究结果并不一致。Bagherian等[9]研究发现HLADRB1*04是龋保护因素;Yildiz等[15]研究表明龋病患者的HLA-DRB1*04和HLA-DRB1*07等位基因频率显著高于无龋组;张瑞涵等[16]报道龋病患者的HLA-DRB1*04和HLA-DRB1*07等位基因频率均显著低于无龋组。本研究通过Meta分析评估了HLA-DQB1和HLA-DRB1与龋病的相关性。
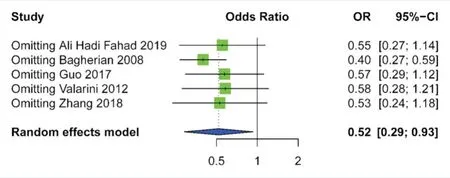
Figure 9 Sensitivity analysis of the correlation between HLA-DQB1*02 and dental caries图9 HLA-DQB1*02与龋病相关性的敏感性分析

Figure 10 Funnel plot of the correlation analysis between HLA-DRB1*04 and dental caries图10 HLA-DRB1*04与龋病相关性分析的漏斗图
在18个特异性等位基因中,HLA-DRB1*13和HLA-DRB1*14等位基因是龋易感因素;HLADQB1*02和HLA-DRB1*09则是龋保护因素,该结果与所纳入研究结果一致。
HLA-DQB1*02等位基因异质性过高,敏感性分析排除Bagherian等[9]的文献后,I2=0%;发现Bagherian等[9]与Fahad等[8]的结论恰好相反,出现这种情况的原因可能与样本的采集方法及样本的分类有关,Bagherian等[9]采集的是血液样本,而Fahad等[8]采集的是唾液样本。
结论有争议的等位基因包括HLA-DRB1*04、HLA-DRB1*07和HLA-DRB1*11。对于HLADRB1*04,张瑞涵等[16]、Bagherian等[9]及Fahad等[8]认为其是龋保护因素;而Yildiz等[15]和郭冉等[10]认为其是龋易感因素;余下纳入研究均认为其与龋病无相关性;经Meta分析(病例组459例和对照组561例)发现差异没有统计学意义,进行敏感性分析排除张瑞涵等[16]的研究后,合并ORs为1.48(1.00,2.18),P=0.008。HLA-DRB1*04与龋病无相关性,分析原因,除遗传因素外,龋病的发生、发展还依赖于其他的因素,如牙齿解剖结构、饮食和整体健康状况以及所处地域医疗环境等;目前尚需更多的研究来阐明两者间的关系。
对于HLA-DRB1*07,共纳入6篇文献,张瑞涵等[16]报导其为龋保护因素,而Yildiz等[15]报导其在土耳其人群中是龋易感因素;经Meta分析(病例组314例和对照组483例)发现并没有统计学意义(I2=56%,P=0.4)。按照研究人群所处国家进行亚组分析发现差异具有统计学意义(I2=47%,P=0.04);提示地域差异性可能是异质性的来源。
对于HLA-DRB1*11,共纳入4篇文献,经Meta分析发现差异并没有统计学意义(I2=85%,P=0.4)。根据采集样本的不同进行亚组分析,在唾液组样本中HLA-DRB1*11是龋保护因素(I2=0%,P<0.001),而在血液组样本HLA-DRB1*11是龋易感因素。分析差异原因,对于样本采集,目前尚无统一的标准,无法排除采样时的混杂因素;其次血液组样本量过少(病例组16例,对照组49例),结果仅具有参考性。后期应扩大样本量进行分析,进一步验证所得结果。
本研究局限性在于:纳入人群的年龄范围较大,同时包括了乳牙列、混合牙列及恒牙列,可能存在混杂偏移;生活、饮食习惯对龋病的发生也有一定的影响,HLA基因多态性与龋病之间的关系还需高质量的研究来进一步验证。本文的优势在于:纳入的10篇文献NOS评分均为6~8分,属于高质量文献,一定程度上减小了发表偏移;本篇Meta分析从样本的种类、种族、地域等方面对HLADQB1和HLA-DRB1基因多态性与龋病相关性进行分析,为龋病的预防提供依据。
综上,本Meta分析研究结果显示,HLA-DRB1*13和HLA-DRB1*14等位基因是龋易感因素,HLADQB1*02和HLA-DRB1*09是龋保护因素。
【Author contributions】 Chen Y,Wu ZY analyzed the data and wrote the article;Mo YL,Jing YH assisted in the data analysis,Liu YS revised the article.All authors read and approved the final manuscript as submitted.

