猪IGFBP3基因生物信息学分析
许金根, 胡 洪, 王重龙, 李庆岗*
(1.安徽科技学院 动物科学学院,动物营养调控与健康安徽省重点实验室,安徽 凤阳 233100;2.安徽省农业科学院 畜牧兽医研究所,安徽 合肥 230031)
胰岛素样生长因子结合蛋白(Insulin-like growth factor binding proteins,IGFBPs)的生物学作用主要是与胰岛素样生长因子(insulin-like growth factor,IGF)结合,通过延长IGF的半衰期而调节IGF的功能,以及独立于IGF的内在活性。作为IGFBP3
家族的一个重要成员,IGFBP3
是血清中与IGF结合的主要蛋白,通过调控IGF的生物活性而对机体生长发育起作用。因此,IGFBP3
是影响猪生长发育的功能候选基因。研究发现,大白猪IGFBP3
基因G3695A位点不同基因型个体的生长速度和背膘厚存在显著差异。在长白猪×蓝塘猪F资源群中,IGFBP3
基因A2670G位点与体长、眼肌面积、皮与脂肪率、胴体瘦肉量、瘦肉率存在显著或极显著相关。在蓝塘猪×长白猪杂交 群体中,IGFBP3
不同合并基因型TAT/TAT与GGC/GGC个体的背膘厚、肉色存在显著差异。猪IGFBP3
基因序列已被克隆出来,包括5个外显子和4个内含子。但目前关于猪IGFBP3
基因序列及其编码蛋白质结构和特征的研究比较少。因此,本试验综合利用生物信息学分析猪IGFBP3
基因序列及其编码蛋白质的结构和特征,为研究该基因在猪生长发育中的调控作用提供参考。1 材料与方法
1.1 供试材料
从美国国立生物技术信息中心(NCBI)数据库中(https://www.ncbi.nlm.nih.gov/),下载猪IGFBP3
基因mRNA序列(登录号:NM001005156)及其编码的蛋白质序列(登录号:NP001005156)。同时,下载其它种属黄牛(登录号:NP_776981)、水牛(登录号:XP_006051369)、绵羊(登录号:NP_001152748)、山羊(登录号:NP_001301148)、猫(登录号:XP_023105911)、狗(登录号:XP_003639605)、小鼠(登录号:NP_032369)、大鼠(登录号:NP_036720)、鸡(登录号:NP_001094504)和鸭(登录号:XP_027307091)的IGFBP3蛋白序列。
1.2 试验方法
用DNASTAR软件的EditSeq分析猪IGFBP3
基因编码区碱基组成。用ExPASy数据库的ProtParam(Https://web.expasy.org/protparam/)分析猪IGFBP3蛋白理化特性。用SOPMA软件(Https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)分析猪IGFBP3蛋白二级结构。用SMART数据库(Http://smart.embl-heidelberg.de/)分析猪IGFBP3蛋白功能域。用CBS数据库(Http://www.cbs.dtu.dk/services/)的SignalP 4.1分析猪IGFBP3蛋白的信号肽,用TMHMM 2.0分析猪IGFBP3蛋白的跨膜结构。用NetPhos 3.1和NetNGlyc 1.0分别预测猪IGFBP3蛋白的磷酸化位点和N-糖基化位点。
用DNAStar软件的MegAlign模块(Jotun Hein法,PAM设为250),分析不同物种IGFBP3氨基酸序列的同源性。用MEGA 5.0软件的邻接(Neighbor-Joining)法,构建不同物种IGFBP3氨基酸序列的系统进化树,自展值(Bootstrap)设为1 000次。
2 结果与分析
2.1 猪IGFBP3基因序列分析
猪IGFBP3
基因mRNA序列,编码区长882 bp(位于107~988处),共编码293个氨基酸。猪IGFBP3
编码区的A、G、C、T含量分别为17.57%、34.47%、35.83%、12.13%,A+T含量(29.70%)低于G+C含量(70.30%)。2.2 猪IGFBP3蛋白理化性质分析
经ProtParam软件分析,猪IGFBP3蛋白分子式为CHNOS,相对分子质量为31 722.27,理论等电点为8.97,不稳定系数为48.17,脂肪系数为61.06,平均亲水系数为-0.592。猪IGFBP3蛋白的氨基酸组成中,Val含量最高(占10.6%),Trp含量最低(占0.7%),正电荷(Asp和Glu)数31个,负电荷(Arg和Lys)数43个(表1)。
表1 猪IGFBP3蛋白氨基酸组成
2.3 猪IGFBP3蛋白二级结构和结构域分析
经SOPMA程序分析,猪IGFBP3蛋白二级结构包含61个α螺旋(占20.82%)、24个延伸链(占8.19%)、15个β转角(占5.12%)和193个无规卷曲(占65.87%)(图1)。经SMART软件分析,猪IGFBP3蛋白存在1个位于第38~118处的胰岛素生长因子结合蛋白同源物(IB)结构域和1个位于第239~291处的甲状腺球蛋白Ⅰ型重复(TY)结构域(图2)。

2.4 猪IGFBP3蛋白信号肽和跨膜结构分析
经SignalP 4.1软件预测,猪IGFBP3蛋白信号肽切割位点位于第27~28处(图3)。经TMHMM 2.0软件预测,猪IGFBP3蛋白无跨膜结构(图4)。

图3 猪IGFBP3蛋白信号肽预测
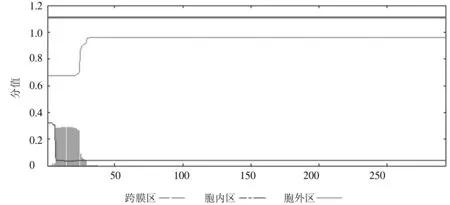
图4 猪IGFBP3蛋白跨膜结构预测
2.5 猪IGFBP3蛋白磷酸化和糖基化位点分析
经NetPhos 3.1预测,猪IGFBP3蛋白含26个磷酸化位点,分别为17个丝氨酸位点(位于第23、29、140、142、147、150、158、169、185、194、196、203、204、206、233、256和289处)、5个苏氨酸位点(位于第159、172、199、210和222处)和4个酪氨酸位点(位于第192、212、248和270处)(图5)。经NetNGlyc 1.0预测,猪IGFBP3蛋白含3个N-糖基化位点,分别位于第118、138和201处(图6)。

图5 猪IGFBP3蛋白磷酸化位点预测
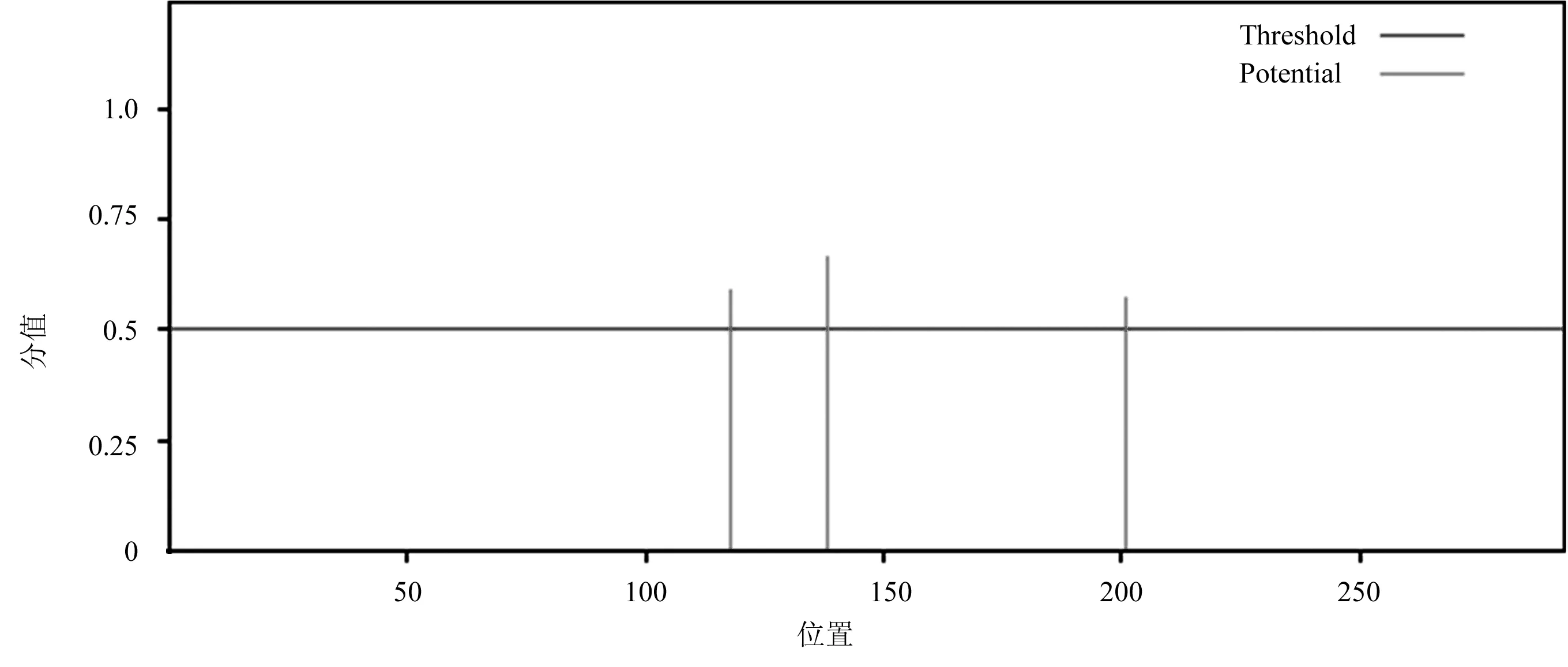
图6 猪IGFBP3蛋白N-糖基化化位点预测
2.6 不同动物IGFBP3蛋白同源性和系统进化分析
经DNASTAR软件的MegAlign分析,猪IGFBP3蛋白与水牛、黄牛、山羊、绵羊、猫、狗、小鼠、大鼠、鸡和鸭的氨基酸序列同源性分别为91.2%、90.8%、87.8%、87.8%、87.2%、86.9%、82.3%、81.2%、73.9%和73.3%(图7)。IGFBP3蛋白氨基酸序列在不同动物中的同源性较高,表明IGFBP3蛋白在进化中较保守。

图7 IGFBP3氨基酸序列同源性分析
利用MEGA软件对不同动物IGFBP3氨基酸序列构建系统进化树(图8)。猪IGFBP3蛋白与同属于偶蹄目的水牛、黄牛、山羊和绵羊亲缘关系最近,与鸡和鸭亲缘关系最远,这与氨基酸同源性分析结果一致,表明IGFBP3的发育进化符合物种进化规律。
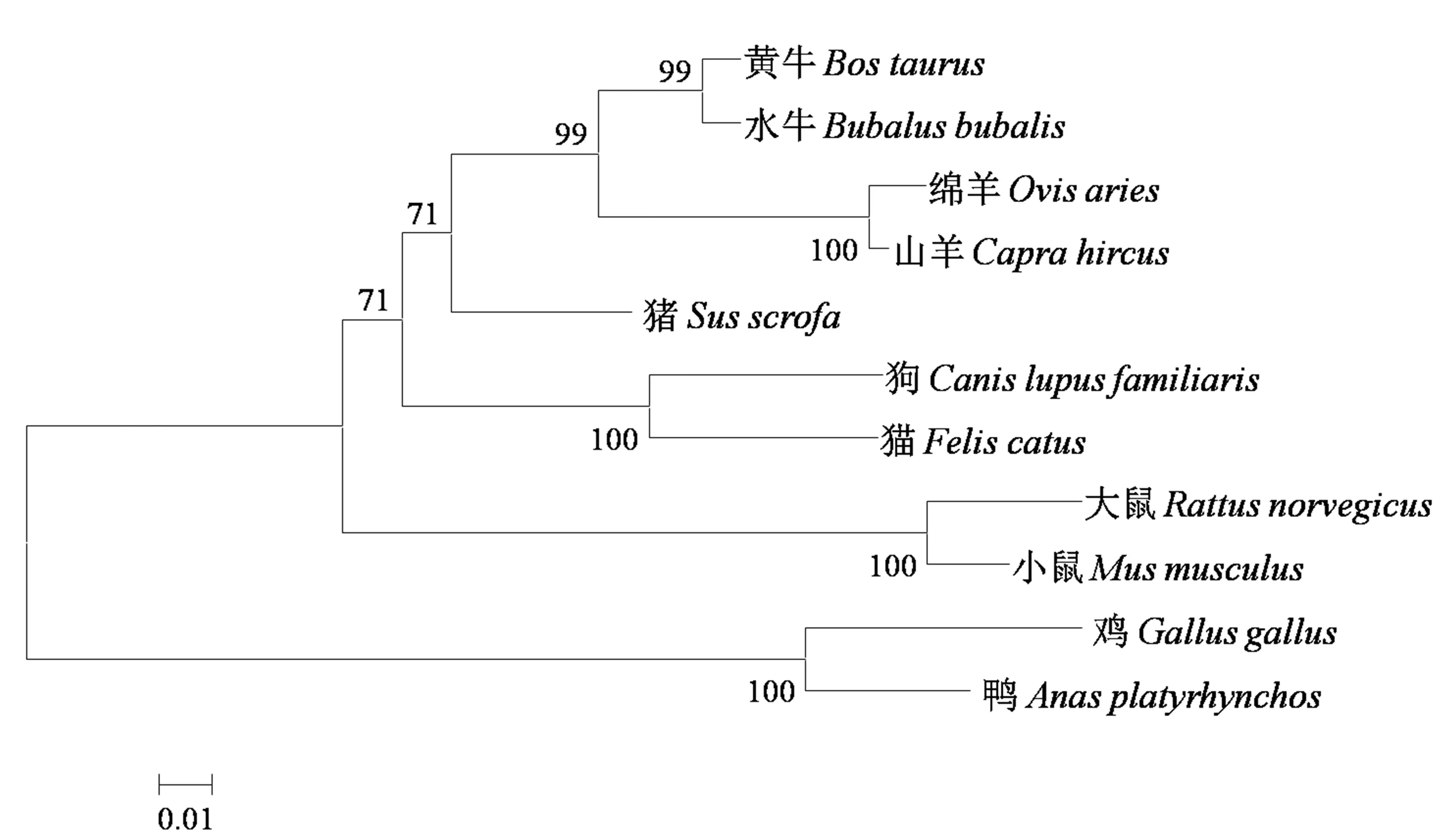
图8 基于IGFBP3氨基酸序列的系统进化树(邻接法)
3 结论与讨论
研究发现,150日龄的肥胖型太湖猪IGFBP3 mRNA表达量极显著低于瘦肉型长白猪。西藏小型猪肝脏、肾脏、肌肉、心脏IGFBP3 mRNA表达量均高于军牧一号猪,肾脏中IGFBP3蛋白含量显著高于军牧一号猪。提示IGFBP3
基因与猪的脂肪沉积、生长发育相关。因此,研究IGFBP3
基因及其编码蛋白序列的结构和特征,为进一步研究IGFBP3
基因对猪生长发育的调控提供参考。本研究通过生物信息学分析了猪IGFBP3
基因及其编码蛋白质序列和结构特征。猪IGFBP3
基因编码区长882 bp,共编码293个氨基酸,G+C含量(70.30%)高于A+T含量(29.70%)。猪IGFBP3蛋白的分子式为CHNOS,相对分子质量为31 722.27,理论等电点为8.97,不稳定系数为48.17(>40),为不稳定蛋白,脂肪系数为61.06,平均亲水系数为-0.592(<0),为亲水性蛋白。猪IGFBP3蛋白的二级结构主要由无规卷曲(65.87%)和α螺旋(20.82%)构成,而延伸链(8.19%)和β转角(5.12%)比例较低。猪IGFBP3蛋白存在1个IB结构域和1个TY结构域,这与山羊IGFBP3蛋白的结构域预测结果一致。猪IGFBP3蛋白信号肽切割位点位于第27~28处,即存在一个长27个氨基酸残基的信号肽,但不存在跨膜结构,这与绵羊IGFBP3蛋白预测结果一致。磷酸化和糖基化是蛋白质翻译后修饰的重要方式。本研究预测猪IGFBP3蛋白含26个磷酸化位点(包括17个丝氨酸位点、5个苏氨酸位点和4个酪氨酸位点)和3个N-糖基化位点,表明这些氨基酸残基是猪IGFBP3蛋白潜在的修饰位点。猪IGFBP3氨基酸序列与水牛、黄牛、山羊和绵羊的同源性最高,与猫、狗、小鼠和大鼠的同源性>80%,表明IGFBP3蛋白序列在哺乳动物中的保守性较高。IGFBP3氨基酸序列构建的系统进化树结果显示,猪与水牛、黄牛、山羊、绵羊亲缘关系近,这与氨基酸同源性分析结果一致。

