犊牛腹泻粪便样本中纽布病毒VP1和RNA依赖性RNA聚合酶基因的序列分析
李斯熠,岳 华,2,汤 承,2*
(1.西南民族大学畜牧兽医学院,成都 610041; 2.动物医学四川省高校重点实验室,成都 610041)
纽布病毒(Nebovirus,NeV)是嵌杯病毒科(Caliciviridae)纽布病毒属(Nebovirus)的唯一成员。基因组长度为7 453~7 460 bp,分为两个开放阅读框(ORFs),编码RNA依赖性RNA聚合酶(RdRp)、主要衣壳蛋白(VP1)和次要衣壳蛋白(VP2)等结构[1]。由于缺乏类似其他嵌杯病毒的双命名系统,因此,NeV的基因分型仍基于RdRp核苷酸或完整VP1氨基酸序列分别表述。目前,NeV可分为3种RdRp型(NB-like、NA1-like和SC-yak)和3种VP1型(基因1~3型),其中,基因1型可进一步划分为4个 谱系(谱系1~4)[1]。
VP1是杯状病毒重要的结构蛋白,由S结构域和P结构域组成,P结构域进一步分为P1和P2子结构域,其中,P2结构域外表面含有组织血型抗原(histo-blood group antigens, HBGAs)的结合位点和抗原中和表位[1-2]。最近研究证实,NeV MA415-KOR毒株(基因1.3型)的VP1可与多种HBGAs结合,在病毒的感染与免疫过程中发挥重要作用[3]。RdRp与RNA的复制有关,它是一种易出错的酶,从而导致病毒快速进化[4]。
最近,本实验室证实NeV在我国的奶牛和青藏高原牦牛中广泛流行[5]。国内NeV流行毒株的RdRp基因型有 NB-like和SC-yak型;VP1基因型有1型(包括1.1、1.3和 1.4 3个谱系)、基因2型和3型,表明我国NeV VP1蛋白具有丰富的遗传多样性。本研究从2019年9—11月宁夏和河南的奶牛腹泻样本检测到VP1基因1.2型毒株,旨在通过分析其完整VP1和RdRp的分子特征,为进一步了解国内NeV的遗传多样性提供参考。
1 材料与方法
1.1 样本信息
2019年9—11月,从宁夏(8个场)和河南(5个场)分别采集了53份和58份犊奶牛腹泻粪便样本,共111份。所有样本于-80 ℃保存备用。
1.2 核酸的提取和反转录
用Trizol法提取样本总RNA,并使用PrimeScriptTM试剂盒(TaKaRa公司)反转录成cDNA,-20 ℃ 保存备用。
1.3 NeV的检测
采用Park等[6]的RT-PCR方法对NeV进行检测,所有扩增产物均由生工生物工程股份有限公司进行双向测序,并进行BLAST比对验证。
1.4 混合感染调查
参照相关文献中有关牛轮状病毒(bovine rotavirus, BRV)、牛冠状病毒(bovine coronavirus, BCoV)、牛病毒性腹泻/黏膜病病毒(bovine viral diarrhea virus, BVDV)和牛诺如病毒(bovine Norovirus, BNoV)的检测方法[7-10],进一步调查NeV阳性样本的混合感染情况。
1.5 完整VP1和RdRp的扩增
利用Primer 5.0设计引物用于扩增阳性样本VP1和RdRp,引物信息见表1。阳性PCR产物胶回收后连接至pMD19-T,再转入DH5α,增菌后筛选质粒,送至生工生物工程(上海)有限公司进行双向测序。
1.6 序列分析
使用Seqman7.0对序列进行拼接;使用MegAlign对序列进行同源性分析;使用MEGA 6.0中ClustalW进行多序列对比,并采用最大似然法构建核苷酸或氨基酸系统发育树。
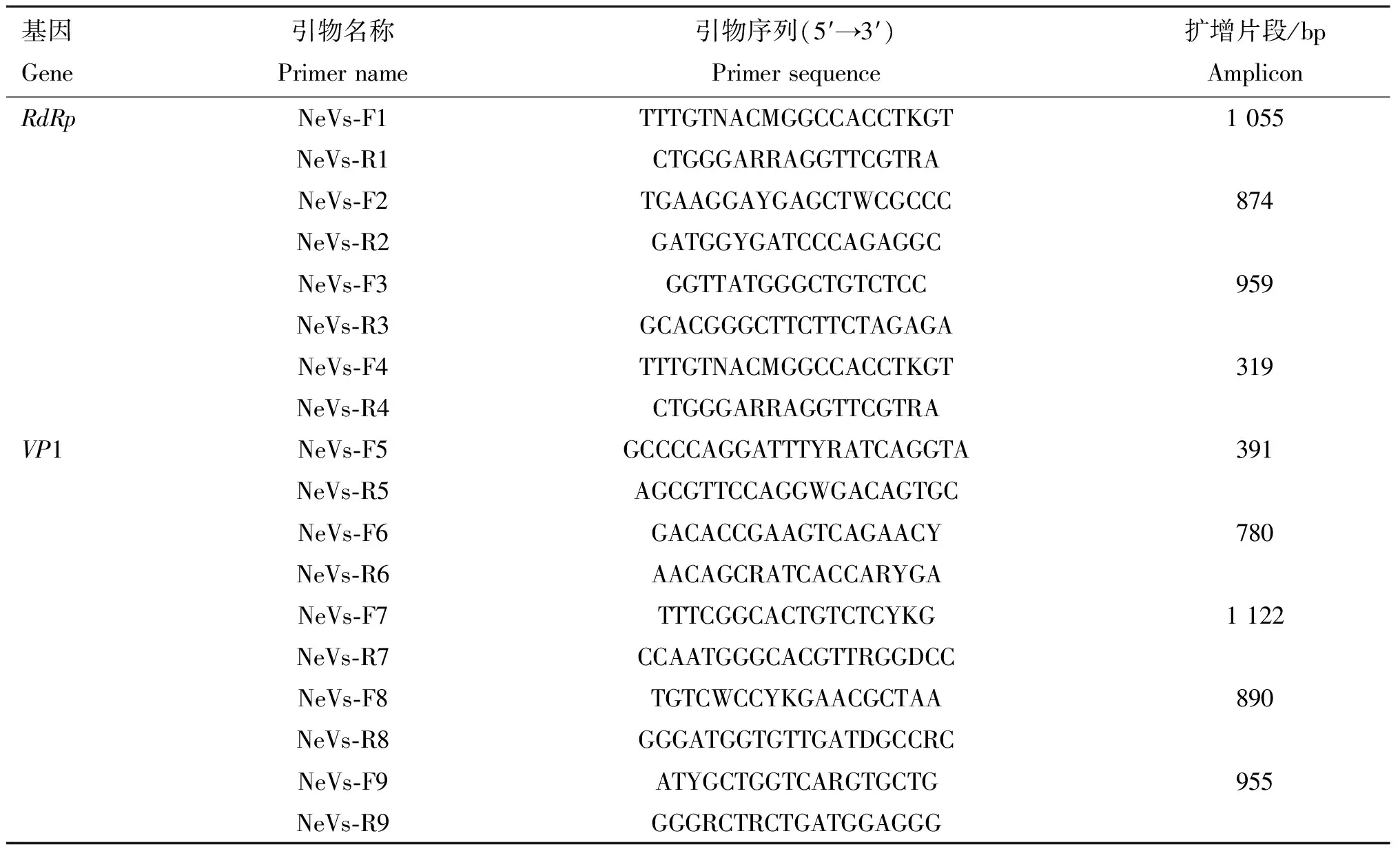
表1 NeV RdRp和VP1的扩增引物
2 结 果
2.1 NeV的RT-PCR检测结果及混合感染情况
从111份腹泻样本中检出11个NeV阳性,平均阳性率为9.91%(11/111),平均场阳性率为61.54%(8/13),其中,宁夏NeV阳性率为11.32%(6/53),场阳性率为50%(4/8);河南的阳性率为8.62%(5/58),场阳性率为80%(4/5)。11份NeV阳性样本中4份为NeV单独感染,7份为混合感染,见表2。
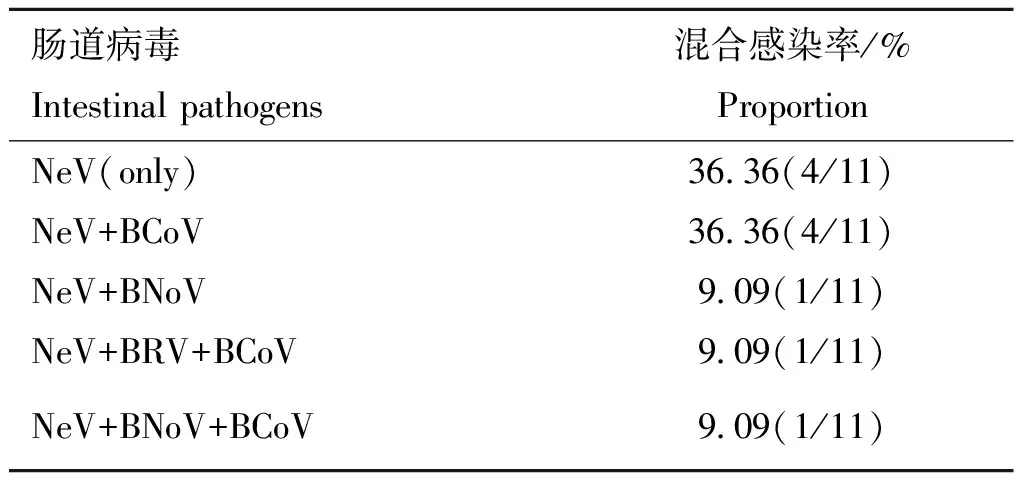
表2 NeV与其他腹泻病毒的混合感染
2.2 基因1.2型毒株完整VP1序列的分析
对11份NeV阳性样本进行了完整VP1的扩增,只从宁夏和河南样本中各成功扩增出2个完整的VP1序列,扩增结果见图1。4个样本完整VP1长度均为1 647 bp,编码549 aa (登录号为MW218190~MW218193),核苷酸相似性为86.7%~99.9%,氨基酸相似性为96.2%~100.0%。与GenBank中36个基因1.1型NeV毒株的核苷酸相似性为83.5%~89.8%,氨基酸相似性为92.0%~95.1%;与仅有的3个基因1.2型NeV毒株的核苷酸相似性为86.5%~89.5%,氨基酸相似性为95.1%~97.8%;与16个基因1.3型NeV毒株的核苷酸相似性为84.4%~87.3%,氨基酸相似性为92.0%~94.4%;与14个基因1.4型NeV毒株的核苷酸相似性为79.9%~86.8%,氨基酸相似性为88.4%~95.1%。与2个基因2型NeV毒株的核苷酸相似性为69.3%~69.8%,氨基酸相似性为75.4%~76.7%。与2个基因3型NeV毒株的核苷酸相似性为71.6%~72.8%,氨基酸相似性为79.1%~80.7%。完整VP1氨基酸进化树显示,本研究的4个毒株与美国毒株TCG、CV519-OH和日本毒株OKY共同聚为一个独立的分支,属于基因1.2型(图2)。
本研究的河南毒株与宁夏毒株相比,有21个共同的氨基酸突变,13个位于P2区(S280T、T/M288Q、A290V、N291E、I297M、T323V、E355G、N369S、D378 N、N383H、A404S、R416Q、E418D),3个位于P1区(T219S、L229Q、G454A),5个位于S区(H11Q、H12 N、A56T、I80V、N95H)。4个毒株与GenBank中仅有的3个基因1.2型毒株相比,有3个相同的氨基酸突变,1个位于P2区(D380E),2个位于P1区(G453S和S477T)。与21条国内基因1.1型毒株相比,有12个相同的氨基酸突变,9个位于P2区(H/C/R292V、V298A、G326E、G341S、T384A、L389V、R/S401Q、A/T/V402S和I420V),2个位于P1区(T493 N和L523I),1个位于S区(V/M/T154I)。与4条国内基因1.3型毒株相比,有21个相同的氨基酸突变,18个位于P2区(E293 N、G294R、L311F、T322S、T325G、A326P、V332I、G341S、N342D、I353V、Y368F、A377I、T382V、V384T、Y398F、H401Q、A403T和I420V),2个位于P1区(C268S和S476T),1个位于S区(V154I)。与2条国内1.4型毒株相比,有17个相同的氨基酸突变,14个位于P2区(E293 N、G294R、L311F、A322S、S325G、T326P、A327D、V332I、G341S、V350T、A377I、Q380E、T382V和Y398F),2个位于P1区(C268S和S476T),1个位于S区(V154I)。
M. DL2000 DNA相对分子质量标准;1~5.VP1分段扩增结果M. DL2000 DNA marker;1-5.Amplification result of VP1 fragments图1 VP1扩增电泳图Fig.1 Electrophoregram of VP1 amplification
2.3 完整RdRp基因序列的分析
成功获得4条NeV完整的RdRp序列,与VP1序列来自相同的样本,扩增结果见图3。RdRp长度均为1 455 bp,编码485 aa。4个完整RdRp序列之间的核苷酸相似性为95.1%~100.0%,氨基酸相似性为99.4%~100.0%;与GenBank中5个完整NB-like毒株的核苷酸相似性为85.3%~94.8%,氨基酸相似性为95.9%~99.4%;与1个完整NA1-like毒株的核苷酸相似性为78.4%~78.7%,氨基酸相似性为89.7%~89.9%;与2个完整SC-yak毒株的核苷酸相似性为67.2%~67.8%,氨基酸相似性为75.1%~75.9%。完整RdRp核苷酸遗传进化显示,本研究的4个完整RdRp序列与国内毒株Bo/LZB-1/17/CH和Bo/YLA-2/17/CH的遗传关系最近,属于NB-like型(图4)。
对完整RdRp序列进行进一步分析,与其他3个NB-like型毒株相比,国内6个NB-like型毒株(包括本研究的4株)有36个相同的核苷酸突变,其中28个为无义突变(G60A、A168G、G174A、C192T、C291A、C309T、G348A、G363A、A/G/C405T、G435A、A474G、T/A519G、A525G、G546A、C582T、A/G735C、A/C753C、A798G、T/G/C879A、C1002T、C1068T、T/A1116C、T1125C、A/G1158C、G1221A、T1227C、T1305A和T/C1317G),8个为有义突变(G97A、C/T511A、G862A、A1036G、T1037C、T1228A、T1230C和T1421C),这些有义突变导致了6个氨基酸突变(G33S、L171M、V288M、M346A、S410T和M474T)。
3 讨 论
NeV是犊牛腹泻的重要病原,在世界各地有广泛的地域分布[11-16]。NeV是国内犊牛腹泻的新发病原,在新疆、陕西、河南、四川等地犊牛腹泻粪便均有较高的检出率,该病毒在我国的流行情况值得进一步被监测。本研究首次在宁夏犊牛腹泻粪便检出NeV,检出率为11.32%,为当地犊牛腹泻的诊断和防治提供了参考。本试验在河南地区的检出率低于前期对2017—2018年采集的河南样本的检出率,可能是由于采样的时间和地点存在差异。此外,NeV既存在单独感染,也存在混合感染,这与先前报道的结果相似[5]。
迄今为止,GenBank中共登录了30条国内NeVVP1序列(21条基因1.1型,4条基因1.3型,2条基因1.4型,1条基因2型,2条基因3型),表明我国NeV VP1具有丰富的遗传多样性,但目前还未见基因1.2型毒株的报道。本试验成功地从河南和宁夏地区各获得了2条基因1.2型毒株的完整VP1序列,河南毒株与宁夏毒株在P2区有13个共同的氨基酸突变。前期研究显示,与国外NeV毒株相比,国内毒株有共同的进化趋势,可能和地域环境有关[5];本试验结果提示,国内不同地区的NeV毒株间VP1重要功能结构域氨基酸也可能存在较大的差异。鉴于VP1 P2区含有受体结合位点和中和抗原表位,这些氨基酸突变的生物学意义值得进一步关注。尽管目前没有关于NeV不同VP1基因型或基因亚型与HBGAs的结合能力差别的报道,但诺如病毒不同VP1基因型或亚型与HBGAs的结合存在差异[17-18],从而导致不同基因型的流行程度不同。如:The Norwalk virus(GI.1型)能与H或A抗原结合[17],VA207毒株(GⅡ.9型)能与Lex和Ley抗原结合[19],而大流行GⅡ.4型毒株似乎能够结合更广谱的HBGAs,从而获得更多的易感人群[18]。因此,NeV不同基因型或亚型的受体结合能力值得进一步研究。与国内流行毒株基因1.1型毒株相比,本试验获得的4个基因1.2型毒株在P2区有12个共同的氨基酸突变,这些突变导致的抗原漂移可能引起病毒的免疫逃避现象[20]。
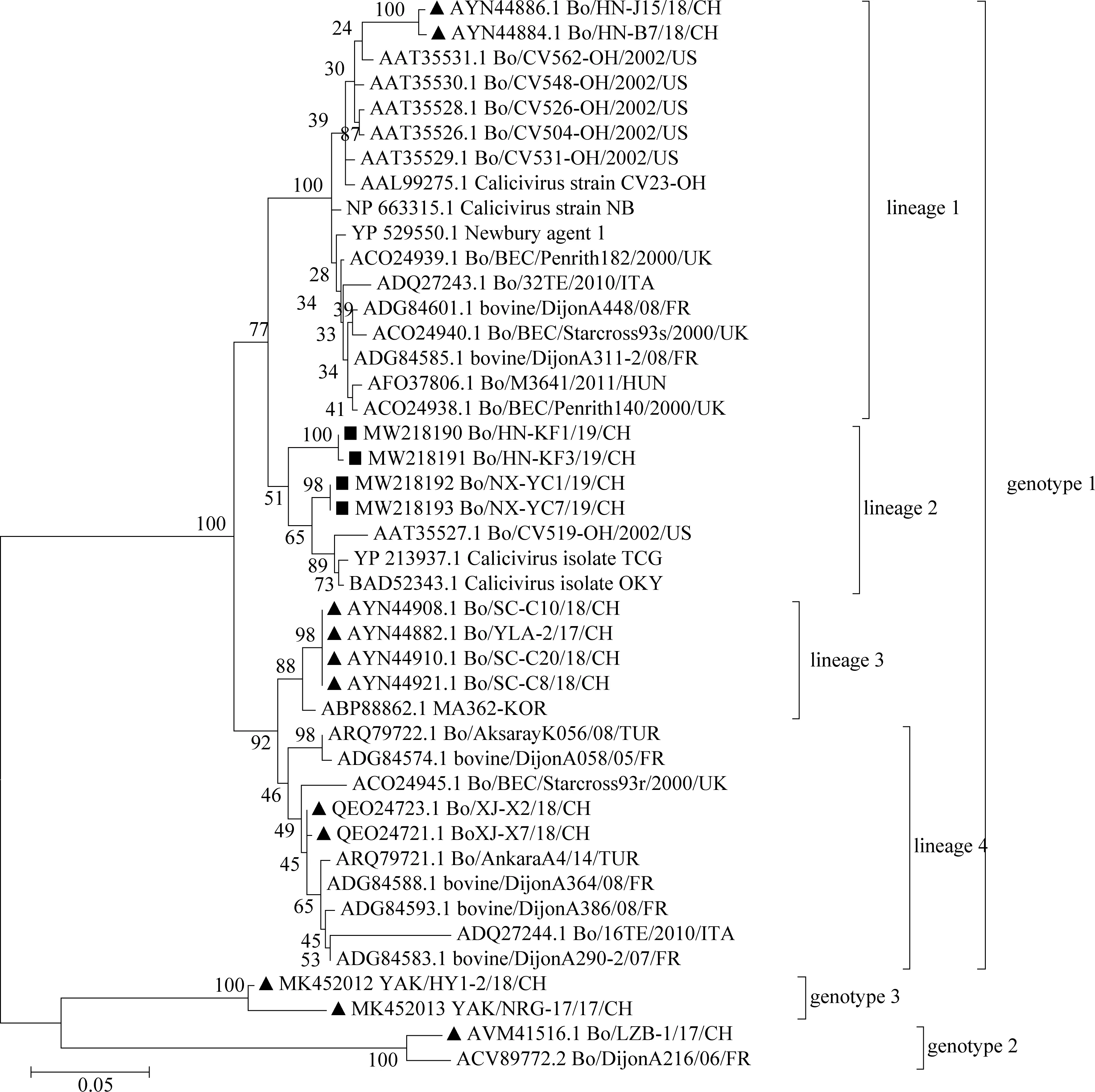
▲表示我国NeV毒株,■表示本研究的毒株▲ marks the NeV strains in China, ■ marks the strains in this study图2 完整VP1氨基酸的最大似然法进化树Fig.2 The maximum likelihood phylogenetic tree based on complete VP1 amino acids
M. DL2000 DNA相对分子质量标准;A~D. RdRp分段扩增结果M. DL2000 DNA marker;A-D. Amplification result of RdRp fragments图3 RdRp扩增电泳图Fig.3 Electrophoregram of RdRp amplification
RdRp对杯状病毒的复制起着重要作用,复制的保真度可能影响病毒的传播能力[21-22]。例如,在诺如病毒中,大流行毒株GⅡ.4的RdRp具有较高的突变率,显示出较低的复制保真度,表明RdRp的单点突变对病毒的复制和传播产生了影响[22-23],这种由RdRp驱动的遗传多样性对病毒的适应性至关重要[21]。本试验成功获得了4个完整RdRp序列,使得GenBank中NeV完整RdRp序列由8个增加到12个。目前,NB-like型为主要的流行毒株,与GenBank中其他3个NB-like型毒株相比,国内6个NB-like型RdRp序列(包括本研究的4株)共有36个相同的核苷酸突变,显示国内NeV毒株具有共同的进化趋势。这种地域相关的RdRp进化现象也在韩国的NeV毒株中观察到[12]。与NA1-like型毒株和SC-yak型毒株相比,NB-like型毒株在RdRp区分别有110个和222个核苷酸突变,并且大多数核苷酸为无义突变。尽管目前关于RdRp无义突变的生物学意义并不清楚,但RdRp是NeV的检测靶基因,核苷酸突变会影响已有PCR检测方法的检出效果。因此监测RdRp核苷酸序列的遗传变异,验证或调整NeV的PCR检测方法,对保证检测结果的准确性具有重要意义。
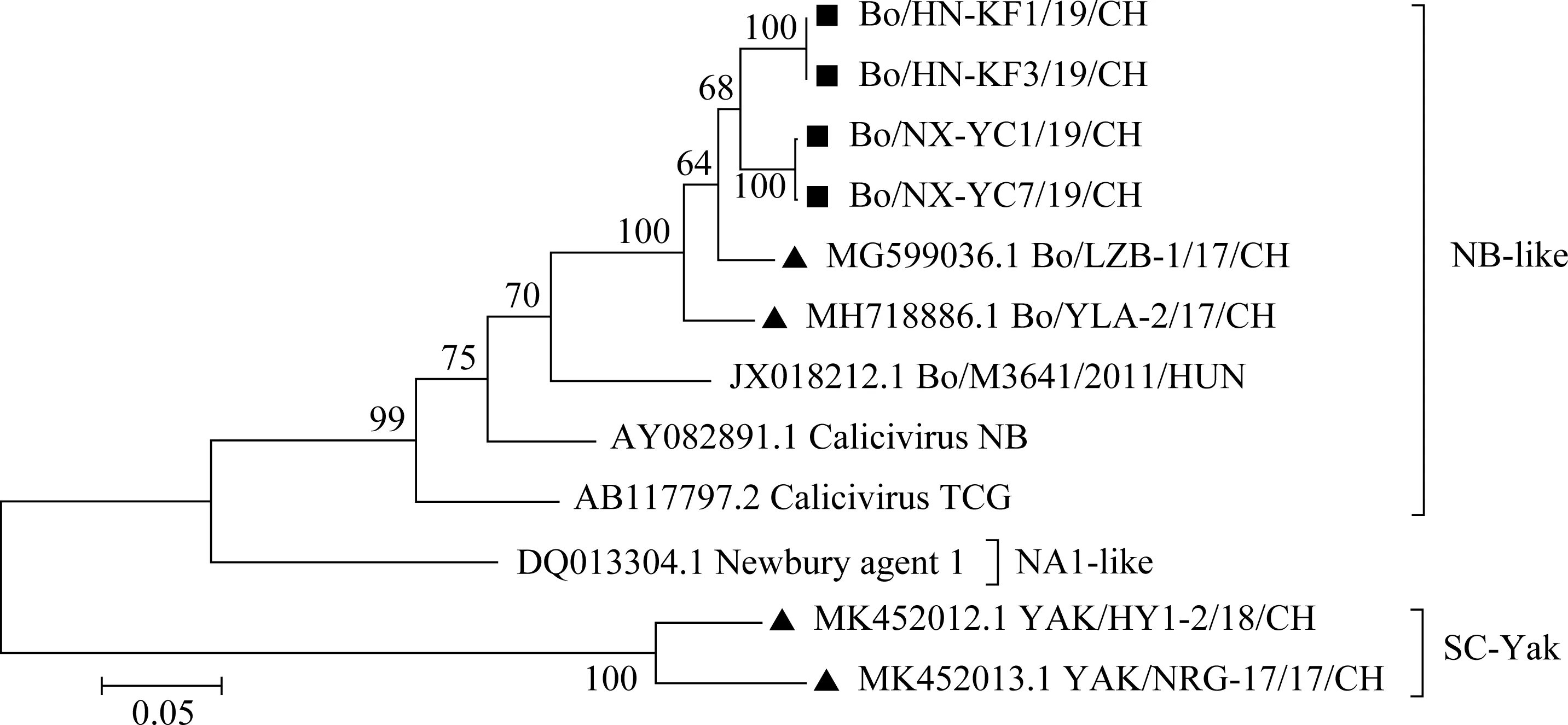
▲表示我国NeV毒株,■表示本研究的毒株▲ marks the NeV strains in China, ■ marks the strains in this study图4 完整RdRp核苷酸的最大似然法进化树Fig.4 The maximum likelihood phylogenetic tree based on complete RdRp nucleotide
4 结 论
本研究调查了2019年宁夏和河南地区NeV的流行情况,首次在我国检测到VP1基因1.2型毒株并从4个样本中同时获得基因1.2型毒株的完整VP1和RdRp序列,为国内NeV分子流行病学调查和遗传进化研究等提供有用信息。

