水稻粒型调控机制及相关基因在育种中应用研究进展
贾修齐 薛超 龚志云

摘要:粒型是影响稻米产量和的品质的重要数量性状之一。其遗传调控网络包括信号通路、泛素-蛋白酶体通路、丝裂原活化蛋白激酶(MAPK)信号通路、G蛋白信号通路及转录因子等多个调控通路。目前已经定位到的与粒型相关数量性状位点(QTL)有600多个,并克隆了70多个基因,这些基因间相互作用并于其他调控通路共同构成了水稻粒型调控网络。笔者对粒型调控网络和相关基因进行了总结和梳理并阐明其在育种上的应用前景,以期为水稻高产育种提供有利的理论和材料基础。
关键词:水稻;粒型;调控途径;QTL;育种应用
中图分类号:S511.03 文献标志码: A
文章编号:1002-1302(2021)12-0029-10
收稿日期:2021-03-21
基金项目:国家自然科学基金(编号:31871232);中国博士后科学基金(编号:2019M651981)。
作者简介:贾修齐(1995—),男,陕西澄城人,硕士,主要从事水稻遗传育种研究。E-mail:731150785@qq.com。
通信作者:龚志云,博士,教授,主要从事水稻基因组学研究。E-mail:zygong@yzu.edu.cn。
水稻是世界三大主要粮食作物之一,全球半数以上人口以稻米为口粮[1]。经过矮杆和杂种优势的广泛应用,水稻产量有了极大提高,但随着人类活动的影响使得耕地面积逐年下降和人口增加,水稻高产育种仍然是水稻育种的主要目标之一。水稻的产量由3个重要因素决定:有效穗数、每穗粒数和千粒质量。其中,有效穗数由水稻的分蘖能力决定,每穗粒数取决于一次、二次枝梗数和结实率,而千粒质量则受粒型性状的影响[2]。水稻粒型性状主要由水稻种子的粒长、粒宽和粒厚等组成,因此分离和克隆粒型相关基因是提高水稻产量的重要理论基础之一。已有研究表明,水稻粒型相关性状是受多个基因控制的数量性状,其遗传机制十分复杂。发现、评价和利用这些数量性状基因是水稻遗传育种学家研究的主要目标,其中数量性状位点(QTL)作图是实现该目标的重要手段之一。
1 QTL常用作图群体
数量性状一般是受2个或2个以上基因控制的性状,后代单株间差异只能用数量来区别,该性状的变异呈连续性易受环境条件影响,因此研究人员构建了多种作图群体对其进行分析。
1.1 暂时群体
暂时性群体不能永久保存,在经过自交或者近交后遗传特性及群体就发生改变。主要包括F2群体和回交群体(BC)。F2群体在短时间内易于构建且包含所有基因型,具有丰富的遗传信息,一般采用含目标性状的籼粳杂交构建F2分离群体对目标基因进行遗传连锁分析以及进行QTL定位。吕勇利用AZU和 9311构建的F2代群体结合高通量测序在QTL位点qGW2-1区间找到与粒宽完全连锁的候选基因Os02g0202950[3]。回交群体是在F1与亲本之一杂交产生的分离群体,其优点是作图效率比较高,构建时间短,可以有效减少微效基因的干扰,但所能提供的遗传信息的量没有F2群体丰富。这2个群体的遗传背景都很复杂,不利于纯系后代鉴定,所以利用这2个群体进行定位的精确性较低,更多用于QTL初定位[4]。
1.2 永久群體
1.2.1 CSSL群体
染色体片段代换系(CSSL)是受体亲本与供体亲本经过多次杂交、回交、自交然后结合分子标记辅助选择构建的少数来自供体亲本的基因片段,其余遗传背景与轮回亲本一致的永久群体。通过染色体片段代换系可以把复杂的数量性状分解成简单的单个孟德尔基因进行研究,是理想的遗传分析材料。其中染色体单片段代换系(SSSL)指与受体亲本间有且只有一个来自供体亲本的染色体片段[5]由于可以排除试验过程中遗传背景的干扰,具有单一性和稳定性,因此成为水稻中QTL鉴定、基因精细定位、克隆及基因聚合的优良材料,已经被广泛应用于水稻的分子设计育种并取得了较多的研究成果。Wang等以受体亲本华粳籼74及供体亲本Basmati385、Basmati370构建了SSSL,通过精细定位以及高精度连锁分析,将控制粒宽的QTL-GW8定位在8号染色体7.5 kb区间内[5]。Wu等以非洲野生稻W1411作为供体,非洲栽培稻IRGC102305作为受体构建了渗入系,利用大粒GIL25系和小粒IRGC102305系进行杂交,最后利用后代纯合的分离株系将控制粒长的QTL-GL4定位在M3和M4之间的5.9 kb区域内[6]。qGS10[7]及qGL12[8]也分别被定位到并将其范围缩小至300 kb和87 kb。
1.2.2 DH群体
DH群体是对 F1代材料通过花药离体培养后用秋水仙素进行染色体加倍得到的群体,其优点是遗传背景简单、植株都是纯合体,适合用于研究QTL与环境因素之间的互作,但缺点是所含的重组交换的信息较少不能够区分显隐性。Li等为定位GS5以珍汕97和H94构建DH群体,再通过多次回交将DH27(含有H94染色体片段)导至珍汕97构建群体最终将GS5定位在5号染色体[9]。
1.2.3 NIL群体
近等基因系(NIL)是结合分子标记通过多代回交筛选构建的群体。其优点是遗传背景干净只在目标区域内存在差异,检测灵敏度较高,可以用来对目标基因及性状进行QTL的定位、QTL之间互作以及效应性分[3],但NIL群体的构建往往需要花费更多的时间精力。Wang等用珍汕97和Nip(宽粒)构建了NIL定位并克隆了粒长QTL-qGL11[10]。
1.2.4 RILs群体
重组自交系群体是对F2 代群体进行筛选后,对目标系进行多次多代的自交而产生的群体,所以重组自交系群体每个单株都是纯合的,可以作为永久性的群体使用[3],RIL群体受环境因素影响较小,家系间的基因型不同但家系内纯合,定位结果的精确性相对较高,可以从该群体中挑选出纯合的、优良的株系用作研究品种。调控粒型的基因GW5/qSW5分别通过Asominori/Nip和IR24/Kasalath(细粒)杂交产生的不同重组自交系(RILs)被鉴定出来[11-12],Fu等利用粳稻春优84,以及叶片形态差异显著的籼稻春恢84构建的重组自交系(RIL)群体,检测到1个主要的叶长QTL-qLL9被定位到C-1640和C-1642这2个标记之间的16.17 kb区域内,包含3个开放阅读框(ORFs)[13]。
2 水稻粒型相关QTL的研究进展
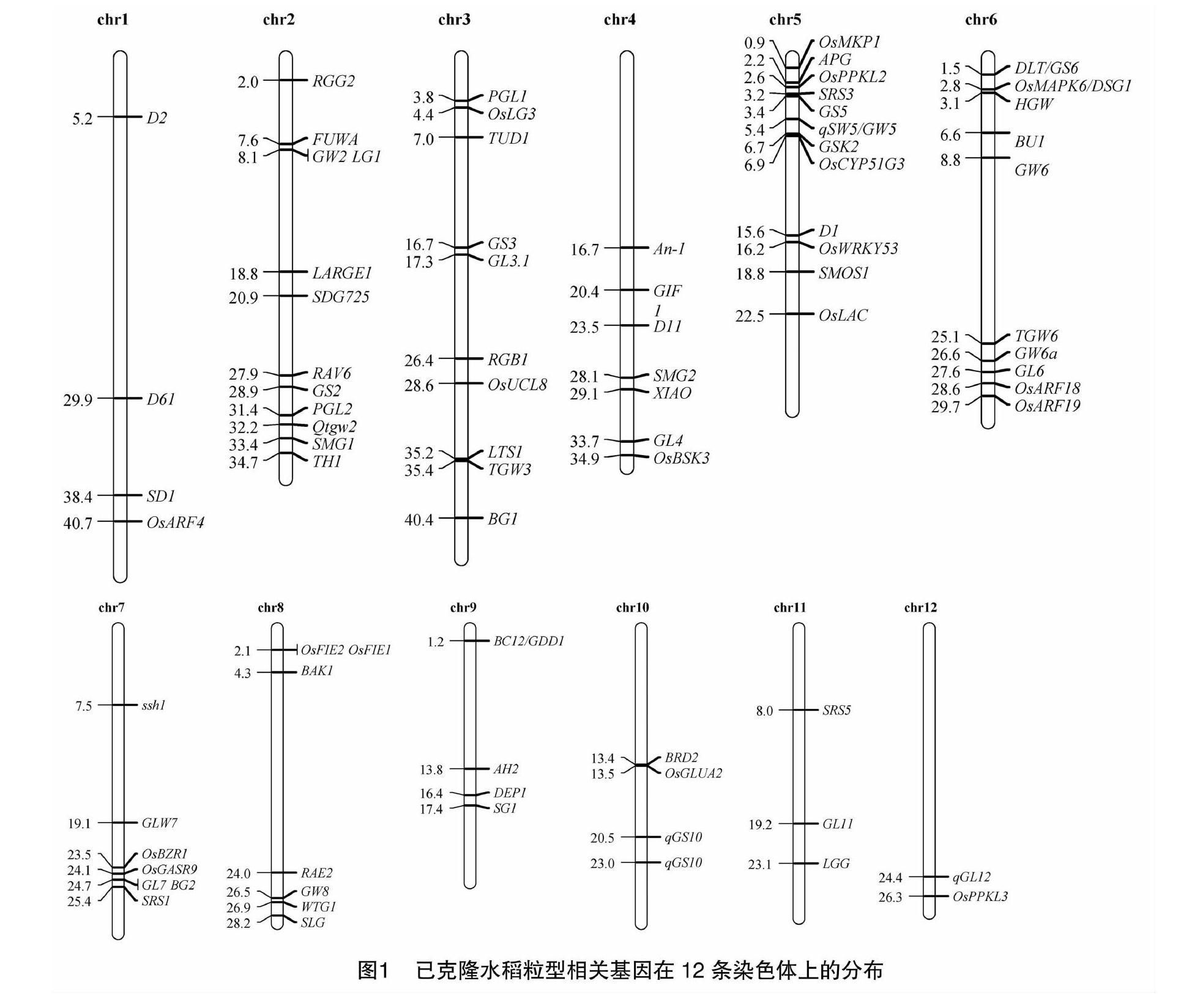
随着水稻基因组测序的完成,研究人员利用目标表型间差异明显的品种通过F2群体、BC群体、DH群体、RLIs群体、CSSL等遗传群体,在水稻12条染色体上定位了超过600个与粒型相关的QTL,与粒长、粒宽相关的QTL最多,而与粒厚相关的QTL最少,并克隆了一些对水稻粒型具有显著作用但不影响植株形态基因的主效QTL[14-15],经整理后见图1。
2.1 调控水稻粒宽相关QTL研究进展
粒宽是水稻粒型构成的主要因素,与其相关的QTL研究对提高种子千粒质量具有重要意义。Song等利用小粒品种丰矮占1号和千粒质量较高的WY3,将GW2定位在2号染色体W024和W004 2个标记之间的8.2 kb区间内[16],GW2的功能缺失会导致颖壳细胞分裂速度加快、颖壳变宽,有利于胚乳灌浆,增加粒质量。Shomura等以窄粒品种Kasalath和宽粒测序品种Nip为亲本构建的分离群体将qSW5定位于5号染色体负调控粒宽;Nip在该范围内有1 212 bp的碱基缺失以及16个SNP突变,而将Kasalath品种qSW5位点中ORF1表达量下调使粒宽变大[17];Li等以珍汕97为轮回亲本和H94构建DH群体在5号染色体短臂端的11.6 kb区间内定位到的GS5编码丝氨酸羧肽酶正调控籽粒大小[9,18]。Ruan等利用9311和培矮64S克隆出控制粒宽和粒质量的TGW2,该基因编码调控因子OsCNR1,通过影响颖片细胞增殖和扩张来调控粒宽和粒质量,TGW2基因启动子上游1 818 bp处的碱基替换(G→A)可增强TGW2的表达[19]。
2.2 调控水稻粒长相关QTL研究进展
水稻粒长相关QTL是目前鉴定并克隆最多的,GS3是水稻中首个被克隆的粒长和粒质量QTL[22-21],Fan等以大粒品种明恢63以及小粒品种川7构建的NIL群体,将GS3定位在第3号染色体上着丝粒附近的7.9 kb区间内,明恢63 GS3密码子TGC 突变成终止密码子TGA,使蛋白翻译终止,产生一部分氨基端具有类γ结构域的蛋白与DEP1或GGC2競争性结合Gβ,使粒长缩短 [20]。Zhang等选用千粒质量较轻的N643[千粒质量=(17.80±0.82) g]和千粒质量较高的N411[千粒质量=(72.13±2.32) g]进行杂交,经过高精度连锁分析将qGL3定位到标记XJ39和XJ26之间的46.6 kb区域内[22],qGL3编码含有2个Kelch功能域的蛋白磷酸酶OsPPKL1,负调控水稻粒长[23-24]。Wu等以野生稻W1411为供体,非洲栽培稻IRGC102305为受体构建了一套基因导入系,利用纯合的分离株系进行高精度连锁分析将调控内外颖纵向细胞的伸长来调控粒长的GL4定位在M3和M4之间的5.9 kb区域内 [6]。Ishimaru等以Kasalath作供体亲本,Nip为受体亲本构建NIL,利用重组纯合植株进行高分辨率定位,将TGW6定位在标记G214和G232之间的4.9 kb区域内,候选基因为Os06g0623700,TGW6编码一个具有吲哚乙酸(IAA)水解酶活性的蛋白,在库器官中Nip的tgw6等位基因通过控制IAA供应来限制细胞数量和粒长,在Kasalath中等位基因tgw6功能的丧失会对源器官产生多效性影响进而提高粒质量[25]。Yu等采用新方法Ho-LAMap克隆了OsLG3,编码APETALA2/乙烯反应元件结合蛋白,通过促进细胞数目的增加正向调控粒长且不影响稻米品质[26];康艺维试验所用的长粒品种IRAT109、SLG-1、Haobuka三者的启动子序列相同但与短粒品种存在差异,因此OsLG3致使籽粒变长千粒质量增加的主要原因是启动子区的自然变异[15]。TGW3/qTGW3/GL3.3的克隆分别来自亲本JZ1506和小粒品种黄华占、CW23和PA64、珍汕97和南洋占,通过编码类SHAGGY41激酶OsSK41/OsGSK磷酸化OsARF4负调控颖壳的细胞影响粒长[27-29]。Wang等以野生稻W1943为供体亲本,广陆矮4号为受体亲本,利用2 181个BC1F5个体进行高精度定位,最终将GL6定位到GL4350和GL4411这2个标记之间的6.1 kb区域内,GL6编码的PLATZ转录因子通过促进幼穗细胞的增殖正调控粒长,该转录因子与RPC53和OsTFC1之间存在互作,通过参与到RNA聚合酶Ⅲ转录机制,调节参与水稻籽粒发育基因的表达[30]。Si等利用籽粒间差异明显的380多个水稻品种进行GWAS(genome-wide association studies)分析,将控制粒长的主效基因QTL-GLW7定位在7号染色体上,其编码的转录因子OsSPL13通过调控颖壳细胞正调控籽粒大小[31]。
2.3 调控水稻粒型的多效性QTL研究进展
水稻中已经克隆的一些基因如GL2/GS2、GW7、GW6等具有一因多效,不仅调控粒型还会影响稻米品质。Hu等以浙江省特大粒地方品种宝大粒(BDL)、中花11、9311和武运粳7号为亲本,构建分离群体将GS2定位在2号染色体BG82和BGS83标记间的7.4 kb区间内[32];GL2/GS2通过编码转录因子OsGRF4促进细胞膨大和细胞分裂调控籽粒大小[33]。BG2编码细胞色素P450家族成员OsCYP78A13蛋白,OsCYP78A13的表达上调可促进细胞增殖使籽粒变大[34]。Wang等利用泰丰A和华粳籼74为亲本构建作图群体,最终将GW7基因准确定位于7号染色体标记S5和S6约2.6 kb的区域内,GW7编码1个TONNEAU1募集基序蛋白,该蛋白表达上调,通过促进纵向细胞分裂抑制横向细胞分裂使籽粒细长[35],也提高了稻米的外观品质[36]。Song等用籼稻品种Kasalath和粳稻品种Nip为亲本构建的作图群体,将GW6定位在第6号染色体上,GW6a编码具有组蛋白乙酰转移酶活性的OsglHAT1蛋白,该蛋白通过增加颖壳细胞数目及灌浆速率使粒质量得到提高[37]。GS9通过控颖壳细胞的分化负调控籽粒大小,Zhao等用清芦占11和Nip构建NIL群体初步将GS9定位到标记IN0927和IN0929之间的5.9 kb范围内,该区域只有1个候选基因LOC_Os09g27590被鉴定具有预测的开放阅读框,GS9控制颖壳中纵向细胞分裂加快细胞数目增加而横向细胞分裂速度变慢[38-39]。
3 水稻粒型调控网络
进一步理解水稻粒型相关基因调控方式是合理利用它们的前提与基础。在被子植物中受精卵、受精卵中央细胞和母体珠被分别发育成种子的胚、胚乳以及种皮,构成了种子基本结构[40]。因此,种子大小是由母体组织和合子组织协调控制的[41];通过母体组织控制籽粒大小的信号通路包括泛素-蛋白酶体通路、G蛋白信号通路、丝裂原活化蛋白激酶(MAPK)信号通路、多种植物激素信号通路、转录调节因子[42]以及表观遗传调控途径。而合子组织生长受到植物激素以及HAIKU途径调控[43],上述途径构成的调控网络见图2。
3.1 植物激素信号通路
植物激素通过对细胞周期的调控在籽粒大小发育方面发挥重要作用。研究表明,生长素通过浓度依赖性对种子发育进行调控,生长素的时空分布受生长素合成动态调节:极性运输、信号转导、生长素偶联以及生长素分解代谢来维持生长素在种子发育的最佳水平[44]。microR167在拟南芥、番茄、亚麻荠中被证实可以靶向调控生长素反应因子ARF6和ARF8,这些因子能调节植物种子发育[45];Liu等研究发现的植物生长素原初响应基因BG1参与生长素运输和转运,是植物特异的控制器官大小的调节因子,其突变体增加了生长素含量,改变了生长素的分布,而敲除材料则减少了生长素的运输,在水稻和拟南芥中使用BG1基因均可提高植株生物量、种子质量和产量[46]。在甘蓝型油菜中,通过正向遗传学鉴定出的ARF18被证实在角果发育过程中可以通过调控生长素信号通路来决定粒质量[47];一些生长素介导的与种子大小相关的基因如SMOS1编码的一个生长素调控的APETAL2类转录因子,在水稻中受OsARF1的正調控;SMOS1使细胞变小、数目增加表现为种子变小[48-49];种子发育是一个复杂的动态过程,受到多种植物激素的严格调控,生长素与其他植物激素之间协同或拮抗调节种子发育,如:SMOS1可以与GRAS转录因子SMOS2/DLT相互作用,通过油菜素内酯(BR)信号正向调控种子发育来调节其转录活性,这一发现证明生长素和BR信号通路的相互作用可以调节作物的种子大小[44]。
3.2 G蛋白信号通路
在水稻中异源三聚体G蛋白参与生长、 发育的多个信号通路,G蛋白由1个α亚基基因(RGA1)、4个GTP结合蛋白基因(XLGs)、1个β亚基基因(RGB1)和5个γ亚基基因(命名为RGG1、RGG2、RGG3/GS3、RGG4/DEP1和RGG5/OsGGC2)组成[51]。Gα和Gβ蛋白都是细胞分裂和籽粒大小的正调控因子,RGA1/D1编码G蛋白的α亚基,突变体d1中G蛋白失活细胞分裂减少籽粒变小,同时还发现G蛋白途径与植物激素途径存在影响,突变体d1 Gα亚基的缺陷会影响赤霉素(GA3)和油菜素内酯(BR)的信号传导[51]。RGB1编码Gβ亚基,其突变体会导致籽粒变小[52],Sun等结果表明,RGB1在整个生长过程中发挥着重要作用,RGB1为3种Gγ蛋白粒型调控通路提供了基础[53]。DEP1编码G蛋白的γ亚基,是水稻穗长、株高、粒型等的负调控因子[54],当RGA1、DEP1和GCC2单独或共同存在时,可以使粒型增大。GS3是栽培稻自然群体和育种群体中最重要的粒长负调控因子[55-56];野生型的等位基因N端有OSR(organ size regulation)结构域会产生中等粒,功能缺失会形成长粒;而C端结构域功能缺失会丧失对OSR结构域的抑制产生极短粒[53];GS3通过与DEP1和GCC2竞争结合Gβ从而使粒型变小,与野生型相比敲除RGA1后的籽粒变的极短,籽粒长度减少约35%,而DEP1和GCC2敲除后的表型基本一致;敲除DEP1或GCC2或过表达GS3均可导致中短粒(减少7.47%~11.18%),相反过表达DEP1或GCC2或敲除GS3,籽粒长度和粒质量均适度增加(6.85%~13.03%),在GS3的敲除突变体中过表达DEP1可使粒长进一步增加(约增加20%),也使粒质量大幅增加(28.45%)[53],DEP1的自然变异体会改变穗型结构和氮素利用效率。Gγ亚基的RGG1和RGG2参与非生物胁迫的调控,Miao等通过测定赤霉素(GA3)处理后第二叶鞘的长度和GA诱导的种子α-淀粉酶活性,发现RGG2也参与了GA信号传导,可以通过GA途径调控负调控水稻的籽粒和器官大小[57]。
3.3 丝裂原活化蛋白激酶(MAPK)信号通路
丝裂原活化蛋白激酶(MAPK)信号通路已被证明在植物的防御反应和与生长发育相关的多个过程中发挥重要作用。MAPK通路是1个三级激酶级联反应,包含至少3个激酶:MAPK激酶激酶(MKKK),MAPK激酶(MKK)和MAPK[58]。Xu等提出生长信号可能激活OsMKKK10-OsMKK4-OsMAPK6级联反应,丝裂原活化蛋白激酶OsMAPK6主要分布在细胞核和细胞质中,并广泛分布于各个器官中,主要分布在小穗和小穗壳中,激活的OsMAPK6通过促进小穗壳细胞增殖来调节籽粒大小,该信号通路对水稻籽粒大小和质量有正向调控作用[59];而GSN1是级联反应的负调控因子,通过编码丝裂原活化蛋白激酶磷酸酶OsMKP1与OsMAPK6相互作用,使OsMAPK6去磷酸化失活[60]。另外,MAPK信号通路同时也会影响植物激素油菜素内酯(BR),激素敏感性试验表明,dsg1突变体对油菜素内酯(BR)的敏感性较低。在dsg1突变体中内源BR水平降低,一些BR信号通路基因和反馈抑制基因的表达均发生改变,表明OsMAPK6可能影响了BR的动态平衡和信号转导[61];SMG1编码1个OsMKK4通过影响BR的应答以及BR相关基因表达,在籽粒生长中可能作为MAPK通路和油菜素内酯途径间的连接因子,抑制OsMKK4表达,会使籽粒变小[62]。
3.4 泛素-蛋白酶体途径
泛素化途径是将泛素偶联到底物蛋白内部的赖氨酸残基上,从而使靶蛋白被蛋白酶体降解,泛素通过影响蛋白质运输、信号转导、降解等直接或间接地调控粒长[63]。泛素化过程需要多个酶参与:泛素活化酶E1,泛素结合酶E2,泛素连接酶E3。泛素分子被E1激活连接到E2,E3帮助E2识别靶蛋白,将泛素转移到靶蛋白进行泛素化[64],被蛋白酶体降解[65-66],泛素化是一个可逆的过程可以通过去泛素酶( DUBs )[61]释放底物上的泛素;WTG1编码一种具有去泛素活性的Otubain-like蛋白酶,它通过影响细胞伸长来决定粒型大小[68]。GW2编码1个E3泛素连接酶负调节细胞分裂,影响粒宽及粒质量[16]。LG1编码去泛素化特异性蛋白酶OsUBP15,OsUBP15功能丧失和表达下调会产生更窄、更小的籽粒,OsUBP15和GW2在调控籽粒大小上存在可能的相互作用,通过聚合OsUBP15和GW2有可能提高粮食产量[69]。
3.5 转录因子调控
水稻中已成功分离并鉴定了一批控制籽粒的主要数量性状位点如GS3、GS5[5]、GS9 [9]、GW2[16]、GW5[17-18]、GW7[25]、GW8[35]和TGW6[38]。研究表明,这些编码转录因子的QTL是调节籽粒大小的关键,GLW7编码转录因子OsSPL13,该转录因子通过小穗壳内细胞的增殖来增加籽粒大小[31]。GW8编码转录因子OsSPL16,通过促进细胞分裂及灌浆速率来增加籽粒宽度和粒质量[5];同样,GS2/PT2/LGS1编码的转录调节因子OsGRF4通过增加小穗壳中的细胞生长分裂来调节籽粒大小[32,70-71]。GL4编码MYB类转录因子,通过调控内外颖纵向细胞伸长使籽粒变长[6],而OsGBP1转录因子抑制粒长,OsGBP3则提高粒长[72]。籽粒大小不仅受穗壳的限制还受到胚乳生长及籽粒灌浆控制,转录因子MADS29通过调控母体组织的降解进而影响籽粒的灌浆[73],转录因子OsNF-YB1的表达下调会显著延缓籽粒灌浆,导致小粒[74]。qLGY3编码Gβ关键的下游效应因子MADS结构域转录因子OsMADS1[75],通过外表皮细胞分裂使籽粒变长,而OsMADS1的转录活性受Gγ亚基GS3和DEP1与MADS的角蛋白结构域互作正调控[76]。
3.6 表观遗传调控途径
植物表观遗传主要涉及DNA甲基化、组蛋白修饰(乙酰化、甲基化、磷酸化和泛素化等)、RNA甲基化、基因組印迹和母性效应等调控类型[77]。miRNA-OsmiR397 正调控水稻产量,Zhang等研究发现,OsmiR397通过下调其靶基因OsLAC的表达增加水稻产量,OsLAC的产物是一种漆酶类蛋白,参与了植物对BR的敏感性[78]。Duan等报道,GS2编码的生长调控因子OsGRF4受OsmiR396的调节,当GS2发生2 bp的替换突变后,会影响OsmiR396对其的调控,导致籽粒增大、粒质量增加,且GS2与转录辅激活子OsGIF1/2/3相互作用,OsGIF1过表达时会使粒质量增加[79];他们发现的另一个miRNA-OsmiR408通过调控OsUCL8正调控植株的光合效率及产量性状[80]。SDG725编码1个H3K36甲基转移酶,其表达下调导致种子变小[81]。GW6a编码具有组蛋白乙酰转移酶活性的类GNAT蛋白OsglHAT1,通过促进细胞分裂调控粒长;GW6a表达提高会使组蛋白H4的乙酰化水平提高,增加颖壳细胞数目和灌浆速率达到增产目的[37,65,82]。LARGE1编码的OML4在发育中的穗粒中表达且GFP-OML4融合蛋白定位于细胞核中,OML4通过限制小穗壳内细胞的扩张来调节颗粒大小,负调控水稻的粒级和粒质量;OML4可被GSK2磷酸化调控OML4蛋白稳定性,OML4功能缺失导致大粒和重粒,而OML4过表达导致小粒和轻粒[83]。
4 水稻粒型基因间的互作及在育种中的应用
水稻粒型是多基因控制的数量性状,不同粒型基因之间互相影响,育种的重要目的之一是得到高产优质的水稻新品种,通过以上分析表明,目前水稻粒长、粒宽、粒厚相关QTL已经定位很多,也有70多个水稻粒型相关基因已被克隆,但这些克隆出来的基因在育种上的应用却相对较少。
4.1 水稻粒型基因间互作
水稻粒型相关的不同位点非等位基因之间也存在相互作用,最终影响籽粒发育,Yan等通过对GS3-RNAi、GW2-RNAi系和qSW5的CSSL系的基因表达分析研究了GW2、GS3、GIF1和qSW5/GW5四者间的互作关系,并用180个品种的自然群体分析了GW5/qSW5和GS3的等位基因作用[84]。研究表明,GW5/qSW5和GW2正调控GS3的表达,GW2能抑制GW5/qSW5的表达,GW5/qSW5正向调控GIF1的表达,而GW2和GS3负向调控GIF1的表达,另外GS3可以消除GW5/qSW5的粒长效应,GS3对粒宽的作用受到qSW5的影响[84];HGW编码1个含有泛素相关结构域的上游调控蛋白,可以直接通过GIF1控制水稻籽粒和质量,在HGW突变体中GIF1、GW2、GW5和GS3等基因的表达降低[85]。
GW7是调节粒长、粒宽的水稻籽粒品质数量性状基因,GW8是包含SBP结构域的转录因子,与GW7启动子结合抑制其表达,GW8通过细胞分裂对粒宽进行调控[86]。转录抑制因子OsARF4负调控粒长而TGW3编码的GSK3/SHAGGY-Like家族蛋白激酶OsSK41与OsARF4互作会发生磷酸化使OsARF4的转录抑制功能增强从而负调控水稻的粒型和粒质量[87]。相关研究发现,GL3.3(TGW3)与GS3互作也会使水稻籽粒显著增大[27]。
4.2 水稻粒型基因在育种中的应用
GS3是一个在育种中应用较广的基因,在增加籽粒质量的同时也能改善外观品质,为更方便地聚合目的基因,通常采用遗传背景一致,目标基因选择效率高,育种周期短的SSSL进行选育。如张剑霞将抗白叶枯病基因Xa23和粒长基因GS3基因聚合到珍汕97B和Ⅱ-32B中,使其在改良后对白叶枯病具有明显的抗性且在粒长方面也得到了增长[88];杨梯丰等通过分子聚合育种将携带粒长基因GS3的华粳籼74与携带其他优良基因的SSSL进行基因聚合,聚合后籽粒变得更长[89];Wang等通过聚合GS3等位基因和OsSPL16(GW8)发现,可以在提高籽粒外观品质的同时保证产量[5]。Zeng等对GS3、Ghd7、qSW5、DPE1等基因进行聚合育种研究,成功选育出优良品种两优培九,其在增产和提升品质方面都更有优势[90]。
Wang等研究发现,GL7在不影响产量和食用品质的前提下可以用来改良籽粒外观品质,遗传背景含GL7的NIL相比稻米垩白度和垩白率显著降低而单株产量、直链淀粉含量、胶稠度和蛋白质含量均不受影响[36],因而将GL7和其他与产量和品质相关的有益等位基因聚合更有助于优良水稻品种的选育。
Zhao等发现,新粒型基因GS9的缺失突变体通过改变细胞分裂来增加籽粒长度,GS9与GS3聚合会获得更长更细的粒型,表明GS9不管单独还是与其他粒型基因结合,都是育种的良好材料[38]。Song等发现,含有GW2基因的丰矮占1号其粒宽、粒厚和千粒质量都分别显著提高26.2%、10.5%、49.8%,而外观及食用品质均未改变[16]。
5 结论与展望
水稻粒型调控涉及很复杂的遗传网络,根据目前研究已經鉴定出的通路包括植物激素、泛素-蛋白酶体通路、G蛋白信号通路、MAPK信号通路及转录因子调控等,再加上各个控制粒型基因间的相互作用共同构成了籽粒大小的调控网络,但各个通路之间互作关系有待更深入研究。基因克隆是研究粒型大小调控网络的基础,今后仍需要尽可能多地克隆出粒型相关基因,与传统的图位克隆相比,近年新的技术方法:基因组编辑技术(CRISPR/Cas9)、GWAS等可以更高效便捷地鉴定出更多粒型调控相关基因,同时有助于了解并完善粒型调控网络。
CRISPR/Cas9等基因组编辑工具给水稻分子设计育种及品种改良带来了革命性的巨变,其在相同的遗传背景中快速高效地创制功能缺失型突变体[91]的特点使得水稻分子育种的研究及应用前途光明。最近开发的新型基因编辑工具PE(Primer Editors),实现了从C-T/G-A的简单转换到12种单碱基的转换以及多碱基的精准插入与删除[92-93],这些都将有助于完善调控水稻粒型大小的遗传网络[94]。此外由于基因编辑过程涉及转基因,因此在应用时需要通过所选材料的背景,亲本与阳性株进行回交去除转基因成分;其次基因编辑导致的突变和脱靶现象,会使等位基因在不同遗传背景下突变表型不完全一样,因此可以在特定遗传背景下利用等位基因突变来提高产量。
GWAS虽然在全基因组范围内可以通过自然群体或构建的人工群体快速高效地查找并筛选出与目标性状相关联的SNP、InDel等变异位点[95-98],但在涉及 QTL 的克隆时仍需与图位克隆二者相结合才能确定候选基因。基于二代测序的BSA法(bulk segregant analysis)是通过在群体中挑选目标性状极端的个体,构建DNA混池进行全基因组重测序及差异分析,快速实现对单一性状的初定位[99]。
目前一些已经克隆的基因如GW2、GW5、GS5、GW8等在生产应用时仍然要先通过田间生产试验及在不同遗传背景下试验才能进一步明确其育种利用价值。目前许多被鉴定克隆出的基因不能直接通过分子设计育种用于改良已有品种,对提高水稻产量和稻米品质的作用并不大,因此在鉴定新功能基因的同时可以用老基因讲出新故事,将研究工作落实到实际生产应用中;相对于粒长、粒宽的研究已经定位并克隆了多基因,但是关于水稻粒厚方面的研究报道仍然比较少,粒厚基因的遗传研究仍有待进一步加强[100]。
参考文献:
[1]Wang A H,Garcia D,Zhang H,et al. The VQ motif protein IKU1 regulates endosperm growth and seed size in Arabidopsis[J]. The Plant Journal,2010,63(4):670-679.
[2]Xing Y Z,Zhang Q F. Genetic and molecular bases of rice yield[J]. Annual Review of Plant Biology,2010,61(1):421-442.
[3]吕 勇. 水稻产量相关性状的QTL定位与分析[D]. 泰安:山东农业大学,2017:52-53.
[4]陈 曦. 多群体定位水稻粒形数量性状位点[D]. 南宁:广西大学,2020:6-8.
[5]Wang S K,Wu K,Yuan Q B,et al. Control of grain size,shape and quality by OsSPL16 in rice[J]. Nature Genetics,2012,44(8):950-954.
[6]Wu W G,Liu X Y,Wang M H,et al. A single-nucleotide polymorphism causes smaller grain size and loss of seed shattering during African rice domestication[J]. Nature Plants,2017,3(6):17064.
[7]Zhu Y J,Sun Z C,Niu X J,et al. Dissection of three quantitative trait loci for grain size on the long arm of chromosome 10 in rice (Oryza sativa L.)[J]. PeerJ,2019,7:e6966.
[8]Qi L,Ding Y B,Zheng X M,et al. Fine mapping and identification of a novel locus qGL12.2 control grain length in wild rice (Oryza rufipogon Griff.)[J]. Theoretical and Applied Genetics,2018,131(7):1497-1508.
[9]Li Y B,Fan C C,Xing Y Z,et al. Natural variation in GS5 plays an important role in regulating grain size and yield in rice[J]. Nature Genetics,2011,43(12):1266-1269.
[10]Wang D W,Sun W Q,Yuan Z Y,et al. Identification of a novel QTL and candidate gene associated with grain size using chromosome segment substitution lines in rice[J]. Scientific Reports,2021,11(1):189.
[11]Shomura A,Izawa T,Ebana K,et al. Deletion in a gene associated with grain size increased yields during rice domestication[J]. Nature Genetics,2008,40(8):1023-1028.
[12]Weng J F,Gu S H,Wan X Y,et al. Isolation and initial characterization of GW5,a major QTL associated with rice grain width and weight[J]. Cell Research,2008,18(12):1199-1209.
[13]Fu X,Xu J,Zhou M Y,et al. Enhanced expression of QTL qLL9/DEP1 facilitates the improvement of leaf morphology and grain yield in rice[J]. International Journal of Molecular Sciences,2019,20(4):866.
[14]尉 鑫,曾智鋒,杨维丰,等. 水稻粒形遗传调控研究进展[J]. 安徽农业科学,2019,47(5):21-28.
[15]康艺维,陈玉宇,张迎信. 水稻粒型基因克隆研究进展及育种应用展望[J]. 中国水稻科学,2020,34(6):479-490.
[16]Song X J,Huang W,Shi M,et al. A QTL for rice grain width and weight encodes a previously unknown RING-type E3 ubiquitin ligase[J]. Nature Genetics,2007,39(5):623-630.
[17]Shomura A,Izawa T,Ebana K,et al. Deletion in a gene associated with grain size increased yields during rice domestication[J]. Nature Genetics,2008,40(8):1023-1028.
[18]Liu J F,Chen J,Zheng X M,et al. GW5 acts in the brassinosteroid signalling pathway to regulate grain width and weight in rice[J]. Nature Plants,2017,3(5):17043.
[19]Ruan B P,Shang L G,Zhang B,et al. Natural variation in the promoter of TGW2 determines grain width and weight in rice[J]. The New Phytologist,2020,227(2):629-640.
[20]Fan C C,Xing Y Z,Mao H L,et al. GS3,a major QTL for grain length and weight and minor QTL for grain width and thickness in rice,encodes a putative transmembrane protein[J]. Theoretical and Applied Genetics,2006,112(6):1164-1171.
[21]Mao H L,Sun S Y,Yao J L,et al. Linking differential domain functions of the GS3 protein to natural variation of grain size in rice[J]. Proceedings of the National Academy of Sciences of the United States of America,2010,107(45):19579-19584.
[22]Zhang X J,Wang J F,Huang J,et al. Rare allele of OsPPKL1 associated with grain length causes extra-large grain and a significant yield increase in rice[J]. Proceedings of the National Academy of Sciences of the United States of America,2012,109(52):21534-21539.
[23]Qi P,Lin Y S,Song X J,et al. The novel quantitative trait locus GL3.1 controls rice grain size and yield by regulating Cyclin-T1;3[J]. Cell Research,2012,22(12):1666-1680.
[38]Zhao D S,Li Q F,Zhang C Q,et al. GS9 acts as a transcriptional activator to regulate rice grain shape and appearance quality[J]. Nature Communications,2018,9(1):1240.
[39]趙冬生. 水稻幼苗白化致死基因AL1和粒形基因GS9的克隆与功能分析[D]. 扬州:扬州大学,2016:19-28.
[40]刘晓青. 玉米百粒重主效QTL qHKW3的精细定位[D]. 武汉:华中农业大学,2018:24-28.
[41]Li N,Li Y H. Signaling pathways of seed size control in plants[J]. Current Opinion in Plant Biology,2016,33:23-32.
[42]Li N,Xu R,Duan P G,et al. Control of grain size in rice[J]. Plant Reproduction,2018,31(3):237-251.
[43]曹 维,赵 静,禹艳坤,等. 调控植物种子大小的分子机制综述[J]. 江苏农业科学,2020,48(6):1-7.
[44]Cao J S,Li G J,Qu D J,et al. Into the seed:auxin controls Seed development and grain yield[J]. International Journal of Molecular Sciences,2020,21(5):1662.
[45]Na G N,Mu X P,Grabowski P,et al. Enhancing microRNA167A expression in seed decreases the α-linolenic acid content and increases seed size in Camelina sativa[J]. The Plant Journal,2019,98(2):346-358.
[46]Liu L C,Tong H N,Xiao Y H,et al. Activation of Big Grain1 significantly improves grain size by regulating auxin transport in rice[J]. Proceedings of the National Academy of Sciences of the United States of America,2015,112(35):11102-11107.
[47]Liu J,Hua W,Hu Z Y,et al. Natural variation in ARF18 gene simultaneously affects seed weight and silique length in polyploid rapeseed[J]. Proceedings of the National Academy of Sciences of the United States of America,2015,112(37):E5123-E5132.
[48]Hirano K,Yoshida H,Aya K,et al. SMALL ORGAN SIZE 1 and SMALL ORGAN SIZE 2/DWARF AND LOW-TILLERING form a complex to integrate auxin and brassinosteroid signaling in rice[J]. Molecular Plant,2017,10(4):590-604.
[49]Aya K,Hobo T,Sato-Izawa K,et al. A novel AP2-type transcription factor,SMALL ORGAN SIZE1,controls organ size downstream of an auxin signaling pathway[J]. Plant & Cell Physiology,2014,55(5):897-912.
[50]Matsuta S,Nishiyama A,Chaya G,et al. Characterization of heterotrimeric G protein γ4 subunit in rice[J]. International Journal of Molecular Sciences,2018,19(11):3596.
[51]Wang L,Xu Y Y,Ma Q B,et al. Heterotrimeric G protein alpha subunit is involved in rice brassinosteroid response[J]. Cell Research,2006,16(12):916-922.
[52]Utsunomiya Y,Samejima C,Takayanagi Y,et al. Suppression of the rice heterotrimeric G protein β-subunit gene,RGB1,causes dwarfism and browning of internodes and lamina joint regions[J]. The Plant Journal,2011,67(5):907-916.
[53]Sun S Y,Wang L,Mao H L,et al. A G-protein pathway determines grain size in rice[J]. Nature Communications,2018,9(1):851.
[54]Zhou Y,Zhu J Y,Li Z Y,et al. Deletion in a quantitative trait gene qPE9-1 associated with panicle erectness improves plant architecture during rice domestication[J]. Genetics,2009,183(1):315-324.
[55]Feng Y,Lu Q,Zhai R R,et al. Genome wide association mapping for grain shape traits in indica rice[J]. Planta,2016,244(4):819-830.
[56]Zhao K,Tung C W,Eizenga G C,et al. Genome-wide association mapping reveals a rich genetic architecture of complex traits in Oryza sativa[J]. Nature Communications,2011,2(1):467.
[57]Mao H L,Sun S Y,Yao J L,et al. Linking differential domain functions of the GS3 protein to natural variation of grain size in rice[J]. Proceedings of the National Academy of Sciences of the United States of America,2010,107(45):19579-19584.
[58]Xu J,Zhang S Q. Mitogen-activated protein kinase cascades in signaling plant growth and development[J]. Trends in Plant Science,2015,20(1):56-64.
[59]Xu R,Duan P G,Yu H Y,et al. Control of grain size and weight by the OsMKKK10-OsMKK4-OsMAPK6 signaling pathway in rice[J]. Molecular Plant,2018,11(6):860-873.
[60]Guo T,Chen K,Dong N Q,et al. GRAIN SIZE AND NUMBER1 negatively regulates the OsMKKK10-OsMKK4-OsMPK6 cascade to coordinate the trade-off between grain number per panicle and grain size in rice[J]. The Plant Cell,2018,30(4):871-888.
[61]Liu S Y,Hua L,Dong S J,et al. OsMAPK6,a mitogen-activated protein kinase,influences rice grain size and biomass production[J]. The Plant Journal,2015,84(4):672-681.
[62]Duan P G,Rao Y C,Zeng D L,et al. SMALL GRAIN 1,which encodes a mitogen-activated protein kinase kinase 4,influences grain size in rice[J]. The Plant Journal,2014,77(4):547-557.
[63]杨维丰,詹鹏麟,林少俊,等. 水稻粒形的遗传研究进展[J]. 华南农业大学学报,2019,40(5):203-210.
[64]谢旺清. MERIT40-Tankyrase1相互作用对纺锤体组装影响的研究[D]. 深圳:深圳大学,2017:29-31.
[65]刘 喜,牟昌铃,周春雷,等. 水稻粒型基因克隆和调控机制研究进展[J]. 中国水稻科学,2018,32(1):1-11.
[66]Li N,Xu R,Li Y H. Molecular networks of seed size control in plants[J]. Annual Review of Plant Biology,2019,70(1):435-463.
[67]南 玲. UCH-L5對TGFβ-1信号转导通路中Smad2/Smad3蛋白去泛素化和稳定性的研究及对肺纤维化的作用[D]. 长春:吉林大学,2017:39-41.
[68]Huang K,Wang D K,Duan P G,et al. WIDE AND THICK GRAIN 1,which encodes an otubain-like protease with deubiquitination activity,influences grain size and shape in rice[J]. The Plant Journal,2017,91(5):849-860.
[69]Shi C L,Ren Y L,Liu L L,et al. Ubiquitin specific protease 15 has an important role in regulating grain width and size in rice[J]. Plant Physiology,2019,180(1):381-391.
[70]Sun P Y,Zhang W H,Wang Y H,et al. OsGRF4 controls grain shape,panicle length and seed shattering in rice[J]. Journal of Integrative Plant Biology,2016,58(10):836-847.
[71]Li S,Tian Y H,Wu K,et al. Modulating plant growth-metabolism coordination for sustainable agriculture[J]. Nature,2018,560(7720):595-600.
[72]Ji X,Du Y,Li F,et al. The basic helix-loop-helix transcription factor,OsPIL15,regulates grain size via directly targeting a purine permease gene OsPUP7 in rice[J]. Plant Biotechnology Journal,2019,17(8):1527-1537.
[73]Yin L L,Xue H W. The MADS29 transcription factor regulates the degradation of the nucellus and the nucellar projection during rice seed development[J]. The Plant Cell,2012,24(3):1049-1065.
[74]Xu J J,Zhang X F,Xue H W. Rice aleurone layer specific OsNF-YB1 regulates grain filling and endosperm development by interacting with an ERF transcription factor[J]. Journal of Experimental Botany,2016,67(22):6399-6411.
[75]葛慧雯. OsRhoGAP2基因过表达改变水稻粒形的分子机制研究[D]. 新乡:河南师范大学,2018:41-43.
[76]Liu Q,Han R,Wu K,et al. G-protein βγ subunits determine grain size through interaction with MADS-domain transcription factors in rice[J]. Nature Communications,2018,9(1):852.
[77]王静宇,陈晓慧,赖钟雄. 植物表观遗传修饰的分子机制及其生物学功能[J]. 热带作物学报,2020,41(10):2099-2112.
[78]Zhang Y C,Yu Y,Wang C Y,et al. Overexpression of microRNA OsmiR397 improves rice yield by increasing grain size and promoting panicle branching[J]. Nature Biotechnology,2013,31(9):848-852.
[79]Duan P,Ni S,Wang J,et al. Regulation of OsGRF4 by OsmiR396 controls grain size and yield in rice[J]. Nature Plants,2015,2(1):15203.
[80]Zhang J P,Yu Y,Feng Y Z,et al. MiR408 regulates grain yield and photosynthesis via a phytocyanin protein[J]. Plant Physiology,2017,175(3):1175-1185.
[81]Sui P,Shi J,Gao X,et al. H3K36 methylation is involved in promoting rice flowering[J]. Molecular Plant,2013,6(3):975-977.
[82]張昌泉,赵冬生,李钱峰,等. 稻米品质性状基因的克隆与功能研究进展[J]. 中国农业科学,2016,49(22):4265-4283.
[83]Lyu J,Wang D R,Duan P G,et al. Control of grain size and weight by the GSK2-LARGE1/OML4 pathway in rice[J]. The Plant Cell,2020,32(6):1905-1918.
[84]Yan S,Zou G H,Li S J,et al. Seed size is determined by the combinations of the genes controlling different seed characteristics in rice[J]. Theoretical and Applied Genetics,2011,123(7):1173-1181.
[85]Li J,Chu H W,Zhang Y H,et al. The rice HGW gene encodes a ubiquitin-associated (UBA) domain protein that regulates heading date and grain weight[J]. PLoS One,2012,7(3):e34231.
[86]余海平. 水稻颖壳缺陷基因DG1和粒型基因GR5的克隆与功能分析[D]. 沈阳:沈阳农业大学,2018:68-71.
[87]Hu Z,Lu S J,Wang M J,et al. A novel QTL qTGW3 encodes the GSK3/SHAGGY-Like kinase OsGSK5/OsSK41 that interacts with OsARF4 to negatively regulate grain size and weight in rice[J]. Molecular Plant,2018,11(5):736-749.
[88]张剑霞. 利用分子标记辅助选择转移野生稻增产QTL和聚合水稻优良基因[D]. 武汉:华中农业大学,2009:25-27.
[89]杨梯丰,曾瑞珍,朱海涛,等. 水稻粒长基因GS3在聚合育种中的效应[J]. 分子植物育种,2010,8(1):59-66.
[90]Zeng D L,Tian Z X,Rao Y C,et al. Rational design of high-yield and superior-quality rice[J]. Nature Plants,2017,3(4):17031.
[91]姚祝平,程 远,万红建,等. CRISPR/Cas9基因编辑技术在植物基因工程育种中的应用[J]. 分子植物育种,2017,15(7):2647-2655.
[92]Zong Y,Song Q N,Li C,et al. Efficient C-to-T base editing in plants using a fusion of nCas9 and human APOBEC3A[J]. Nature Biotechnology,2018,36(10):950-953.
[93]Anzalone A V,Randolph P B,Davis J R,et al. Search-and-replace genome editing without double-strand breaks or donor DNA[J]. Nature,2019,576(7785):149-157.
[94]Mieulet D,Jolivet S,Rivard M,et al. Turning rice meiosis into mitosis[J]. Cell Research,2016,26(11):1242-1254.
[95]Sun D W,Cen H Y,Weng H Y,et al. Using hyperspectral analysis as a potential high throughput phenotyping tool in GWAS for protein content of rice quality[J]. Plant Methods,2019,15(3):54.
[96]Korte A,Farlow A. The advantages and limitations of trait analysis with GWAS:a review[J]. Plant Methods,2013,9(1):29.
[97]Wang H R,Xu X,Vieira F G,et al. The power of inbreeding:NGS-Based GWAS of rice reveals convergent evolution during rice domestication[J]. Molecular Plant,2016,9(7):975-985.
[98]周玲,熊 威,胡俏強,等. 基于温带和热带玉米群体全基因组选择和杂种优势候选位点的鉴定[J]. 江苏农业科学,2021,49(4):19-26.
[99]Michelmore R W,Paran I,Kesseli R V. Identification of markers linked to disease-resistance genes by bulked segregant analysis:a rapid method to detect markers in specific genomic regions by using segregating populations[J]. Proceedings of the National Academy of Sciences of the United States of America,1991,88(21):9828-9832.
[100]宫李辉,高振宇,马伯军,等. 水稻粒形遗传的研究进展[J]. 植物学报,2011,46(6):597-605.

