医学知识图谱构建关键技术及研究进展
谭玲,鄂海红,匡泽民,宋美娜,刘毓,陈正宇,谢晓璇,李峻迪,范家伟,王晴川,康霄阳
1. 北京邮电大学,北京 100876;2. 首都医科大学附属北京安贞医院,北京 100029
1 引言
人工智能的发展已经进入快车道,作为新一轮科技革命和产业变革的重要驱动力量,人工智能技术正在深入各行各业,悄无声息地改变着人们日常生活的方方面面[1]。知识图谱是由谷歌(Google)公司在2012年提出的一个概念,本质上是语义网的知识库。知识图谱由节点和边组成,节点表示实体,边表示实体与实体之间的关系,这是最直观、最易于理解的知识表示和实现知识推理的框架,奠定了第三代人工智能研究的基础[1]。
目前,医学是知识图谱应用较广的垂直领域之一,也是目前国内外人工智能领域研究的热点。医学知识图谱在临床诊断、治疗、预后等方面均可发挥较大的作用。高效地将知识图谱应用于医学领域将给人类的医疗卫生带来革命性的变化[1]。由于医学领域数据的特殊性,医学知识图谱的构建也面临不少机遇与挑战。
本文对医学知识图谱构建的关键技术及应用进行了全面的梳理,对各类公共数据集、处理医学问题的特异性难点及现有解决办法进行了综述。通过阅读本文,可以了解医学知识图谱的发展现状、未来发展方向以及面临的挑战,便于医学知识图谱研究者参照对比,加快医学知识图谱领域的研究及临床落地应用。
本文主要按照医学知识图谱构建的流程来阐述,主要框架如图1所示。

图1 医学知识图谱构建框架
2 医学本体构建
网络上文本数据的爆炸式增长,以及对本体需求的增加,促进了语义网络的发展,使得基于文本的本体自动构建成为一个非常有前途的研究领域。文本本体学习是一种以机器可读形式(半)自动地从文本中提取和表示知识的过程。本体被认为是在语义网络上以更有意义的方式表示知识的主要基石之一。
2.1 本体构建定义及任务
万维网联盟(World Wide Web Consortium,W3C)将本体论定义为用于描述和表示知识领域的术语。本体是一个数据模型,它表示一组概念以及一个域中这些概念之间的关系。
本体构建可以定义为从头创建本体或重用现有本体以丰富或填充现有本体的迭代过程。构建本体的过程包括以下6个任务:
● 指定一个域以创建定义良好的术语和概念;
● 识别域中的关键术语、概念及其关系;
● 建立或推断描述域结构属性的规则和公理;
● 使用支持本体的表示语言(如资源描述框架(resource description framework,RDF)、资源描述框架模式(resource description framework schema,RDFS)或网络本体语言(Web ontology language,OWL))对构建的本体进行编码(表示);
● 将构建的本体与现有本体结合(如果现有本体可用);
● 通过使用通用和特定的评估度量来评估构建的本体[2]。
2.2 医学本体构建难点及现有技术
随着对许多医学本体构建研究的深入,目前医学本体库的构建主要存在以下难点。
首先应该尽可能减少在本体构建过程中的人为干预。目前实现本体构建过程的完全自动化是不现实的,怎样减少人为干预是目前医学本体构建的一个难点和热点。2018年,Mazen A等人[3]提出了一种新的本体自动生成框架,即链接开放数据项目授权的生物医学本体自动生成(linked open data approach for automatic biomedical ontology generation,LOD-ABOG)方法。与现有框架相比,参考文献[3]的评估结果显示,大多数本体生成任务的结果有所改善。该参考文献提出的LOD-ABOG框架表明,现有的LOD源和技术是一个很有前途的解决方案,可以在更大程度上实现生物医学本体生成和关系提取过程的自动化。另外,与现有的框架在本体开发过程中需要领域专家的参与不同,该参考文献提出的方法只要求领域专家在本体构建周期结束时参与到本体的改进中。
2019年,Lytvyn V等人[4]提出了从自然文本中提取知识的方法和算法(包括一个基于本体引入的概念、关系、谓词和规则的多层次过程),建立了一种基于本体的本体开发方法,该方法利用现有本体对文本文档进行分析,构建了命名和本体术语体系。这使得本体开发过程自动化成为可能。
再者,由于医学信息的特殊性,对医学信息的匿名化处理在本体构建过程中也是一个难点。2017年,Polsley S等人[5]提出一种可识别被映射到本体论术语的受保护健康信息(protected health information,PHI)的方法,临床专家使用数百份医学文献对该方法进行了评价,F1分数达98.8%,在后续处理中保留语义信息具有一定的前景。但该方法仍有较大的局限性,需要不断地进行优化。
2.3 医学本体常用数据集
医学本体较常用的数据集主要有以下几种,见表1。

表1 医学本体常用的数据集
2.4 挑战及未来研究方向
首先,由于医学数据的多样性,在设计医学本体构建系统时,无论是来自小的静态文本集合的数据,还是万维网上的海量异构数据,都需要进行数据转换。目前,针对此问题的文献较少,有待后续研究的推进。
其次,医学的临床数据会不断变化,如何根据患者的当前情况创建动态的最佳保护服务,为患者提供个性化的实时医疗护理也是医学实体构建过程中的一大问题[6]。
3 医学命名实体识别
3.1 命名实体识别定义
命名实体识别(named entity recognition,NER)又称专名识别,指识别文本中具有特定意义的实体(主要包括人名、地名、机构名、专有名词等)。通常包括两部分:一是识别实体边界;二是确定实体类别(人名、地名、机构名或其他)。英语中的命名实体具有比较明显的形式标志(即实体中的每个词的第一个字母要大写),因此识别实体边界相对容易,任务的重点是确定实体的类别。和英语相比,汉语命名实体识别任务更加复杂,实体边界的识别更加困难。
3.2 医学命名实体识别难点及现有技术
与传统的命名实体识别相比,医学名词实体一般比较长,长实体名词常常包含多个名词实体,造成医学实体边界识别的难度较大。此外,医学名词存在大量的同义词替换、缩写以及一词多义现象,加大了确定实体类别的难度。
针对医学实体中大量同义词替换以及大量缩写的问题,2020年Kato T等人[7]提出了一种共享和学习标签组件嵌入的方法,通过对英语和日语细粒度NER进行实验,证明了该方法比标准序列标记模型性能更好,特别是在低频标签情况下。
为了解决医学名词实体较长、识别边界困难的问题,2020年,Tan C Q等人[8]提出了边界感知的神经网络模型来预测实体的类别信息。该模型可以先定位出实体的位置, 然后在对应的位置区间内进行实体类型的预测。在公开的嵌套NER数据集上,该模型取得了超越以往方法的效果,并在预测上取得了更快的速度。
另外,大多数NER系统只处理平面实体,忽略了内部嵌套实体,导致无法捕获底层文本中的细粒度语义信息。为了解决这个问题,2018年Ju M Z等人[9]提出了一种新的神经模型,通过动态叠加平面NER层来识别嵌套的实体。模型将长短时记忆(long short term memory,LSTM)层的输出合并到当前的平面NER层中,为检测到的实体构建新的表示,并将它们提供给下一个平面NER层。模型动态地堆加平面NER层,直到没有提取任何外部实体。该模型针对特定数据集(具有多种类别和嵌套的实体)具有较好的实验效果。
对于医学实体中常见的一词多义现象,2019年Pham T H等人[10]在细粒度NER任务中进行了多任务学习和语境化单词表征的有效性研究,并研究了多任务序列标记的不同参数共享方案、神经语言模型学习和不同单词表示设置下的学习。最终得到的最佳模型不需要任何额外的人工操作来创建数据和设计特征,F1分数达到83.35%。Luo Y等人[11]提出了一个增加了上下文表示层次的模型:句子级表示和文档级表示。在句子级,考虑到单个句子中单词的不同贡献,通过标签嵌入注意机制来增强从独立的双向长短时记忆(bidirectional long short term memory,BiLSTM)学习到的句子表征。在文档级,采用键值存储网络记录对上下文信息相似度敏感的单个单词的文档感知信息。在基准测试的实验结果数据集(CoNLL-2003和Ontonnotes 5.0英语数据集,CoNLL-2002西班牙语数据集)上获得了最先进的结果。
3.3 医学命名实体识别常用数据集
医学命名实体识别较常用的数据集主要有以下几种,见表2。
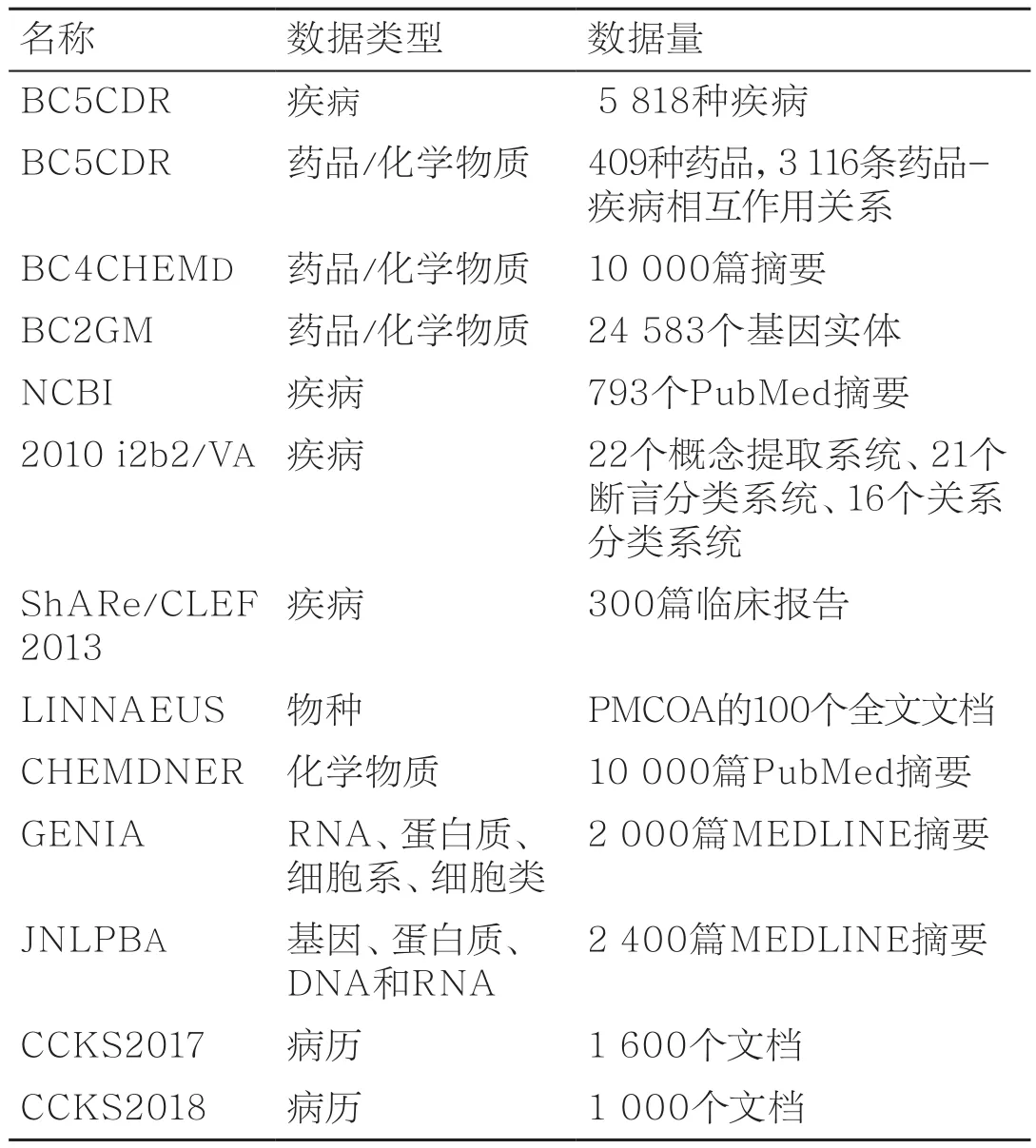
表2 医学命名实体识别常用的数据集
3.4 挑战及未来研究方向
(1)多类别实体在不同语境、不同词性、不同类别下的应用
语言的博大精深、丰富多彩正是语言的魅力所在,但对于机器来说,丰富多彩的语言使语言的使用规则变得更加复杂,很难归纳和总结。将机器语言变得更加智能,理解多类别的实体在不同语境、不同词性及不同类别下的应用是一个重要的研究方向。
(2)嵌套实体的研究
在医学领域中,实体嵌套的现象非常常见,绝大部分医学长实体中会存在实体嵌套,如何更有效地识别实体嵌套是医学命名识别实体领域必须面对且具有重要意义的问题。
(3)实体识别与实体关系抽取的结合
输入一个句子,通过实体识别和关系抽取联合模型,直接得到有关系的实体三元组。这可以克服实体识别模块的错误引起的错误传播,重视两个子任务之间存在的关系,使信息抽取任务完成得更加准确高效,但同时也可能会有更复杂的结构,因此如何用更简单的结构实现实体识别和实体关系抽取的结合将是之后的研究重点。
4 医学实体关系抽取
4.1 实体关系抽取定义
实体关系抽取是指从一个句子中抽取出关系三元组,主要目的是从文本中识别实体并抽取实体之间的语义关系。实体关系抽取解决了原始文本中目标实体之间的关系分类问题,它也是构建复杂知识库系统的重要步骤,如文本摘要、自动问答、机器翻译、搜索引擎、知识图谱等。随着近年来信息抽取的兴起,实体关系抽取进一步得到广泛的关注和深入的研究。
4.2 医学实体关系抽取难点及现有技术
与一般的实体关系抽取相比,生物医学领域语料库的建设很复杂,且需要大量的人力、物力,对参与人员的专业背景要求高,因此使用仅有的医学知识来自动构建大规模的语料库对于医学实体关系的抽取十分重要。此外,医学实体之间普遍存在重叠关系,这给关系抽取的准确性带来较大的干扰。现有的医学关系抽取方法大多需要复杂的特征工程,越来越多的学者采用深度学习方法进行关系的抽取,但大多采用的是流水线的方法,没有充分利用实体信息,且容易导致错误的传递。最后,医学关系的跨度较大,句子级的抽取不能满足要求。
为了自动构建大规模的语料库,2019年Li Y等人[12]提出了一种全新的轻量级神经网络框架来解决远程监督关系抽取问题,以弥补以往选择的不足,使用《纽约时报》(New York Times,NYT)数据集进行实验,结果表明该方法在AUC和Top-n精度指标方面都达到了较先进的性能。2020年He Z Q等人[13]设计了一个新的状态表示形式,它考虑了句子嵌入、关系嵌入以及所选的正向实例的嵌入,该方法解决了远程监督方法中的错误标签问题,同时提升了词袋水平的关系提取效果。Chen D Y等人[14]提出了通过多代理强化学习模型来重新标记噪声训练数据,并共同提取实体和关系的新方法。他们在两个真实的数据集上对该方法进行了评估,结果证明,该方法可以显著提高提取器的性能,并实现有效的学习。
针对医学实体间普遍存在重叠关系这一问题,2019年Zeng D J等人[15]重新研究了基于复制机制的关系抽取模型,提出了使用序列到序列(Seq2Seq)方法共同提取实体和关系的多任务学习复制模型(copy mechanism for multi-task learning,CopyMTL)。该模型利用多任务的学习框架来识别多词实体,通过提高实体识别精度来提升关系抽取的效果,从而达到了较理想的效果。2020年Nayak T等人[16]提出了使用编码器-解码器体系结构共同提取实体和关系的方法。该方法使用一种用于关系元组的表示方案,使解码器能够像机器翻译模型那样一次生成一个单词,并且仍然可以找到句子中存在的所有元组,它们具有不同长度的完整实体名称,并且具有重叠的实体。对NYT数据集进行的实验表明,该方法明显优于所有以前的模型。
为了减少深度学习方法关系抽取中错误的传递,2019年Eberts M等人[17]提出了一种混合模型,包括基于转换器的编码层、LSTM实体检测模块、基于强化学习的关系分类模块。实验结果表明,与基线方法相比,该混合模型在关系和实体提取方面表现更好。2019年Bansal T等人[18]提出了一个新的模型——同时神经实体-关系连接器(simultaneous neural entityrelation linker,SNERL)。首先使用自注意力机制来捕获文本中每个实体提及的上下文表示;然后使用这些上下文表示来预测提及水平的实体分布和提及对水平的关系分布;最后针对每个提及对,将这些预测概率进行组合,并合并到文档级别,以获得预测关系三元组的最终概率。实验结果表明,SNERL模型在CDT和CDR这两个生物医学数据集上的表现达到了最优的效果,并且可以大大改善系统的整体召回率,同时避免了级联错误。
针对医学关系跨度大的问题,2020年Nan G S等人[19]提出潜在结构优化(latent structure refinement,LSR)模型,以端到端的方式构造一个文档级图谱来推理句间关系,通过迭代优化策略,模型能够动态构建潜在结构,以改善整个文档中的信息聚合。该模型在生物医学领域的两个文档级关系抽取数据集上取得了较好的效果。
4.3 医学实体关系抽取常用数据集
医学实体关系抽取较常用的数据集主要有以下几种,见表3。

表3 医学实体关系抽取常用的数据集
4.4 挑战及未来研究方向
(1)加强语料库建设
相对于无监督学习方法,有监督学习方法有更好的准确性和稳定性,而构建良好的语料库是有监督学习方法得以开展的关键前提。
(2)利用联合学习方法更好地提取文本中的关系
现有的联合学习方法大多存在不同的问题,例如不能很好地识别医学文本中的重叠嵌套关系,但是联合学习方法可以充分利用实体与关系之间的交互信息,且普遍证明比流水线方法更有效,因此应该着力提升联合学习方法中识别重叠嵌套关系的能力,使联合学习方法更有效。
(3)实现跨句子或文档级关系抽取
医学文本中的关系往往不在一个句子中,而是跨句子的,因此关系抽取模型不应该仅仅满足于句子级的抽取,应该进行更广范围的关系抽取。
(4)解决远程监督学习的问题,提升远程监督的效果
医学领域语料库较小,远程监督方可以有效地解决这个问题,但是远程监督方法中存在错误标签等问题,会影响模型效果。未来可以着重解决远程监督中的错误标签问题,使用远程监督方法可以省去人工标注数据的工作。
5 实体对齐
5.1 实体对齐定义
实体对齐是判断多源异构数据中的实体是否指向真实世界同一对象的过程。如果多个实体表征同一个对象,则在这些实体之间构建对齐关系,同时对实体包含的信息进行融合和聚集。由于目前将实体对齐应用于医学领域的研究文章较少,因此本节主要介绍实体对齐,而不是医学实体对齐。
5.2 实体对齐难点及现有技术
(1)综合利用知识图谱的多种信息,如关系三元组、属性三元组、摘要等
传统的实体对齐任务直接将实体进行对齐,由于没有考虑到与实体相关的背景信息(如关系三元组、属性三元组、摘要等),实体对齐任务准确率不高,容易出现较多的噪声和错误数据,利用背景信息进行实体对齐是目前研究的一个难点。
2020年,E H H等人[20]尝试将关系和属性三元组结合起来进行实体对齐。采用参数共享联合方法和基于翻译的知识嵌入方法将它们联合嵌入。实验结果表明,该方法对实体对齐任务有明显的改进。Munne R F等人[21]提出了一种基于嵌入的实体对齐方法。针对实体对齐任务,提出了一种汇总与属性嵌入的联合方法。当实体具有较少的属性或关系结构,无法捕获实体的有意义的表示时,实体摘要嵌入会很有用。他们在真实世界的数据集上进行了实验,结果表明,所提方法显著优于当时最先进的实体对齐模型。
(2)多语言知识图谱的实体对齐
随着信息全球化的进一步发展,一种语言的知识图谱已经不能满足信息的沟通与交流,因此多语言知识图谱间的实体对齐方法是计算机研究的必然趋势。
2020年,Chen M H等人[22]提出了一种新的模型JEANS,在一个共享的嵌入方案中联合表示多语种的知识图谱和文本语料库,并试图通过文本附带的监督信号来改善实体对齐效果。在基准数据集上的实验结果表明,JEANS在伴随监督的实体对齐方面有很好的改善,并且显著地优于只提供知识图谱内部信息的最新方法。KANG S Z等人[23]利用本体提出了一种基于TransC的嵌入模型。该模型首先采用TransC和参数共享模型,将知识图谱中的所有实体和关系映射到一个基于对齐实体集的共享低维语义空间,然后迭代地使用重新初始化和软对齐策略来执行实体对齐。实验结果表明,与基准算法相比,该模型能有效地融合本体信息,取得了较好的效果。
(3)数据异构实体对齐
医学知识的表现方式复杂多样,在数据异构的知识图谱之间进行实体对齐也是当前研究的一个难点。
针对不同类型实体的对齐,2020年,Zhu Q等人[24]提出了一个集合图谱网络——多类型实体对齐的集合图神经网络(collective graph neural network for multitype entity alignment,CG Mualign)。与以前的工作不同,CG Mualign联合对齐不同类型的实体,集中利用邻域信息并概括未标记的实体类型。在真实世界知识图谱百万计的实体实验中,该方法的实体对齐效果超过了现有的方法。但是,该方法的运行效率没有超过当前最先进的深度学习方法。
针对邻域结构的非同构性,Sun Z Q等人[25]提出了一种新的知识图谱对齐网络AliNet,旨在以端到端的方式减轻邻域结构的非同构性。该方法采用一种注意机制来突出有用的远距离邻居,并减少噪声,然后使用门控机制控制直接邻域信息和远程邻域信息的聚合。他们进一步建议使用关系损失来重新定义实体表示,并对5个实体对准数据集进行了详细的研究和分析,证明了AliNet的有效性。
针对知识图谱之间的结构异构性,Wu Y T等人[26]采用一种新的图谱采样策略来识别面向实体对齐的信息最丰富的邻居,利用基于交叉图谱注意力的匹配机制,联合比较两个实体的区分子图,以实现稳健的实体对齐。在3个实体比对数据集上进行的大量实验表明,该方法可以在更困难的情况下很好地估计邻域相似度,显著优于12种现有方法。
(4)大规模知识图谱间的实体对齐
在信息化高速发展的今天,数据达到了空前规模,这对技术提出了更多的挑战,大规模知识图谱间的实体对齐也成为研究难点和重点。
2019年,Zhang F J等人[27]将两个有上亿级别节点的网络——AMiner和微软学术进行了对齐,这项研究综合利用了LSTM、灰色神经网络(gray neural network,GNN)、哈希等技术,能够高效处理多种类型的节点以及不同类型的信息,并且使对齐效果达到了可以应用的级别(总体F1分数为96.81%)。
2020年,Flamino J等人[28]提出了一个可解决大规模对齐问题的多步骤通道。在这个通道中,引入了具有鲁棒时间属性的可伸缩特征提取,并使用了聚类算法,以便在图上找到相似节点的分组。这些特征和它们的集群被输入一个通用的对齐阶段,在数百万个可能的匹配中准确地识别伙伴节点。实验结果表明,该管道可以处理大数据集,在内存限制下实现高效的运行。
5.3 实体对齐常用数据集
实体对齐较常用的数据集主要有以下几种,见表4。
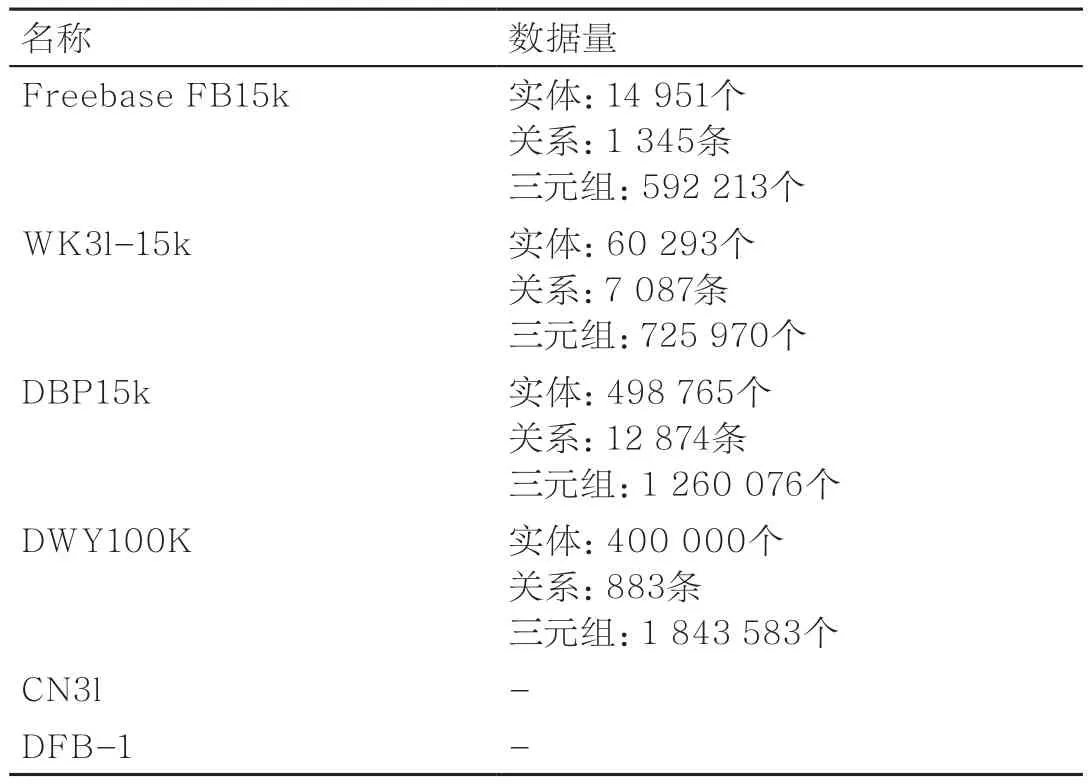
表4 实体对齐常用的数据集
5.4 医学实体对齐挑战及未来研究方向
目前医学实体对齐研究尚处于起步阶段,根据医学数据的特点,医学实体对齐未来的研究方向主要包括以下方面。
● 医学实体存在较多同义词、缩略词,导致实体对齐的精确性受到影响,但是医疗领域要求的精度非常高,使得在医疗领域实现实体对齐这项工作的开展和进行非常艰难,这将是之后医疗领域需要重点解决的问题。
● 数据质量良莠不齐,存在数据壁垒。由于不同医疗知识库的构建目的和方式不同,数据质量不一,并且不同医疗机构的数据一般不能互相开放,如何打破数据壁垒,解决可能存在的相似重复数据、孤立数据、数据时间力度不一致等问题,是未来的一个重点研究方向。
● 医疗数据庞大复杂,标签数据有限,且医学数据精度要求高,需要领域专家手工对数据进行操作,这是一个耗费极大的工程。如何在较少的标签数据中进行训练,实现高效的实体对齐,也是后续研究要关注的问题。
6 医学实体链接
6.1 实体链接定义
由于语言表达的多样性、歧义性以及上下文关联,语言理解面临巨大的挑战。语言理解主要包括语法解析、语义解析和特定的知识表示或其中的某个片段。而在知识图谱中主要涉及的技术即实体理解或实体链接技术,将现实世界中的知识映射到现有知识图谱中的实体,进而用现有知识图谱进行表示,达到理解的目的。在实体链接任务中输入的是实体的指代和上下文以及待链接的知识库,输出的是指代所对应的知识库中的实体。
实体链接(或实体规范化、实体消歧)指将文本中的短语(提及范围)映射到结构化源(如知识库)中的概念。提及范围通常是一个词或短语,描述一个单一的、连贯的概念。
6.2 医学实体链接的难点及现有技术
(1)联合在命名实体识别和实体链接中建模
在知识库构建中,实体识别是实体链接的前提,实体识别可为实体链接提供更多有效的信息。实体链接与实体识别联合学习可减少工作量。实体识别与实体链接任务联合解决既能提高命名实体识别的性能,也能提高实体链接的性能,是当前研究的重点和难点。
2017年,Lou Y X等人[29]提出了一种基于转换的联合疾病实体识别与规范化模型,将输出构造过程转化为一个渐进的状态转换过程,允许使用非局部特征。实验表明,与其他方法分开执行任务相比,联合框架实现了更高的性能。与其他先进的方法相比,该方法更具优势。
2019年,Zhao S D等人[30]提出了一个新的具有显式反馈策略的深层神经多任务学习框架,用于联合实体识别和实体规范化建模。该方法利用多任务学习对两个任务进行一般表示,在保持任务之间相互支持的同时,成功地将跨体系结构的任务转换为并行的多任务设置。实验结果表明,在两个公开的医学文献数据集上,该方法比当时最先进的方法表现得更好。
2020年,Luo Z H等人[31]开发了pyMeSHSim软件包,这是一个用于生物医学文本挖掘的集成、轻量级和数据丰富的Python包。作为第一个一站式医学主题词(medical subject heading,MeSH)工具包,它集成了生物NER、规范化和比较功能。pyMeSHSim嵌入了一个自制的数据集,其中包含主标题(main heading,MH)、补充概念记录(supplementary concept record,SCR)及其在MeSH中的关系。基于该数据集,pyMeSHSim实现了4种基于信息内容的算法和一种基于图谱的算法,可用于度量两个网格术语之间的语义相似度。结果表明,使用pyMeSHSim识别的网络术语和以前手工识别的网络术语的语义相似度高达0.89~0.99。PyMeSHSim有望在生物信息学、计算生物学和生物医学研究中作为一种强大的工具得到广泛的应用。
(2)医学实体语义模糊
基于研究和医学文献分析发现,相同疾病名可能以多种不同的形式出现,比如同义词替换(如“脑中风”“脑卒中”)、疾病名称前的简短描述修饰语(如“大面积心脏病发作”),这些均会造成医学实体语义的复杂多变。近年来针对这个问题的实体链接研究较多。
2017年,Cho H等人[32]联合解析同义词和缩写词的领域特定词典及基于神经网络算法组合的大量未标注数据,该联合方法的精确度显著提高。
2018年,Gorrell G等人[33]提出了一个新的系统Bio-YODIE。Bio-YODIE有两个主要的组成部分,首先,资源准备步骤将运行时所需的UMLS和其他信息资源处理为高效的形式,尽可能多地提前完成工作,以尽量减少运行时的处理;其次,流程本身对文档进行了注释,这些文档包括UMLS概念唯一标识符以及来自UMLS的其他相关信息。基于文本工程的通用结构(general architecture for text engineering,GATE),YODIE最初是一个通用的域系统,引用了DBpedia。Bio-YODIE是该系统的生物医学版本,它继承了一般领域的研究历史。与MetaMapLite的不同之处在于, 消除歧义是Bio-YODIE中的优先事项。Bio-YODIE已被集成到CogStack中,并在大规模临床应用中得到广泛应用。
2019年,Wright D[34]提出了一个深度连贯模型NormCo,它考虑了实体提及的语义,以及单个文档中提及的主题连贯性。NormCo在两个疾病标准化语料库上的预测质量和效率方面优于当时最先进的基线方法,并且至少在准确性和标记文档的F1分数方面表现同样出色。
2019年,Mondal I等人[35]提出了一种基于候选知识库条目与疾病描述相似度的排序方法,探讨了域内子词级信息处理疾病规范化任务的能力。该方法利用由疾病描述m、阳性候选qp、阴性候选qni组成的三元组(qp,m,qni)进行候选排序,引入了一个稳健的、可移植的候选生成方案,该方案不使用手工编制的规则。在标准基准NCBI疾病数据集上的实验结果表明,该系统在很大程度上优于先前的方法。
2020年,Zhu M等人[36]提出了一种潜在类型实体链接模型LATTE,该模型通过对实体提及和实体的潜在细粒度类型信息进行建模来改进实体链接。与以前直接在实体提及和实体之间执行实体链接的方法不同,LATTE在没有直接监督的情况下联合执行实体对齐和潜在的细粒度类型学习。大量的实验结果表明,该模型比几种先进的技术具有显著的性能改进。
(3)公开医学数据集较小
在医学领域,对数据进行标签标注是一项费时费力的大工程。因此目前所有的实体链接公开数据集都是小规模的,如何在小规模数据集上进行高质量的实体链接是目前研究的一个难点。
2017年,Rajani N F等人[37]提出使用精确聚焦的辅助特征来克服医学领域的这些挑战,这些辅助特征可以从少量数据中形成分类边界。该模型优于多个基线水平,并在多个医学数据集上更新了最优结果。
6.3 医学实体链接常用数据集
医学实体链接较常用的数据集主要有以下几种,见表5。

表5 医学实体链接常用的数据集
6.4 未来展望
(1)别名实体候选生成问题
在医学领域中相同的语义往往可以有多种不同的叫法,医学实体的多词同义现象十分普遍,在判断别名实体时很难将所有对应实体的候选实体全部找出,导致实体链接的准确率下降,因此解决别名实体候选生成是未来的研究重点。
(2)不完整数据集的实体链接
在实体链接中,实体、实体的类别信息、关系信息以及上下文信息对实体对齐非常重要,医学数据经常存在数据不完整的情况,使得实体链接效果不是很好,通过仅有的实体相关信息进行链接是医学领域实体对齐面临的又一大挑战。
(3)基于多种语言的实体对齐
目前实体链接系统主要针对的是英文语料,中文或者其他语言的链接系统非常缺乏。中文以及其他语言与类似英语的语言不同,使得实体链接难度增加。对于中文和其他语言的实体链接系统,也需要重点研究。
7 医学知识图谱存储
7.1 知识图谱存储方式
现有知识图谱数据的存储方式主要分为两种:基于关系模型的存储方式和基于图模型的存储方式。
基于关系模型的知识图谱存储方式包括三元组表、水平表、属性表、垂直划分、六重索引和DB2RDF。
目前,基于图数据库的知识图谱存储方法是学术界研究的主流。图数据库的优点在于其天然能表示知识图谱结构,图中的节点表示知识图谱的对象,图中的边表示知识图谱的对象关系。其最大的优点是可以用来处理复杂的关系问题,提供完善的图查询语言,支持各种图挖掘算法。采用图数据库存储知识图谱,能有效利用图数据库中以关联数据为中心的数据表达、存储和查询。基于图模型的存储方式见表6。

表6 基于图模型的存储方式
知识图谱的存储方式应考虑其后续的使用效率,应根据自己的应用场景、数据情况来具体设计。可参考表7选择最适用的存储方式。

表7 知识图谱存储方式比较
基于医学知识图谱更侧重于实体之间的关系(例如药物-疾病、疾病-表征、药物-药物及药物-表征)的特点,医学知识图谱的存储基本采用图数据库,其中应用最广泛的为Neo4j系统。曹明宇等人[38]开发的基于知识图谱的原发性肝癌知识问答系统、吴嘉敏[39]构建的肺癌知识图谱都将Neo4j作为知识图谱的存储系统。Deng W等人[40]利用Neo4j图形数据库构建医学图谱,包含医院科室、疾病和症状之间的关系,并基于图谱提供医学指导。
张崇宇[41]提出了基于知识图谱的医疗自动问答系统,考虑到知识库问答应用中知识存储与检索的效率问题,采用三元组表示与图数据库存储(Neo4j)以及JSON表示与键值对文档型数据库存储(MongoDB)两种形式的混合数据库存储的方式对构建的临床医疗知识图谱进行表示和存储。同时,通过对医疗实体进行归一化处理,将标准化后的实体作为节点存储到知识图谱中。
7.2 医学知识图谱存储的难点及现有技术(以图数据库为例)
(1)复杂关系的可视化
在医学知识中,实体之间的关系经常是错综复杂的,这使得将复杂关系能够更好地可视化成为研究的一个难点。
当前,新的蛋白质和基因序列的数量呈爆炸式增长,这使得对其生物学特性的有效表征和分析变得越来越复杂。2019年,Hu G M等人[42]提出了一个基于网络的图数据库工具SeQuery,通过整合序列结构和功能信息,直观地可视化蛋白质组/基因组网络。用GPCR2841数据集进行的序列测试表明,SeQuery能正确识别查询到的100个蛋白质序列中的99个。SeQuery非常适用于其他生物网络,可以通过添加更多的生物数据库来扩展SeQuery。
(2)用户友好的查询方式
知识图谱的存储是为了让用户更好地使用和查询知识,让用户的查询更简单便捷一直是知识图谱存储的关键和难点。
结直肠癌(colorectal cancer,CRC)是常见的癌症类型之一,它的发生与基因和细胞表观遗传机制的放松有关。2017年,Balaur I等人[43]提出了图数据库EpiGeNet,用于存储和查询在结直肠癌发生的不同阶段观察到的分子事件(遗传和表观遗传)之间的条件关系。EpiGeNet增强了探索与结直肠癌进展相关的研究方面的查询能力,EpiGeNet框架提供了更好的管理和可视化数据的能力,特别是针对结直肠癌的发生和发展的分子事件。
基因组技术的最新进展使得从结核分枝杆菌分离物中产生大量成本效益高的“组学”数据成为可能,然后可以通过许多异构的公开可用的生物数据库共享这些数据。尽管碎片化管理很有用,但它对研究人员联合查询利用数据的能力产生了负面影响。2020年,Lose T等人[44]提出了抗结核病NeoDB(一个整合的结核分枝杆菌经济学知识库)。基于Neo4j,将标签属性图模型绑定到合适的本体,从而创建抗结核病NeoDB。抗结核病NeoDB使研究人员能够通过链接著名的生物数据库和发表文献中的结核分枝杆菌变体数据来执行复杂的联合查询。
(3)认证和加密形式的安全保障
隐私是医院在发布涉及个人敏感信息的数据时应保留的一个重要因素。研究寻求在不侵犯个人信息保密性的情况下向公众发布数据的解决方案。对数据进行处理,可以在维护基本信息的同时安全地发布数据。2020年,Saranya K等人[45]提出了一种基于事务图的自适应概率安全处理方法,用于医疗环境中的安全处理。该方法首先为每个用户交互生成交互图,并在此基础上估计每个交互项的收敛性和偏差测度。基于这些值,该方法计算了一个概率矩阵,并在这个矩阵的基础上生成本体。实验结果表明,所提方法可以产生有效的安全处理和数据发布结果。
7.3 挑战及未来研究方向
● 医疗数据类型种类繁多,现有图数据库系统支持过多数据组织的形式,但不清楚在一些情景中哪个是最好的。如何根据数据的不同选择合适的系统和图模型是未来一个很重要的问题。
● 医疗数据大多独立分布在不同的医疗机构,数据的分布式存储对医疗数据的存储与分析至关重要。目前还没有为图数据库开发拓扑感知或路径感知的数据分布方案,特别是在最近提出的数据中心、高性能计算网络拓扑和路径体系结构的背景下。因此,未来数据的分布式处理将是一个亟待解决的问题。
● 很少有研究使用不同类型的硬件结构、加速器和硬件相关设计(如FPGA、与网络接口卡相关的设计、硬件交互等),但这对于大规模医疗数据的存储也是不可缺少的重要一环。
8 医学知识图谱应用
8.1 基于医学知识图谱的问答
医学知识图谱与问答系统的融合是目前极具挑战性的研究方向,同时也是典型的应用场景。基于知识图谱的医疗问答系统可以快速响应医患用户提出的问题,并给出准确、有效的解答。下面将从问答系统的实现方法、实际应用、关键挑战3个方面进行阐述分析。
(1)实现方法
本文参考了近3年的研究进展,总结出医疗领域基于知识图谱的问答系统主要有两种实现方法:检索式和生成式。其中,检索式主要面向系统构建的知识图谱,生成式主要面向系统收集的问答库数据,表8列出了可用于构建基于知识图谱的医疗问答系统的数据来源。

表8 基于知识图谱的医疗问答系统使用的数据来源
检索式方法就是将用户的问句转化为知识库的查询语句,再将查询的结果转化成自然语言返回给用户,其一般流程由语义提取、问题匹配以及答案查询3个部分组成,如图2所示。

图2 检索式方法的一般流程
语义提取指从用户提出的问句中提取出涉及的医学实体、关系等语义信息,主要包括实体识别和关系抽取两部分,可以采用词典匹配、传统机器学习、神经网络甚至平台工具(如哈尔滨工业大学语言云平台)等方法。参考文献[46]基于自定义词典的Jieba分词匹配获得问句中的实体。
参考文献[47]中的DIK-QA系统使用BiLSTM-CRF神经网络模型抽取问句中的医疗实体,并在该模型中引入注意力机制,以提高实体识别的准确度。参考文献[48]借助哈尔滨工业大学语言云平台的LTPParser接口进行句法分析,将结果与词库内的实体进行比对,从而获取比对成功的实体和关系。
问题匹配旨在识别问句的意图,将问题进行分类,匹配预先制定的问题模板,一般采用匹配算法、TextCNN分类算法、SVM分类器等方法。Huang M X等人[47]采用AC多模式匹配算法将问句匹配到不同的问题类型上。
参考文献[38]结合术语频率-逆文档频率(term frequency–inverse document frequency,TFIDF)算法和word2vec词向量生成句子向量,匹配最相似的问题模板,根据模板的语义及问题中的实体到知识图谱中检索答案。参考文献[49-50]均采用TextCNN分类算法实现问句类型的分类。谢刚等人[51]利用支持向量机模型对问题进行主题分类和意图识别。
答案查询即根据问题模板将问题转化成查询语句,然后在知识图谱中查询问题的答案,主要通过查询语句直接检索答案或者通过推理规则得出答案。曹明宇等人[38]使用Cypher语言在Neo4j图形数据库中查询答案。参考文献[48]根据问题模板生成完整的SPARQL语言,并在甲状腺知识图谱内进行查询。Bo L等人[52]使用Elasticsearch查询语言,配合简单的辅助推理算法,给用户匹配相关症状,搜索可能的疾病,并推荐适当的诊断方法。
而生成式方法则利用相关模型,根据输入的问题生成答案或者直接检索问答库,其既需要医疗领域问答对语料数据,也需要知识图谱的实体及关系数据,主要采用神经网络进行模型训练。参考文献[53]使用基于LSTM的Seq2Seq模型构建答案生成模型。参考文献[54]将记忆神经网络作为智能问答的算法模型,将知识库的知识存储在模型中,可在网络中直接调用。
(2)实际应用
虽然我国医疗问答系统起步较晚,但国内已有不少科技公司在市面上推出自主研发的医疗问答系统。如诺华制药携手腾讯合作推出的“护心小爱(AI)”,该平台以微信小程序为载体,通过对话机器人为心衰患者提供针对常规医疗问题及日常生活问题的答疑解惑,以及科学的健康资讯。再如北京慧医明智科技有限公司旗下的“慧医大白”,其使用知识图谱、语义理解和对话管理等技术手段,通过与用户进行多轮问答,了解用户的具体病症,最终提供健康评估和健康行为建议。
而在问答系统起步较早的国外市场,最出名的面向医学领域的智能问答系统是IBM的“沃森医生(Dr. Watson)”,其学习了海量的医疗数据,包括领域内的顶尖文献、诊断报告、电子病历甚至医学影像等医疗信息,利用自身庞大的知识库为患者提出的医学问题提供最佳的答案。
(3)关键挑战
目前,国内医疗问答系统的研究发展仍然存在许多的挑战,下面列举了3个主要的关键挑战。
一是针对非医学专业人员的信息需求问题,由于他们的医学专业知识不强,无法准确描述具体问题,在获取答案时会存在一定程度的困难。
二是中文领域问答系统研究不足,主要体现在3个方面:①缺乏高质量医学领域的语料资源;②国内医学名词术语标准化还存在整体规划缺乏、权威术语标准数量不足以及更新不及时等问题;③构建中文领域的医学智能问答系统的工具和方法不成熟。
三是医疗问答准确性问题,提高问答系统的准确性仍然是研究的热门方向。
8.2 医疗用药推荐系统
(1)简介
医学上的用药推荐与一般的推荐算法不同,一般的推荐算法是根据用户的历史记录,利用数学算法推测出用户可能的需求,已被广泛应用于电商等互联网场景。而用药推荐则是基于循证医学的原则,结合患者的具体患病情况以及医学专业知识,推荐适合的用药方案。一般的推荐算法的推荐结果对准确率的容忍度较高,即使部分推荐结果与用户需求不符,也能够接受。但用药推荐在实际应用中要求达到百分之百的准确率,即药品一定能够起到作用,且不能产生不良反应或药品间的相互作用。
知识图谱能够更加清晰准确地表达疾病与药品之间的适应关系以及药品间的相互作用,基于知识图谱的用药推荐与其他人工智能方法相比,能够取得更好的效果。目前基于知识图谱的用药推荐研究进展与其他基线水平相比有所提升,但还无法达到实际应用的要求。
(2)方法
目前医疗用药推荐系统使用的方法主要有以下两种。
第一种是图卷积网络的方法,即在图上使用卷积神经网络。2018年Shang J Y等人[55]通过一个存储模块将药物相互作用(drug-drug interaction,DDI)的知识图谱集成为一个图形卷积网络,并将纵向患者向量建模作为查询,该方法在所有有效性度量方面都优于所有基线方法,并且在现有电子健康记录(electronic health record,EHR)数据中实现了3.60%的DDI率降低(即推荐药品之间有相互作用的概率降低3.6%)。2019年,Wang S S等人[56]提出了一种针对药物组合预测(medicine combination prediction,MCP)的图卷积强化学习模型。其将MCP任务转换为无序马尔可夫决策过程(Markov decision process,MDP)问题,并设计了一个深度强化学习机制来学习药物之间的相关性和不良相互作用。相比于GAMENet,CompNet在Jaccard和F1分数标准上分别提高了3.74%、6.64%。2020年,Kwak H等人[57]构造了一个药物疾病图谱,使用图神经网络学习节点表示,根据学习到的节点表示来预测药物节点和疾病节点是否具有药物不良反应(adverse drug reaction,ADR)关系。与其他算法相比,该模型的接受者工作特征曲线下的面积(area under curve of receiver operating characteristic,AUROC)和精度-召回率曲线下的面积(area under curve of precision recall curve,AUPRC)性能分别提高到0.795和0.775。
第二种是知识图谱嵌入的方法,包括将实体和关系转化为连续的向量空间,从而简化操作,同时保留知识图谱的原有的结构。2017年Wang M等人[58]构建了患者-疾病-药品图谱,将其嵌入低维空间后,进行用药推荐。首先构建疾病和药品图谱,通过EHR分别连接疾病和药品图谱,形成两个二分图,通过患者数据将两个二分图连接起来,并构建了一个高质量的异构图,该方法的预测准确度(即Jaccard系数)、药物相互作用发生率、冷启动(即没有患者数据时的使用)、临床专家评分均高于基线水平。2019年Wang X Y等人[59]构建了疾病-药品图谱,将其嵌入低维空间后,进行用药推荐,并提出一种基于知识图谱嵌入增强主题模型(knowledge graph enhanced topic model,KGETM)的中药推荐模型。在中药基准数据集上的实验结果表明,该方法优于当时最新的方法,中药知识图嵌入在中药推荐中有很好的应用前景。
(3)研究方向
医学知识图谱在用药推荐系统应用领域的未来研究方向主要有以下几方面。
① 构建完整的医学知识图谱。人类对疾病与药品的认识是动态变化的,结合疾病、症状、药品、药品间的相互作用及患者的临床数据、患病的时间序列信息等,构建一个完整的医学动态知识图谱,确保知识的完整性、准确性和时效性。
② 知识图谱嵌入学习是将实体和关系映射到低维连续向量空间的表示方法,在保留知识图谱结构信息的同时,还能够改善数据稀疏问题,提高计算效率,因此在进行后续用药推荐任务之前,先对知识图谱进行表示学习是很有必要的。
③ 考虑到构建动态医学知识图谱的必要性,而目前大多数知识嵌入表示研究建立在静态的知识图谱上,如何对动态知识图谱进行有效的知识表示是一个待解决的问题。将图时空网络与动态知识图谱相结合的知识嵌入表示用于用药推荐是一个颇具价值的研究方向。
9 医学知识图谱未来展望
构建医疗领域的知识图谱,可以从海量数据中提炼出医疗知识,并合理高效地对其进行管理、共享及应用,这对当今的医疗行业具有重要意义,也是很多企业和研究机构的研究热点。本文对医学知识图谱构建过程中的研究热点、现有技术、挑战及未来发展方向进行了综述,具体见表9。医学知识图谱将知识图谱与医学知识结合,定会推进医学数据的自动化与智能化处理,为医疗行业带来新的发展契机。医学知识图谱未来总的发展方向应该体现以下几个方面。

表9 医学知识图谱构建关键技术及研究进展汇总
(1)多语言医学知识图谱
国内外医学知识的相互融合促进更有利于医学领域的发展,而实现不同国界医学知识的相互沟通和交流,多语言医学知识图谱技术是关键,这会成为未来医学知识图谱发展的一个重要趋势。
(2)大规模多模态多源医学知识库
受到多方面因素的影响,现有的医学知识图谱规模大多有局限,表现方式也较为单一,大多以文本和图数据的形式呈现,但声音、影像、图片等也蕴含大量的医学信息,在医学临床中也存在大量的医疗影像、X光等多模态信息,医学知识的来源也可以来自书本、文献、网页、视频等。因此未来医学知识图谱研究的一个热点是构建大规模多模态多源的医学知识库[60]。
(3)基于时空特性的知识演化和多粒度知识推理
研究基于深度学习与逻辑推理相互约束的大规模多粒度知识推理模型与方法,研制基于本体、规则与深度学习相结合的大规模知识推理系统,使其能够对包含10亿级RDF三元组的知识库和万级规则进行推理,平均响应时间在秒级,并具有良好的可伸缩性。在此基础上,研究基于时空特性的知识演化模型与预测方法,研制知识演化系统,使其能够实时地对知识库进行更新,平均响应时间为秒级。

