基于SSR标记的海南栽培槟榔遗传多样性分析
齐兰 王世正 黄丽云 周焕起 刘立云

摘 要:檳榔是海南重要的热带特色经济作物,具有很高的药用价值,被列为“四大南药”之首。但其基础研究落后,缺乏对种质资源的系统研究,遗传多样性尚不明确。利用SSR分子标记技术对海南岛58份栽培槟榔资源进行遗传多样性分析,旨在明确槟榔栽培种的亲缘关系,为槟榔杂交育种、功能基因的挖掘提供理论依据。结果表明:从500对SSR引物中筛选出32对多态性好、条带清晰的引物,在58份种质资源中共检测到79个等位基因,每个SSR位点2~5个,平均2.4688个,其中有效等位变异占观测等位变异的58.92%。各引物PIC值变幅为0.0169~0.5969,平均值为0.2254,Shannon信息指数(I)为0.0496~1.2552,平均值为0.4496;Neis遗传多样性指数(Nei)变幅为0.0171~0.6492,平均值为0.2596,说明所选SSR引物在供试样品中检测等位基因遗传多样性偏低。利用UPGMA构建58份栽培槟榔的遗传聚类树状图,第V组材料遗传多样性最丰富,其他分组群体遗传多样性较低,聚类结果说明海南栽培槟榔品种无明显的区域划分,表明海南槟榔不同地区间的种苗交流频繁,总体遗传多样性水平较低,该研究为槟榔遗传育种亲本选择提供依据,也为今后引进种质增补差异资源提供参考。
关键词:槟榔;SSR标记;聚类分析;遗传多样性
中图分类号:S792.91 文献标识码:A
Abstract: Arecanut (Areca catechu L.) is an important tropical economic and social importance plant in Hainan Prov-ince. It has high medicinal value and is listed as the first of the four south China medicinal plants. Areca is mostly planted in underdeveloped areas, and the basic research is weak. The germplasm diversity of areca from Hainan remains unclear due to lack of systemic research of germplasm. We analyzed the genetic diversity among 58 arecanut accessions from Hainan by SSR molecular marker technology to clarify the genetic relationship and provide a theoretical basis for the hybrid breeding and functional gene mining. The results showed that 32 primers with high polymorphism and clear bands were screened out from 500 SSR primers. A total of 79 alleles were detected from the 58 arecanut germplasm resources, and there were 2-5 loci per SSR, with an average of 2.4688 alleles at each marker, and the number of effective allele was accounted for 58.92% of the observed one. The range of PIC was 0.0169-0.5969, with an average of 0.2254. The Shannon index (I) of all primers ranged from 0.0496 to 1.2552, with an average of 0.4496. Neis genetic diversity index (Nei) was ranged from 0.0171 to 0.6492, with an average of 0.2596. The genetic diversity of alleles detected by SSR primers was low. UPGMA Cluster analysis showed that the genetic diversity of group V was the richest. Genetic analysis showed that the 58 arecanut were separated from the effect of regional isolation. The conclusion of frequent exchanges among different regions of arecanut in Hainan and lower genetic diversity in Hainan cultivars would provide theoretical guidance for the selection of parents in the genetic breeding of arecanut, while reference for collecting germplasm to supplement different resources in the future.
Keywords: arecanut; SSR markers; clustering analysis; genetic diversity
DOI: 10.3969/j.issn.1000-2561.2021.05.014
槟榔(Areca catechu L.)是棕榈科槟榔属多年生常绿植物,原产于马来西亚,我国主要栽培于海南、云南以及台湾等地,其中海南产量占全国总产的99%(未计台湾省)[1]。槟榔是海南重要的热带特色经济作物,具有很高的药用价值,被列为“四大南药”(槟榔、砂仁、益智、巴戟)之首。含有多种人体所需的营养元素和有益物质,槟榔原果的主要成分为酚类、多糖、脂肪、粗纤维和生物碱,槟榔种子总生物碱含量达0.3%~ 0.6%,主要为槟榔碱,并含有少量槟榔次碱,是驱虫的有效成分,还具有消积导滞、行气利水、降血压的功能[2-3]。
我国槟榔栽培历史悠久,尤其是在海南,形成了一些各具特色的地方品种,不同类型的槟榔在产量、品质、果实形状等方面存在很大差异[4],但是槟榔种质资源基础研究相对薄弱,对种质资源的多样性缺乏系统的比较和研究,存在种质混杂,同名异物或者同物异名等情况[1]。随着槟榔产业规模的不断扩大,农户在引种时偏向于栽培商品喜好型品种,栽培品种单一,一些特有种质正在逐步消失,遗传基础可能存在逐年变窄的趋势。种质资源的收集、保存和鉴定评价对作物新品种选育具有重要作用,种质资源遗传多样性是槟榔遗传育种的基础和前提,因此,对来源于不同生态区域的槟榔种质资源开展系统遗传多样性研究,对槟榔种质资源评价和创新利用具有重要意义。
DNA分子标记目前广泛应用于作物种质资源遗传多样性研究,其中SSR标记具有共显性、多态性丰富、可重复性好等优点,已被广泛应用于水稻[5]、小麦[6]、甘薯[7]、绿豆[8]、新麦草[9]等大田作物及牧草,以及椰子[10-11]、油棕[12]等棕榈科植物的遗传多样性评价研究。我国槟榔种质资源的遗传多样性研究较少,任军方[13]利用ISSR标记对海南保亭的槟榔栽培居群进行遗传多样性分析,结果显示海南保亭的槟榔多样性水平较低。
本研究调查收集海南不同地区、不同果形的58份栽培槟榔资源,利用SSR分子标记技术进行海南栽培槟榔遗传多样性研究,明确目前海南栽培槟榔的遗传基础,阐明不同材料间的亲缘关系,为我国槟榔种质资源鉴定评价、遗传育种与改良奠定基础。
1 材料与方法
1.1 材料
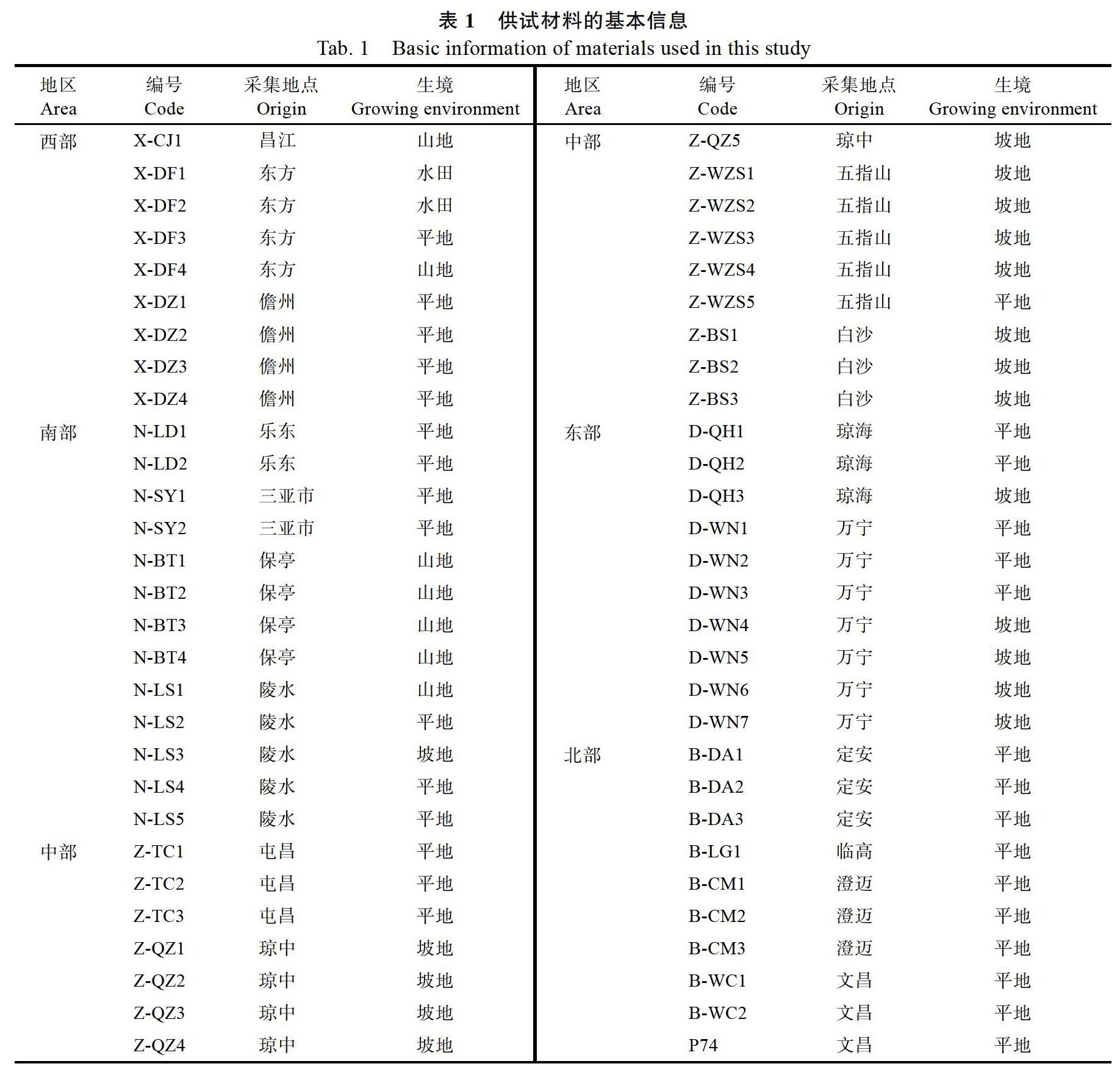
试验材料共58份,来自海南省各市县栽培槟榔园,选取槟榔树株型、果型中有差异的作为试验样品,其中P74为从台湾引进的槟榔种质资源,栽培在中国热带农业科学院椰子研究所槟榔种质资源圃,资源圃内编号为P74,其余样品编号规则:按照各市县在海南省的地理位置分为:西部地区(X)、中部地区(Z)、南部地区(N)、北部地区(B)、东部地区(D),加上样品采集地市县名称拼音首字母缩写,样品基本信息见表1。
1.2 方法
1.2.1 DNA的提取 采集槟榔成龄植株叶片,装入自封袋,置于放冰袋的泡沫盒带回实验室。叶片剪碎后,放入研钵加液氮磨样,采用植物基因组DNA快速提取试剂盒[生工生物工程(上海)股份有限公司]提取叶片基因组DNA,风干后,加入100 μL TE缓冲液溶解。吸取1 μL DNA原液,利用NanoDrop 2000微量紫外分光光度计检测提取DNA质量和浓度,工作液浓度稀释至20 ng/μL,DNA原液和工作液储存于?20 ℃冰箱备用。
1.2.2 引物筛选 利用12份地理来源不同,形态差异较大的材料进行引物筛选,从500多对SSR引物序列中筛选特异性好,条带清晰的引物32对,用于海南栽培槟榔品种基因型检测(表2)。本研究用到的SSR引物是利用‘热研一号槟榔品种的叶片、雌花和果实,通过转录组测序技术开发的SSR引物,引物序列由中国热带农业科学院椰子研究所杨耀东博士提供,引物由生工生物工程(上海)股份有限公司合成。
1.2.3 PCR反應和产物检测 SSR分子标记试验PCR反应体系参考齐兰等[14]的方法,PCR反应程序为:94 ℃预变性5 min;94 ℃变性30 s,58 ℃退火复性30 s,72 ℃延伸30 s,33个循环;72 ℃延伸10 min,最后设置4 ℃保存。用已经筛选出的多态性高且稳定性好的SSR引物进行SSR-PCR扩增。扩增产物用8%聚丙烯酰胺凝胶电泳检测,银染显色,条带清晰后在胶片观察灯上进行拍照。
1.3 数据处理
对照Marker(D 2000)的分子量大小根据条带有无来记录跑胶结果,将SSR引物扩增出的DNA片段按照从大到小的顺序依次排列,相同大小的分子量迁移处,有带记为“1”,无带记为“0”,缺失数据记为“9”,建立0-1形式的二进制数字矩阵,通过DataFormater软件将0-1数据转换成Popgen、Powermarker等软件所需的数据模式[15]。
采用Popgene 1.32软件[16]计算槟榔种质资源的观测等位基因数(Na)、有效等位基因数(Ne)、期望杂合度(He)和观测杂合度(Ho)、Shannons信息指数(I)和Neis遗传多样性指数(Nei)等遗传参数。并计算分组群体间的遗传多样性和遗传距离;利用Powermarker V3.25软件[17]计算每对引物的多态性信息含量(polymorphic information content, PIC);采用Ntsys 2.11软件[18],利用Clustering的SAHN程序中的非加权类平均法(UPGMA)进行聚类分析,通过Tree-plot程序生成遗传聚类图。
2 结果与分析
2.1 SSR标记多态性分析
槟榔DNA提取原液通过微量紫外分光光度计检测其浓度,各检测样品DNA浓度范围为610~1053 ng/μL,OD260/280比值介于1.8~2.0之间,说明提取样品DNA的纯度高,满足SSR-PCR扩增要求。
利用筛选的32对SSR引物,对58份槟榔种质资源进行基因型检测,结果显示,32对引物共检测到79个等位基因,每个SSR位点2~5个,平均2.4688个,其中有效等位变异占观测等位变异的58.92%。观测杂合度(Ho)变幅为0.0000~ 0.7586,平均值为0.1886,说明所检测等位基因位点杂合度稍低;期望杂合度(He)变幅为0.0172~0.6549,平均值为0.2618,PIC值变幅为0.0169~0.5969,平均值为0.2254,中度及以上多态性信息位点(PIC>0.25)有13个。Shannons信息指数(I)的范围为0.0496~1.2552,平均为0.4496;Neis遗传多样性指数(Nei)变幅为0.0171~0.6492,平均值为0.2596,说明SSR引物在供试样品中检测等位基因遗传多样性偏低(表3)。
2.2 不同槟榔种质资源遗传聚类分析
利用SSR标记检测基因型数据进行聚类分析,构建亲缘关系聚类图(图1)。从聚类分析树状图可以看出,遗传相似系数矩阵(GS)变化范围为0.63~0.94。在遗传相似系数为0.70处可以将海南栽培槟榔与台湾种槟榔区分开。在遗传相似系数0.82处(图1中参考线所示),根据离散程度,将供试样本分为5个组,其中第I组划分为2个亚组,I-1和I-2组,将未聚类在一起的与其他样品亲缘关系较远的4份材料划为第Ⅴ组,分别是来自中部琼中、五指山,北部定安以及1份台湾种质。
第I组分为2个亚组,I-1组主要包括中部、北部和东部的材料,其中还有3份来自南部陵水的材料。亚组I-2组的材料来源相对分散,大多数是来自西部和中部地区的样本,该组的材料来源相对复杂,占供试材料的2/3,基本覆盖了海南岛各地的样品。其中有几份来自不同地区的样本
遗传距离很接近,遗传相似系数达到0.95,推测这些材料可能来自相同的亲本,说明海南栽培槟榔在不同地区的种苗交流很频繁。第Ⅱ组和第Ⅲ组包含的材料相对分散,无明显的区域性划分,第IV组的3份材料来自南部的陵水和乐东。第V组的材料1份来自台湾,另外3份与其他海南本地种资源遗传差异较大,这4份材料各自成为1个分支,推测可能是早期从周边东南亚国家引进的种质。
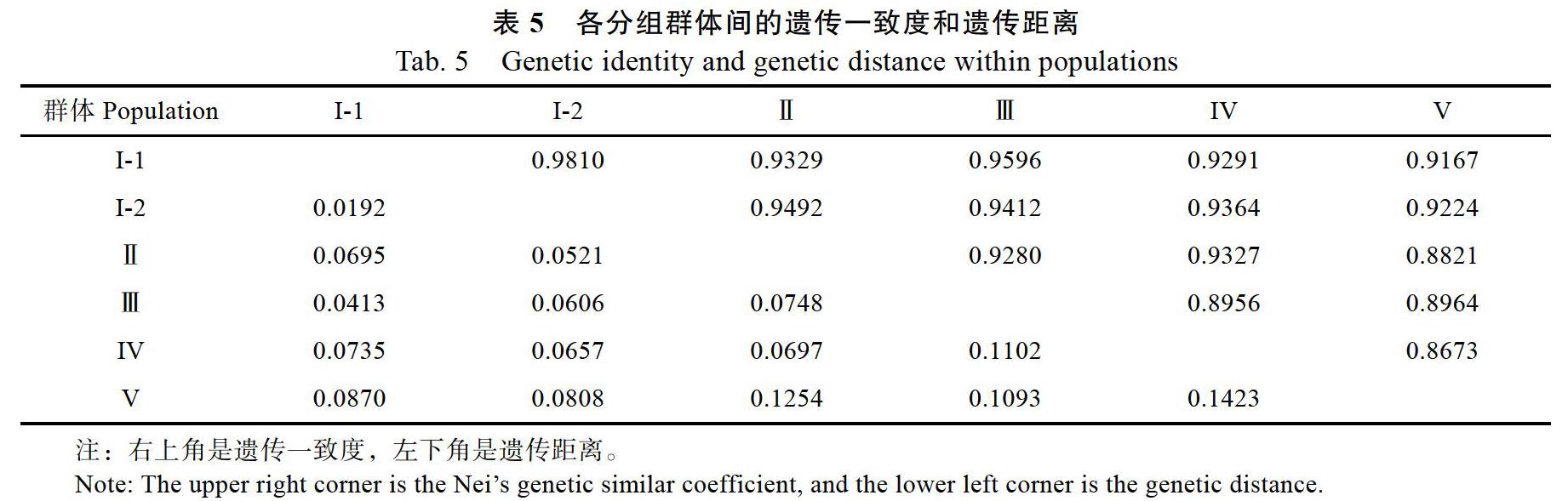
2.3 基于SSR标记的槟榔分组群体遗传多样性与遗传距离
根据SSR标记基础上的遗传聚类结果,将58份海南栽培槟榔种质资源分为5组,由于第I组
成员较多,明显分为2个亚组,因此按照6个分组,即I-1、I-2、Ⅱ、Ⅲ、IV和V组,利用Popgene 1.32软件分析槟榔种质分组群体内的遗传多样性参数(表4)。对分组群体SSR标记多态性分析结果显示,第V组的观测等位基因数(Na)最高(2.2188),最低为第IV组(1.5312);有效等位基因数(Ne)最高为第V组(1.8001),最低为第Ⅱ组(1.2909);Shannons指数(I)最高的是第V组(0.6106),最低为第Ⅱ组(0.2753);Neis遗传多样性指数(Nei)最高为第V组(0.3828),最低为第Ⅱ组(0.1704);观测杂合度(Ho)和期望杂合度(He)分别为0.1328~0.3047和0.1818~0.4375,均为第V组最高。结果表明,第V组的遗传多样性最丰富,其他几组遗传多样性参数值稍低,说明海南本地栽培槟榔群体遗传多样性水平偏低。
利用Popgene 1.32软件计算6个分组群体间的遗传一致度和遗传距离,群体I-1和I-2组间的遗传距离最小(0.0192),遗传一致性系数最大(0.9810);群体IV和群体V的遗传距离最大(0.1423),遗传一致性系数最小(0.8673);总体来讲不同分组群体的遗传距离差异不大(表5),表明海南槟榔不同分组群体遗传差异较小,需要引进国外种质资源以拓宽其遗传基础。遗传一致度和遗传距离分别是从相同和相反方面度量群体间遗传关系的远近,遗传距离系数大,相应的遗传一致性系数小,材料间的亲缘关系比较远;因此开展杂交育种时建议选择亲缘关系远的組合,更有利于产生杂种优势。
3 讨论
槟榔是热带亚热带地区一种重要的经济作物,栽培地区多为不发达地区,基础研究薄弱。印度是最早对槟榔开展系统性研究的国家,自1957年开始先后从中国等世界各地收集槟榔种质,已经达到117份,保存在印度的维塔槟榔实验站[19]。国内外已开展槟榔种质资源遗传多样性的分析研究,刘慧娟[20]利用10对RAPD标记对海南20个槟榔主要种植县市的74份样品进行聚类分析,在遗传相似系数为0.78左右槟榔群体被分为2组,第1组包括印度、柬埔寨样品,还有部分中国云南与海南的样品等,第2组样品包括来自中国云南和海南样品,说明海南栽培槟榔与周边东南亚国家存在种苗交换现象。任军方[13]利用ISSR标记对海南保亭的槟榔居群进行遗传多样性分析,结果显示保亭槟榔12个居群总的遗传多样性Neis指数为0.2878,表明海南保亭槟榔的多样性水平比较低,可能是由于采样区域太小。台湾学者Hu等[21]开发了9对SSR引物,对台湾屏东县的36份槟榔材料进行了群体遗传分析,预期杂合度和观测杂合度分别为0.71~0.94和0.00~0.88。Sankaran等[22]利用11对RAPD标记对10份印度槟榔种质材料进行遗传多样性分析,PIC值范围为0.17~0.60,UPGMA聚类分析显示野生种槟榔与栽培种槟榔遗传差异大。印度农作物研究中心(Central Plantation Crops Research Institute, CPCRI)的Bharath等[23]利用RAPD标记对60份槟榔种质资源进行遗传多样性分析,遗传相似系数介于0.68~0.93之间,印度本地收集材料聚类在一起,其他国家收集资源聚类在一起,聚类分析结果与地理来源有显著相关性。RAPD标记技术的一个缺点是实验重复性差,反应条件的变化常常导致扩增结果带形的变化,结果可靠性较低[20]。而SSR标记具有重复性高,可靠性强且品种间多态性丰富等特点。Ramaswamy等[24]利用槟榔叶片开展转录组测序,在槟榔基因组上鉴定到6853个含SSR位点的区域,可开发更多的SSR标记用于槟榔种质资源研究。
本研究筛选了32对多态性SSR引物,在58份栽培槟榔材料中共檢测到79个等位基因,每个SSR位点2~5个,平均2.4688个。PIC值变幅为0.0169~0.5969,平均值为0.2254。Shannons信息指数(I)范围为0.0496~1.2552,平均为0.4496;Neis遗传多样性指数平均值为0.2596,说明本研究所用SSR引物在供试样品中检测等位基因遗传多样性偏低,需要更多的SSR标记来评估更多的遗传资源。
根据SSR分子标记进行UPGMA遗传聚类分析,从聚类分析图可以看出,遗传相似系数矩阵(GS)变化范围为0.63~0.94。在遗传相似系数为0.70处可以将海南栽培槟榔与台湾槟榔种区分开,表明海南本地槟榔与台湾槟榔种的遗传差异较大。第V组的材料1份来自台湾,另外3份与其他海南本地种资源遗传距离较远,该组的Shannons信息指数和Neis遗传多样性指数均为最高,表明该组样品间遗传差异较大。海南在20世纪90年代引种过东南亚部分国家或地区的槟榔品种,后期由于果实品质不能满足市场需求,在海南的种植面积越来越少,本次采集的这3份槟榔材料是否为从国外引种遗留下来的资源有待于进一步验证。遗传分析结果显示海南栽培槟榔无明显的地域聚类,说明海南岛槟榔栽培种苗交流比较频繁,且海南栽培槟榔遗传基础相对狭窄,其原因可能是农民选种时喜欢商品性较好的品种,主栽优良品种由于长时间的人工驯化、不同地区间资源共享,导致遗传多样性水平降低。因此,在今后的育种工作中应积极引进国内外的槟榔种质资源以拓宽其遗传背景。本研究明确了海南栽培槟榔品种的遗传多样性差异,为槟榔种质资源保存利用及遗传育种、改良、育种亲本选择提供理论指导,也为槟榔种质资源保护和今后引进种质增补差异资源提供参考。
参考文献
[1] 晏小霞, 王祝年, 王建荣. 槟榔种质资源研究概况[J]. 中国热带农业, 2008(5): 34-36.
[2] 郑锦星, 李忠海, 袁列江, 等. 槟榔生理效应研究进展[J]. 食品科技, 2006(9): 302-305.
[3] 张春江, 吕飞杰, 陶海腾. 槟榔活性成分及其功能作用的研究进展[J]. 中国食物与营养, 2008(6): 50-53.
[4] 黄丽云, 刘立云, 李 艳, 等. 海南主栽槟榔品种鲜果性状评价[J]. 热带作物学报, 2014, 35(2): 313-316.
[5] 从夕汉, 施伏芝, 阮新民, 等. 东南亚62个籼型水稻亲本SSR遗传多样性分析[J]. 核农学报, 2016, 30(5): 859-868.
[6] 李正玲, 胡 琳, 王会伟, 等. 河南省同名小麦地方品种SSR遗传多样性分析[J]. 麦类作物学报, 2016, 36(5): 564-570.
[7] 聂立圆, 李爱贤, 秦 桢, 等. 基于SSR标记的132份甘薯种质指纹图谱的构建及遗传多样性分析[J]. 植物遗传资源学报, 2018, 19(5): 904-911.
[8] Chen H L, Qiao L, Wang L X, et al. Assessment of genetic diversity and population structure of mungbean (Vigna ra-diata) germplasm using EST-based and genomic SSR mark-ers[J]. Gene, 2015, 566(2): 175-183.
[9] 张 晨, 云 岚, 李 珍, 等. 新麦草种质的SSR遗传多样性及群体结构分析[J]. 植物遗传资源学报, 2019, 20(1): 48-59.
[10] Xia W, Xiao Y, Liu Z, et al. Development of gene-based simple sequence repeat markers for association analysis in Cocos nucifera[J]. Molecular Breeding, 2014, 34(2): 525-535.
[11] Xiao Y, Luo Y, Yang Y D, et al. Development of microsa-tellite markers in Cocos nucifera and their application in evaluating the level of genetic diversity of Cocos nucifera[J]. Plant Omics, 2013, 6(3): 193 -200.
[12] 周丽霞, 吴 翼, 肖 勇. 基于SSR分子标记的油棕遗传多样性分析[J]. 南方农业学报, 2017, 48(2): 216-221.
[13] 任军方. 利用ISSR标记对海南保亭槟榔遗传多样性的研究[D]. 海口: 海南大学, 2010.
[14] 齐 兰, 黄丽云, 王 泽, 等. 正交设计优化槟榔SSR-PCR反应体系及引物筛选[J]. 分子植物育种, 2019, 17(4): 1264-1269.
[15] 樊文强, 盖红梅, 孙 鑫, 等. SSR数据格式转换软件DataFormater[J]. 分子植物育种, 2016, 14(1): 265-270.
[16] Yeh F C, Boyle T J. Population genetic analysis of co-dominant and dominant markers and quantitative traits[J]. Belgian Journal of Botany, 1997, 129: 157.
[17] Liu K, Muse S V. Powermarker: An integrated analysis environment for genetic marker data[J]. Bioinformatics, 2005, 21(9): 2128-2129.
[18] Rohlf F J. NTSYS-pc: Numerical taxonomy and multi-variate analysis system[M]. New York: Exeter Software, Setauket, 1992.
[19] Balasimha D, Rajagopal V. Arecanut[M]. Kasaragod: COCRI, 2004.
[20] 刘慧娟. 海南槟榔种质遗传多样性与槟榔黄化病发生关系研究[D]. 海口: 海南大学, 2010.
[21] Hu C H, Huang C, Hung K H. Isolation and characterization of polymorphic microsatellite loci from Areca catechu (Arecaceae) using PCR-based isolation of microsatellite arrays[J]. Molecular Ecology Resources, 2009, 9(2): 658-660.
[22] Sankaran M, Chandrasekar P, Singh D R, et al. Assessment of genetic diversity among arecanut accessions by using RAPD markers[J]. Indian Journal of Horticulture, 2013, 70(3): 428-430.
[23] Bharath B G, Ananda K S, Rijith J, et al. Studies on genetic relationships and diversity in arecanut (Areca catechu L.) germplasm utilizing RAPD markers[J]. Journal of Plantation Crops, 2015, 43(2): 117-125.
[24] Ramaswamy M, Smita N, Naganeeswaranb A, et al. Tran-scriptome sequencing and de novo assembly in arecanut, Areca catechu L elucidates the secondary metabolite path-way genes[J]. Biotechnology Reports, 2018, 17: 63-69.
責任编辑:谢龙莲

