8个玉米ZmDOFs基因对非生物胁迫响应的表达分析
贾利强 赵秋芳 陈曙


摘 要:转录因子家族是由含有锌指结构的DOF结构域组成,构成植物特有的转录因子家族之一,在植物的生长发育进程中诸如信号转导、形态建成、抵御环境胁迫等方面都发挥着重要的作用。本研究利用定量PCR技术,探测了8个ZmDOFs基因在玉米6个不同组织的表达模式,结果表明,8个ZmDOFs基因具有不同的组织特异性表达模式,4个主要在幼穗中表达,2个主要在雄花中表达,ZmDOF28主要在苞叶中表达,而ZmDOF38在多种组织中表达,表明在玉米进化的进程中8个ZmDOFs基因表达模式的保守性和特异性。盐胁迫和干旱胁迫时,分别有7个和6个ZmDOFs基因受到明显的诱导,表明ZmDOFs基因廣泛参与玉米应答盐胁迫或干旱胁迫响应途径。在铵态氮胁迫和硝态氮胁迫时,分别有7个和8个ZmDOFs基因的表达受到了明显的改变,表明ZmDOFs基因参与玉米应答氮胁迫响应途径,体现了在其历史进化进程中的表达模式和功能的保守性与独特性。本研究为后续研究ZmDOFs基因的功能提供了参考。
关键词:DOF转录因子;进化树;组织表达;非生物胁迫
中图分类号:S513 文献标识码:A
Abstract: The DNA-binding one finger (DOF) transcription factor family is a major family of plant-specific transcrip-tion factors containing the DOF domain. These transcription factors are involved in a variety of functions of importance for different biological processes in plants. In the current study, an overview of 8 DOFs genes in maize is presented, including the gene structure, chromosome location, phylogeny, proten motif and expression pattern in responses to various abiotic stresses. Expression patterns indicated that the eight ZmDOFs had tissue specific expression patterns, indicating these genes are crucial in maize growth and development. Moreover, we examined the transcriptional levels of the eight ZmDOFs under salt and PEG6000 stress treatments, seven ZmDOFs and six ZmDOFs were up-regulated after salt and PEG6000 treatment, respectively. We also analyzed the eight ZmDOFs profilling under ammonium and nitrate stress treatment, seven and eight ZmDOFs significantly responded to these two stresses, indicating they are associated with nitrogen stresses responses. The results would provide a very useful reference for the cloning and functional analysis of the members of this gene family in maize and other species.
Keywords: DOF transcription factor; phylogenetic tree; tissue expression; abiotic stress
DOI: 10.3969/j.issn.1000-2561.2021.05.006
DOF蛋白是含有保守的DOF结构域的一类植物特有的转录因子。其保守的结构域一般位于DOF蛋白的N端,由保守的52个氨基酸组成的含有C2-C2锌指结构的元件组成,能特异性地结合下游基因启动子区域的保守元件5-T/AAAG-3来调控下游基因的表达[1]。DOF蛋白的C端一般有高度不保守具有调控基因表达的氨基酸序列组成[2]。第一个报道的DOF基因来自于玉米[3],第一个报道DOFs基因家族的植物是拟南芥[4]。之后,植物DOFs基因家族在许多植物包括低等植物藻类或苔藓中开展了研究[5]。相比较而言,高等植物编码的DOFs基因数目增多。已报道的有拟南芥(36个)和水稻(30个)[4]、大麦(26个)[5]、小麦(96个)[6]、短柄草(27个)[7]、番茄(34个)[8]、大豆(28个)[9]、甘蔗(25个)[10]、白菜(76个)[11]、玉米(46个)[12]、马铃薯(35个)[13],西瓜(36个)[14],以及坚果(25个)和蓖麻(24个)[15]。植物DOFs基因家族的广泛研究为后续揭示DOFs基因的功能提供了基础信息。
植物DOFs基因家族参与调控植物生长发育的诸多进程,包括种子萌发和成熟、激素响应等[16]。玉米的DOF1和DOF2通过调控磷酸烯醇式丙酮酸羧化酶(PEPC)、谷氨酰胺合成酶(AS)以及谷氨酸合成酶等活性来参与碳氮代谢调控[17-18]。进化树分析结果显示,植物DOF蛋白主要分成4个亚家族(A、B、C、D)[4]。D亚家族中的植物DOF蛋白作为细胞周期调控因子,通过节律性地调控CONSTANS(CO)和FLOW?E-RING LOCUS T(FT)基因的表达来影响光周期诱导的植物开花[19]。一些研究表明,番茄的CDFs蛋白作为转录调控因子还有其他的一些功能。在拟南芥中超表达番茄SICDF1和SICDF3会提高拟南芥的抗旱和抗盐胁迫的能力[20]。另外在拟南芥中超表达SICDF3会延迟开花,表明CDFs蛋白在植物的抗逆性以及光周期诱导的植物开花等方面发挥着重要的作用。拟南芥的CDF3基因会被干旱、盐、高低温以及激素ABA等逆境胁迫诱导,超表达CDF3能提高植物的抗逆性[21]。这些研究结果增进了对DOFs基因在植物中的生物学功能的认识。
玉米是粮、经、饲综合利用为一体的高效益大田作物,对于保障我国粮食安全起着重要作用。玉米生长周期内经常遭受各种逆境胁迫,诸如干旱胁迫、盐胁迫以及氮胁迫的影响,玉米产量不稳定,常遭受重大损失,严重影响玉米产业的可持续发展和粮食安全。因此研究玉米抵御各类逆境胁迫的分子生理机制显得尤为迫切。本研究系统地研究了8个ZmDOFs基因应答200 mmol/L NaCl、20% PEG6000胁迫以及氨态氮/硝态氮胁迫的响应表达模式,为后续进一步深入研究ZmDOFs的生物学功能提供参考。
1 材料与方法
1.1 材料
试验材料为玉米自交系B73。总RNA提取采用Trizol试剂(Invitrogen, Carlsbad, CA, USA)。利用NanoDrop2000(Thermo?Scientific, Massa-chusetts, USA)进行RNA浓度与纯度测定。残留的核酸用DNase I(TaKaRa, Tokyo, Japan)除去。cDNA第一链的合成用PrimeScriptTM RT试剂盒(TaKaRa)。获得的cDNA进行后续的荧光定量PCR(Light Cycler 480 System, Roche, Basel, Switzerland)。荧光染料用SYBR Premix Ex TaqTM kit (Roche, Mannheim, Germany)。
1.2 方法
1.2.1 处理方法 为了探测8个ZmDOFs基因在玉米不同组织器官的表达模式,玉米B73自交系种植于中国热带农业科学院玉米育种基地(广东湛江),生长至扬花授粉期,分别采集玉米的根系、茎、叶、雄花、授粉2周的幼穗和苞叶等组织器官,立即进行液氮冷冻处理保存。对于胁迫试验,玉米B73種子经过5%次氯酸钠溶液表面消毒5 min,放于浸润的滤纸上在黑暗28 ℃下催芽,挑选出芽一致的种子在Holland营养液中置于生长箱中生长至3叶期,生长箱设定条件为长日照(16 h光照,28 ℃;8 h黑暗,25 ℃),然后进行各种胁迫处理。正常营养液中生长至15 d后选择生长整齐一致的玉米3叶期幼苗分别转移至200 mmol/L NaCl溶液中进行盐胁迫处理,转移至20% PEG6000溶液中模拟干旱胁迫处理,转移至硝态氮或铵态氮缺乏的营养液中进行不同氮形态缺乏胁迫处理,分别在胁迫处理0、1、6、24 h后取叶片,快速液氮保存,备用。
1.2.2 RNA的提取及定量PCR分析 取0.1 g嫩叶组织,用Trizol试剂对总RNA进行提取。逆转录获得cDNA,进行后续的荧光定量分析。扩增体系为10 μL体系:1 μL cDNA、上下游引物各1 μL、SYBR反应MIX为5、2 μL水。设定的PCR程序为95 ℃ 30 s;95 ℃ 5 s;60 ℃ 20 s;72 ℃ 10 s;共循环45个。玉米Actin 1(GRMZM?2?G?126010)用作内参。用于进行定量分析8个基因的引物序列见表1。反应结束后分析荧光值变化曲线及熔解曲线,并采用2?CT算法计算基因的相对表达量[22]。
1.2.3 8个ZmDOFs的生物信息学分析 36个拟南芥DOFs和8个玉米DOFs的蛋白和核酸序列从TAIR(https://www.arabidopsis.org/)和Phyt?oz?o???me(https://jgi.doe.gov/data-and-tools/phytozome/)数据库下载。利用Pfam和SMART(http://smart. embl-heidelberg.de/)检测候选蛋白结构域,获得拟南芥和玉米的DOF蛋白序列。利用ProtParam (http://expasy.org/tools)在线工具对8个ZmDOFs蛋白进行理化性质分析。使用在线软件MAFFT(https://mafft.cbrc.jp/alignment/software/)进行多序列比对。利用在线软件MEME(http://meme- suite.org/tools/meme)对ZmDOFs保守基序元件进行分析。利用软件MEGA 7(http://www.ma-gasoftware.net/)中的NJ(neighbor-joining)法对DOF蛋白进行进化树分析,构建系统发育进化树。进化树分析参数设定为p-distance方法,bootstrap值设定为1000,其他选项采用默认设置。根据这8个ZmDOF基因在玉米染色体上的位置,利用Mapinspect绘制其在染色体上的分布(http:// www.plantbreeding.wur.nl/uk/software-mapinspect.html)。下载8个ZmDOFs基因的CDS和基因组序列,利用在线软件GSDS 2.0(http://gsds.cbi. pku.edu.cn)进行内含子-外显子基因结构分析。
2 结果与分析
2.1 8个ZmDOFs的生物信息学分析
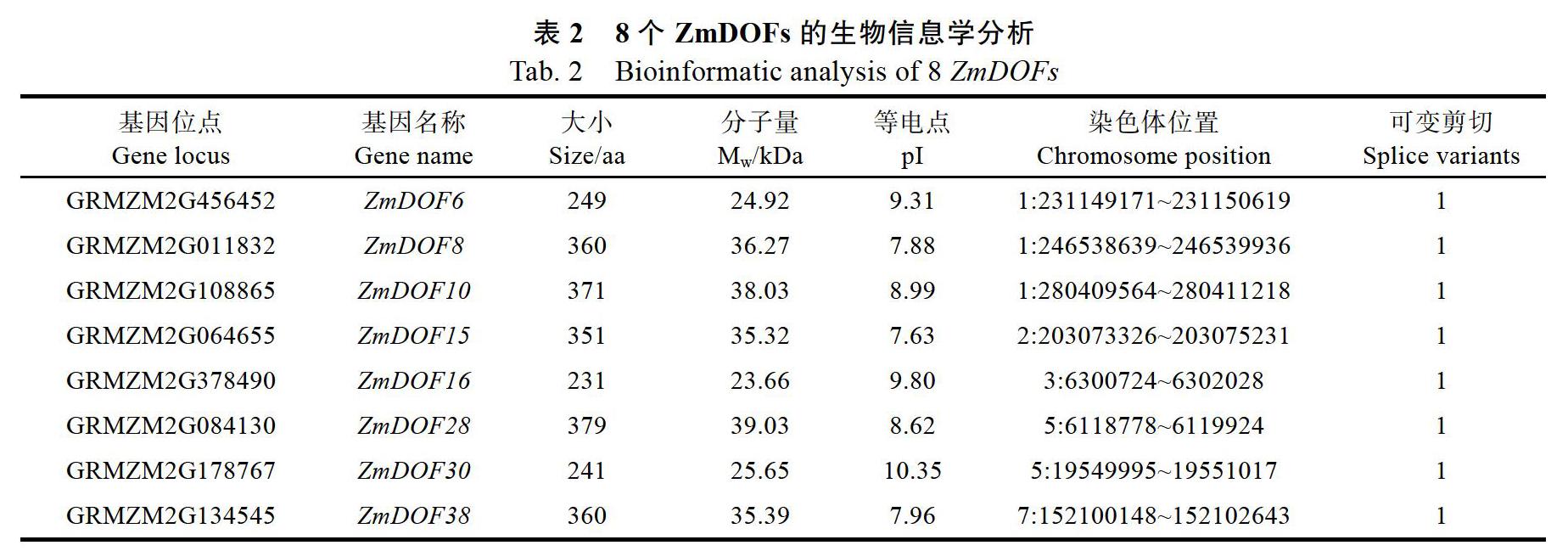
8个ZmDOFs分布于5条染色体上,其中Chr1有3个ZmDOFs,Chr5有2个,Chr 2、Chr 3和Chr 7上各含有1个(图1A)。8个ZmDOFs氨基酸数目为231~379,对应的Mw值为23.66~ 39.03 kDa。pI值为7.63~10.4。8个基因的基本信息见表2。为了更好地理解8个ZmDOFs的结构特征,利用在线软件GSDS 2.0,用基因组序列和cDNA序列进行比对,绘制基因的外显子和内含子的位置,结果显示3个ZmDOFs含有1个内含子,而其他5个ZmDOFs没有内含子(图1B),表明这5个基因可能是通过转座机制获得的新基因。ZmDOF8和ZmDOF30以及ZmDOF10和ZmDOF28由片段复制机制形成(图1C)。8个ZmDOFs和拟南芥36个AtDOFs构成的蛋白序列数据矩阵进行进化树分析,结果表明(图1D),8个ZmDOFs分布于3个亚家族Group Ⅰ、Group Ⅱ和Group Ⅲ。其中Group Ⅰ中有4个ZmDOFs,而另2个亚家族各含有2个ZmDOFs,说明各家族内发生了不同的基因扩增或缺失进化历程。
2.2 8个ZmDOFs的结构域分析
为了探测8个ZmDOFs功能蛋白基序的分布情况,利用在线软件MEME预测了功能基序的分布,鉴定了6个Motifs(图2A)。6个Motifs在8个基因中的分布情况见图2B。Motif1存在于所有8个ZmDOFs蛋白的N端,是构成DOF结构域的核心蛋白基序。其他的基序分布于部分ZmDOFs蛋白中。位于同一家族的ZmDOF 蛋白具有类似的基序分布模式,例如ZmDOF8和ZmDOF30都只有保守的Motif1,Group Ⅲ中的ZmDOF10和ZmDOF28都含有Motif1、Motif3、Motif5和Motif6 四个基序。不同蛋白家族中含有不同的基序分布模式预示着在ZmDOF进化历程中功能分化的来源。
2.3 8个ZmDOFs的组织特异表达分析
为了检测8个ZmDOFs在玉米不同组织器官中的表达模式,为揭示其生物学功能提供信息,利用定量PCR技术探测了在玉米根(R)、茎(S)、叶(L)、花(F)、授粉15 d的小穗(E)和苞叶(CB)等不同组织的表达模式(图3)。8个ZmDOFs具有明显的组织特异性表达模式。其中4个ZmDOFs(ZmDOF6、ZmDOF8、ZmDOF10和ZmDOF15)主要在授粉15 d的幼穗中表达,呈现明显的组织特异性表达模式。ZmDOF6、ZmDOF8、ZmDOF10和ZmDOF15在幼穗中的表达丰度分别是根部的87 278、113、42、7倍,表明这4个基因在幼穗的生长发育进程中发挥着重要的作用。ZmDOF16和ZmDOF30主要在雄花中表达,其表达丰度分别是根部的342倍和34倍。ZmDOF28主要在苞叶中表达,其丰度是根部的27倍。ZmDOF38呈组成性表达模式。结果表明,8个ZmDOFs在玉米幼穗、雄花和苞叶的生长发育进程中发挥着重要的作用,也表明在其进化历程中,8个ZmDOFs的表达模式和蛋白功能具有保守性和分化性。
2.4 8个ZmDOFs对盐和干旱胁迫的响应模式
为了研究8个ZmDOFs在玉米应答盐和干旱等胁迫时的响应表达模式,用200 mmol/L NaCl和20% PEG6000分别模拟盐胁迫和干旱胁迫,分别在处理玉米3叶期幼苗0、1、6、24 h时,取样并利用定量PCR技术检测了8个ZmDOFs的响应表达模式(图4)。在盐胁迫条件下,ZmDOF15没有发生明显的变化,其余7个ZmDOFs的表达在胁迫处理的1 h发生明显的诱导增强,其中ZmDOF10表现最为明显,其表达丰度增加了45倍,其他的增加幅度在2~4倍之间,表明这些基因为盐胁迫早期响应基因,在抵御盐胁迫的响应途径中发挥作用。在整个盐胁迫进程中,ZmDOF6的表达逐渐增强,在24 h时达到高峰,预示着ZmDOF6在参与玉米应答盐胁迫的响应途径中所发挥的生物学功能与其他的ZmDOFs有异。在干旱胁迫处理时,ZmDOF6和ZmDOF28没有发生明显的变化,表明这2个基因可能不参与干旱胁迫途径。其他6个ZmDOFs基因在干旱胁迫处理1 h时发生了明显的诱导,ZMDOF16的表达水平提高了46倍,并且一直维持到胁迫处理结束,表明这些基因是干旱胁迫早期响应基因,参与应答干旱胁迫响应途径。
2.5 8个ZmDOFs对铵态氮和硝态氮胁迫的响应模式
由图5可知,在氨态氮胁迫处理1 h时,有4个基因(ZmDOF8、ZmDOF10、ZmDOF16和ZmDOF30)受到氨态氮胁迫的明显诱导,其中ZmDOF16的表达增强10倍以上,并且一直持续到试验结束,表明这4个基因是参与玉米应答氨态氮胁迫时的早期响应基因,在早期参与调控玉米应答铵态氮胁迫途径。有3个基因(ZmDOF6、ZmDOF15和ZmDOF38)在胁迫处理的后期才出现明显的诱导,表明这3个基因为晚期响应基因。而ZmDOF28的表达在整个胁迫处理过程中没有出现明显的变化,表明该基因可能不参与氨态氮胁迫响应途径。研究表明,8个ZmDOFs基因广泛地参与调控玉米应答氨态氮胁迫的响应途径,同时在其进化历程中表现出不同的表达模式,在调控氨态氮胁迫途径上功能多样。硝态氮胁迫处理时,8个ZmDOFs的表达模式可分为2大类。有4个基因(ZmDOF6、ZmDOF15、ZmDOF28和ZmDOF38)從胁迫处理开始时一直下调,在胁迫试验结束时下调了至少10倍,表明这4个基因在硝态氮胁迫响应途径中可能起负调控作用。另外4个基因(ZmDOF8、ZmDOF10、ZmDOF16和ZmDOF30)在处理1 h时表达明显上调,例如ZmDOF8、ZmDOF16和ZmDOF30分别上调了34、42、43.5倍,之后其表达逐渐下降,到试验结束时,其表达受到抑制,例如ZmDOF10在24 h的表达丰度只有处理前的5%。研究表明,8个ZmDOFs都参与了调控硝态氮胁迫响应途径,并且发挥着不同的作用。
3 讨论
进化树蛋白家族成员起源相同,在蛋白序列、基因结构和功能基序上高度相似,同时在表达模式和蛋白功能上往往体现了保守中的多样性。Group Ⅰ中有4个基因,其中ZmDOF8和ZmDOF30聚合在一起,ZmDOF15和ZmDOF38聚合在一起。ZmDOF8和ZmDOF30的表达模式都具有高度的组织特异性,体现了基因的保守性,前者主要在幼穗中表达,而后者主要在苞叶中表达,表达模式出现了分化,体现了基因的特异性。这一特点在Group Ⅱ和Group Ⅲ中也有体现,说明ZmDOFs在进化进程中的保守性和特异性(或者说分化)的统一。这一特点在不同物种间表现得更加明显。Group Ⅱ家族中的成员ZmDOF6和ZmDOF16都是特异表达基因,体现了基因进化的保守性,前者在幼穗中表达,而后者在雄花中表达,表达模式出现了分化。同一家族成员拟南芥At1G28310调控细胞分裂周期,主要是在分生组织部位表达[23]。Group Ⅲ有3个成员(ZmDOF10、ZmDOF28和At5G65590),ZmDOF10和ZmDOF28表达具有组织特异性,而拟南芥At5G65590主要参与调控气孔形态建成,其表达主要集中在叶片中[24]。这些数据表明在进化历程中,这些基因的表达模式都出现了分化,可以预测其蛋白功能也发生了分化。亚家族Group Ⅰ中的ZmDOF10和ZmDOF30,Group Ⅲ中的ZmDOF8和ZmDOF30分别位于玉米染色体的同源序列区域,表明这2对基因是通过片段复制形成[25-26]。
近年來,许多研究表明植物DOFs基因家族参与调控植物的抗逆性。逆境胁迫会改变植物DOFs的表达模式,表明植物DOFs在植物抗逆性中的作用。辣椒DOFs应答不同逆境胁迫时呈现不同的表达模式[27]。香蕉DOFs对盐和干旱胁迫的表达模式有异,在整个胁迫处理期间,大部分受到盐和干旱胁迫的抑制,只有MaDOF23和MaDOF52受到诱导上调[28]。SICDFs同时受到盐和PEG6000的强烈诱导,特别是SICDF2和SICDF4在胁迫处理24 h时达到最高峰[20]。SICDF3同时受到盐、干旱或者温度胁迫的强烈诱导,似乎SICDF3受逆境胁迫的诱导具有普遍性,而与受胁迫的类型无关[21]。Chen等[12]研究结果表明,ZmDOF16受到盐胁迫的强烈诱导,比对照上调了25倍,而对干旱胁迫不敏感,ZmDOF6同时受到盐胁迫和PEG6000胁迫的强烈诱导。而在本研究中,ZmDOF16受盐胁迫上调了3.88倍,受PEG6000胁迫诱导上调了接近50倍,而ZmDOF6只受到盐胁迫的强烈诱导,上调了近30倍,对PEG6000胁迫响应不敏感。这2种结果的差异可能是由于试验方法的差异,本研究中用的是三叶期的玉米幼苗,而前人的研究中没有说明试验材料的苗龄,另一方面前人的干旱胁迫试验用的是水培玉米苗离体自然干旱,本研究的是PEG6000模拟干旱,以及NaCl的浓度差异和取样时间的差异等。
氮素是植物生长发育必需元素之一,阐述植物氮高效吸收利用的分子生理调控机制是选育资源友好型新品种的基础工作。近年来,一些研究表明DOFs参与调控植物碳氮代谢平衡[29]。ZmDOF1可以促进拟南芥、马铃薯或水稻对土壤中氮素的吸收[17-18]。小麦TaDOF1的超量表达可促进谷氨酰胺合成酶和谷氨酸酶的表达,进而影响了小麦的氮利用效率[30]。水稻OsDOF18通过影响氨氮的运输和氮的分布,进而影响植物对氮素的使用效率[29]。番茄CDF3过量表达会促进氮同化进程[20]。这些基因的表达模式也同时受到氮胁迫的改变。茶树CDF1受到氮饥饿的明显诱导[31]。水稻DOF18受到铵态氮胁迫的诱导,而受到硝态氮的抑制,表现出相反的作用[20]。小麦DOF1也受到氮胁迫的明显诱导。本研究结果和前人的大多一致,例如8个ZmDOFs对铵态氮胁迫大部分受到明显的诱导,而一部分ZmDOFs的表达受到硝态氮胁迫的抑制,表明玉米DOFs应答不同氮形态胁迫时的保守性和多样性,提示ZmDOFs广泛地参与调控玉米氮素代谢平衡并发挥着各自独特的作用。
参考文献
[1] Yanagisawa S, Schmidt R J. Diversity and similarity among recognition sequences of Dof transcription factors[J]. Plant Journal, 1999, 17(2): 209-214.
[2] Yanagisawa S. The Dof family of plant transcription fac-tors[J]. Trends in Plant Science, 2002, 7(12): 555-560.
[3] Yanagisawa S, Izui K. Molecular cloning of two DNA-binding proteins of maize that are structurally dif-ferent but interact with the same sequence motif[J]. The Journal of Biological Chemistry, 1993, 268(21): 16028-16036.
[4] Lijavetzky D, Carbonero P, Vicente-Carbajosa J. Ge-nome-wide comparative phylogenetic analysis of the rice and Arabidopsis Dof gene families[J]. BMC Evolutionary Biology, 2003, 3(1): 17.
[5] Moreno-Risueno M ?, Martínez M, Vicente-Carbajosa J, et al. The family of DOF transcription factors: from green un-icellular algae to vascular plants[J]. Molecular Genetics and Genomics, 2007, 277(4): 379-390.
[6] Liu Y, Liu N N, Deng X, et al. Genome-wide analysis of wheat DNA-binding with one finger (Dof) transcription fac-tor genes: evolutionary characteristics and diverse abiotic stress responses[J]. BMC Genomics, 2020, 21(1): 276.
[7] Hernando-Amado S, González-Calle V, Carbonero P, et al. The family of DOF transcription factors in Brachypodium distachyon: phylogenetic comparison with rice and barley DOFs and expression profiling[J]. BMC Plant Biology, 2012, 12(1): 202.
[8] Cai X, Zhang Y, Zhang C, et al. Genome-wide analysis of plant-specific Dof transcription factor family in tomato[J]. Journal of Integrative Plant Biology, 2013, 55(6): 552-566.
[9] Guo Y, Qiu L J. Genome-wide analysis of the Dof transcription factor gene family reveals soybean-specific duplicable and functional characteristics[J]. PLoS One, 2013, 8(9): e76809.
[10] Gupta S, Kushwaha H, Singh V K, et al. Genome wide in silico characterization of Dof transcription factor gene family of sugarcane and its comparative phylogenetic analysis with Arabidopsis, rice and Sorghum[J]. Sugar Technology, 2014, 16(4): 372-384.
[11] Ma J, Li M Y, Wang F, et al. Genome-wide analysis of Dof family transcription factors and their responses to abiotic stresses in Chinese cabbage[J]. BMC Genomics, 2015, 16: 33.
[12] Chen Y, Cao J. Comparative analysis of Dof transcription factor family in maize[J]. Plant Molecular Biology Reporter, 2015, 33(5): 1245-1258.
[13] Venkatesh J, Park S W. Genome-wide analysis and expres-sion profiling of DNA-binding with one zinc finger (Dof) transcription factor family in potato[J]. Plant Physiology and Biochemistry, 2015, 94: 73-85.
[14] Zhou Y, Cheng Y, Wan CP, et al. Genome wide characteri-zation and expression analysis of the Dof gene family related to abiotic stress in watermelon[J]. Peer Journal, 2020, 8(1-2): e8358.
[15] Zou Z, Zhang X C. Genome - wide identification and com-parative evolutionary analysis of the Dof transcription factor family in physic nut and castor bean[J]. Peer Journal, 2019, 7(17): e6354.
[16] Noguero M, Atif R M, Ochatt S, et al. The role of the DNA-binding One Zinc Finger (DOF) transcription factor family in plants[J]. Plant Science, 2013, 209: 32-45.
[17] Yanagisawa S. Dof domain proteins: plant-specific transcription factors associated with diverse phenomena unique to plants[J]. Plant Cell Physiol-ogy,2004,45(4):386-391.
[18] Kurai T, Wakayama M, Abiko T, et al. Introduction of the ZmDof1 gene into rice enhances carbon and nitrogen assimilation under low-nitrogen conditions[J]. Plant Biotechnology Journal, 2011, 9(8): 826-837.
[19] Goralogia G S, Liu T K, Zhao L, et al. CYCLING DOF FACTOR 1 represses transcription through the TOPLESS co-repressor to control photoperiodic flowering in Arabidopsis[J]. Plant Journal, 2017, 92(2): 244-262.
[20] Corrales A R, Nebauer S G, Carrillo L, et al. Characteriza-tion of tomato Cycling Dof Factors reveals conserved and new functions in the control of flowering time and abiotic stress responses[J]. Journal of Experimental Botany, 2014, 65(4): 995-1012.
[21] Corrales A R, Carrillo L, Lasierra P, et al. Multifaceted role of cycling DOF factor 3 (CDF3) in the regulation of flower-ing time and abiotic stress responses in Arabidopsis[J]. Plant Cell & Environment, 2017, 40(5): 748-764.
[22] Livak K J, Schmittgen T. Analysis of relative gene expres-sion data using real-time quantitative PCR and the 2ΔΔCT method[J]. Methods, 2001, 25(4): 402-408.
[23] Skirycz A, Radziejwoski A, Busch W, et al. The DOF tran-scription factor OBP1 is involved in cell cycle regulation in Arabidopsis thaliana[J]. Plant Journal, 2008, 56(5): 779-792.
[24] Castorina G, Fox S, Tonelli C, et al. A novel role for STOMATAL CARPENTER 1 in stomata patterning[J]. BMC Plant Biology, 2016, 16(1): 172.
[25] Wei K F, Chen J, Wang Y M, et al. Genome-wide analysis of bZIP-encoding genes in maize[J]. DNA Research, 2012, 19(6): 463-476.
[26] Rueda-López M, Ca?as R A, Canales J, et al. The overex-pression of the pine transcription factor PpDof5 in Arabi-dopsis leads to increased lignin content and affects carbon and nitrogen metabolism[J]. Physiology Plant, 2015, 155(4): 369-383.
[27] Kang W H, Kim S, Lee H A, et al. Genome-wide analysis of Dof transcription factors reveals functional characteristics during development and response to biotic stresses in pep-per[J]. Scientific Reports, 2016, 6: 33332.
[28] Dong C, Hu H G, Xie J H. Genome-wide analysis of the DNA-binding with one zinc finger (Dof) transcription factor family in bananas[J]. Genome, 2016, 59(12): 1085-1100.
[29] Wu Y, Yang W, Wei J, et al. Transcription factor OsDOF18 controls ammonium uptake by inducing ammonium trans-porters in rice roots[J]. Molecules and Cells, 2017, 40(3): 178-185.
[30] Kumar R, Taware R, Gaur V S, et al. Influence of nitrogen on the expression of TaDof1 transcription factor in wheat and its relationship with photo synthetic and ammonium assimilating efficiency[J]. Molecular Biology Reports, 2009, 36(8): 2209-2220.
[31] 胡 娟, 王麗鸳, 韦 康, 等. 茶树CsCDF1基因克隆及表达分析[J]. 茶叶科学, 2015, 35(5): 501-511.
责任编辑:谢龙莲

