中文医疗命名实体识别方法研究综述
王 红 王彩雨
( 山东师范大学信息科学与工程学院,250358,济南 )
1 引 言
目前,医疗领域的信息化建设稳步开展,现代化的医疗信息系统已经积累了海量医疗数据,包括电子病历(Electronic Medical Record,EMR)、医学报告和医疗文本等[1].信息技术在医学领域的广泛使用不仅促使EMR数据急剧增加,而且使得大规模EMR的自动分析成为可能.如何将这些医疗数据变成更有价值的学术资源,是国内外研究者共同关注的问题.随着数据的不断积累,利用自然语言处理技术(Natural Language Processing,NLP)和深度学习(Deep Learning,DL)的方法挖掘电子病历,已经成为医学领域和人工智能领域交叉研究的热点.
EMR是描述患者在临床治疗过程中产生的医疗活动的记录,包括:所患疾病、药物、检查和治疗结果等.这些信息是重要的临床数据,对其进行精确高效地分析和挖掘,能够更好地支持临床辅助诊疗系统、精准医学研究和疾病监控等应用[2].但是,由于临床医学信息大多存储在非结构化或半结构化文本中,许多应用程序无法直接使用这些文档,造成大量资源的流失.调查结果表明,阻碍临床数据使用的主要原因是数据的非结构化程度.因此,人们迫切需要发现信息抽取与挖掘方法,将非结构化文本转化为高质量、便于计算机理解的知识.由于NLP技术的不断发展和成熟,其在医学领域也显示出了巨大的使用价值.因此,可以采用NLP技术挖掘电子病历中蕴含的丰富医学知识,为医务工作者和科研人员提供有效的数据资源,进而造福患者.
医学文本挖掘的第一步即是医疗命名实体识别(Medical Named Entity Recognition,NER)[3],旨在提取电子病历中的重要医学术语,比如:疾病、手术和药物等.利用这些提取出来的信息可以发现新的医疗命名实体和分析这些实体之间的关系,从而给临床决策提供数据支持,提升医院的整体医疗质量[4].不仅如此,医疗命名实体的结果可以辅助人们识别并解释晦涩难懂的医疗术语,方便人们了解和认识医学知识.因此,医疗命名实体识别对医学研究、医疗诊断和患者都具有重要意义.
2 中文医疗命名实体识别研究的热点
2.1医疗命名实体识别研究NER是医学领域NLP任务的重要一步,如基因名称的提取,可以为后期的药物预测、疾病预测等提供基础;药物和疾病的识别,可以为后期的关系抽取、知识图谱的构建等提供基础;症状和治疗方式的识别,可以为后期的智能问答系统提供基础.生物NER的常见方法是将该任务表述为一个序列标记问题,从基于特征的方法到基于神经网络方法,各种方法已经被成功地应用于该领域的研究中[5].医疗命名实体识别是人工智能与医学交叉领域的重要课题,虽然人工智能技术已广泛应用于医学领域,但是在医疗命名实体识别方面仍然存在很多亟待解决的问题.首先,大多数的医疗文本数据存储方式为结构化或半结构化,如何更好地将数据转化为计算机所能理解的方式,并且不损失大量的语义信息,仍是目前研究者们关注的问题之一;其次,使用监督学习技术来实现高性能的NER系统需要一个手动注释的语料库,其中,文本中每次提到所需的语义类型都需要注释.然而,构建和维护注释语料库需要大量的人力和精力;另外,单个医疗平台上的标记数据通常是有限的,虽然标记数据集存在于许多不同的医疗平台中,但由于医疗数据对隐私高度敏感,这些数据不能直接共享.因此,如何从大量的医学数据中挖掘出有意义的信息为医疗领域提供支持并帮助人们的生活,是一直以来人们关注的热点之一.
2.2中文医疗命名实体识别研究虽然人工智能[6]技术已广泛应用于医学领域,但是在医疗命名实体识别方面仍然存在很多亟待解决的问题,尤其是中文医疗文本的命名实体识别.首先,目前大多数命名实体识别工作集中在英文数据集,受中英文语言表达不同的限制,英文医疗文本NER方法不能直接应用于中文医疗文本NER任务,模型的泛化能力严重不足;其次,语义特征提取不够充分.早期的命名实体方法多采用独热(One-hot)向量表示字词嵌入,一方面会导致特征向量嵌入的高维度和稀疏性;另一方面会忽略字词之间隐含的语法和语义关系,无法充分融合多维信息[7];另外,由于涉及患者隐私问题,很多医疗数据不公开,因此开源的医疗数据资源相对较少,无法很好地进行大规模的模型训练.如何准确把握中文医疗数据的特点,充分表达医疗文本的语义特征,解决中文医疗文本数据样本不够丰富问题,以及提高模型的泛化能力都是目前研究的挑战性问题.
2.2.1 中文医疗文本的语义缺失问题 近年来,医疗NER研究持续开展,早期广泛采用特征工程的方法,并取得了一些成果.随着深度学习时代的来临,神经网络模型则为医疗NER带来新的突破.尽管如此,医疗命名实体识别依然存在很多难点.第一,医疗文本通常不规范,很难一致地识别疾病和症状;第二,语义信息提取不完整.众所周知,英语文本中单词之间存在明显的间隔,中文文本中字符之间没有间隔.一方面,可以采用基于字符的分割方式,简单直观,且不会存在单词边界信息错误的问题,但是会缺失语义信息,也就是说,由于没有单词、位置、语法结构以及领域知识等其他类型的信息,很难正确表达一句话的正确含义,甚至还会产生歧义.另一方面,可以采用基于词的分割方式.虽然单词所包含的信息比字符丰富,但是同一个词在不同上下文中的含义也可能不同.因此,只采用单词序列,也会出现NER结果的偏差;第三,分词错误.中文分词一直是中文自然语言处理领域的瓶颈.如上所述,基于单词的分割方式可能会存在分词错误,进而直接导致实体识别效果的下降.这个问题在中文医疗NER中格外突出.并且在电子病例中含有很多专业术语,例如:“乙状结肠癌根治性切除术”和“奥沙利铂”等,如果没有医学领域知识的指导,对这些专业术语的分词将变得非常困难.针对此问题,可以采取融合多语义字典和多模态树的医疗命名实体识方法.具体来说:
1) 融合多语义字典.即分别将字典和词典应用于模型中.在字符基础上,把词汇信息和位置信息合并到字符识别中,达到更加精细的医疗语义理解.加入一种单词位置信息的提取方法,更好地获取单词的边界信息,减少中文单词分割错误的不良影响.
2) 不同粒度特征的提取.多模态树由句子中的字符和单词构成.字符和单词分别来自不同的字典和词典,因此称为多模态树.同时,采用不同的路径方式从多模态树中动态提取不同长度的单词序列,确保语义信息不丢失.
2.2.2 中文医疗文本的资源不足问题 随着诊断工具和医疗决策系统的使用,产生了大量的医疗电子病历数据,它们是居民在访问医疗机构期间生成的有价值的数据,为发现疾病的规律和预测疾病的发生提供了强大的帮助[8].利用挖掘出的信息,可以生成关于患者或治疗的相关知识,进而为后续诊疗提供决策支持,为患者提供更好的个性化诊疗服务[9,10].但是,同时也带来了医疗参与者隐私丧失的风险[11],因此只有少量的数据被公开利用.
受到新闻和社交等领域的命名实体识别研究的启发,基于深度学习的自然语言处理也成为医疗NER的主要方法,许多研究已经证明了它在临床实践和研究中的成功应用.但是,采用深度学习方法进行命名实体识别时,一般需要大规模的标注数据,但由于人工标注代价高昂,在中文医疗命名实体识别中并没有大规模的标注数据,所以,基于小规模标注语料的命名实体识别也成为热点研究问题.针对此问题,可以采取基于图神经网络和跨语言的医疗命名实体识别方法.具体来说:
1) 通过图神经网络解决实体冲突问题.通过图神经网络建立实体词典与医疗文本之间的关联关系,通过实体词典指导医疗文本内容中的实体类型,从而解决实体冲突的问题.
2) 实现跨语言知识迁移.将高资源语言知识迁移到中文医疗文本中,进行知识补充,借助外部语言知识监督中文NER任务.在提出的方法中中引入双语词典,由于不同的语言通常包含关于实体的互补线索,因此可以通过双语词典,将资源丰富的语言映射到低资源中的语言,提取跨语言知识,获取不同语言之间的含义,实现高资源向低资源的知识迁移.
3) 学习外部知识.外部知识是通过外部知识库获得的比较隐含的语义信息,可以实现用一个有限的向量集合去表示一个词汇,不但可以提取细粒度信息,还可以进一步捕捉到词之间的关系,解决歧义的问题.
2.2.3 中文医疗文本的命名实体嵌套问题 医学数据的大规模研究,推动了医学领域的迅速发展.采用文本挖掘方式,提取存储在数据库中的大量可用医学报告信息,可以获得丰富的医学知识,为医学研究和应用带来巨大的好处[12].在医学数据挖掘任务中,医疗命名实体的识别和规范化是最基本的任务.然而,从这些数据中提取临床信息并不容易,因为这些数据是用自然语言编写,充满医学术语、缩写词、速记符号、拼写错误和句子片段的记录[13].除此之外,还面临着许多挑战和难点,比如:命名实体识别在识别的过程中会存在实体彼此嵌套的问题.像“直肠癌根治术”这一类的实体,既包含了疾病类的实体“直肠癌”,又包含了手术类的实体“直肠癌根治术”.但是有关命名实体识别工作几乎完全忽略了嵌套实体,而是选择专注于最外层的实体,并且在命名实体中,分词和词性标注都是在词的级别上进行处理的,因为词汇信息被视为解决语义歧义的关键,但词汇化的统计信息稀疏且难以直接估计[14].因此,语义注释资源尤为重要,比如句法结构分析在命名实体识别任务上也起着重要的作用.由于中文表达和英语表达不同,中文存在分词的问题,使得目前现阶段的句法分析大多数都集中在英语上.针对此问题,可以基于依存句法分析和动态堆叠网络的医疗命名实体识别方法,具体来说:
1) 通过动态堆叠网络解决实体嵌套的问题.动态堆叠网络是根据实体嵌套的层数进行网络的叠加,通过动态堆叠网络,对句中的嵌套实体进行动态堆叠式识别,利用内嵌实体的特征帮助外部实体的识别,从而解决实体嵌套的问题.
2) 获取长距离的语义信息句法分析.句法分析是分析句子中成分之间的关系.通过提取文本数据中的句法结构,分析各个单词的词性,捕获整个句子中主语、宾语和谓语等之间的依存关系.可以获取长距离的语义信息.
3 医疗命名实体识别研究的现状
医疗命名实体识别是人工智能与医学交叉领域的重要课题和前沿方向.纵观NER相关前沿研究方法,本文将其分为4类:基于机器学习的NER方法、基于深度学习的NER方法、基于预训练模型的NER方法和可解释的NER方法.
3.1基于规则和词典的医疗命名实体识别早期的NER方法绝大部分是基于规则和词典的,即科研人员通过分析文本数据的书写规则和语言特色,结合相关词典手工设计相应的规则,然后通过模式匹配的方式完成NER任务.其中,规则包括词的位置、关键词、指示词、中心词、方位词、标点符号和统计信息等;词典是由两部分组成:外部词典和特征词词典,外部词典是指众所周知的常识词典;特征词词典是指对文本解析后提取的特征词集合.人工制定规则和词典后,利用相匹配的方法对文本实现命名实体识别[15].
Carol等人[16]开发了一种通用的自然语言处理器,该处理器可识别叙事报告中的临床信息并将其映射为包含临床术语的结构化表示形式.Fukuda等人[17]提出了一种从生物学论文中识别蛋白质名称的方法, 该方法在这些字段中使用专有名词描述的特征,并且不需要任何预先准备的特定术语词典.无论是已知的还是新定义的,无论是单个单词还是复合单词,都可以高精度地提取句子中的材料名称.此类方法的共同问题是,第一,专家需要花费巨大的时间和精力编写规则,成本较高;第二,编制过程中不能解决未登录词汇的问题,因此容易出现差错;第三,不同的系统需要书写不同的规则,可移植性差,难以快速复用.
3.2基于机器学习的医疗命名实体识别近年来,主要医疗命名实体识别方法是基于传统机器学习的.常用的机器学习模型有隐马尔可夫模型[18](Hidden Markov Models,HMM)、最大熵型[19](Maxim Entropy,ME)、支持向量机[20](Support Vector Machine,SVM)和条件随机场[21](Conditional Random Field,CRF)等.
Lafferty[22]在2001年首次提出CRF,并由McCallum[23]最先成功应用于命名实体识别中.Settles[24]提出了CRF结合特征集合的方法,用于生物医学的NER任务.Ju[25]使用SVM从生物医学文本中识别指定类型的名称.Tang[26]开发了基于SVM的NER系统来识别医院收费摘要中的临床实体.此外,他们还提取了两种不同类型的词表示特征(基于聚类的表示特征和分布表示特征),并将它们与基于SVM的临床NER系统集成在一起.Liu[27]首先建立了一个医学词典,然后基于条件随机场CRF算法,研究了不同类型特征在中文临床文本NER任务中的作用.Wang[28]提出了一种基于CRF的中药NER方法.结果表明,该方法对明清时期提出的古代医学症状和发病机制的鉴定具有明显的优势.Wang[29]对中医临床文本中的症状名称识别进行了初步研究.
尽管研究取得很大进展,但是,研究人员不得不花费大量精力进行特征工程[30],超过80%的工作是数据预处理,合并、定制和清理数据集[31],以获得更好的识别结果.上下文特征词或词性等,均需要手动标记,成本非常高,而且,还存在数据高维稀疏、扩展性差、冷启动及用户偏好建模难等问题,使得基于传统机器学习的医疗命名实体识别效果无法令人满意[32].
3.3基于深度学习的医疗命名实体识别深度学习在语音和图像等领域取得了巨大成功,也被越来越多地应用到自然语言处理任务中[33].由于基于深度学习的方法不需要特征工程,而且能够找到更深层次和更加抽象的特征,因此,基于深度学习的NER方法受到研究者的广泛关注.最典型的神经网络有深度神经网络 (Deep Neural Networks,DNN)[34]和循环神经网络(Recurrent Neural Network,RNN).尤其是基于RNN-CRF的方法在中文命名实体识别任务中取得了很好的性能.Wu等人[35]基于最小特征工程策略研究了一种深度学习方法来识别中文临床文献中的临床实体.Wang等人[36]将字典纳入中文NER的深层神经网络.Xu等人[37]提出了一种基于双向LSTM[49](Long Short-term Memory, LSTM)和CRF的医学NER模型.Tang等人[38]提出了基于注意力的CNN-LSTM-CRF,用于识别中国临床文本中的实体.Liu等人[39]研究了词嵌入对药物名称识别的影响,并与传统的语义特征进行了比较.Tang等人[40]研究了基因和DNA等识别任务中的单词嵌入.此外,Wu等人[41]研究了神经词嵌入在临床缩写消歧中的应用.Liu等人[42]开发了面向任务的资源来学习单词嵌入,用于临床缩写扩展.一些研究侧重于联合提取生物医学实体和关系[43,44].
上述方法大都集中在英文数据集.尽管研究人员在NER问题上取得了突破性的进展,但是将深度学习应用于中文医疗NER仍然存在许多难点,包括数据格式多样化、数据缺乏、模型可解释性和可靠性等问题.
3.4基于预训练模型的医疗命名实体识别命名实体识别是自然语言处理中的一项基本任务,近几十年来人们对此进行了大量的研究.传统的命名实体识别方法将字映射为One-hot编码,这样无法表征字的多义性.为了解决这一问题,研究者们提出了一种利用预训练语言模型进行单词表示的方法.
早期最常见的预训练模型采用了Word2Vec工具训练词向量,该工具由Mikolov等人[45]提出,可以在捕捉语境信息的同时压缩数据规模.Word2Vec工具包含了CBOW和Skip-gram 两种语言模型.由于Word2Vec只考虑到了词的局部信息,因此,Peters等人[46]提出了ElMo算法,对这一点进行优化,提供了词级的动态表示,能有效地捕捉语境信息,解决一词多义的问题.Pennington等人[47]提出了一种基于全局共生矩阵的单词表示算法——GloVe算法.
近年来,预训练模型受到了越来越多的关注,2018年,Devlin等人[48]提出的BERT(Bidirectional Encoder Representations From Transformers,BERT)模型采用了具有更强表达能力的Transformer结构.2019年Yang[49]等人提出了XLnet模型,弥补了BERT模型的很多不足.研究者已经将这两种模型用于NER任务,Yang等人[50]提出基于BERT嵌入的中文NER方法,利用具有双向Transformer结构的BERT预训练模型,增强字的语义表示,根据其上下文动态生成语义向量.Ding等人[51]提出了基于BERT嵌入和残差连接的中文电子病历NER方法,在增加网络表征能力的同时解决了堆叠多层网络时出现的神经网络退化问题.Zhang等人[52]提出基于BERT-BiLSTM-CRF的中文电子病历NER模型,该模型利用BERT预训练语言模型来增强单词的语义表示,然后将BiLSTM网络与CRF(Conditional Random Field,CRF)层结合起来,以词向量作为训练的输入.Cai等人[53]提出了基于BERT嵌入的中文NER方法,该方法通过预训练的BERT语言模型增强单词的语义表示.Li等人[54]提出了基于变异BERT结构的中国临床命名实体识别,利用未标记的特定领域知识,预先训练了未标记的中文医疗文本.Yang等人[55]提出了基于XLnet与字词融合编码的中文命名实体识别研究.此模型不仅克服了中文文本分词困难的局限,而且能够兼顾对输入文本中词与词之间关联性的关注.Huang等人[56]采用XLnet构造了一种新的医疗文本表示形式,并完成临床数据的建模.Carvallo等人[57]提出了一种应用在医学上的文本分类方法,并在实验中证明了XLnet的效果最好.Sharma等人[58]提出了一种用于生物医学序列标记任务的预先训练的合并上下文嵌入,其中也采用了XLnet预训练模型作为嵌入工具.
4 医疗命名实体识别数据集和效果评估
4.1数据集常用的医疗命名实体识别数据集有CCKS 2018和CCKS 2019等,均采用了全国知识图谱与语义计算大会(CCKS)提供的数据,数据为一组记录了病人在医院诊断治疗的全过程的临床病例电子文档,任务的要求给出文档中与医学相关的命名实体名字的字符串边界.数据中包括六种类型的实体,分别为:
1) 疾病和诊断:医学上定义的疾病和医生在临床工作中对病因、病生理、分型分期等所作的判断.
2) 检查:影像检查(X线、CT、MR、PETCT等)+造影+超声+心电图,未避免检查操作与手术操作过多冲突,不包含此外其它的诊断性操作,如胃镜、肠镜等.
3) 检验:在实验室进行的物理或化学检查,本期特指临床工作中检验科进行的化验,不含免疫组化等广义实验室检查.
4) 手术:医生在患者身体局部进行的切除、缝合等治疗,是外科的主要治疗方法.
5) 药物:用于疾病治疗的具体化学物质.
6) 解剖部位:用指疾病、症状和体征发生的人体解剖学部位.
关于数据的详细介绍如下:
CCKS2018:训练集包括600份病历,测试集包括400份病历.
CCKS2019:训练集包括1 600份病历,测试集包括379份病历.
这些不同类型实体的数量统计如图1所示.
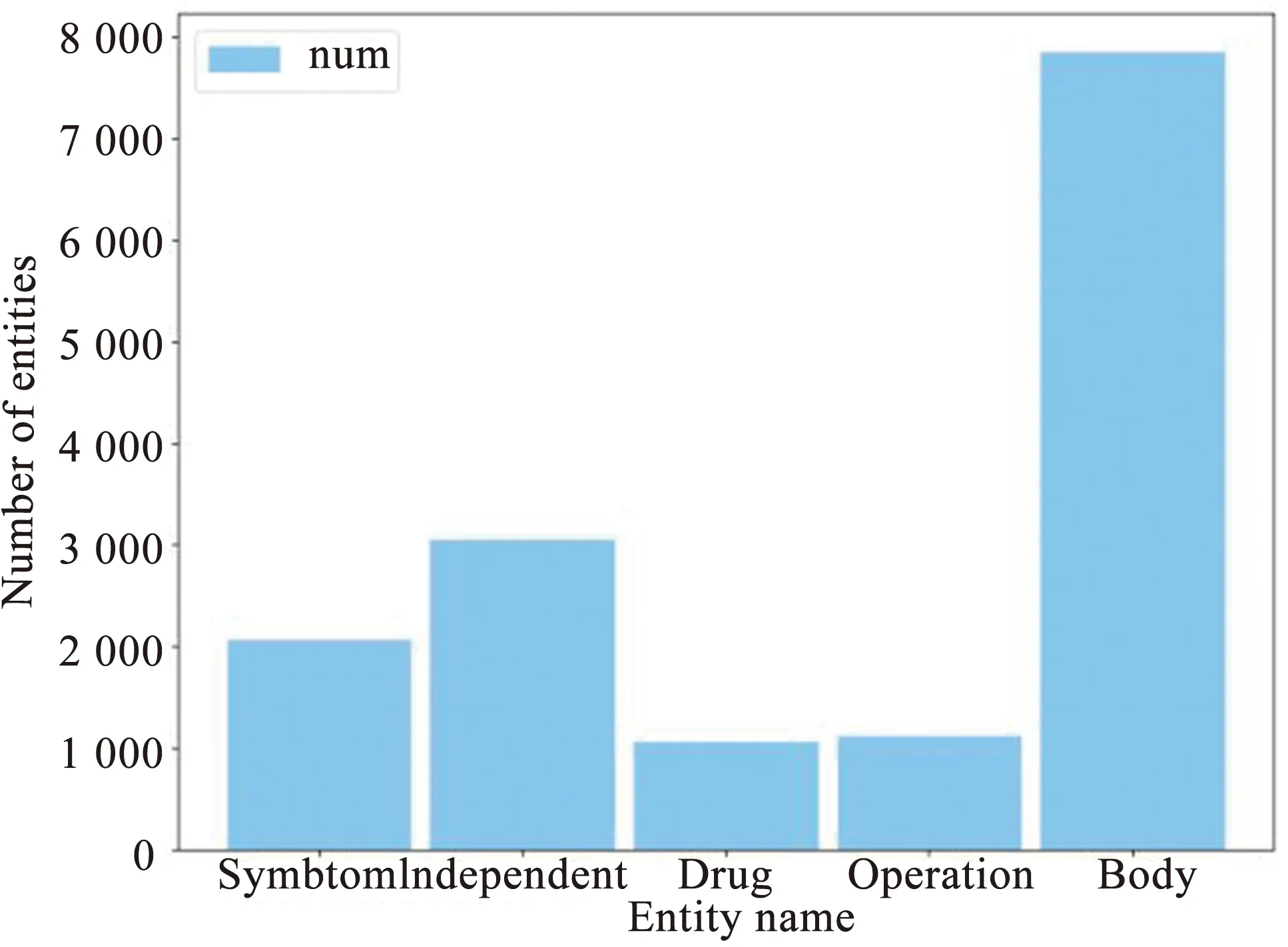
(a)CCKS2018
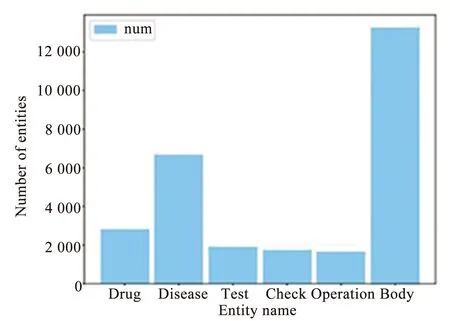
(b) CCKS2019
4.2数据集标注策略命名实体识别任务中常用的标注模式有BIO,BIOE和BIOES等,其中B表示实体开始,I表示实体中间,E表示实体结束,S表示单个实体,O表示不是实体.由于BIOES标注模式的标签种类最多,其表达能力最强,因此,大多数数据的标注均选择BIOES模式.一些工作不仅要预测实体边界,还要预测实体类型,因此,数据的标注格式为“实体类型-B/I/E/S”.数据集中共有23种标签结果,分别为:“O”, “影像检查-B”,“影像检查-I”,“影像检查-E”,“解剖部位-B”,“解剖部位-I”,“解剖部位-E”, “解剖部位-S”,“手术-B”,“手术-I”,“手术-E”,“实验室检验-B”,“实验室检验-I”,“实验室检验-E”,“实验室检验-S”,“疾病和诊断-B”,“疾病和诊断-I”,“疾病和诊断-E”,“疾病和诊断-S”,“药物-B”,“药物-I”,“药物-E”,“药物-S”.如“患者诊断为直肠癌”的标注结果如图2所示.

图2 实体的标注模式
4.3文本预处理为了获得更好的实验结果,需要对输入的原始数据进行预处理,使得数据更符合标准.主要操作过程如下:
1) 有些数据在标注的过程中,存在标注方式不标准的问题,比如,在标注实体时,实体的位置出现了偏差,针对这一问题,对数据进行了手动调整.
2) 为了将数据更好地输入到模型中,在保证句内语义信息相对完整的前提下,句子以句号进行分割,并且在训练数据时,将句子中的标点符号“;”和“:”等删除.
3) 统一数据文本的拼写方式,英文大小写转换,不可见字符的处理等.
当然还有其他处理方式,应该通过观察数据的特点来确定.
4.4评价指标为了评估模型效果,需要进行对比实验.目前,命名实体识别任务常采用的评价指标有精确率(Precision)、召回率(Recall)和F1值 ( F1-Measure) 等.具体计算公式如下:
(1)
(2)
(3)
其中,TP表示将正例预测为正,FP表示将负例预测为正,FN表示将正例预测为负.
评价指标又分为严格指标和松弛指标.如果标记的实体类型和边界均正确,则严格指标认为该实体标记正确.如果实体边界正确,实体类型至少有一个正确,则松弛指标将实体标记确定为正确.如“直肠癌”的两种评价指标标记结果如表1所示.

表1 标记结果的正确评估举例
5 医疗命名实体识别未来研究的方向与建议
近年来,随着人工智能技术的高速发展,医疗健康与人工智能的交叉研究成为炙手可热的研究方向,出现了大量的新型智能医疗设备和电子医疗系统.电子医疗系统中包含着丰富的医疗健康数据,为医学研究和医疗实践奠定了强大的数据基础.但是,如何充分利用这些数据进行探索和分析,更好地支持临床决策和大众健康仍然面临着诸多挑战.其中,医疗文本中的NER,尤其是中文医疗文本的NER即是关键挑战之一.尽管命名实体识别在各个领域都引起了极大关注,但是由于医学文本的复杂性、资源稀少和实体嵌套等问题,医学领域的NER更加具有挑战性.下一步的研究工作可以考虑从以下二个方面开展:
1) 融合领域知识.随着信息技术的飞速发展,知识在各个领域中发挥着越来越重要的作用,但是却也面临着“数据爆炸而知识贫乏”的困境.专业的领域知识可以帮助研究者们更好地进行数据分析.因此,在未来的工作中,可以进一步改进模型,将领域知识加入到模型中.例如,疾病与药物之间的关系等,使更多的知识来指导和约束模型完成识别任务.
2) 数据增强.现在深度学习技术通常需要大量的标记数据,然而,标记庞大的数据集根本不现实,生物医学领域尤其如此,因为标注数据需要更多的领域专家知识,需要消耗大量时间.因此,针对医疗数据资源稀少的问题,可以采用数据增强的方式,比如同义词替换,句法变换等方式,增加数据资源,实现大规模的数据训练,使用更广泛的数据集来优化模型,增加模型的泛化能力.

