基于多视角的脑胶质瘤分级模型研究
郝惠惠 吴亚平 赵国桦 王梅云 林予松,2*
1(郑州大学互联网医疗与健康服务河南省协同创新中心 河南 郑州450052) 2(郑州大学软件学院 河南 郑州450052) 3(河南省人民医院影像科 河南 郑州450003)
0 引 言
脑胶质瘤是颅脑最常见的恶性肿瘤[1]。世界卫生组织根据肿瘤的恶性程度将脑胶质瘤分为低级别脑胶质瘤(Low Grade Glioma, LGG)和高级别脑胶质瘤(High Grade Glioma,HGG),并分别给出处理治疗的建议方案[2]。因此,精确的脑胶质瘤分级对治疗方案的确定或者预后有着重大意义[3]。磁共振成像(Magnetic Resonance Imaging,MRI)在软组织中具有敏感性和卓越的图像对比度,是大脑诊断和治疗过程中的首选检查方法[4]。通常头部MRI检查均包含液体衰减反转恢复(Flair)成像、T1加权成像(T1)、增强T1加权成像(CET1)和T2加权成像(T2)等序列[5-6]。每个MRI序列构成了一个视角,从不同的方面对肿瘤病灶进行描述,为脑胶质瘤诊断提供多个视角的互补信息。
随着机器学习的快速发展,以影像组学(Radiomics)为代表的脑胶质瘤分级研究,通过高通量地从医学影像中提取大量特征训练预测模型,取得了显著的研究成果[7-9]。但现有研究主要从单视角出发基于某个序列进行特征计算和模型训练[10],或将多个序列计算的特征进行简单组合进行训练[11],难以有效利用脑胶质瘤多个视角之间的互补信息。
多视角学习以训练目标不同观测数据作为研究对象,每个观测数据作为一个视角,通过多个视角的协同训练,能有效提升模型的泛化性能[12-13]。传统的多视角学习主要采用共同训练算法[14],如co-EM[15]、co-testing[16]等。最大熵判别(MED)模型是一种判别式和生成式的集成框架,能有效应用于疾病诊断[17]。文献[18]验证了MED在多视角显微神经损伤影像上的诊断效果显著。基于边缘一致性的算法以MED作为基础模型框架,充分考虑了多个视角分类结果的潜在一致性,代表算法有多视角最大熵判别(MVMED)[19]、灵活的多视角最大熵判别(AMVMED)[20]等。AdaBoost算法由于泛化错误率低被广泛应用于集成机器学习算法实践中,与Adaboost结合成为多视角学习的一种新的研究方法[21]。文献[22]将多视角AdaBoost算法和SVM算法结合实现了汉语组块识别,文献[23]利用AdaBoost思想增强了视角的多样性,文献[24]通过改进传统的AdaBoost算法实现了多视角人脸检测方法,均取得了较好效果。
脑胶质瘤磁共振影像的每个序列均可作为单一视角进行肿瘤描述,多个视角的互补性可以为脑胶质瘤分级提供更多的信息,应用多视角学习进行脑胶质瘤分级将有助于提升模型的泛化性能,如何利用脑胶质瘤多视角信息的互补性是一项具有挑战性的任务[25]。本文基于多视角AdaBoost算法设计了一种以最大熵判别(MED)作为基本分类器的多视角AdaBoost最大熵判别模型(Multi-view AdaBoost Maximum Entropy Discrimination,MBMED),能够充分利用不同视角的信息互补性及目标一致性,实现脑胶质瘤的精准分级。
1 模型设计
本文结合多视角、MED模型及AdaBoost的思想,提出了MBMED模型。AdaBoost是一种将弱分类器以迭代的方式形成一个强分类器的集成模型,模型权重和样本权重不断地被迭代更新。MED模型则是借鉴正则化理论的概念,通过对目标函数施加大间隔约束对KL散度进行最小化求解,具有较好的灵活性,同时满足了判别性的分化约束[26]。利用多视角学习的特点,本文提出的MBMED模型采用类似AdaBoost的思想,引入了新的参数αk,以多个视角的MED模型作为基础分类器并在此基础上对脑胶质瘤进行分级处理,实现脑胶质瘤多视角的信息互补性。为了满足多视角MED学习的一致性需求,还需要令各个视角的分类间隔相等。在MBMED模型训练过程中,通过调整不同样本、不同视角在模型中的权重,更好地平衡了各个视角对于模型的贡献,既利用了多个视角之间的互补性,又考虑了各个视角的多样性和差异性。MBMED模型的框架如图1所示。
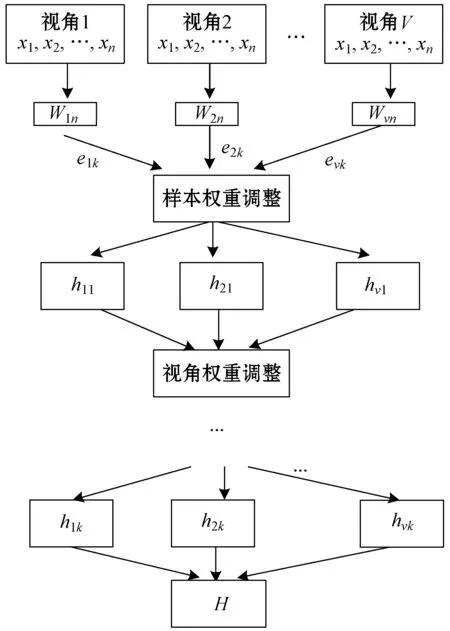
图1 MBMED模型框架
在MBMED模型中,令给定的数据集T={Xvt,yt},v∈{1,2,…,V},t∈{1,2,…,N},其中v表示视角数量,t表示输入样本数量。yt∈{±1}分别表示脑胶质瘤的高低级别。首先以视角为单位,给视角中的样本赋予一个权重W={w1,w2,…,wn},且初始化权重为1/N,该权值向量对样本点的权值进行记录。接下来按W对样本分布D进行K次迭代运算,迭代规则如下:
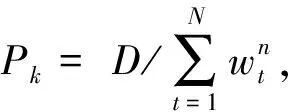
minp(Θ,γ)KL(p(Θ,γ)‖p0(Θ,γ))
(1)
1≤t≤N
式中:γ={γ1,γ2,…,γN}表示分类间隔向量;L(Xt|Θ)为判别函数(Θ表示分类器参数);p(Θ,γ)为需要求解的联合分布;p0(Θ,γ)为目标数据集的一个理论分布。优化目标是使得p(Θ,γ)接近p0(Θ,γ),求解后得到弱分类器优化问题的解为:
(2)
Z(λ)为归一化常数:
(3)
通过对公式进行实例化求解得到λ后,将其代入式(2),求出p(Θ,γ)并将λ积分掉得到最终的概率分布p(Θ),并对新样本标签进行预测:
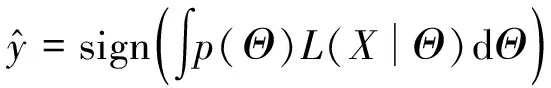
(4)
然后根据弱分类器hv(xt)对脑胶质瘤的预测结果计算误差率:
(5)
接下来的权重更新中,引入αk变量来表示各视角在脑胶质瘤分级中的关系:
(6)
式中:⊗为同或运算,表示当V个弱分类器相同时为0,不同时为1;αk表示了不同视角对于脑胶质瘤预测结果不同的比例。
为了表示各个视角在脑胶质瘤分级中的作用与差异性,令各个视角的权重βk表示为:
(7)
则新的样本权重表示为:
(8)
以此不断迭代,模型训练共得到V×K个弱分类器。最后对脑胶质瘤的新样本高低级别进行预测:
(9)
迭代过程中,若样本在MED基础学习器中的预测错误,将导致ek增大,在下一轮迭代过程中,样本权重将增加,从而达到调整各个视角及视角内部权重的作用,同时兼顾了每个视角在模型中的联系与作用。
2 实 验
为了验证MBMED模型对于脑胶质瘤分级的有效性,实验针对脑胶质瘤MRI数据的四个视角,分别计算相应的影像组学特征得到训练集,然后对训练集进行预处理,最后使用多视角AdaBoost-MED得到最终的输出,并分析模型的各项性能指标,实验流程如图2所示。
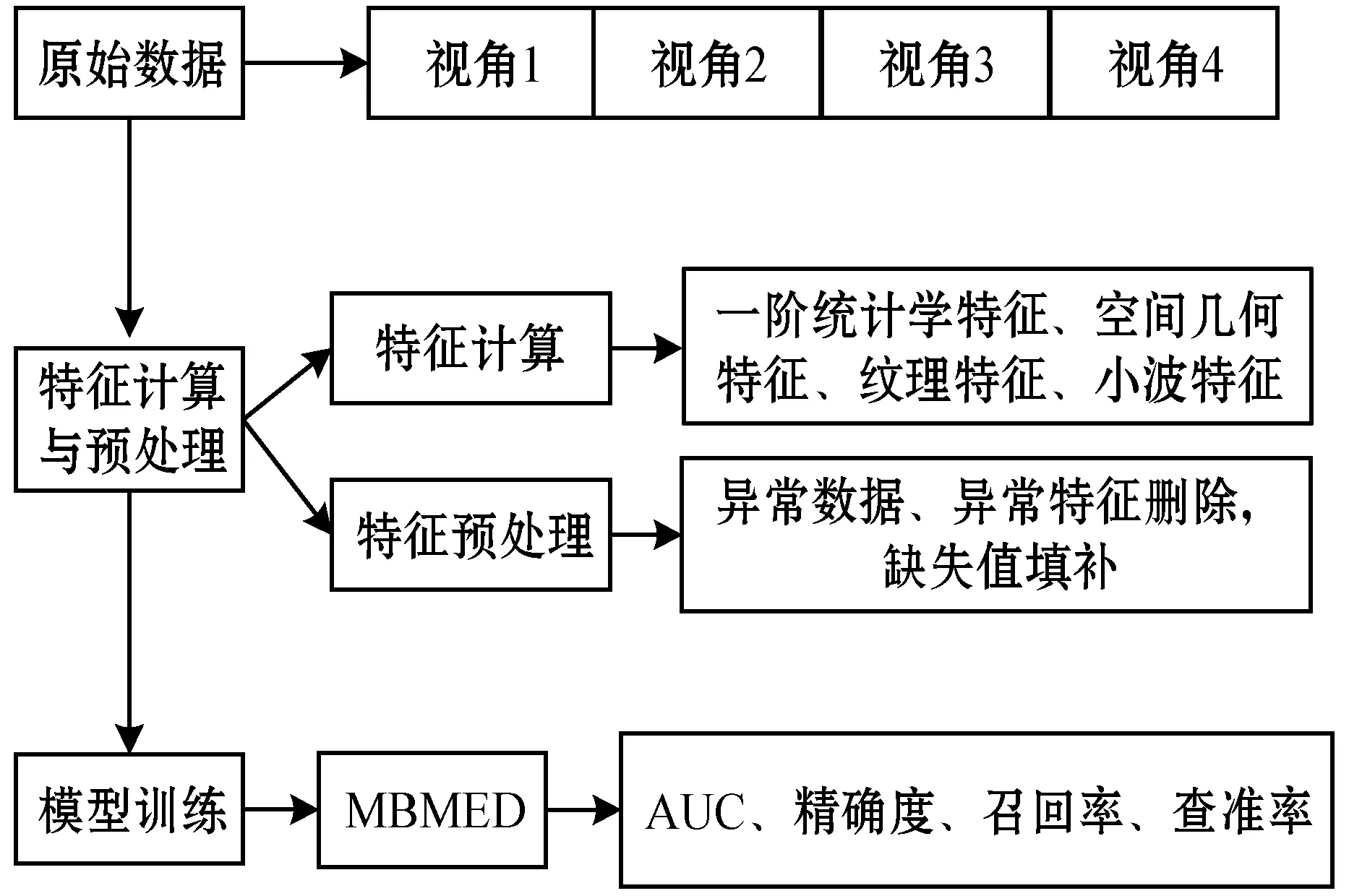
图2 实验流程图
2.1 数据集及实验平台
本文采用的实验数据集来自公开数据集BraTS2017脑胶质瘤数据集和河南省人民医院PACS系统中2012年到2018年的脑胶质瘤数据集(GliomaHPPH2018数据集),具体信息如表1所示。两组数据集均包括脑胶质瘤分级的最常用MRI影像序列,即T1加权成像、T2加权成像、液体衰减反转恢复(FLAIR)成像和增强T1加权成像,其中:T1可以提供脑胶质瘤病灶的解剖信息;T2对水肿区域敏感,可以提供肿瘤边界、水肿程度等信息;CET1可以反映病灶的血流信息;而FLAIR成像中的水分子信号被抑制,强化了边缘外邻近皮层异常信息,有助于区分水肿区域脑脊液[5],它们从不同视角对脑胶质瘤进行了观测和评价。
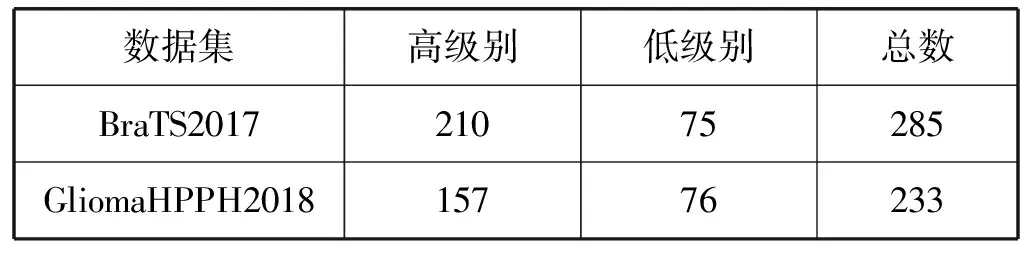
表1 脑胶质瘤数据集
实验平台:处理器为Intel(R) Core(TM) i5- 6500 CPU @ 3.20 GHz,内存为8 GB,采用Microsoft Windows 7操作系统,主要在MATLAB R2015a上完成实验。
2.2 特征工程
针对数据集BraTS2017和GliomaHPPH2018中的四个视角分别计算影像组学特征。本文使用开源数据包Pyradiomics[27]工具进行影像组学特征计算,提取的特征包括一阶统计特征、空间几何特征、纹理特征及小波特征四类,共计558个特征,如表2所示。
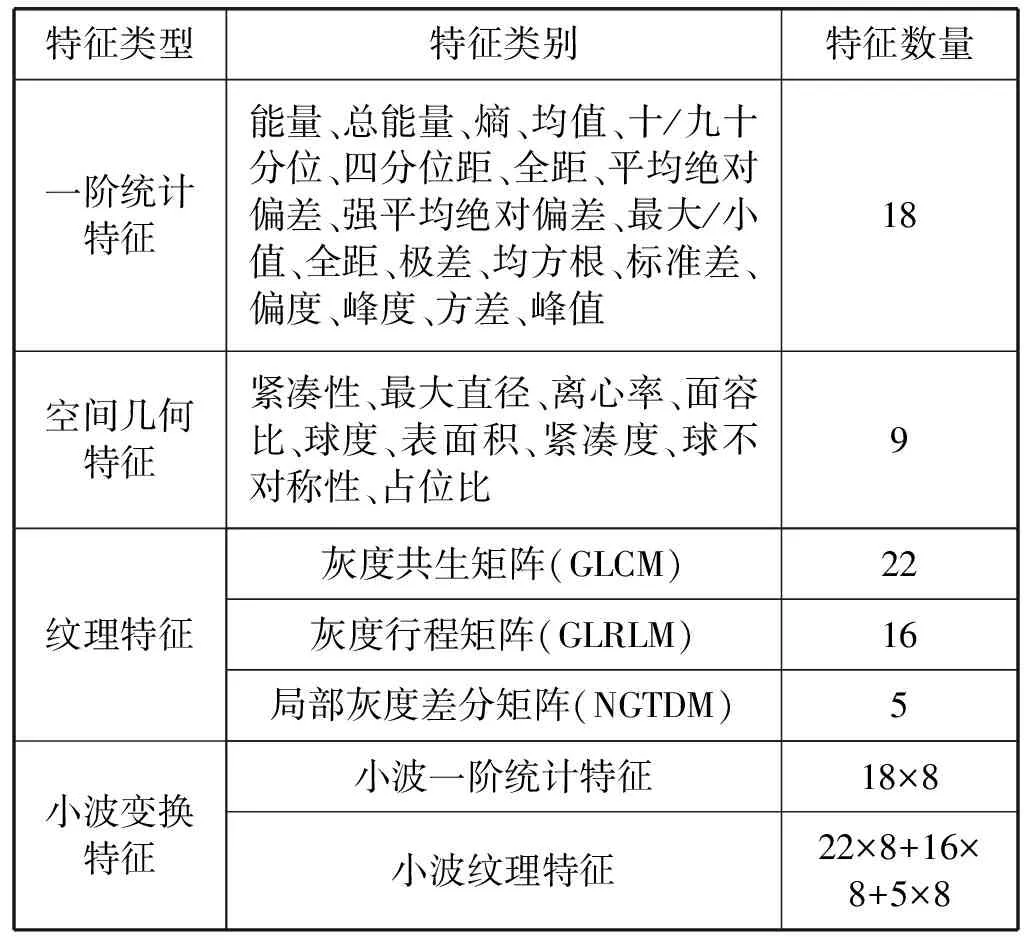
表2 脑胶质瘤提取特征信息
由于提取的脑胶质瘤影像组学特征中存在部分数值缺失或无效,采用以下原则进行缺失值处理:对于大规模特征缺失的样本,通过移除该训练样本进行处理;对于存在大量缺失的特征,对特征评估确认后予以移除;若特征缺失的数量相对较少,采用特征均值进行缺失值填补处理。最终每个视角包含549个特征。
2.3 模型训练
对脑胶质瘤四个视角对应的数据集,采用十折交叉验证进行模型训练,平均结果作为模型的评价指标。训练过程中,首先将脑胶质瘤四个视角的训练集输入到模型中,并对样本权重进行初始化;然后使用MED模型对各个视角进行第一轮训练,得到四个基础分类器,根据错误率分别实现样本及视角的权重更新;其次对上述过程不断迭代(本文取K=50),从而得到权重更新后的基本分类器;最后,将基本分类器按照规则组合得到最终分类器进行脑胶质瘤分级。MED模型训练时需要设置超参数C,本文采用网格搜索法进行优化参数的寻找。前期实验已经证实,C过小时会出现不可分的情况,因此网格搜索范围为20至210,并以log2(C)每次增加1为步长。
2.4 评价指标
为了全面评价模型的分级性能,本文采用ROC曲线下面积(AUC)、精确度(Acc)、召回率(Rec)、查准率(Pre)来对模型进行综合评价。根据数据的实际分类和模型的预测分类构建混淆矩阵,如表3所示。
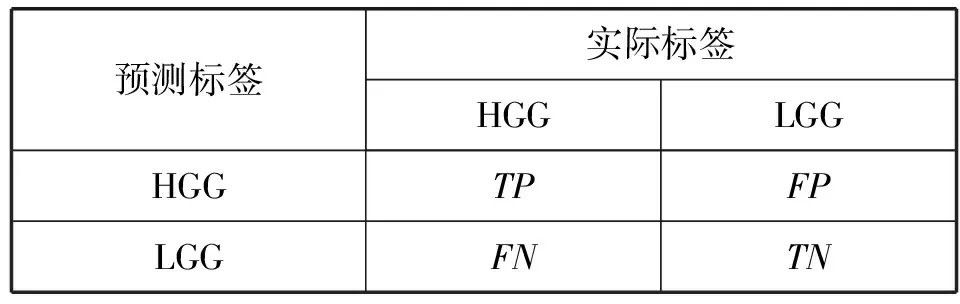
表3 脑胶质瘤分级混淆矩阵
表3中,TP表示实际标签和预测标签都为HGG的样本数,FP表示实际标签为LGG而预测为HGG的样本数,FN表示实际标签为HGG而预测为LGG的样本数,TN表示实际标签和预测标签都是LGG的样本数。评价指标AUC表示以FP为横坐标TP为纵坐标的ROC曲线下面积。评价指标Acc、Rec、Pre的计算公式分别为:
Acc=(TP+TN)/(TP+TN+FP+FN)
(10)
Rec=TP/(TP+FN)
(11)
Pre=TP/(TP+FP)
(12)
2.5 实验结果
对于基本分类器MED模型而言,超参数C一定意义上表明了数据对线性不可分的容忍度,由于当超参数C过小时,模型可能没有有效解;当C大于一定值时,对模型的精度影响较小。因此在模型训练时首先需要对超参数C进行设置,最终通过网格搜索选择一个最优参数值。实验以AUC作为评价指标,选择平均性能最好的超参数C作为最终模型超参数。图3和图4分别展示了BraTS2017数据集和GliomaHPPH2018数据集在不同C值情况下,MBMED模型和其他单视角MED模型的AUC均值变化情况。可以看出多视角学习MEMED模型要优于单视角MED模型,在MBMED模型训练过程中,随着C值增加,相应的AUC值也在增加,当增加到一定值时,AUC略有下降并保持稳定。对于BraTS2017数据集和GliomaHPPH2018数据集,当C=23时,模型平均性能最好。
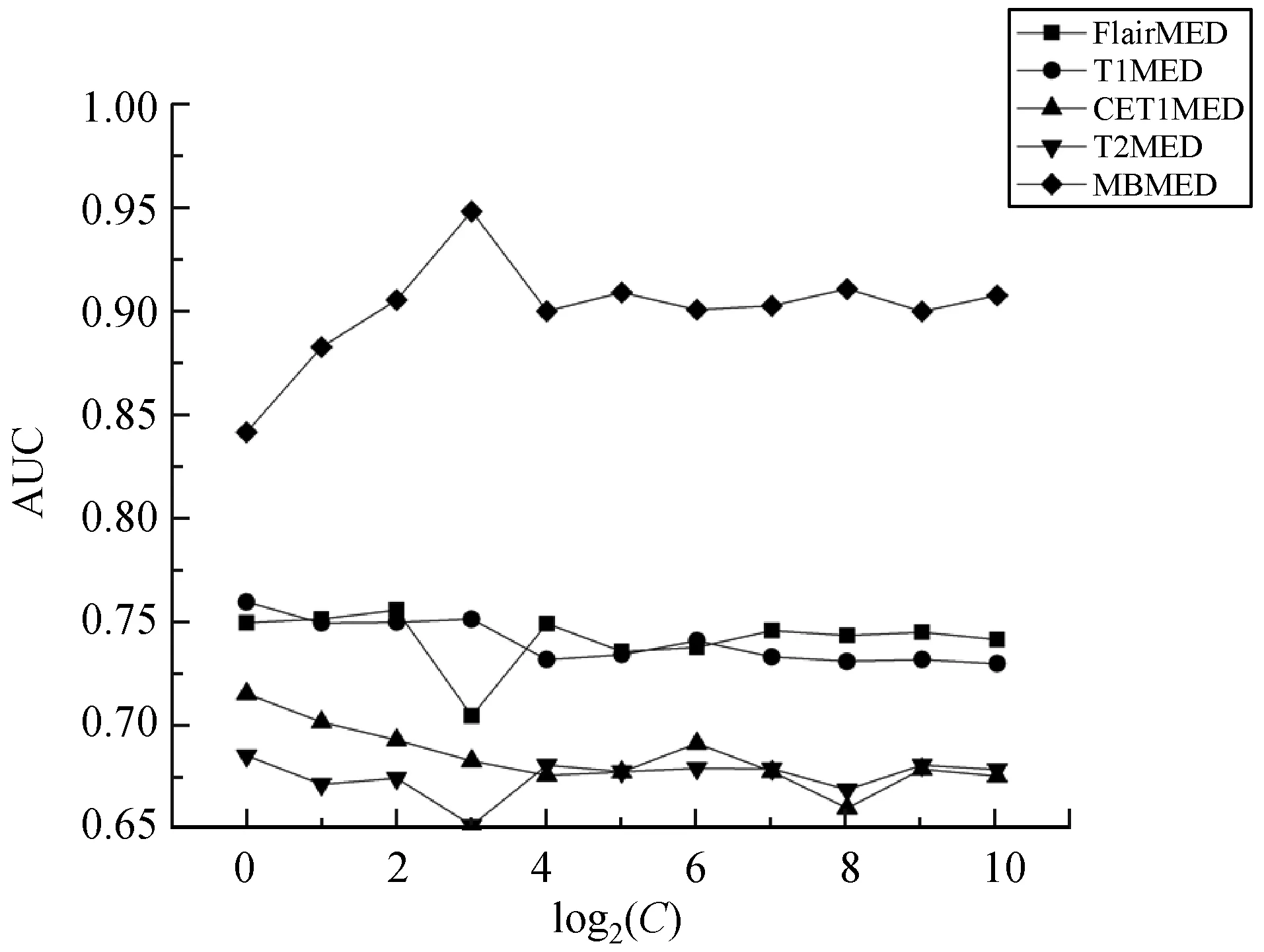
图3 BraTS2017上不同C值的AUC变化图
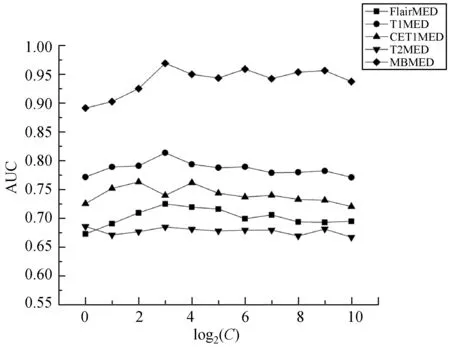
图4 GliomaHPPH2018上不同C值的AUC变化图
BraTS2017和GliomaHPPH2018数据集在最优参数时对应的平均AUC值分别为0.948 5和0.961 2。本文采用了AUC、Acc、Rec和Pre作为模型的评价指标,详细实验结果如表4所示。

表4 与单序列对比实验结果
可以看出,MBMED模型的总体性能指标明显优于使用单视角的MED模型,在BraTS2017和Glioma-HPPH2018上的AUC分别提升了大约20%和15%以上。同时,MBMED在GliomaHPPH2018数据集的整体性能要优于BraTS2017数据集,分析其原因可能是GliomaHPPH2018数据集都来源于同一家医院的PACS系统,高低级别脑胶质瘤所占比例相差较小。而BraTS2017数据集则来源于多个中心,数据规格具有不统一的问题,在预处理过程中可能导致了整体数据的质量下降,其样本中高低级别脑胶质瘤的比例相差较大,数据更加不均衡。
将本文的方法MBMED模型与基于影像组学(Radiomics)的脑胶质瘤分级方法[9,28],以及多视角学习的MVMED[19]、EMV-AdaBoost[21]方法进行对比,采用AUC值作为评价指标,并在两个数据集上进行实验,实验结果如图5所示。
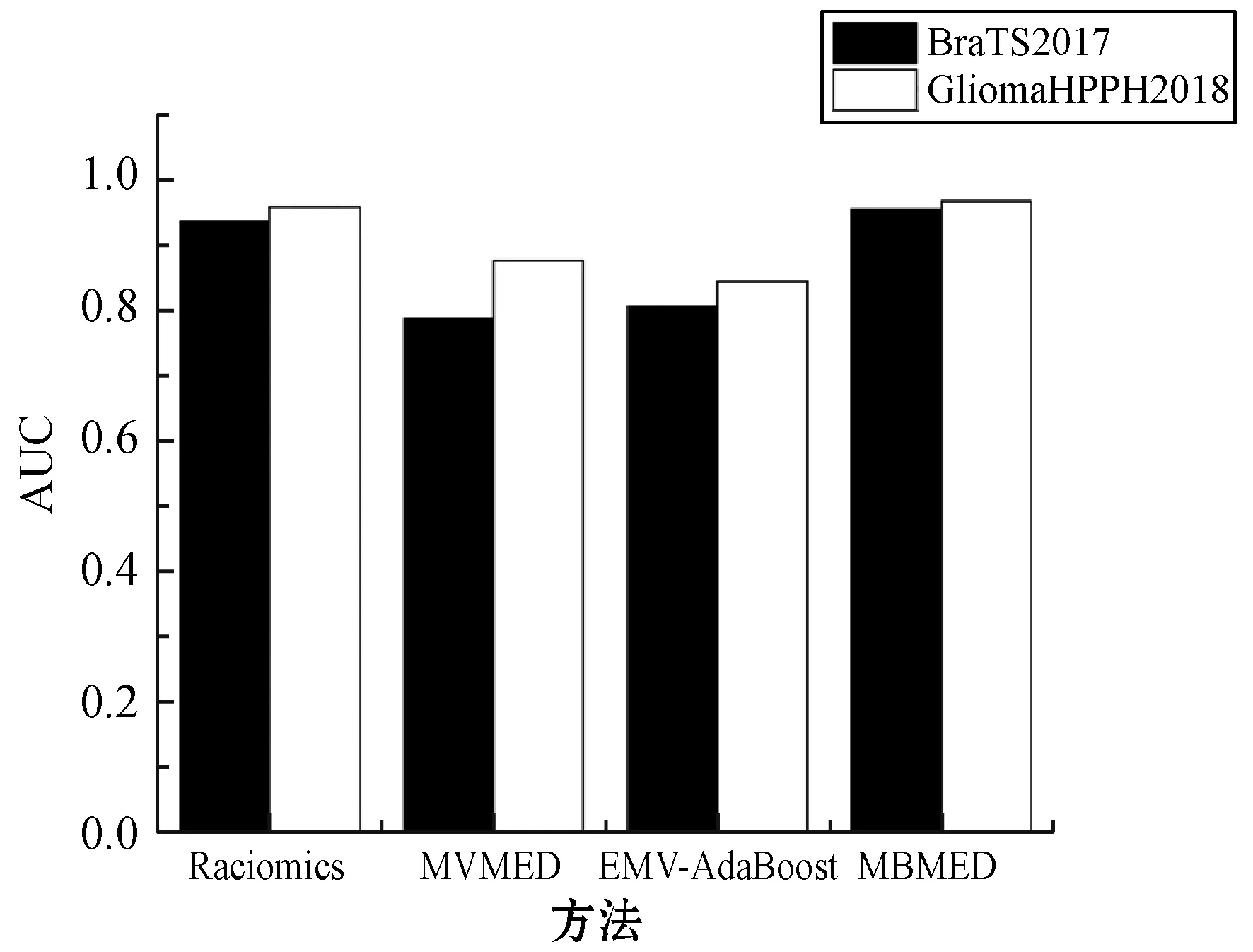
图5 对比实验结果
可以看出,在本实验中,MBMED算法的性能优于另外两种多视角学习方法。同影像组学的脑胶质瘤分级方法相比,本文提出的MBMED模型与影像组学的方法对脑胶质瘤进行分级的结果相当。基于影像组学的方法在脑胶质瘤分级过程中进行了有效的特征选择,而本文提出的MBMED模型在没有进行特征选择的情况下,依然可以取得与影像组学方法相当的结果,这也说明了本文方法在没有数据损失的情况下对于脑胶质瘤分级的临床有效性。本文模型的缺点在于多个视角同时训练时存在噪声问题,有待进一步解决。
3 结 语
为了实现多视角AdaBoost和MED模型结合的优势,本文针对脑胶质瘤MRI影像四个视角,提出了多视角AdaBoost-MED模型(MBMED),实现了脑胶质瘤的精准分级。通过实验对比证明了该模型对于脑胶质瘤分级的有效性。由于多视角影像数据存在噪声影响,下一步工作将研究多视角脑胶质瘤数据的消歧和去噪问题。

