基于DNA条形码技术的连云港基岩岸常见帽贝物种识别
郑 霞,张梦秋,刘 军
(连云港师范高等专科学校,连云港市生物技术重点实验室,江苏连云港 222006)
世界海洋物种目录数据库(World Register of Marine Species,WoRMS)将软体动物门(Mollusca)腹足纲(Gastropoda)物种分为10个亚纲。然而,目前学术界对这些亚纲的分类与该数据库并不完全一致。以其中的亚纲Patellogastropoda为例,该数据库中按“亚纲”来分,而在一些研究中仍按“目”来对待[1-2]。但较为统一的是,学术界将属于Patellogastropoda的物种统称为“真帽贝”(true limpets),共包含4个科,即帽贝科(Patellidae)、花帽贝科(Nacellidae)、笠贝科(Lottiidae)和无鳃笠螺科(Lepetidae)[3]。不属于Patellogastropoda的其他类似真帽贝形态的物种则被称为“假帽贝”[1,4]。
帽贝喜欢附着于坚硬的基底,如岩石、贝壳等。该类物种通常具有类似“帽子”或“斗笠”形状的外壳,其作为腹足纲物种中最原始的种类,具有重要的研究价值[3]。比如,由于其分布广泛(广泛的生存适应性),且兼备取样的易获得性,帽贝已成为学者们研究生物适应性进化史与生物分布变迁史的重要模型生物[5-7]。
此外,随着社会经济的发展,沿海地区环境污染问题日益严重,世界各国对此采取了一系列应对措施。其中,海洋环境污染的生物评价法是了解污染水平的有效手段之一[8]。而帽贝因其自身特点已成为重要的污染评价指示生物[9]。据记载,中国沿海分布的真帽贝有26种,其中,中国大陆沿海的真帽贝物种主要分布于山东、浙江、福建沿海[10]。连云港地处中纬度,东临黄海,具有江苏省唯一的基岩海岸(长约22 km)[11],然而,目前连云港地区帽贝的分布情况尚不清楚[1,12],因此有必要对连云港地区沿岸分布的帽贝种类进行研究。由于同一种帽贝在形态上可能存在较大差异,仅基于形态特征的识别具有一定的不确定性。因此,本研究主要利用DNA分子标记(细胞色素c氧化酶I,COⅠ)对所采集的帽贝进行鉴别[13],以期为连云港沿海帽贝物种的分布研究补充资料,并为今后开展有关无脊椎动物进化研究及环境污染评价等工作提供参考。
1 材料与方法
1.1 样本采集与统计
由于江苏省连云港市的连岛东侧为旅游景区(连岛景区),人为开发程度较高,而连岛西侧基岩岸较为原始,受人为干扰较少,因此本研究以连岛西侧基岩岸作为采样点(图1)。2016年3月—2019年5月,课题组按4个季节(春季:3—5月,夏季:6—8月,秋季:9—11月,冬季:12—2月)分别在连岛西侧的基岩岸进行了帽贝样本的采集,每个季节采集1~2次。采集过程中,主要采用样线法,以潮上带外缘某点为起点,向大海方向呈放射状样线采样。每次采样均从高潮退潮开始采。用游标卡尺测量帽贝贝壳的长、宽、高。利用python 3.8.3的scipy包进行组间形态特征的差异显著性检验(独立样本t检验)。
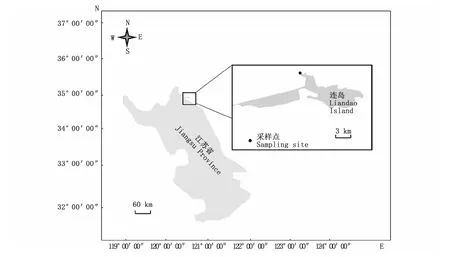
图1 样本采集的地理位置Fig.1 Geographical coordinates of sampling site
1.2 DNA提取及聚合酶链式反应(PCR)
帽贝肌肉组织DNA的提取参照经典的酚氯仿抽提法[8]。物种识别中用到的无脊椎动物线粒体COⅠ基因的通用引物为:上游引物,5′-TAAA CTTCAGGGTGACCAAAAAATCA-3′;下游引物,5′-GGTCAACAAATCATAAAGATATTGG-3′[14]。扩增体系设定为50μL,见表1。

表1 PCR体系配置情况Tab.1 Materials used in PCR
程序设置为:94℃,30 min;94℃,40 s,72℃,1 min(5个循环);94℃,40 s,46℃,40 s,72℃,1 min(30个循环);72℃,10 min;4℃下储存。将PCR产物送至南京擎科生物科技有限公司进行测序。
1.3 物种识别
利用世界海洋物种目录数据库(WoRMS)比对本实验采集的全部样本,并基于帽贝外壳的形状、颜色、大小,挑取形态差异较大的样本进行基于DNA条形码的识别。利用NCBI数据库对COⅠ序列进行比对分析(Blast)。在所有比对到的序列中将相似度≥99%的序列作为目标序列,并采集这些序列进行系统发生分析。系统发生分析采用NJ法,参数选择MEGA软件的默认值[15]。
2 结果与分析
2.1 帽贝的获得情况
本研究在3年内共采集205份帽贝样本。基于帽贝的形态及贝壳颜色将其分为5种类型,分别是X-W-1、X-W-6、X-W-7、X-W-18、X-W-20(图2)。其中,类型X-W-6的帽贝形态与其他帽贝差异较大,表现为其贝壳近乎圆形,而其他帽贝都为卵圆形。类型X-W-1的样本,其贝壳内有一圈白色的环,贝壳边缘分布有很粗的黑白相间的条纹。X-W-7的样本仅发现了2个,明显比其他样本要小(表2)。X-W-20的样本贝壳内部有明显的黑色光泽。从采样情况来看,X-W-20及X-W-1类帽贝较为常见,分别占样本总数的52.68%和28.30%;其次是X-W-18类帽贝,占比为16.59%(表2)。由于X-W-6类型与X-W-7类型的帽贝数量未达到统计学分析的基本要求,因此仅利用独立样本t检验对其他3种类型帽贝进行了两两间的形态差异显著性分析,结果显示,各类型间均不存在显著性差异(P>0.05)。
2.2 DNA提取与PCR扩增
从上述5类帽贝中各选择1~3个个体,均成功提取总DNA。利用通用引物对其进行PCR扩增,成功率均为100%,测序峰图单一,扩增获得了650 bp左右的序列(图3)。
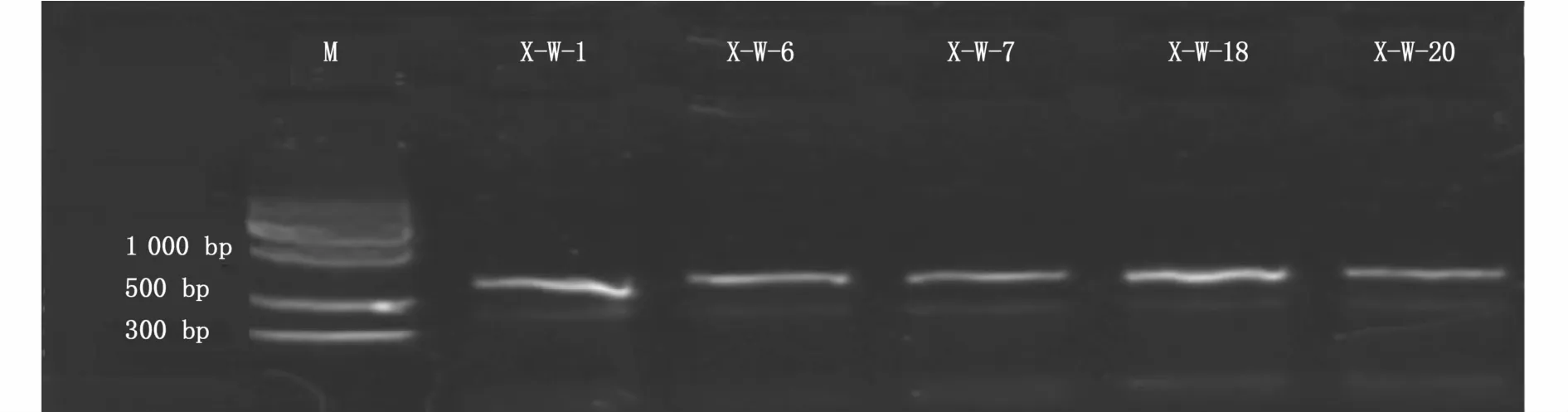
图3 COⅠ基因PCR产物电泳图Fig.3 Prolife of agarose gel electrophoresis for PCR products of COⅠ
2.3 物种识别及系统发生分析
利用COⅠ序列通过数据库进行比对分析(Blast),初步识别出这些帽贝隶属于4个属,共5个种,其中4种为“真帽贝”,分别是圆锥拟帽贝(Patelloidaconulus)、史氏背尖贝(Nipponacmeaschrenckii)、齿舌新笠贝(Nipponacmearadula)、笠贝(Lottiagoshimai),1种是“假帽贝”,为日本菊花螺(Siphonariajaponica)(表3)。与以往研究相比,除日本菊花螺与笠贝在连云港海岸未见报道外,其余3种均已有报道(表3)[1,12]。本研究结果为江苏连云港沿海海域帽贝的物种多样性提供了新的记录。
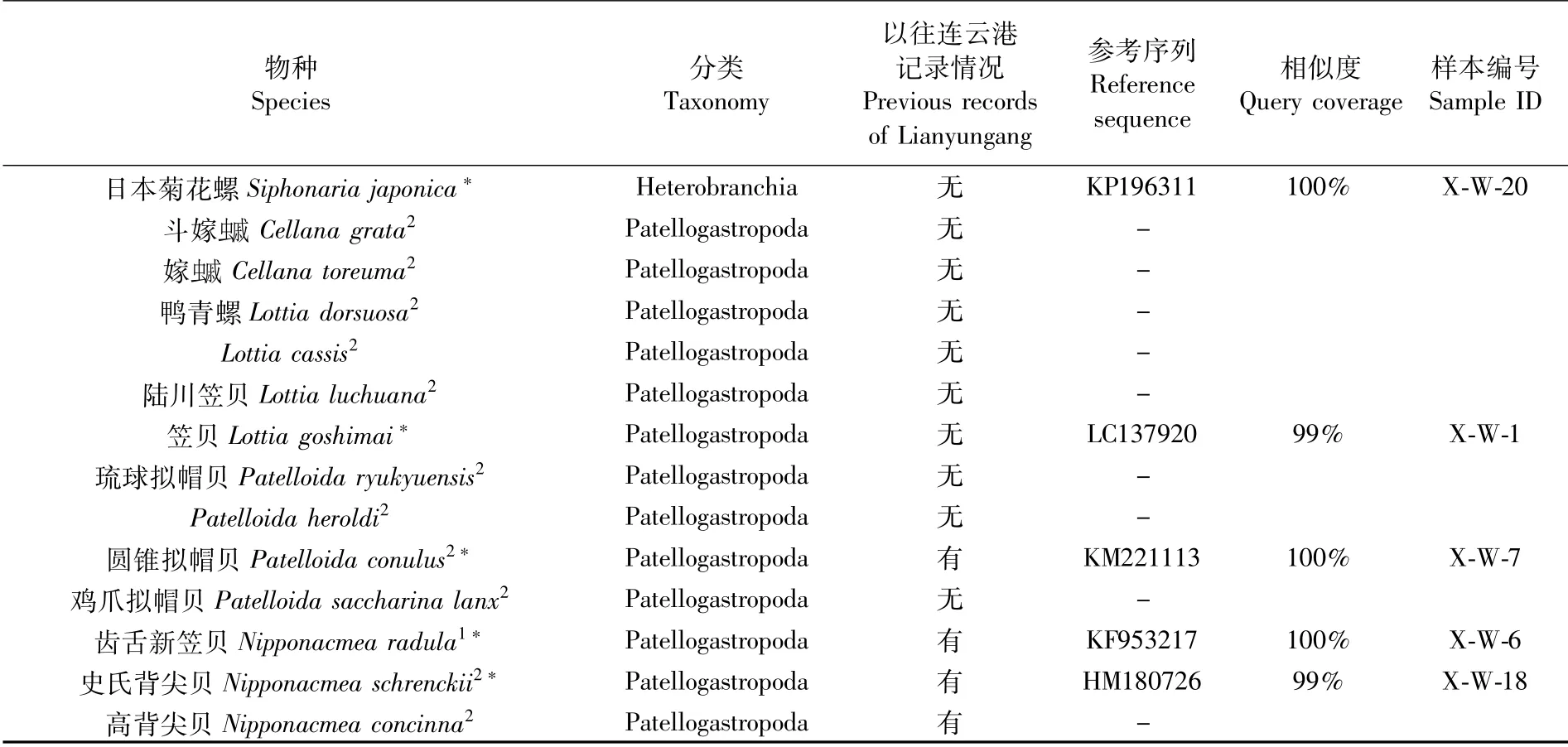
表3 帽贝的COⅠ序列识别结果Tab.3 Results of species identification by COⅠbarcoding
基于Blast结果,利用这5个种的代表序列(样品编号为X-W-1、X-W-6、X-W-7、X-W-18,XW-20),结合NCBI中25条COⅠ基因序列(每个种各5条序列),构成一个系统发生分析数据集(图4),以此进一步佐证物种识别的结果。从系统发生关系来看,本次识别的所有“真帽贝”均来自笠贝科,“假帽贝”来自松螺科(Siphonariidae)。Bootstrap计算结果显示,齿舌新笠贝(X-W-6)、笠贝(X-W-1)、日本菊花螺(X-W-20)的识别有着较高的可信度(图4)。同时,本研究基于世界海洋物种目录数据库还进行了图片检索,结果发现,X-W-20的外部形态与该数据库中的日本菊花螺有着较高的相似性(图1,图5)。此外,X-W-1的外部形态与NAKAYAMA等[16]报道的笠贝具有较高的相似性(图1,图5),该物种于2017年首次被日本学者命名。研究还发现,X-W-6的外部形态与SASAKI和OKUTANI[17]报道的齿舌新笠贝有着较高的相似性(图1,图5)。
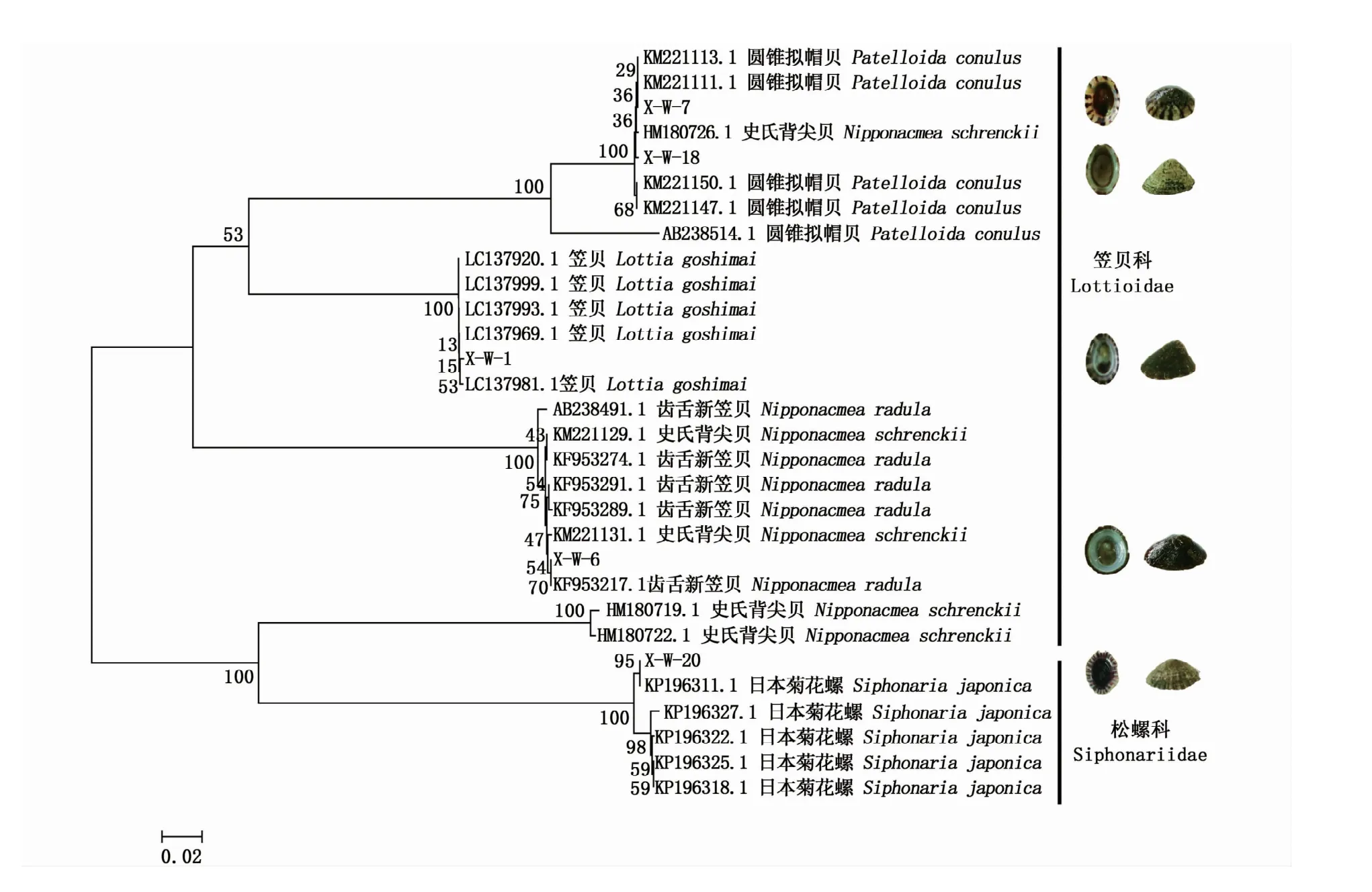
图4 基于COⅠ基因的系统发生分析Fig.4 Phylogenic analysis by COⅠgenes
然而,系统发生结果显示,X-W-7和X-W-18混杂于包含圆锥拟帽贝与史氏背尖贝的支系中(图4)。对于此种现象,本研究比较了X-W-7、XW-18与数据库中的史氏背尖贝。通过图片比较发现,这二者在形态上与数据库中的史氏背尖贝差异都较大(图1,图5),基本上可排除样本XW-7和X-W-18为史氏背尖贝的可能性。结果支持这两种类型的帽贝样本更可能是圆锥拟帽贝。

图5 WoRMS及文献中的帽贝参考图Fig.5 Limpet photos from WoRMSand references
3 讨论
以往基于帽贝贝壳形态的物种识别,存在的主要问题是贝壳形态易受到不同生境及年龄差异的影响,从而增加了识别工作的难度[3]。本研究比较了不同种帽贝间贝壳的长、宽、高指标,结果显示,利用帽贝的此类形态指标进行物种识别具有较大的局限性(组间无显著性差异,P>0.05)。近期,类似研究也提示了相似的现象,比如,中国沿海的同一种的13个矮拟帽贝(Patelloidapygmaea)地理群体间的长、宽、高指标已具有一定程度的差异(部分地理群体的这些指标均值间甚至有2~3倍的差异)[18]。因此,仅利用这些形态指标区别不同物种,得到的结果并不一定准确。
本研究统计样本数时发现,日本菊花螺为优势种,而齿舌新笠贝的数量较少(仅3个),其原因除了与影响其种群变动的因素有关以外[19],还可能与其生境的特征有关。据观察,本次研究中齿舌新笠贝附着在潮湿、阴暗的石缝中,增加了样本获取的难度。ITSUKUSHIMA等[20]也曾报道过齿舌新笠贝的生境喜好,认为该物种偏向于沙质的生境。这种生境特征可能是导致该物种在本次实验中获得量较少的主要原因之一。当然,这还需要在后期的研究中进一步验证。此外,虽然潮间带环境特殊,但帽贝物种已表现出了很好的适应性。温度常被认为是影响物种地理分布范围边界的重要环境变量,而海洋潮间带物种由于有着近乎线性的地理分布模式,已成为生物地理学研究的模式生物[21]。本次实验的主要目的为识别帽贝物种,今后还可进一步开展帽贝分布情况受温度因素影响的研究,并设计更具有针对性的实验方案。
目前,DNA条形码已被越来越多地用于获取未经种类鉴定生物的分类学信息[22]。该技术涉及到对线粒体细胞色素c氧化酶Ⅰ(COⅠ)基因的一个短片段进行测序,即从分类学上尚未鉴定种类的标本中提取DNA条形码,并与已知分类的DNA条形码库进行比对。形态鉴定结合分子生物学手段能增加物种识别的准确性。如今,已被用于物种识别的线粒体基因技术还有12/16S rRNA和Cytb基因等[23]。线粒体16SrRNA序列在帽贝物种识别及种间关系的研究中都有广泛的应用[15]。为了进一步验证X-W-7和X-W-18并非史氏背尖贝,本研究通过扩增这两个样本的线粒体16S rRNA序列后发现,序列相似度最高的前3条序列均为圆锥拟帽贝,且相似度均为96%①源自本实验室未发表数据。由于拟帽贝属内同一物种的形态和生态类型本身差异较大,因此完全基于形态来识别该属内的近源种具有一定的难度[15]。圆锥拟帽贝为矮拟帽贝的近源种,其壳高相对其他的拟帽贝属物种要高,形状近锥形[15]。圆锥拟帽贝和P.heroldi都曾被看作矮拟帽贝的同种异名[15,17],NAKANO和OZAWA[15]利用COⅠ与16SrRNA分子工具成功将这3个种分离。因此,本研究利用16SrRNA得到的补充识别结果具有一定的可靠性。综合来看,基本可以断定X-W-7和X-W-18都应为圆锥拟帽贝。
4 小结
本研究借助DNA条形码以及帽贝物种形态特征的对比,成功识别出了4种帽贝(日本菊花螺、笠贝、齿舌新笠贝、圆锥拟帽贝)。其中,物种日本菊花螺与笠贝为江苏连云港沿海首次报道种。研究结果为了解连云港地区帽贝多样性提供了必要的资料。由于样本采集具有一定的难度(天气、潮汐、生境等因素都会影响采样结果),导致本次研究存在一定的不足之处。比如本应该采集到的矮拟帽贝却未被采到[18]。连云港基岩海岸帽贝物种的多样性及分布情况仍有必要进行进一步调查。
致谢:感谢参与实验调查的苏丽、魏媛媛、李凡、王井之等同学。

