基于线粒体DNA控制区的黄姑鱼养殖群体与野生群体比较研究❋
赵 祥, 郑 建, 高天翔, 宋 娜❋❋
(1.海水养殖教育部重点实验室(中国海洋大学), 山东 青岛 266003; 2. 浙江海洋大学水产学院, 浙江 舟山 316022)
黄姑鱼(Nibeaalbiflora)隶属于鲈形目(Perciforms)石首鱼科(Sciaenidae)黄姑鱼属(Nibea),别名铜罗鱼、黄婆鸡、春仔(鱼)、黄姑子等。黄姑鱼主要栖息于近海中下层水域,属于暖水性鱼类,主要摄食底栖动物[1]。黄姑鱼分布较广,在中国、朝鲜半岛和日本南部海区均有分布,是传统捕捞生产中非常重要的捕捞对象,也是我国重要的经济鱼类之一[2-3]。近年来,由于受到过度捕捞、海洋污染及气候变化等因素的影响,野生黄姑鱼资源量急剧下降[4]。目前中国多地已经开展黄姑鱼人工育苗与网箱养殖[5-6],因其不易受到养殖病害的影响[7],已成为海水养殖经济鱼类中极具潜力的品种。黄姑鱼人工养殖工作的开展极大地缓解了野生资源的枯竭状况。
目前关于黄姑鱼育苗与养殖方面的研究较多[8-14],而群体遗传学方面研究相对较少。韩志强在2006年[15]与2008年[16]先后利用AFLP和线粒体DNA控制区两种标记方法对黄姑鱼群体遗传多样性进行了分析,结果显示群体间存在着较强的基因交流。徐冬冬在2012年[17]与2017年[18]先后利用线粒体DNA COI基因和微卫星标记对4个野生黄姑鱼群体进行比较分析,结果显示野生黄姑鱼群体遗传多样性水平较高,不同群体间遗传差异较小。目前,国内多个省市已经开展黄姑鱼的增殖放流工作[19-20]。养殖群体的遗传多样性现状不仅涉及到养殖品种的种质资源,结合增殖放流工作的开展更会对野生群体产生一定的影响。因此,养殖群体和野生群体的遗传多样性监测工作对了解黄姑鱼种质资源现状及人工养殖发展趋势具有十分重要的作用,对其增殖放流工作的开展也有重要意义。
线粒体DNA(mtDNA)结构较为简单,大多数呈双链环状结构。遵循母系遗传且不易发生重组,具有可溯源性。突变速率快且突变率高,具有一定的多态性。可以作为一种有效的分子标记用于群体遗传分析[21-23],因此在鱼类遗传多样性检测中被广泛采用[24-26]。例如,肖志忠等[27]利用线粒体DNA控制区序列对来自青岛胶南的条石鲷野生与养殖群体进行比较分析,发现条石鲷养殖群体与野生群体之间产生了较为显著的遗传分化。Iguchi等[28]基于线粒体DNA控制区序列对日本香鱼野生与养殖群体进行比较分析,结果表明日本香鱼养殖群体较野生群体遗传多样性水平显著降低。本研究基于线粒体DNA控制区高变区部分序列对黄姑鱼养殖群体和野生群体进行遗传学比较,明确黄姑鱼群体遗传多样性水平与遗传分化程度,评估目前黄姑鱼养殖工作中存在的弊端,以期为黄姑鱼种质资源保护、人工养殖以及增殖放流等提供基础参考资料。
1 材料与方法
1.1 样品采集
本研究中2个养殖群体分别采自青岛国家海洋科学研究中心和宁德市横屿岛水产有限公司(亲鱼分别来自青岛和厦门近海),2个野生群体分别采自乳山和舟山近海。此外从NCBI数据库中下载3个野生群体[15](青岛、舟山、厦门)的序列进行比较分析。具体样品信息以及NCBI登录号见表1。所有采集的样品经过形态学鉴定后取背部肌肉,置于95%酒精中保存备用。
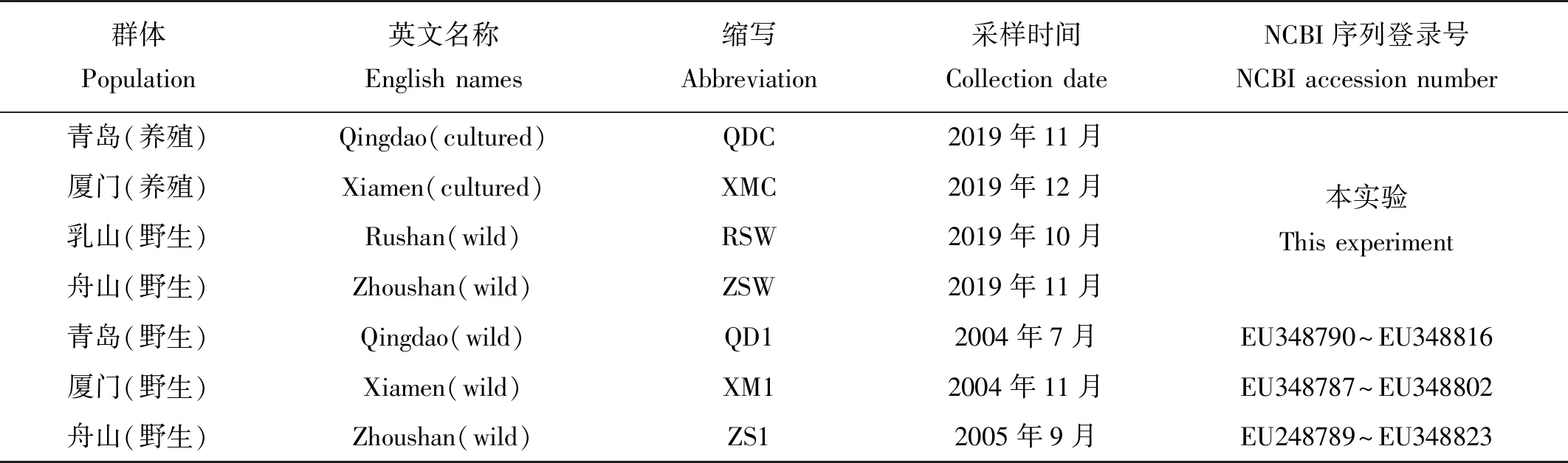
表1 黄姑鱼样品信息及NCBI登录号统计表
1.2 DNA提取及序列扩增
取黄姑鱼样品肌肉组织,经蛋白酶K消化后采用标准酚-氯仿方法提取样品DNA并对线粒体DNA控制区高变区序列进行PCR扩增。扩增所用引物序列根据黄姑鱼线粒体DNA全基因组序列(NCBI登录号: NC_015205),利用Primer Premier6软件[29]设计。正反引物序列分别为dl~h1:TTCTTCCTACTGCCTAACTC;dl~h2:CGCTTATGTATTACCACCAT。PCR反应体系共25 μL,其中包括17.25 μL超纯水、2.5 μL 10×PCR扩增缓冲液、2 μL dNTPs(200 μmol/L)、0.25 μLTaqDNA聚合酶、正反向引物及模板DNA各1 μL。反应条件为95 ℃预变性5 min,94 ℃变性45 s,52 ℃退火1 min,72 ℃延伸1 min,共35个循环,72 ℃延伸10 min。扩增完毕后取2 μL扩增产物进行琼脂糖凝胶电泳检测(U=5 V/cm)后送至擎科生物科技有限公司青岛分公司在ABI3730XL测序仪上使用Sanger法(双脱氧末端终止法)进行双向测序。
1.3 数据分析
测序所得的所有序列利用DNA Star(DNA Star, Inc. Madison, USA)软件包进行序列比对,核对测序胶图并进行人工矫正;利用Arlequin软件[30-31]统计序列碱基含量百分比、单倍型数目、多态位点数目、转换/颠换值、单倍型多样度、核苷酸多样度等。根据单倍型频率分布构建黄姑鱼单倍型网络图。Tajima’D和Fu’sFs中性检验以及核苷酸不配对分布使用Arlequin软件进行分析。采用Arlequin软件计算群体间确切P值和遗传分化指数FST,通过10 000次重抽样检验FST值的显著性。将黄姑鱼DNA控制区序列分为养殖群体(QDC,XMC)和野生群体(ZSW,RSW,QD1,XM1,ZS1)两个组群,并进行AMOVA分析,来检验组群之间的遗传差异。
2 结果
2.1 基于控制区序列片段的群体遗传多样性分析
本研究采集的黄姑鱼4个群体扩增所得到的序列片段长度为425 bp。从NCBI数据库下载3个群体(青岛、舟山、厦门)的线粒体DNA控制区序列,截取425 bp同源片段进行比较分析。本研究中所采集的2个野生群体与2个养殖群体线粒体DNA控制区序列碱基含量差别不大,其中A+T含量(65.29%、 65.69%)均明显大于C+G含量(34.71%、34.31%)。
173个黄姑鱼个体共检测到35个多态位点,定义了62个单倍型(见表2)。养殖群体单倍型数目较少,仅有7个。野生群体与养殖群体之间有1个共享单倍型。2个养殖群体共检测到9个多态位点,1处插入/缺失,转换位点有8个,颠换位点有1个,转换颠换比为8∶1。5个野生群体共检测到35个多态位点,1处插入/缺失,转换位点有32个,颠换位点8个,转换颠换比为4∶1。本研究中的2个野生群体单倍型多样度和核苷酸多样度与NCBI数据库中引用的3个野生群体相似。养殖群体单倍型多样度为0.416 0±0.094 7~0.712 3±0.057 4,明显低于野生群体单倍型多样度(0.913 0±0.030 8~0.992 6±0.023 0)。而养殖群体核苷酸多样度(0.006 1±0.003 7~0.006 4±0.003 9)略低于野生群体核苷酸多样度(0.005 4±0.003 4~0.010 9±0.006 3)(见表3)。综合来看,野生群体遗传多样性指数较高。
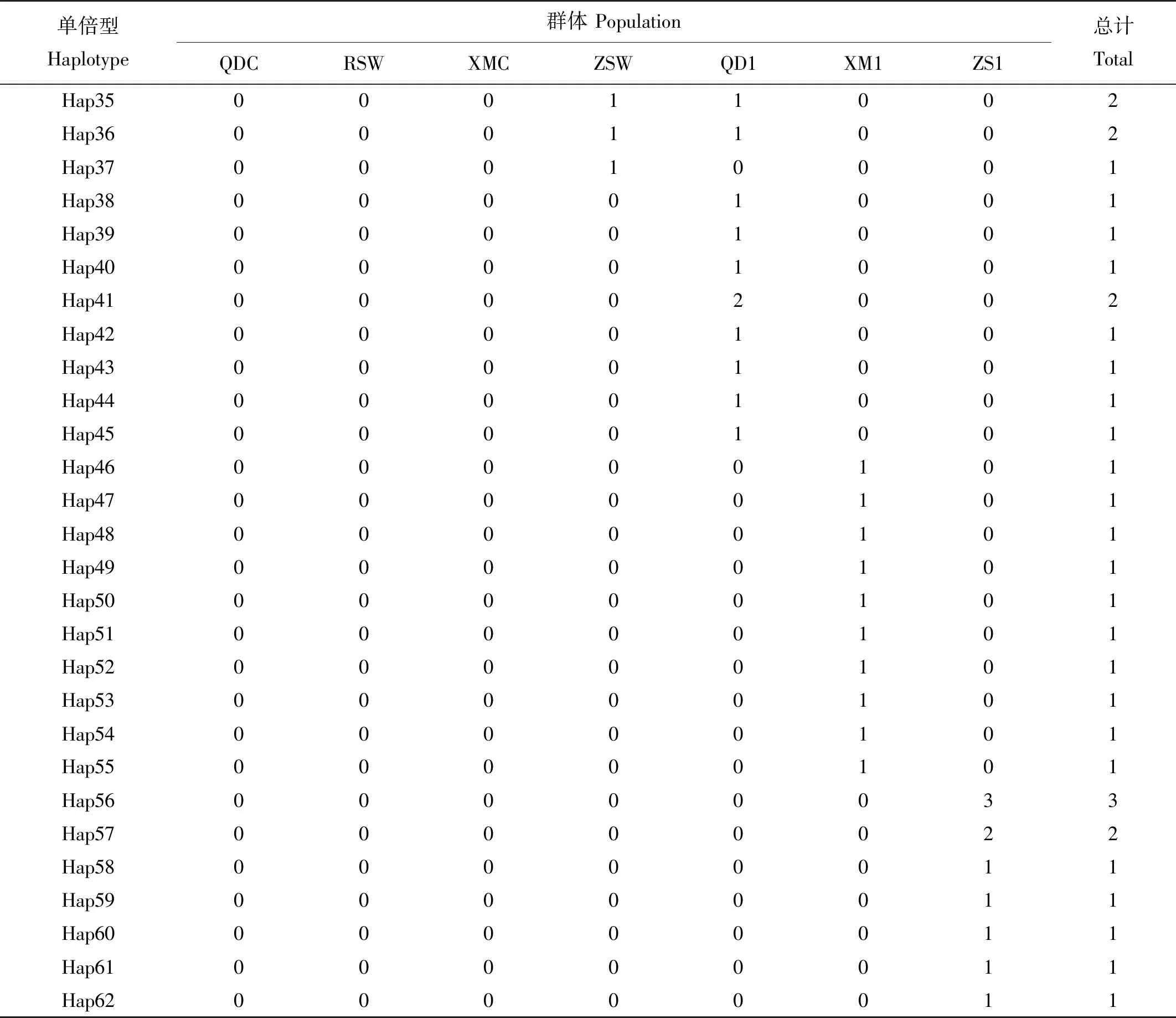
续表2
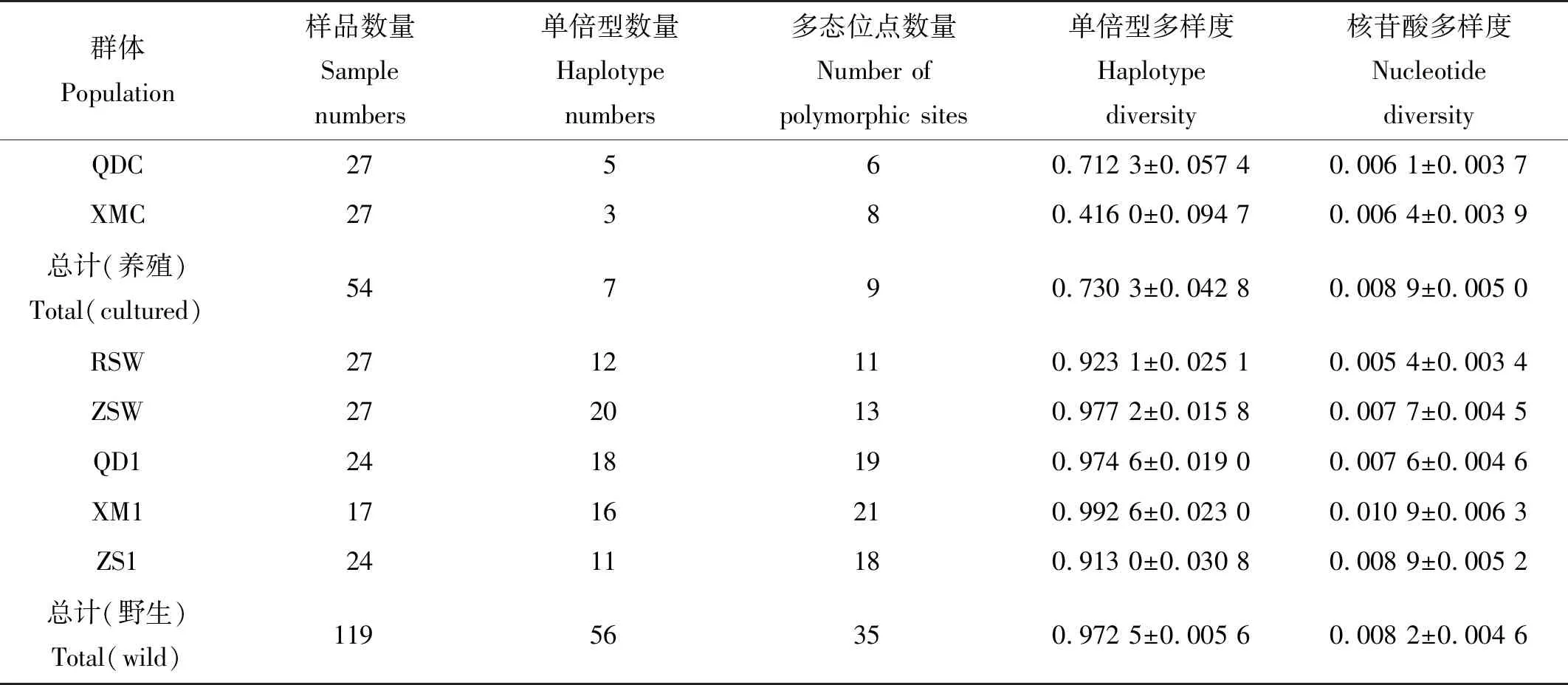
表3 黄姑鱼群体遗传多样性指数
2.2 基于控制区片段的群体遗传结构及系统发育分析
基于线粒体DNA控制区单倍型构建的网络图结构简单,没有发现明显的谱系结构(见图1)。群体间确切P检验显示,本研究中2个养殖群体间以及养殖群体与野生群体间的P值显著(P<0.05)。群体遗传分化指数分析结果显示,黄姑鱼2个养殖群体之间以及养殖群体与野生群体之间遗传分化指数均较大,各FST值均差异显著(P<0.05)。本研究中采集的乳山野生群体与其他野生群体也存在一定程度的遗传分化,但养殖群体之间、养殖群体与野生群体之间的遗传分化大于野生群体之间遗传分化(见表4)。

图1 基于黄姑鱼线粒体DNA控制区序列构建的单倍型网络图
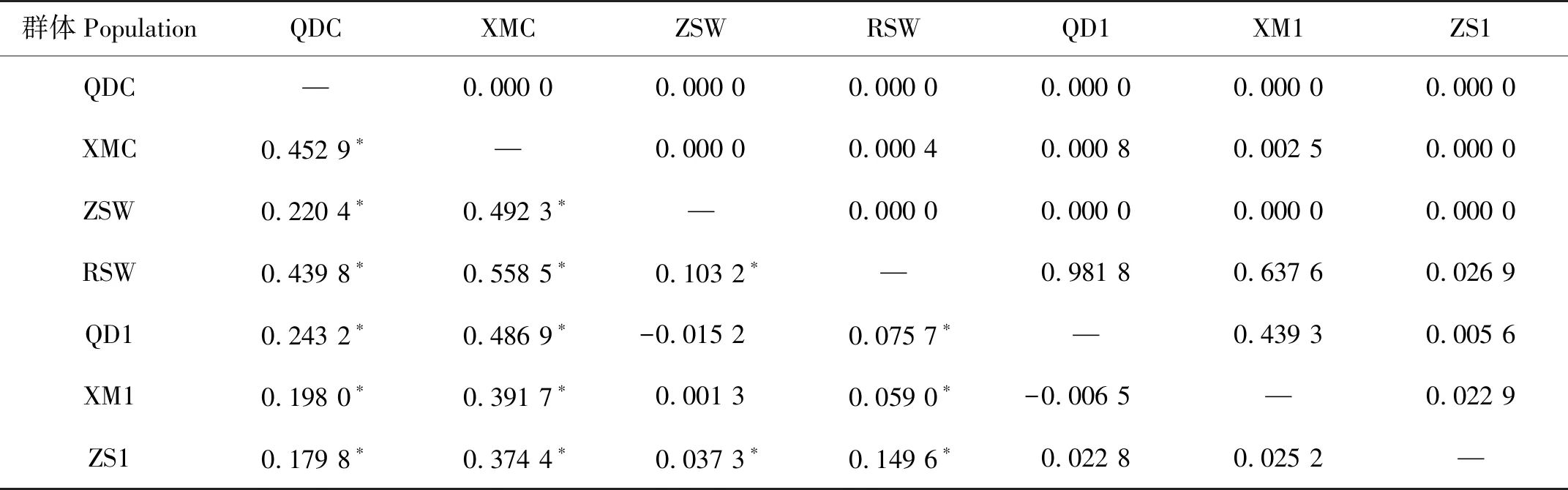
表4 黄姑鱼群体间FST值(下三角数据)以及确切P检验结果(上三角数据)
AMOVA分析结果表明群体内部遗传分化占大部分(65.57%)。组群之间以及组群内群体之间遗传分化占比较小(21.36%、13.07%),组群之间没有显著的遗传分化(FCT=0.213 6,P>0.05)。黄姑鱼野生群体核苷酸不配对分布图呈显著单峰现象(见图2),拟合度检验显示,观测值与模拟值基本吻合(Hri=0.029 8,P>0.05),没有显著偏离群体扩张模型。中性检验显示:Tajima’D值(Tajima’D=-1.466,P>0.05)与Fu’sFs值(Fu’sFs=-26.145,P=0.00)均为负值,根据中性检验原理,可以作为黄姑鱼野生群体扩张的证据。
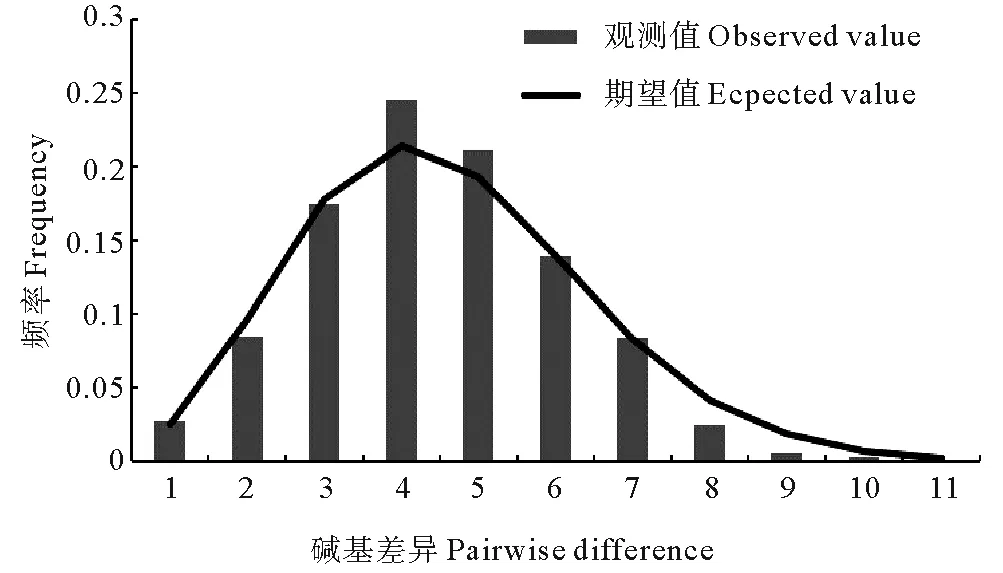
图2 黄姑鱼野生群体核苷酸不配对分布图
3 讨论
遗传多样性作为生物进化的原始材料,是一个物种或群体长期进化过程中的产物。群体遗传多样性研究有助于揭示物种的起源、变异和进化过程,亦可以解释物种濒危原因及过程[32-33]。一般来说,物种遗传多样性水平较高有助于提高物种的环境适应能力、生存能力和进化潜能;遗传多样性贫乏则会导致物种适应和生存能力下降,优良性状丧失,甚至导致物种灭绝[34]。鱼类作为人类赖以生存的自然资源之一,许多种类已经开展人工繁育,但不合理繁育方式可能是导致养殖群体遗传多样性降低的重要原因之一。
3.1 黄姑鱼养殖群体与野生群体遗传多样性分析
以往关于黄姑鱼遗传多样性的研究大多基于野生群体,例如,郭垚示[35]基于线粒体DNA COI序列对我国近海黄姑鱼遗传多样性分析结果表明,野生黄姑鱼单倍型多样度较高但核苷酸多样度较低(h=0.697 0±0.002 2;π=0.001 7±0.000 2);韩志强等[16]通过线粒体DNA控制区分析东黄海黄姑鱼群体遗传多样性时也得到了相似结论(h=0.967 8±0.010 2;π=0.008 1±0.004 6)。本研究中也检测到野生黄姑鱼较高的单倍型多样度与较低的核苷酸多样度。
在黄姑鱼线粒体DNA控制区序列片段中,A+T含量较高,且养殖群体与野生群体差异较小,与其他海水鱼类线粒体DNA控制区片段碱基含量组成相似[36-37]。本研究中黄姑鱼养殖群体单倍型多样度明显低于野生群体,且养殖群体多态位点及单倍型数目较少,遗传多样性水平明显低于野生群体。龚诗琦[38]利用微卫星位点对黄姑鱼野生与养殖群体遗传多样性比较结果表明养殖群体遗传多样性水平低于野生群体。我们推测人工繁育时亲鱼数量少、近亲繁殖、以及遗传漂变和瓶颈效应等可能是导致黄姑鱼养殖群体遗传多样性降低的原因[39-40]。养殖群体遗传多样性低于野生群体的现象在其他鱼类研究中也有报道,宋娜等[41]对香鱼野生与养殖群体的线粒体DNA控制区序列进行比较分析发现,养殖群体单倍型多样度与核苷酸多样度均低于野生群体。由黄姑鱼群体单倍型分布表可以看出:本研究中2个养殖群体单倍型数目均较少,单倍型多样度低。结合线粒体DNA遵循母系遗传的特点,我们推测可能是人工繁育时野生亲鱼数目较少所导致的。
3.2 黄姑鱼养殖群体与野生群体遗传结构与历史动态分析
群体之间遗传分化指数FST结果显示,部分野生群体之间存在遗传分化,但其遗传分化程度较小。韩志强等[15]对青岛和厦门黄姑鱼野生群体的AFLP分析结果显示,两野生群体之间遗传结构相似且不存在显著的遗传分化。值得注意的是,本研究中乳山野生群体与其他野生群体均出现显著的遗传分化。但由于本研究中样品数量较少,且先前的研究中也未见关于乳山黄姑鱼群体与其他群体的比较研究,我们期待在后续的研究中采集更多的样品和使用更加灵敏的分子标记来解释这种现象。群体间确切P检验显示,2个养殖群体间以及养殖群体与野生群体间的P值显著(P<0.05),表明其并非随机交配群体。值得注意的是,本研究中所采集的舟山群体与其他野生群体之间均不是随机交配群体(P=0.00),推测其原因可能是舟山近海地形较为复杂,岛屿众多[43],导致舟山群体与其他海域群体基因交流较少。另一方面,本研究中采集的舟山群体与NCBI数据库中引用的舟山群体采样时间相隔较远可能是导致我们并未检测到两群体之间基因交流的原因。
养殖群体间以及养殖群体与野生群体间均存在显著的遗传分化,但AMOVA分析结果却显示,黄姑鱼两大组群之间并没有显著的遗传分化。丁奎等[44]关于许氏平鲉养殖与野生群体比较的分析中也出现了相似结果,推测可能是2个养殖群体之间亦存在较大的遗传分化,在将其定义为一个组群与野生组群进行AMOVA分析时可能会对结果产生影响。Song等[45]在基于线粒体控制区序列对褐牙鲆养殖与野生群体遗传多样性的比较研究中,同样检测到褐牙鲆养殖群体之间以及养殖群体与野生群体之间存在显著的遗传分化。这一结果暗示我们目前鱼类养殖过程中可能会因为遗传多样性的降低而与野生群体产生较大的遗传分化,在进行增殖放流应注意监测其是否会对野生种群的遗传多样性产生影响。
黄姑鱼野生群体核苷酸不配对分布图呈现明显的单峰状态,Tajima’D值与Fu’sFs值均为负值,支持群体扩张假说。这一结果与郭垚示[35]关于中国近海野生黄姑鱼群体中性检验结果相似,暗示黄姑鱼群体可能由于冰期影响而产生群体扩张。野生群体单倍型多样度较高而核苷酸多样度较低,暗示野生黄姑鱼群体可能是从一个较小的群体经历瓶颈效应后急剧扩张。
研究显示,与黄姑鱼野生群体相比养殖群体的遗传多样性明显降低。在今后的人工繁育工作中,应尽量保证野生黄姑鱼亲本数量足够多,选择遗传多样性丰富的野生黄姑鱼群体作为亲本进行繁育。目前我国多个省市开展黄姑鱼增殖放流工作,且已有关于黄姑鱼增殖放流的报道。放流苗种的遗传多样性状况对于苗种本身以及野生群体种质资源都有影响,因此必须要开展黄姑鱼遗传多样性监测工作。保证用于增殖放流的黄姑鱼群体具有较高的遗传多样性水平,减轻增殖放流对野生黄姑鱼遗传多样性的影响,从而保护优质黄姑鱼种质资源,实现资源可持续利用。

