DNA甲基转移酶1与SLE疾病活动指数的相关性:Meta分析
李子锋 付远芝 周晓静 扶晓兰 杨路焕 王 楚 常彩云
(1. 三峡大学 第一临床医学院[宜昌市中心人民医院] 中医科, 湖北 宜昌 443003; 2. 三峡大学 第一临床医学院[宜昌市中心人民医院] 胸心外科, 湖北 宜昌 443003; 3. 三峡大学 第一临床医学院[宜昌市中心人民医院] 儿科, 湖北 宜昌 443003; 4. 三峡大学 第一临床医学院[宜昌市中心人民医院] 急诊科, 湖北 宜昌 443003; 5. 中国康复研究中心 北京博爱医院 脊柱骨髓神经功能重建科, 北京 100068)
系统性红斑狼疮(systemic lupus erythematosus,SLE)是一种自身免疫介导,以慢性免疫性炎症为突出表现的可复发疾病。主要累及皮肤黏膜、骨骼肌肉、肾和中枢神经系统,同时还可以影响肺、心脏、血液、消化等多个器官和系统,具有不同的临床表现和预后。如果不及时治疗,甚至能够引起肾功能衰竭等致死性损害,严重危害人类健康[1,2]。SLE的全球发病率约为40~70/10万,而我国有估计超过100万SLE患者。目前临床对于SLE的治疗仍然以激素、免疫抑制剂和非甾体抗炎药为主,临床疗效有限[3]。
SLE的发病机制尚不清楚,有研究认为生活环境、激素水平、遗传、表观遗传异常是该病的病因[4-6]。虽然遗传机制在SLE发病中起重要作用,但是仅遗传机制不能解释为何同卵双生子一个发病而另一个不发病[7],且许多为散发病例,没有家族史。近年来大量的研究表明,表观遗传机制在SLE的发生发展中起重要作用[7-10]。表观遗传修饰是可遗传的、可逆的、不改变DNA本身序列的基因修饰方式,主要包括DNA甲基化(DNA methylation,DNMT)、组蛋白修饰和非编码RNA等。表观遗传修饰通过参与调控免疫相关基因的表达,影响免疫细胞分化发育、免疫应答相关分子活化及细胞因子分泌表达等,进而参与免疫调控。许多受表观基因调控的信号分子和受体,在SLE等多种自身免疫性疾病及炎症过程中出现失调[11,12]。
DNA甲基化为DNA化学修饰的一种形式,能够在不改变DNA序列的前提下,改变遗传表现。所谓DNA甲基化是指在DNA甲基化转移酶的作用下,在基因组CpG二核苷酸的胞嘧啶5’碳位共价键结合一个甲基基团。DNA甲基化能引起染色质结构、DNA构象、DNA稳定性及DNA与蛋白质相互作用方式的改变,从而控制基因表达。DNA甲基化由三种不同的DNA甲基转移酶(DNA methyltransferases,DNMTs)催化,分别是DNMT1、DNMT3A和DNMT3B。DNA甲基化的维持是由DNMT1介导的,而从头甲基化则由DNMT3A和DNMT3B主导[13]。
既往研究显示,DNMT1表达水平与SLE疾病活动指数(SLE disease activity index,SLEDAI)有负相关性[14]或无相关性[15]。然而,近年来Mariusz等[16]研究表明DNMT1表达水平与SLEDAI成正相关。为了综合评价DNMT1表达水平与SLEDAI之间相关性,本研究拟进行系统评价和meta分析。
1 资料与方法
1.1 计划书
研究方案在PROSPERO注册,PROSPERO编号:CRD42020177287。
1.2 纳入及排除标准
1.2.1 研究类型
纳入研究类型不做限制。
1.2.2 研究对象
SLE符合《1982年美国风湿病学会修订的SLE标准》[17]或《1997年美国风湿病协会修订的SLE诊断标准》[18]。
1.2.3 结局指标
采用SLE疾病活动指数[19]进行测量,并计算DNMT1与SLEDAI的相关系数。
1.2.4 排除标准
①合并高血压、糖尿病等其他基础疾病患者;②不包含所研究的结局指标;③无法提取数据或未找到全文;④重复发表;⑤数据错误。
1.3 检索策略
本研究使用了三步检索策略。第1步:对Pubmed和知网进行了初步检索,分析标题、摘要及关键词,然后咨询风湿科医师确定相关术语;第2步:由两人分别进行检索;第3步:对所纳入的文献及其参考文献进行检索和审查,用来寻找其他符合纳入标准的文献。
英文数据库:Pubmed、Web of Science、Cochrane Library、Embase;中文数据库:知网、万方、维普。检索时间:数据库建立至2020年4月3日。文献种类:期刊论文、学位论文、会议论文、灰色文献。
以Pubmed为例具体检索策略如下:
#1:Search (((((Search “lupus erythematosus, systemic” [Mesh]) OR systemic lupus erythematosus) OR lupus erythematosus disseminatus) OR libman-sacks disease) OR disease, libman-sacks) OR libman sacks disease
#2:Search ((SLE disease activity index) OR disease activity index) OR SLEDAI
#3:Search (((Search “DNMT1 protein, human [Supplementary Concept]”[Mesh]) OR CXXC9 protein, human) OR DNA (cytosine-5-)-methyltransferase 1, human) OR DNA methyltransferase 1, human
#4:Search (Search “Site-Specific DNA-Methyltransferase (Cytosine-N(4)-Specific)” [Mesh]) OR N (4)-Cytosine-Specific DNA Methylase
#5:Search (((((((((Search “Site-Specific DNA-methyltransferase(adenine-specific)” [Mesh]) OR DNA modification methylases adenine specific) OR DNA modification methylases (adenine-specific)) OR modification methylases (adenine-specific)) OR site-specific methyltransferases (adenine specific)) OR site-specific methyltransferases (adenine-specific)) OR DNA-adenine methylases) OR DNA adenine methylases) OR methylases, DNA-adenine) OR modification methylases (adenine specific)
#6:Search (((((((Search “DNA methyltransferase 3A [Supplementary Concept]”[Mesh]) OR DNA (cytosine-5)-methyltransferase 3A, human) OR DNMT3A protein, rat) OR Dnmt3a2 protein, human) OR DNMT3A protein, human) OR DNA (cytosine-5-)-methyltransferase 3 alpha protein, human) OR Dnmt3a protein, mouse) OR DNA methyltransferase 3A protein, mouse
#7:Search ((((((((((Search “DNA methyltransferase 3B [Supplementary Concept]” [Mesh]) OR DNMT3B protein, human) OR hDNA methyltransferase 3b) OR DNA (cytosine-5-)-methyltransferase 3 beta protein, human) OR DNMT3B protein, rat) OR DNMT3B3DELTA5 protein, human) OR DNMT3B delta5 isoform, mouse) OR DNMT3B4 protein, human) OR DNA (cytosine-5-) methyltransferase 3B1, human) OR Dnmt3b protein, mouse) OR DNA methyltransferase 3B protein, mouse
#8:Search ((((((((((((DNA methyltransferase) OR DNA methyltransferase 1) OR DNA methyltransferase 2) OR DNA methyltransferase 3A) OR DNA methyltransferase 3B) OR DNA methyltransferase 3L) OR DNMT) OR DNMTs) OR DNMT1) OR DNMT2) OR DNMT3A) OR DNMT3B) OR DNMT3L
#9:Search (((“DNA methylation” [Mesh]) OR DNA methylations) OR methylation, DNA) OR methylations, DNA
#10:#3 OR #4 OR #5 OR #6 OR #7 OR #8 OR #9
#11:#1 AND #2 AND #10
1.4 文献筛选和资料提取
使用Endnote X9软件和手动删除重复文献之后,由两位研究者分别进行筛选。首先,两位研究人员通过阅读标题、摘要进行独立筛选,以确定该研究是否保留。所有不确定或可能有相关记录的文献将被保留(比如没有摘要的会议论文)。接下来两位研究人员独立阅读文献全文,严格根据纳入标准进行筛选。对有争议的文献由第三方评价后通过讨论求得统一。筛选出的文献,由两位研究人员独立进行数据提取。数据提取表包含第一作者、发表年限、国家、研究类型、抽样方法、样本量、年龄、检测方法、细胞种类、论文类型等。由一人核对提取的数据,对于不一致的数据再次阅读全文进行确定。
1.5 文献质量评价
由于所纳入研究均为病例对照研究,因此使用Newcastle-Ottawa Scale (NOS)量表[20]对纳入的研究进行质量评价。该量表旨在评估非随机研究的质量,有一个“星级系统”,从3个广泛的角度对研究进行判断:研究对象的选择、组间可比性及病例对照或队列研究的结局指标。两位研究人员独立进行评价,若结果出现不一致,则咨询第三位研究人员进行最终决定。
1.6 统计学方法
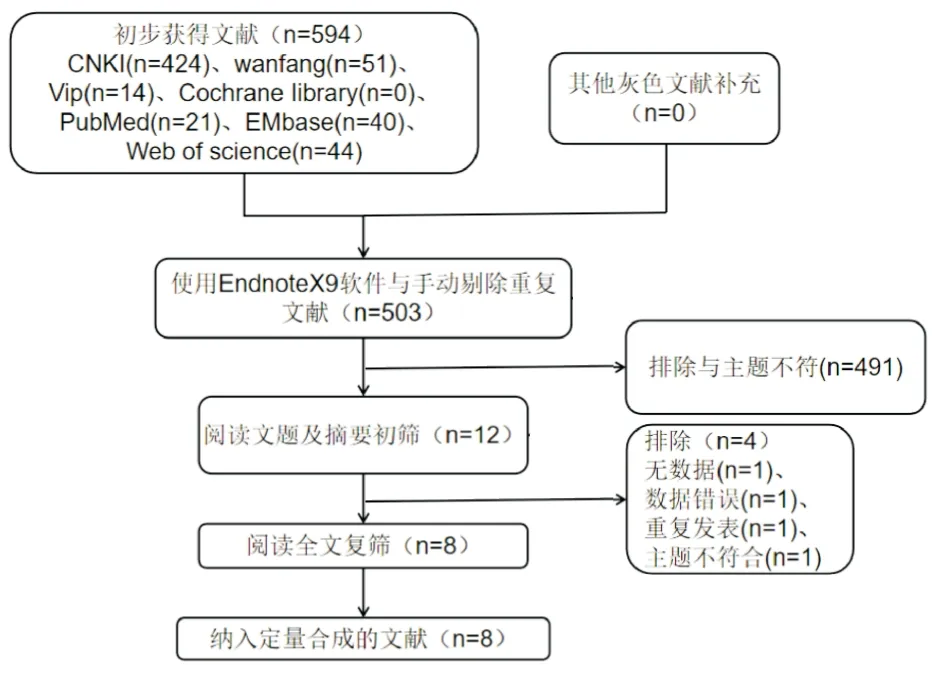
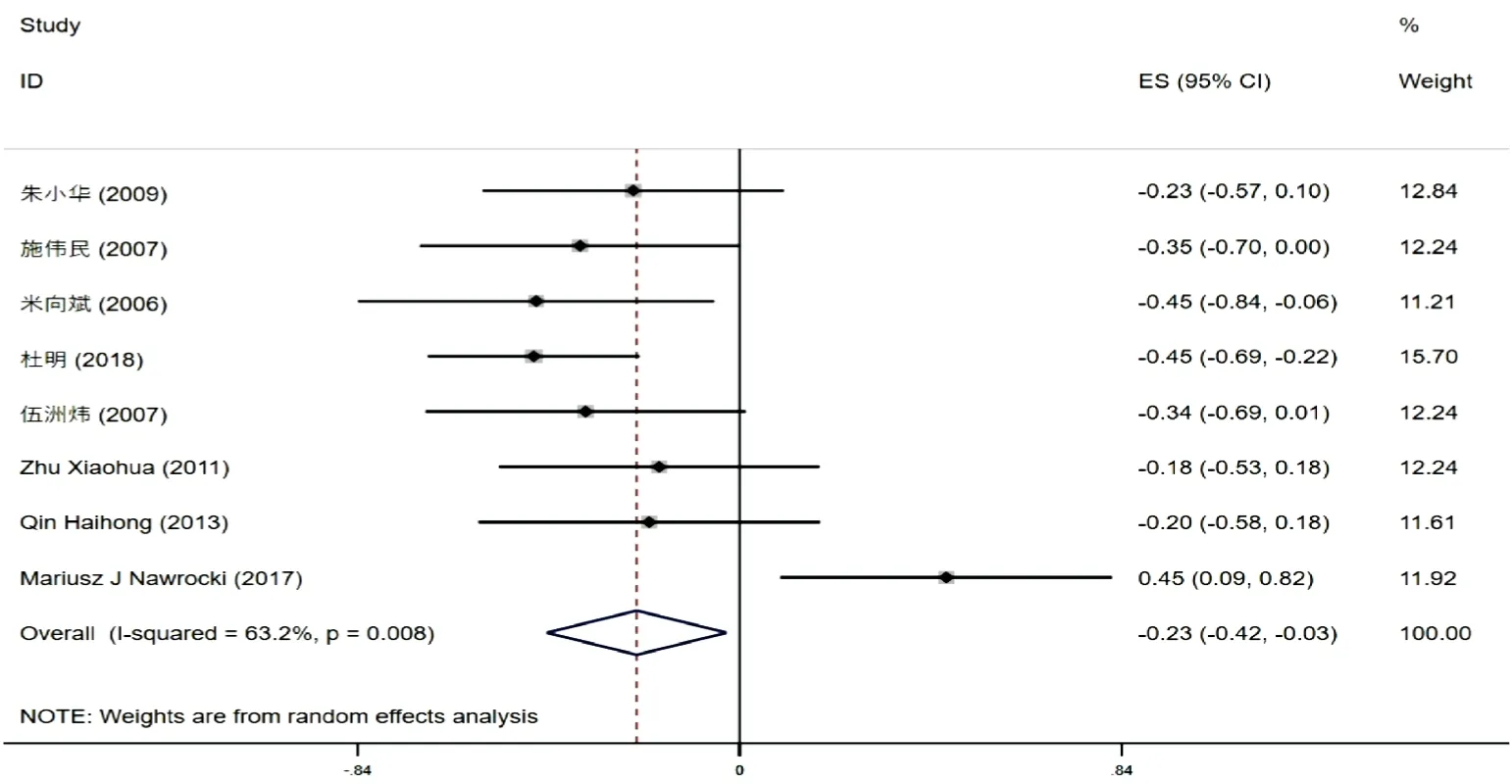
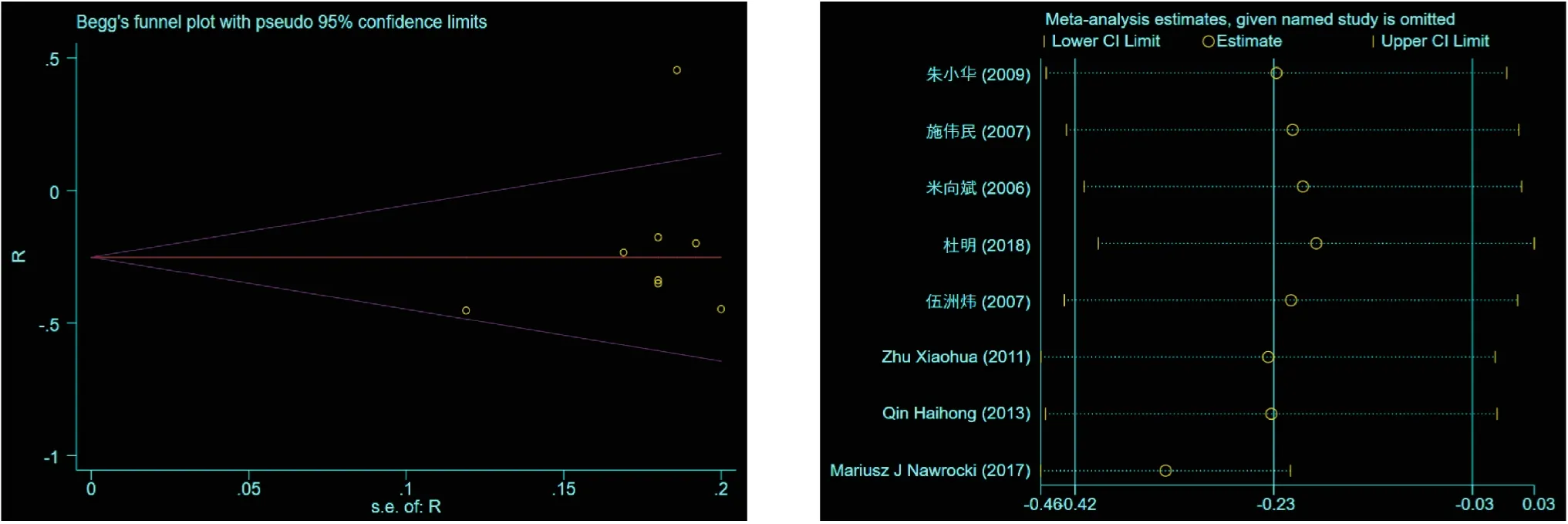
Spearman相关系数(correlation coefficient)又称秩相关系数,是利用两变量的秩次大小进行线性相关分析的,对原始变量的分布不做要求。相关系数r是一个无单位的量值,且-1 最后,输入Stata 15.0进行数据综合,以r值和95%可信区间作为Meta分析的效果指标,使用χ2检验和I2定量分析确定研究间异质性的大小。若P>0.1,I2<50%认为研究间的异质性是可以接受的,使用固定效应模型进行合并;若P<0.1,I2>50%认为研究间的异质性较大,使用随机效应模型进行合并。亚组分析或敏感性分析寻找异质性的来源,使用Begg’s检验与Egger检验来确定是否存在发表偏倚。 共检索出594项研究(英文数据库检索出105项,中文数据库检索出489项),软件自动剔除重复文献39项,手动剔除重复研究52项后剩余503项,评估标题和摘要剔除491项,最后剩余的12项由两位研究人员阅读全文进行筛选。最后,剔除4篇文献(1篇无数据、1篇数据有错误、1篇重复发表、1篇与主题不符合),共8篇纳入了本研究,详见图1。 图1 文献筛选流程图 纳入分析的8项研究均为病例对照研究,有5篇文献报道了外周血单个核细胞(peripheral blood mononuclear cell,PBMC)中DNMT1表达水平与SLEDAI之间的相关性,2篇文献报道了T细胞中DNMT1表达水平与SLEDAI之间的相关性,1篇文献报道了CD4+T细胞中DNMT1表达水平与SLEDAI之间的相关性,详见表1。 所纳入的8项研究均为病例对照研究,使用NOS量表进行评价,均>5颗星。获得6颗星的文献有3篇,获得7颗星的文献有3篇,获得8颗星的文献有2篇,详见表2。 2.4.1 DNMT1与SLEDAI的相关性 纳入分析的8篇文献当中,有3篇文献认为DNMT1表达水平与SLEDAI之间有相关性。8项研究共有304名患者,异质性检验分析显示P=0.008,I2=63.2%,提示纳入的研究间异质性较大,meta分析采用随机效应模型进行合并分析,合并的r=-0.23,95%CI(-0.42,-0.03),认为DNMT1表达水平与SLEDAI之间有低相关性,详见图2。 图2 DNMT1与SLEDAI的相关性森林图 2.4.2 亚组分析 随后进行亚组分析,确定异质性的潜在来源:①细胞类型:PBMCs、T细胞或CD4+T细胞;②PCR引物:β-acting、PBGD;③NOS得分:6星、7星、8星;④样本量(中位数34):n<34、n≥34;⑤平均年龄(中位数33.82):年龄<33.82、年龄≥33.82、未报告;⑥国家:中国、波兰;⑦SLE诊断标准:1982标准、1997标准。亚组分析结果显示:PCR引物与研究的地点可能是异质性的主要来源,详见表3。 表3 亚组分析汇总表 2.4.3 发表偏倚 使用Begg’s法与Egger法进行发表偏倚风险分析,P=0.291(Begg’s)、P=0.610(Egger法)提示发表偏倚不明显,见图3。 图3 发表偏倚Begg’s检验 图4 敏感性分析 2.4.4 敏感性分析 采用逐一剔除所纳入的研究,重新进行合并观察统计量,来评估结果的稳定性.在排除Mariusz等[16]这一项研究后,I2=0.0%,P=0.871,[r=-0.334,95%CI(-0.458,-0.210),P<0.05],说明该研究对于结果的稳定性影响较大,可能是影响异质性的重要来源,结果如图4。 维持甲基化和从头甲基化是由不同DNMTs催化,有研究证明维持甲基化一般是由DNMT1完成[29]。DNMT2为天门冬氨酸tRNA的甲基转移酶,具有微弱的DNA甲基转移酶活性,DNMT3A和DNMT3B基因分别位于2p23.3和20q11.2,两者结构域相似,负责从头合成甲基化,DNMT3A负责修饰裸露DNA,DNMT3B负责核小体核心部位DNA甲基化[30]。DNMT3L缺少C端催化活性区域,没有单独的催化活性,可作为调节因子与DNMT3A和DNMT3B相互作用提高它们的催化活性[31,32]。DNMTs在生物体生长发育不同时期、不同细胞表达不同,其表达异常与SLE的T/B淋巴细胞激活、系统性硬化症的成纤维细胞表型异常以及抑癌基因高甲基化关系密切[33,34],可通过多途径参与自身免疫性疾病、肿瘤、心脑血管等疾病的发生发展[35]。 我们纳入的原始研究中正相关性、负相关性、无相关性的结果都有,不同研究结果之间的差异较大,r在-4.80与0.4087之间。我们综合了这些不同结果的研究之后认为,DNMT1表达水平与SLEDAI之间为负相关性。有研究认为,由于SLE患者DNMT转录下调,可导致CD4+T细胞全基因组的DNA低甲基化和自身免疫反应[36,37]。Farivar等[38]发现,抑制有丝分裂原活化蛋白激酶(mitogen-activatied protein kinase,MAPK)信号通路可降低DNMT1活性,从而引起T细胞DNA的低甲基化,并参与SLE的发病。因此,有可能通过调节DNMT转录,影响DNA低甲基化的状态,从而降低SLEDAI。 异质性检验分析显示P=0.008,I2=63.2%,提示纳入的研究间异质性较大。通过亚组分析和敏感性分析可知,排除Mariusz等[16]这一项研究后异质性明显下降,说明该研究对于结果的稳定性影响较大,可能是影响异质性的重要来源。该项研究所使用的内参蛋白胆色素原脱氨酶(porphobilino gendeaminase,PBGD)与其他研究不一样,可能是造成异质性的原因。其他可能存在异质性的原因有研究人群的不同、研究仪器的不同、试剂的不同、DNMT1测定方法的不同等。 本次研究设计和原始研究本身存在一些局限性和不足之处。每个原始研究的纳入样本量较少,在28~74人,可能会影响本次研究结果。虽然纳入的原始研究阴性和阳性结果均有,但是正相关性的研究只有1个。其原因可能是结果为阴性和不显著的研究未提交或被拒绝,进而造成本次研究结果的偏差。本次研究只检索了7个数据库,这些数据库中所收录的文章只包含了中文和英文出版的文章,可能存在选择偏倚。在meta分析中排除灰色文献的解释可能会导致对干预效果的夸大估计[39]。虽然本研究对灰色文献进行了检索,但并未发现符合纳入标准的文献。 综上所述,根据目前可用的证据表明,DNMT1表达水平与SLEDAI之间存在负相关性。SLE目前仍缺乏判断疾病活动性、预后的特异性生物学标记物,未来可以探究DNA甲基化水平改变的原因和作用机制,为SLE的诊断、治疗、预后、监测等提供有效的分子靶标。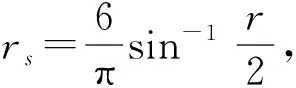
2 结果
2.1 文献检索结果
2.2 纳入研究的基本特征
2.3 纳入研究的偏倚风险评价结果
2.4 Meta分析结果

3 讨论

