黄梁木NcEXPA8基因提高拟南芥种子萌发速度的研究
廖嘉明 包钰韬 陈媛 张登 李华强 欧阳昆唏 陈晓阳

摘 要: 为探究NcEXPA8基因的分子功能,该文以在黄梁木形成层区域中高表达的扩展蛋白基因NcEXPA8为研究对象,研究其在黄梁木种子萌发过程中的表达及其过表达对拟南芥种子萌发的影响。该文以黄梁木和拟南芥野生型(WT)(Col-0)种子以及转NcEXPA8基因的拟南芥T3代纯合体种子为实验材料,利用实时荧光定量RT-qPCR分析NcEXPA8基因在黄梁木种子萌发不同阶段的表达量,并分析NcEXPA8基因和拟南芥种子萌发内源相关基因在拟南芥WT和转基因不同株系萌发种子中的表达量,且对拟南芥WT种子和转基因T3代纯合体种子在不同处理和不同时间的萌发率进行比较。结果表明:NcEXPA8基因在黄梁木种子萌发不同阶段的表达量存在差异,在种壳破裂时表达量最高,随后降低。与拟南芥WT相比,过表达NcEXPA8基因不仅显著提高了种子的萌发速度,而且提高了对赤霉素的敏感性,降低了对脱落酸的敏感性,但未影响拟南芥内源相关结构基因的表达。该研究初步分析了黄梁木NcEXPA8基因在种子萌发中的功能,但其最终确定还需在黄梁木中进行验证。
关键词: 黄梁木, NcEXPA8基因, 种子萌发, 拟南芥
中图分类号: S722
文献标识码: A
文章编号: 1000-3142(2021)04-0654-08
Abstract: In order to explore the molecular function of NcEXPA8 gene, which is highly expressed in the cambium region of N. cadamba, NcEXPA8 gene expression during seed germination of N. cadamba and the effect of its overexpression on seed germination of Arabidopsis thaliana were studied. The seeds of Neolamarckia cadamba, Arabidopsis thaliana wild type (WT, Col-0) and T3 homozygote of A. thaliana overexpressing NcEXPA8 gene were selected as experimental materials. Quantitative real-time polymerase chain reaction (RT-qPCR) was used to analyze the expression of NcEXPA8 gene in different stages during seed germination of Neolamarckia cadamba, and also its expression level and that of endogenous related genes in the germinating seeds of A. thaliana WT and T3 transgenic homozygotes. Furthermore, the germination rates of WT and T3 homozygous seeds of Arabidopsis thaliana were compared at different treatments and different time. The results were as follows: The expression level of NcEXPA8 gene was different among different stages during seed germination of Neolamarckia cadamba, and its expression level was the highest when the seed coat was broken, and then decreased. Compared to wild type of Arabidopsis thaliana, the overexpression of NcEXPA8 gene not only significantly increased seed germination speed, but also increased the sensitivity to GA and reduced the sensitivity to ABA, but did not affect the expression of endogenous related structural genes in A. thaliana. This study preliminarily analyzes the function of NcEXPA8 gene in seed germination, but its final determination still need to be verified in Neolamarckia cadamba.
Key words: Neolamarckia cadamba, NcEXPA8 gene, seed germination, Arabidopsis thaliana
植物擴展蛋白(expansin, 简称EXP)首次在黄瓜下胚轴中被发现,是一类负责酸性诱导细胞壁伸展的细胞壁蛋白,但不具有细胞壁水解活性(Mcqueen-Mason et al., 1992)。对植物而言,细胞壁是一种极其重要的结构,提供植物所需的机械支持,同时为植物组织器官生长发育提供可塑性;植物的生长发育需要改变细胞的大小和形状,这就需要对细胞壁的可塑性进行调节(Marowa et al., 2016)。尽管对于扩展蛋白生化作用机理的了解还不彻底,但大家普遍认为扩展蛋白参与了细胞壁可塑性的调节,即通过打断细胞壁微纤丝与基质间的非共价键而使细胞壁软化,允许细胞壁多聚物在细胞膨压作用下“蠕动”导致细胞快速伸展(Cosgrove, 2000, 2015)。随着研究的深入,认为扩展蛋白对植物各个组织生长发育都有重要作用,例如:根(Yu et al., 2011; Boron et al., 2015)、茎(Boron et al., 2015; Li et al., 2017)、叶(Devi et al., 2015; Zhou et al., 2015)和果实(Minoia et al., 2016)的生长发育。
黄梁木(Neolamarckia cadamba),又名团花树,隶属茜草科(Rubiaceae)团花属(Neolamarckia),常绿大乔木,原产中国广东、广西和云南南部以及越南、缅甸、马来西亚等地,由于其生长十分迅速,在1972年的第七届世界林業大会上,被各国专家誉为“奇迹树”,而且该树种材质好,是胶合板、纤维板和制浆造纸等的理想原料(邓小梅等,2012)。克隆到黄梁木形成层中高表达的扩展蛋白基因NcEXPA8,并构建过表达载体Pbi121-NcEXPA8转化拟南芥,发现NcEXPA8基因的过量表达促进了拟南芥植株生长以及茎段纤维细胞的伸长(Li et al., 2017)。
种子萌发起始于干种子吸水,结束于胚根突破包裹胚的种皮(Nonogaki, 2019)。萌发过程分为2个连续的步骤:种皮沿着已经裂开的缝破裂,然后仅留胚乳帽覆盖胚根(Liu et al., 2005),只有在胚乳帽充分软化后才能被胚根生长穿破,这也标志着萌发过程的结束(Nonogaki, 2019)。前人研究表明,种子萌发过程中,编码修饰细胞壁蛋白的木葡聚糖基转移酶/水解酶(XTHs)、扩展蛋白在拟南芥(Arabidopsis thaliana)、番茄(Lycopersicon esculentum)、芹菜(Lepidium sativum)的胚乳帽区域均为特异表达(Chen et al., 2002; Voegele et al., 2011; Sánchez-Montesino et al., 2019),且GA缺乏的突变体(gib-1)种子只有在外源GA存在时才能萌发,在吸胀12 h内GA诱导LeEXP4的表达(Chen et al., 2002),说明通过细胞壁修饰引起胚乳帽区域软化对种子萌发起到的促进机理具有较高的保守性。
已有研究表明,在拟南芥扩展蛋白家族基因中,AtEXPA2基因在拟南芥萌发的种子中特异高表达,参与种子萌发(Yan et al., 2014; Sánchez-Montesino et al., 2019)。此外,还有其他结构基因,如AtMAN7、AtTZF4等也参与了种子萌发的过程,其中AtTZF4对种子的萌发起到抑制作用;赤霉素诱导AtMAN7表达上调,但下调AtTZF4的表达;ABA诱导AtTZF4表达上调,但对AtMAN7表达无调控作用( Iglesias-Fernandez et al., 2011; Iglesias-Fernandez et al., 2013; Bogamuwa et al., 2013)。因此,本研究分析了NcEXPA8基因在黄梁木种子萌发过程中不同时期的相对表达量,并分析了拟南芥WT和转基因植株中NcEXPA8基因和参与拟南芥种子萌发的内源相关基因AtEXPA2、AtMAN7和AtTZF4在空白处理和植物激素GA3和ABA处理下的相对表达量,以及探究了GA3和ABA对拟南芥WT和转基因种子萌发的影响,试图为后续研究NcEXPA8基因在黄梁木种子萌发中的功能奠定基础。
1 材料与方法
1.1 材料
本研究以当年收集的黄梁木的种子、拟南芥野生型WT(Col-0)以及已获得的转NcEXPA8基因的拟南芥T3代纯合体种子(Li et al., 2017)为材料。
1.2 总RNA的提取与cDNA合成
利用植物RNA试剂盒(Omega)提取总RNA,用40 μL的DEPC水洗脱RNA,于-80 ℃保存备用。PrimeScript TM RT Master Mix试剂盒(Takara,大连)说明书,将总RNA(0.5 μg)逆转录为第1链cDNA,于-20 ℃保存备用。
1.3 荧光定量(RT-qPCR)分析
将合成的第1链cDNA稀释15倍备用,RT-qPCR在罗氏(Roche)LC480定量PCR仪上进行,每个点重复3次,取平均值分析,以ddH2O代替模板作为实验的空白对照。RT-qPCR 20 μL反应体系如下:2×SYBR Premix Ex Taq II 10 μL,引物 F (10 μmol·L-1) 1 μL,引物R (10 μmol·L-1) 1 μL,cDNA 2 μL,ddH2O 6 μL。反应程序如下:95 ℃ 30 s;40个循环(95 ℃ 5 s,58 ℃ 30 s,72 ℃ 30 s);72℃ 2 min;进行58~95 ℃的熔解曲线分析;40 ℃冷却30 s。黄梁木和拟南芥的内参基因分别为NcCyclophilin和AtPP2AA3(Li et al., 2017),拟南芥AtEXPA2、AtMAN7和AtTZF4基因RT-qPCR引物参考已有的文献(Iglesias-Fernandez et al., 2013; Bogamuwa et al., 2013; Yan et al., 2014),所用引物见表1。
1.4 材料处理及萌发试验
黄梁木种子泡于常温的水中并置于37 ℃恒温箱24 h;在培养皿中平铺滤纸并吸足蒸馏水,将浸泡过的种子平铺于滤纸上,盖上盖子,置于光照强度为1 500 lx、16 h光照/8 h黑暗光周期、25 ℃培养箱中培养,分别于培养3、6、7、8 d收集种子。根据Yan et al.(2014)的方法,收集于25 ℃空白处理(水)、10 μmol·L-1 GA3和ABA中浸泡24 h的拟南芥WT和转NcEXPA8基因T3代纯合体种子。每个样品设3个生物学重复,样品采集后放入液氮速冻,置于-80 ℃冰箱储存备用。
根据Wang et al.(2016)的方法,在培养皿中平铺滤纸并各自吸足蒸馏水、10 μmol·L-1 GA3和ABA,将拟南芥WT和转NcEXPA8基因T3代纯合体种子置于滤纸上,每个培养皿80~100粒种子,盖上盖子,4 ℃黑暗中处理3 d,再置于连续光照强度为1 500 lx、温度为25 ℃的培养箱中培养。以胚根出现衡量种子已萌发,分别于12、18、24、48 h统计种子萌发率,每个基因型设4个生物学重复。采用SPSS 19统计软件的t检验进行显著性分析,并计算在18 h时,GA3和ABA对各基因型种子萌发的促进率和抑制率。
2 结果与分析
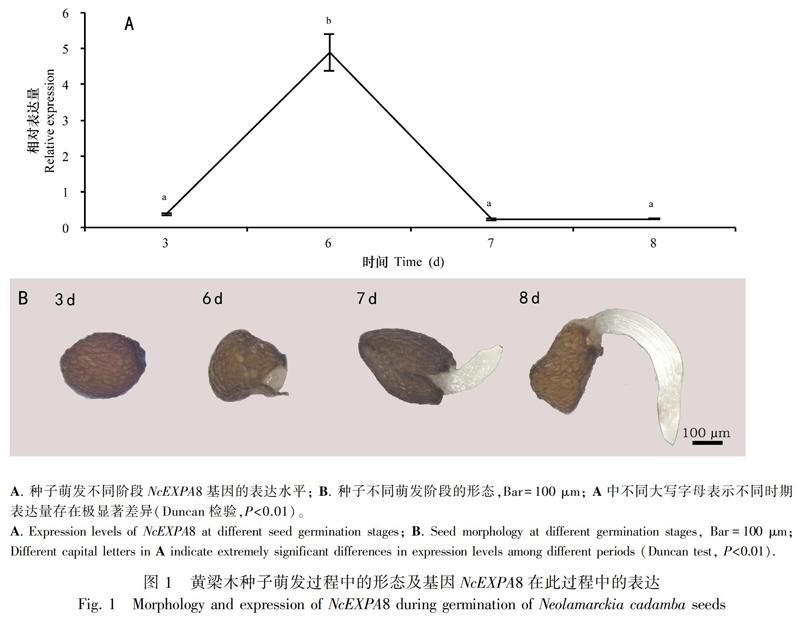
2.1 NcEXPA8基因在黄梁木种子萌发过程中的表达分析
黄梁木种子在培养6 d后,胚根才出现(图1:B),在黄梁木种子萌发过程的不同阶段,NcEXPA8基因的表达变化大。种子在培养3 d后,表达量较低;在培养6 d后,其表达量达到最高,此时胚根刚露白。随后,胚根不断生长,但NcEXPA8基因的表达却显著降低,并维持在较低水平(图1:A)。
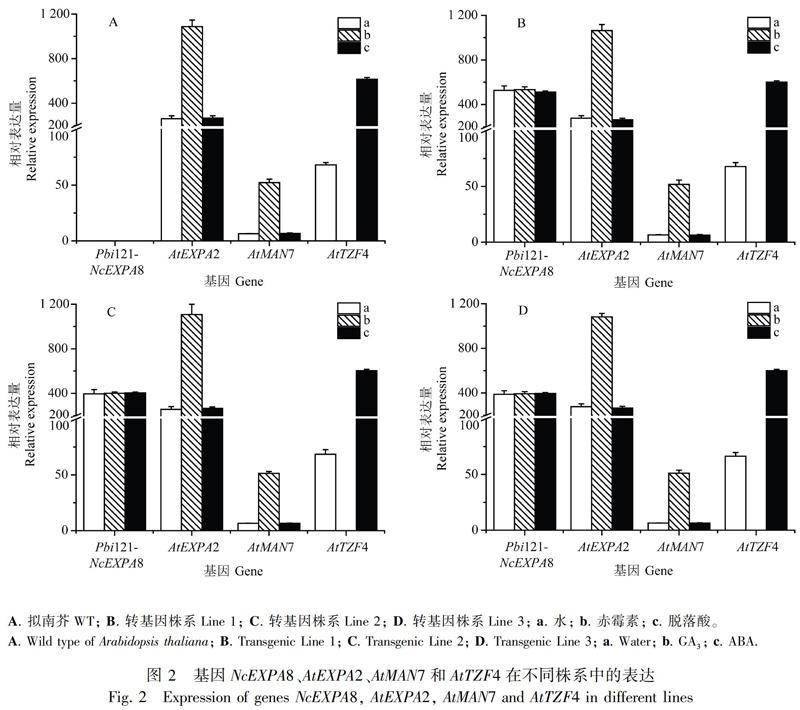
2.2 NcEXPA8、AtEXPA2、AtMAN7和AtTZF4基因在转基因拟南芥中的表达
利用实时荧光定量RT-qPCR技术检测了NcEXPA8、AtEXPA2、AtMAN7和AtTZF4基因在空白处理(水)、10 μmol·L-1 GA3和ABA处理下拟南芥WT和转基因NcEXPA8株系萌发的种子(胚根刚露白阶段)中的表达。由图2可知,在转基因T3代纯合体萌发的种子中,各株系NcEXPA8基因的表达量都很高(图2:B, C, D),而在拟南芥WT中没有表达(图2:A)。在相同处理下,AtEXPA2、AtMAN7和AtTZF4基因在拟南芥WT和各转基因株系间相对表达量无差异。AtEXPA2和AtMAN7基因在拟南芥萌发的种子中特异表达,参与种子萌发(Yan et al., 2014;Sánchez-Montesino et al., 2019)。与空白处理(水)相比,在ABA处理下,AtEXPA2和AtMAN7基因在拟南芥WT和各转基因株系的萌发种子中的表达无明显差异,而AtTZF4则显著提高;但在GA3处理下,AtEXPA2和AtMAN7基因在各基因型萌发种子的表达量显著提高,而AtTZF4则表达量极低。
2.3 拟南芥种子萌发率分析
按照Wang et al.(2016)的方法,进行种子萌发率比较,由表2可知,4 ℃黑暗处理3 d后(0 h),拟南芥WT和3个转NcEXPA8基因T3代纯合体的种子,都未萌发。在25 ℃培养箱中培养12 h后,10 μmol·L-1 ABA处理的WT种子仍未萌发,转基因种子有个别开始萌发,但数量极少,一个培养皿中最多只有4粒,其余处理的种子都有部分开始萌发,且转基因种子的萌发率显著高于WT。在培养箱中培养18 h后,除ABA处理外,其余处理的拟南芥WT和转基因种子大部分都已萌发;此时空白处理(水)的转基因3个株系种子萌发率都极显著地高于拟南芥WT;与空白处理相比,10 μmol·L-1 ABA处理的各基因型种子萌发率低,其中拟南芥WT仅有20.17%,而10 μmol·L-1 GA3处理的各基因型种子萌发率显著提高,且各转基因种子分别在一半的培养皿中已全部萌发。培养24 h后,除了ABA处理,其余处理的各基因型种子全已萌发。培养48 h后,各处理的各基因型种子全都萌发。因此,过量表达NcEXPA8基因只是加速种子萌发。
GA3和ABA对拟南芥WT和转基因种子萌发的影响结果见表2。经10 μmol·L-1 GA3处理的WT和3个转基因株系的种子萌发都显著提高,且3个转基因株系的种子萌发促进率为31.47%~32.09%,大于拟南芥WT的萌发促进率20.92%;而经10 μmol·L-1 ABA处理的各基因型种子萌发率显著降低,但转基因种子的萌发抑制率(47.6%~48.73%)小于拟南芥WT的萌发抑制率70.8%,说明过量表达NcEXPA8基因提高了GA3对种子萌发的促进率,降低了ABA对种子的萌发抑制率。
3 讨论与结论
模式植物基因组含有许多EXP基因(https://homes.bio.psu.edu/expansins/genes.htm),但在不同的组织器官以及不同生长发育阶段,这些基因的表达水平存在差异,因此它们的具体功能也各有差异。如拟南芥中的AtEXPA10基因在幼嫩的叶片中高表达,它的过表达和反义植株在很大程度上增加和降低了叶片的大小(Cho & Cosgrove, 2000);AtEXPA7、AtEXPA18与AtEXPA17以高丰度表达在根毛的起始和根的生长中起着决定性的作用(Cho & Cosgrove, 2002; Lee & Kim, 2013);在叶片保卫细胞特异表达的AtEXPA1,通过改变保卫细胞壁的结构调控气孔开关(Wei et al., 2011; Zhang et al., 2011);在萌发种子中高表达的AtEXPA2,参与种子的萌发(Yan et al., 2014; Sánchez-Montesino et al., 2019)。此外,在其他植物不同的组织器官以及不同生长发育阶段中高表达的EXP基因,都参与相应组织器官的生长发育,如番茄(Rose et al., 2000; Chen et al., 2001)、水稻(Cho & Kende, 1997; Choi et al., 2003; Ma et al., 2013)等。
对于种子萌发和休眠以及相关分子网络已经被广泛地研究,种子萌发分别受植物激素GA和ABA的诱导和打破休眠的调控;种子萌发起始于水分的吸取,导致种壳的破裂,最后胚根伸出(Finch-Savage & Leubner-Metzger, 2006; Holdsworth et al., 2008)。盡管EXP对种子萌发作用的细节尚不清楚,但已有研究证实,在种子萌发中需要细胞壁的软化以及EXP参与珠孔胚乳的软化和胚根生长(Morris et al., 2011; Voegele et al., 2011);而EXP具有软化细胞壁导致细胞快速伸展的功能(Cosgrove, 2000, 2015)。因此,可以推断EXP在种子的萌发中起着重要的作用。在黄梁木种子萌发的过程中,NcEXPA8基因的表达量在种子刚萌发时最高,而后在胚根的生长中极显著降低,并维持在较低水平。这些结果表明,NcEXPA8基因可能参与了黄梁木种子的萌发,尤其是胚乳帽区域的软化。
為了鉴定基因功能,转基因技术是常用的有效手段之一,尤其是对拟南芥模式植物的转化。由于其转化技术成熟、基因组小、生长周期短、植株小等优点,对其容易进行表型观测、生理生化指标测定以及基因表达检测,已被广泛应用于基因功能的检测(Zhang et al., 2011; Lu et al., 2013; Bae et al., 2014; Boron et al., 2015)。在我们的前期研究中,已获得了过表达黄梁木NcEXPA8基因的拟南芥T3代纯合体种子(Li et al., 2017)。
在本研究中,NcEXPA8基因在3个转基因株系萌发的种子中都存在很高的表达,其表达水平显著高于在种子萌发中特异表达的AtEXPA2、AtMAN7和AtTZF4基因,但与拟南芥WT相比,转基因并未改变这些内源基因的表达。参与种子萌发的内源基因AtEXPA2、AtMAN7和AtTZF4,其中AtTZF4对种子的萌发起到抑制作用(Bogamuwa et al., 2013),在GA3处理下,萌发种子中AtEXPA2和AtMAN7的相对表达量提高,AtTZF4的表达被抑制;在ABA处理下,AtTZF4的表达量提高,但对AtEXPA2和AtMAN7的表达无明显影响。此外,在GA3处理下,转基因种子的萌发促进率明显高于拟南芥WT; 而在ABA处理下,转基因种子的萌发抑制率明显低于拟南芥WT。这些结果表明,种子萌发速度的改变,是NcEXPA8基因过表达的直接结果,内源相关基因的表达并未受到转基因的影响;过表达NcEXPA8基因不仅促进了拟南芥种子的萌发,而且提高了其对GA的敏感性,降低了对ABA的敏感性。因此,这些信息进一步加深了NcEXPA8基因参与黄梁木种子萌发的推断。为了进一步了解NcEXPA8基因的功能,对转基因的纯合体植株表型和细胞形态进行观察分析发现,过表达NcEXPA8基因还促进了拟南芥茎叶的生长、纤维细胞的伸长等多效的表型(Li et al., 2017)。这些发现,暗示着NcEXPA8具有促进多类细胞软化伸展,从而促进植物的生长功能。
但要了解NcEXPA8基因在黄梁木中的真正功能,还需在黄梁木自身个体中进行功能验证,利用包括基因过表达以及CRISPR/Cas9基因敲除(Hsu et al., 2014)等技术创造黄梁木NcEXPA8基因的突变体。目前,我们已建立了黄梁木的遗传转化体系(Li et al., 2019),为在黄梁木中开展基因功能验证奠定基础。因此,先前对NcEXPA8基因的研究(Li et al., 2017)以及本研究将为在黄梁木中开展NcEXPA8基因的研究奠定基础。
参考文献:
BAE JM, KWAK MS, NOH SA, et al., 2014. Overexpression of sweetpotato expansin cDNA (IbEXP1) increases seed yield in Arabidopsis[J]. Transgenic Res, 23(4):657-667.
BOGAMUWA S, JANG JC, 2013. The Arabidopsis tandem CCCH zinc finger proteins AtTZF4, 5 and 6 are involved in light-, abscisic acid- and gibberellic acid-mediated regulation of seed germination[J]. Plant Cell Environ, 36(8): 1507-1519.
BORON AK, VAN LOOCK B, SUSLOV D, et al., 2015. Over-expression of AtEXLA2 alters etiolated Arabidopsis hypocotyl growth[J]. Ann Bot, 115(1):67-80.
CHEN F, DAHAL P, BRADFORD KJ, 2001. Two tomato expansin genes show divergent expression and localization in embryos during seed development and germination[J]. Plant Physiol, 127(3):928-936.
CHEN F, NONOGAKI H, BRADFORD KJ, 2002, A gibberellin-regulated xyloglucan endotransglycosylase gene is expressed in the endosperm cap during tomato seed germination[J]. J Exp Bot, 53(367): 215-223.
CHO HT, COSGROVE DJ, 2000. Altered expression of expansin modulates leaf growth and pedicel abscission in Arabidopsis thaliana[J]. Proc Natl Acad Sci USA, 97(17):9783-9788.
CHO HT, COSGROVE DJ, 2002. Regulation of root hair initiation and expansin gene expression in Arabidopsis[J]. Plant Cell, 14(12):3237-3253.
CHO HT, KENDE H, 1997. Expression of expansin genes is correlated with growth in deep water rice[J]. Plant Cell, 9(9):1661-1671.
CHOI D, LEE Y, CHO HT, et al., 2003. Regulation of expansin gene expression affects growth and development in transgenic rice plants[J]. Plant Cell, 15(6):1386-1398.
COSGROVE DJ, 2000. Loosening of plant cell walls by expansins[J]. Nature, 407(6802):321-326.
COSGROVE DJ, 2015. Plant expansins: Diversity and interactions with plant cell walls[J]. Curr Opin Plant Biol, 25:162-172.
DENG XM, ZHAN YL, ZHANG Q, et al., 2012. Study on tissue culture of Neolamarckia cadamba[J]. J S Chin Agric Univ, 33(2):216-219.[鄧小梅,詹艳玲,张倩,等,2012. 黄梁木组培快繁技术研究[J]. 华南农业大学学报,33(2):216-219.]
DEVI MJ, TALIERCIO EW, SINCLAIR TR, 2015. Leaf expansion of soybean subjected to high and low atmospheric vapour pressure deficits[J]. J Exp Bot, 66(7):1845-1850.
FINCH-SAVAGE WE, LEUBNER-METZGER G, 2006. Seed dormancy and the control of germination[J]. New Phytol, 171(3):501-523.
HOLDSWORTH MJ, BENTSINK L, SOPPE WJ, 2008. Molecular networks regulating Arabidopsis seed maturation, after-ripening, dormancy and germination[J]. New Phytol, 179(1):33-54.
HSU PD, LANDER ES, ZHANG F, 2014. Development and applications of CRISPR-Cas9 for genome engineering[J]. Cell, 157(6):1262-1278.
IGLESIAS-FERNANDEZ R, BARRERO-SICILIA C, CARRILLO-BARRAL N, et al., 2013. Arabidopsis thaliana bZIP44: A transcription factor affecting seed germination and expression of the mannanase-encoding gene AtMAN7[J]. Plant J, 74(5): 767-780.
IGLESIAS-FERNANDEZ R, RODRIGUEZ-GACIO MC, BARRERO-SICILIA C, et al., 2011. Three endo-β-mannanase genes expressed in the micropylar endosperm and in the radicle influence germination of Arabidopsis thaliana seeds[J]. Planta, 233(1): 25-36.
LEE HW, KIM J, 2013. EXPANSINA17 up-regulated by LBD18/ASL20 promotes lateral root formation during the auxin response[J]. Plant Cell Physiol, 54(10):1600-1611.
LI JC, HU XS, HUANG XL, et al., 2017. Functional identification of an EXPA gene (NcEXPA8) isolated from the tree Neolamarckia cadamba[J]. Biotechnol Biotechnol Equip, 31(6):1116.
LI JJ, ZHANG D, QUE QM, et al., 2019. Plant regeneration and agrobacterium-mediated transformation of the miracle tree Neolamarckia cadamba[J]. Ind Crops Products, 130:443-449.
LIU P, KOIZUKA N, HOMRICHHAUSEN TM, et al., 2005. Large-scale screening of Arabidopsis enhancer-trap lines for seed germination-associated genes[J]. Plant J, 41(6):936-944.
LU P, KANG M, JIANG X, et al., 2013. RhEXPA4, a rose expansin gene, modulates leaf growth and confers drought and salt tolerance to Arabidopsis[J]. Planta, 237(6):1547-1559.
MA N, WANG Y, QIU S, et al., 2013. Overexpression of OsEXPA8, a root-specific gene, improves rice growth and root system architecture by facilitating cell extension[J]. PLoS ONE, 8(10): e75997.
MAROWA P, DING A, KONG Y, 2016. Expansins: Roles in plant growth and potential applications in crop improvement[J]. Plant Cell Rep, 35(5):949-965.
MCQUEEN-MASON S, DURACHKO DM, COSGROVE DJ, 1992. Two endogenous proteins that induce cell wall extension in plants[J]. Plant Cell, 4:1425-1433.
MINOIA S, BOUALEM A, MARCEL F, et al., 2016. Induced mutations in tomato SlExp1 alter cell wall metabolism and delay fruit softening[J]. Plant Sci, 242:195-202.
MORRIS K, LINKIES A, MULLER K, et al., 2011. Regulation of seed germination in the close Arabidopsis relative Lepidium sativum: A global tissue-specific transcript analysis[J]. Plant Physiol, 155(4):1851-1870.
NONOGAKI H, 2019. Seed germination and dormancy: The classic story, new puzzles, and evolution[J]. J Integr Plant Biol, 61(5):541-563.
ROSE JK, COSGROVE DJ, ALBERSHEIM P, et al., 2000. Detection of expansin proteins and activity during tomato fruit ontogeny[J]. Plant Physiol, 123(4):1583-1592.
SNCHEZ-MONTESINO R, BOUZA-MORCILLO L, MARQUEZ J, et al., 2019. A regulatory module controlling GA-mediated endosperm cell expansion is critical for seed germination in Arabidopsis[J]. Mol Plant, 12(1):71-85.
VOEGELE A, LINKIES A, MULLER K, et al., 2011. Members of the gibberellin receptor gene family GID1 (GIBBERELLIN INSENSITIVE DWARF1) play distinct roles during Lepidium sativum and Arabidopsis thaliana seed germination[J]. J Exp Bot, 62(14):5131-5147.
WANG Z, CHEN F, LI X, et al., 2016. Arabidopsis seed germination speed is controlled by SNL histone deacetylase-binding factor-mediated regulation of AUX1[J]. Nature Comm, 7(1):13412.
WEI PC, ZHANG XQ, ZHAO P, et al., 2011. Regulation of stomatal opening by the guard cell expansin AtEXPA1[J]. Plant Signal Behav, 6(5):740-742.
YAN A, WU M, YAN L, et al., 2014. AtEXP2 is involved in seed germination and abiotic stress response in Arabidopsis[J]. PLoS ONE, 9(1): e85208.
YUZM, KANG B, HE XW, et al., 2011. Root hair-specific expansins modulate root hair elongation in rice[J]. Plant J, 66(5):725-734.
ZHANG XQ, WEI PC, XIONG YM, et al., 2011. Overexpression of the Arabidopsis alpha-expansin gene AtEXPA1 accelerates stomatal opening by decreasing the volumetric elastic modulus[J]. Plant Cell Rep, 30(1):27-36.
ZHOU S, HAN YY, CHEN Y, et al., 2015. The involvement of expansins in response to water stress during leaf development in wheat[J]. J Plant Physiol, 183:64-74.
(責任编辑 李 莉)

